基于數據挖掘分析KEAP1基因在肺癌中的表達及意義


摘要:目的" 利用生物信息學技術對人胞質接頭蛋白(KEAP1)進行分析,并匯總其在肺癌中的表達情況及意義,為后續肺癌相關治療和預后提供有力的依據。方法" 利用GEO數據庫挖掘KEAP1基因表達及染色體定位,利用Kaplan-Meier數據庫分析肺癌中KEAP1表達與患者預后的關系,通過Linkedomics數據庫和String數據庫獲取該基因在不同組織和疾病中的表達數據和蛋白相互作用網絡,分析KEAP1與其他蛋白之間的關系,探索與其他基因的關聯和調控網絡。通過GEPIA2數據庫進行KEAP1表達分析,比較KEAP1在正常肺組織和肺癌組織中的表達情況。結果" KEPA1基因位于19號染色體上;該基因表達量與肺癌患者的預后生存分析有相關性(P<0.05);基因共分析顯示,在肺癌中PTGER4、CD36、KRAS等基因與KEAP1基因表達呈負相關(P<0.05);KEAP1在肺癌中主要通過KEAP1-NFE2L2 通路發揮作用;與正常肺組織相比,KEAP1基因突變后在肺癌組織中表達異常下降(P<0.05)。結論" KEAP1突變與肺癌患者更短的生存期相關,該基因的降低與肺癌的發生和進展有關。在肺癌的驅動基因中,KEAP1在肺癌患者中屬于突變風險較高的基因,有望成為肺癌預后評估和治療的有效靶點。
關鍵詞:肺癌;KEAP1基因;生物信息學;預后;生存期
中圖分類號:R734.2" " " " " " " " " " " " " " " " " " " 文獻標識碼:A" " " " " " " " " " " " " "DOI:10.3969/j.issn.1006-1959.2024.18.003
文章編號:1006-1959(2024)18-0012-07
Abstract:Objective" To analyze the expression and significance of human cytoplasmic adaptor protein 1 (KEAP1) in lung cancer by bioinformatics technology, and to provide a strong basis for the follow-up treatment and prognosis of lung cancer.Methods" GEO database was used to mine the expression and chromosomal localization of KEAP1 gene. Kaplan-Meier database was used to analyze the relationship between KEAP1 expression and prognosis of patients with lung cancer. Linkedomics database and String database were used to obtain the expression data and protein interaction network of KEAP1 gene in different tissues and diseases, the relationship between KEAP1 and other proteins was analyzed, and the association and regulatory network with other genes was explored. The expression of KEAP1 was analyzed by GEPIA2 database to compare the expression of KEAP1 in normal lung tissues and lung cancer tissues.Results" KEPA1 gene was located on chromosome 19, and the expression of this gene was correlated with the prognosis and survival analysis of patients with lung cancer (Plt;0.05). Gene co-analysis showed that PTGER4, CD36, KRAS and other genes were negatively correlated with KEAP1 gene expression in lung cancer (Plt;0.05). KEAP1 played a role in lung cancer mainly through the KEAP1-NFE2L2 pathway; compared with normal lung tissues, the expression of KEAP1 in lung cancer tissues decreased significantly after gene mutation (Plt;0.05).Conclusion" KEAP1 mutation is associated with shorter survival time in patients with lung cancer, and the decrease of this gene is related to the occurrence and progression of lung cancer. Among the driving genes of lung cancer, KEAP1 is a gene with a high risk of mutation in patients with lung cancer, which is expected to become an effective target for prognosis evaluation and treatment of lung cancer.
Key words:Lung cancer;KEAP1 gene;Bioinformatics;Prognosis;Survival period
肺癌(lung cancer)是美國癌癥相關死亡的主要原因,平均5年生存率為15%。吸煙仍然是肺癌的主要危險因素。肺癌分為小細胞癌及非小細胞癌(如腺癌、鱗狀細胞癌、大細胞癌)[1]。盡管目前很容易識別出有患肺癌風險的人群,但現有的證據并不支持對肺癌進行大規模篩查。對于早期診斷肺癌的患者,通過手術切除和術后化療可能治愈,切除的程度取決于腫瘤的大小、位置以及患者的術前肺儲備[2]。然而,在絕大多數肺癌患者中,被發現時已處于晚期階段,可用的治療策略有限。因此尋找有效的分子靶標對肺癌的治療和預后具有重要的臨床意義[3]。KEAP1 是一種高度保守的富含半胱氨酸的624個氨基酸的蛋白質,在哺乳動物中具有大約92%的序列同源性。KEAP1屬于BTB-Kelch家族蛋白,該家族的所有成員都能夠通過其BTB結構域與CUL3結合[4]。Nrf2轉錄因子控制著參與細胞防御氧化應激的數百個基因的表達。Nrf2活性調節的主要途徑是通過與KEAP1蛋白的相互作用。在常氧下,KEAP1結合Nrf2并將其靶向蛋白酶體降解,而KEAP1再生。在氧化應激下,Nrf2和KEAP1之間的相互作用被中斷,Nrf2激活保護基因的轉錄。目前,Nrf2系統激活被認為是治療不同病理的強大細胞保護策略[5]。KEAP1-Nrf2系統就像一把雙刃劍:Nrf2 活性保護細胞并使細胞抵抗氧化和親電應激,而升高的 Nrf2 活性有助于癌細胞的存活和增殖。KEAP1基因的缺失或突變會導致Nrf2蛋白的過度激活,從而促進肺癌細胞的生長和轉移和抵抗化療藥物[6]。為此,本研究通過GEO數據庫、Kaplan-Meier等數據庫分析KEAP1基因在肺癌中的表達和預后關系,旨在為后續研究提供參考。
1資料與方法
1.1數據來源" 在GEO數據庫(https://www.ncbi.nlm.nih.gov/geo/)輸入關鍵詞“KEAP1”,數據集篩選:選擇與肺癌相關的數據集。
1.2生存分析" 在Kaplan-Meier數據庫(http:kmplot.com/)的mRNA gene chip一欄中選擇“Start KM plotter”,Gene Symbol選擇“KEAP1”,Split patients by“Auto”,篩選性能最佳的閾值作為截止值。Survival選擇“OS or PPS”。分析KEAP1基因表達與肺癌患者生存率之間的關系。
1.3相關性分析" 在Linkedomics數據庫(http:www.linkedomics.org/)選擇癌癥類型“TCGA_LUAD”,選擇研究數據“TCGA LUAD HiSeq RNA”,輸入基因“KEAP1”,選擇數據集“TCGA LUADHiSeq RNA ”,確定統計學方法 “Pearson Correlation test”。
1.4 KEAP1相互作用蛋白分析" 在String-db數據庫(http: string-db.org)中,Protein Name輸入“KEAP1”基因,Organisms選擇“Homo sapiens”,獲取KEAP1的蛋白相互作用網絡。
1.5組織表達差異分析" 在GEPIA2數據庫(http://gepia2.cancer-pku.cn/)以“KEAP1”作為關鍵詞進行搜索,選擇與LUAD相關的數據集,基因表達分析點擊“Expression”標簽,選擇腫瘤類型為“LUAD”,觀察KEAP1在LUAD腫瘤與正常組織中的表達水平。
1.6免疫組化表達情況" 在The Human Protein Atlas數據庫(https://www.proteinatlas.org/)中,關鍵詞輸入“KEAP1”,選擇肺組織數據集:點擊“Tissue specificity”,選擇“Respiratory system”,再選擇“Lung”,查看KEAP1在正常肺組織和肺癌組織中的免疫組化圖像。
1.7統計學方法" 采用SPSS 20.0進行統計分析,計量資料以(x±s)表示,配對樣本比較使用配對t檢驗;采用Kaplan-Meier法分析KEAP1表達與預后的關系,應用Log-rank檢驗比較組間生存率,P<0.05為差異有統計學意義,P<0.01為統計學意義顯著。
2結果
2.1 KEAP1基因結構及數據集表達情況" KEAP1基因是人類基因組中的一種重要基因,位于染色體19p13.2位點。KEAP1蛋白質屬于Cullin3-RBX1 E3泛素連接酶復合物的組分之一,并與核因子E2相關因子2(Nrf2)相互作用。該復合物在細胞中通過對Nrf2的調控,參與了氧化應激反應和抗氧化防御系統的調節[7],見圖1。
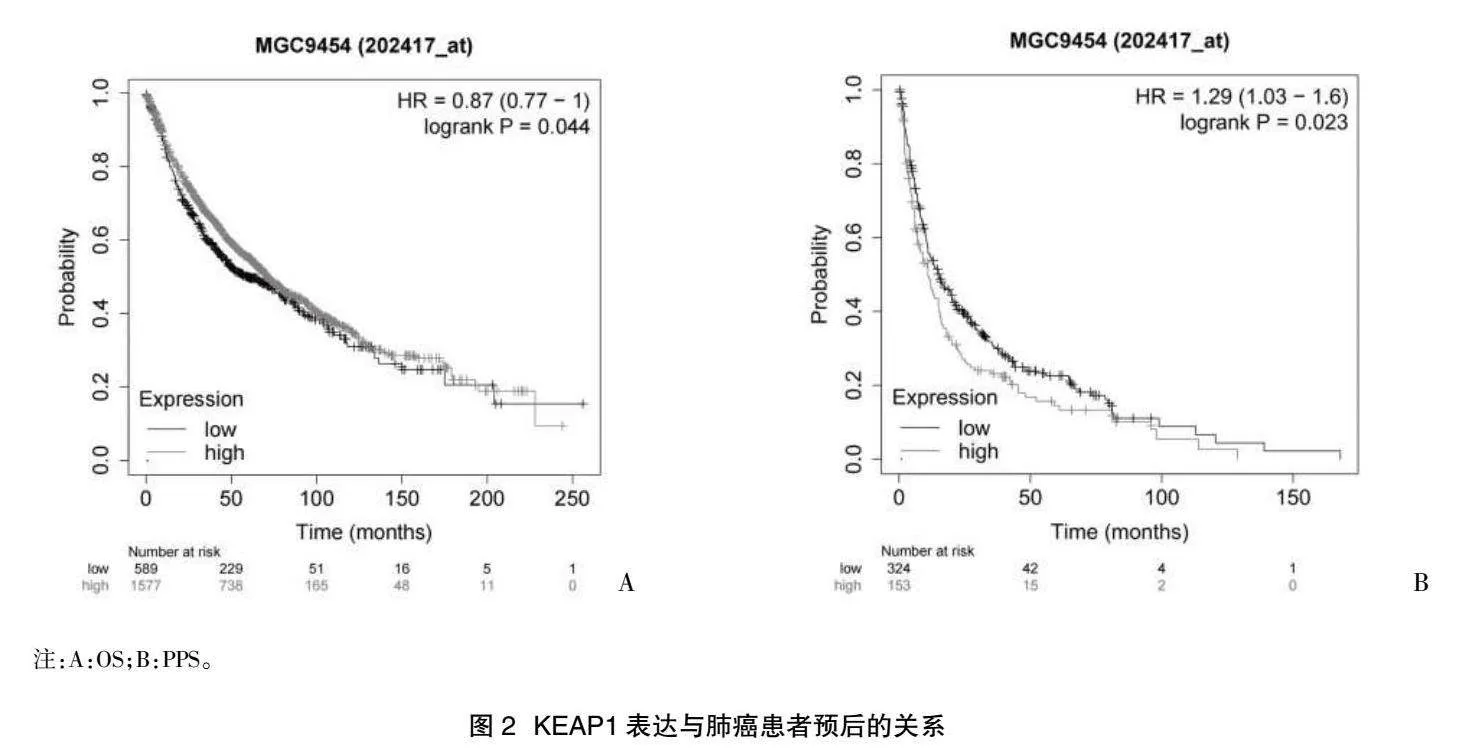
2.2 KEAP1表達與肺癌患者預后的關系" Kaplan-Meier分析顯示,KEAP1 mRNA表達量與肺癌患者的OS和PPS均有相關性(OS:Log-rank P=0.044;PPS:Log-rank P=0.023),見圖2。
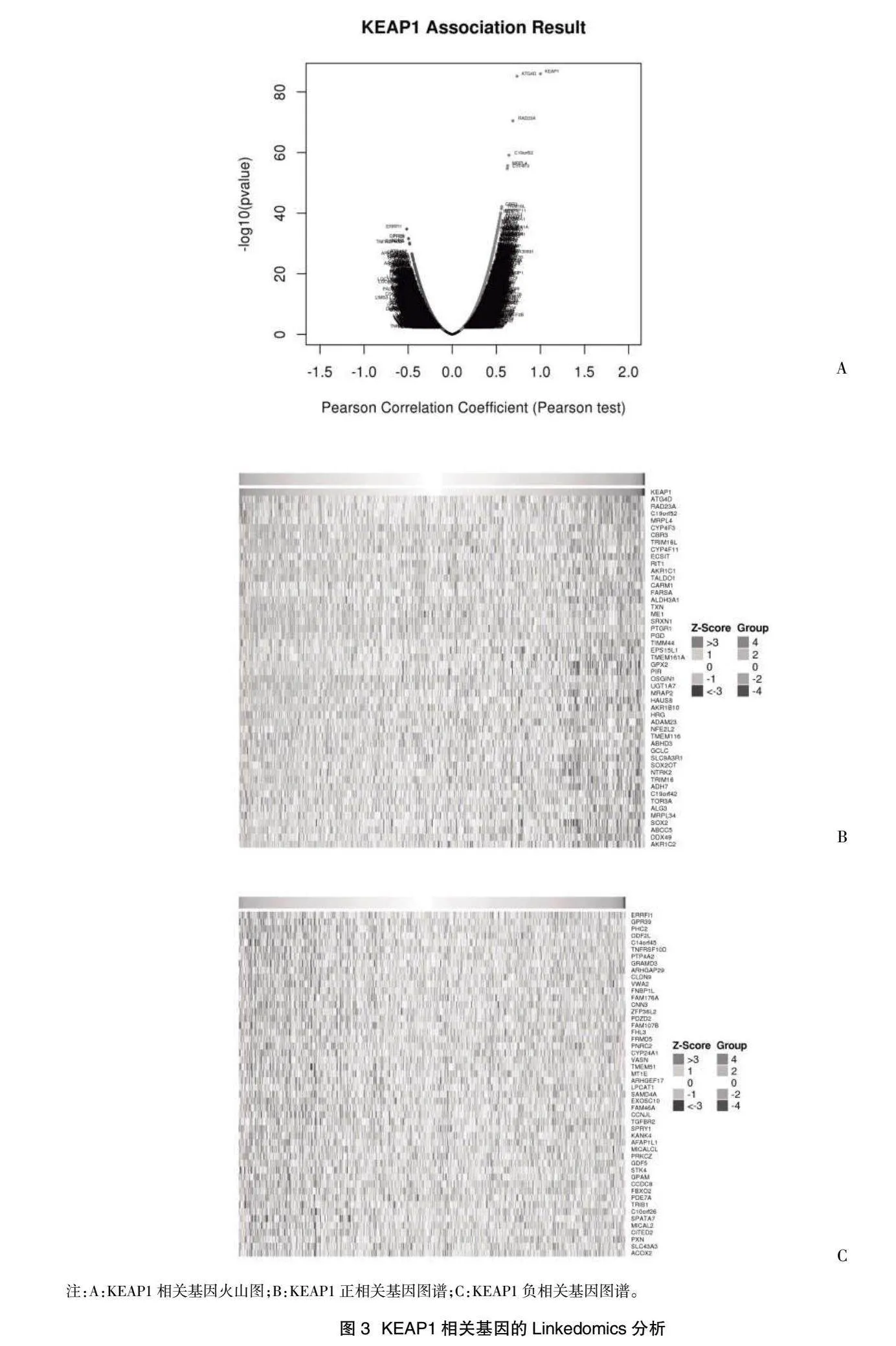
2.3 KEAP1相關基因的Linkedomics分析" 基因共表達分析顯示,與KEPA1呈正相關的基因和與KEPA1呈負相關的基因大概有50多種,見圖3。
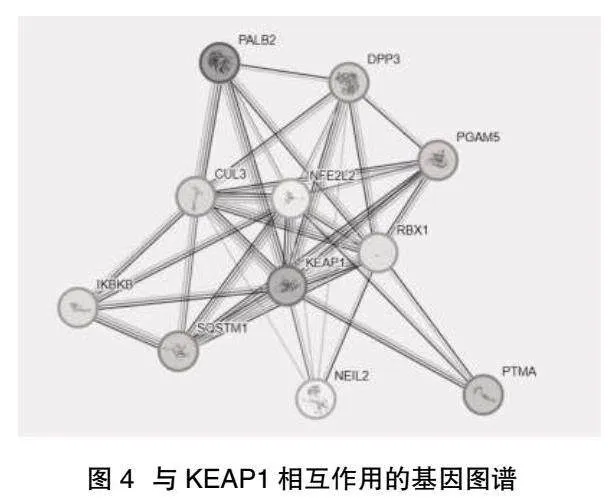
2.4 KEAP1在肺癌組織String數據分析" KEAP1與SQSTM1、CUL3、NEIL2、NFE2L2、IKBKB、RBX1等基因關系密切,見圖4。根據通路分析發現,KEAP1在肺癌中主要通過KEAP1-NFE2L2 通路發揮作用。
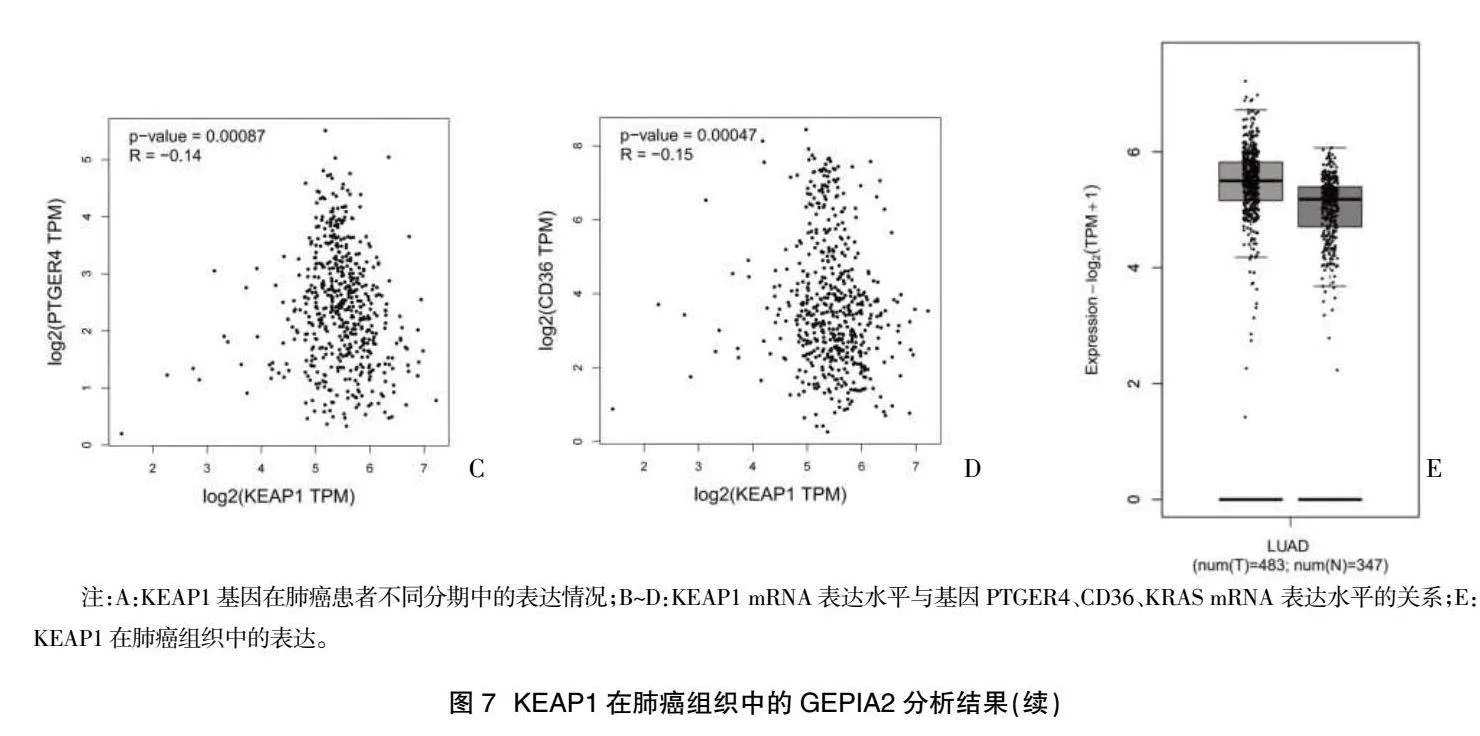
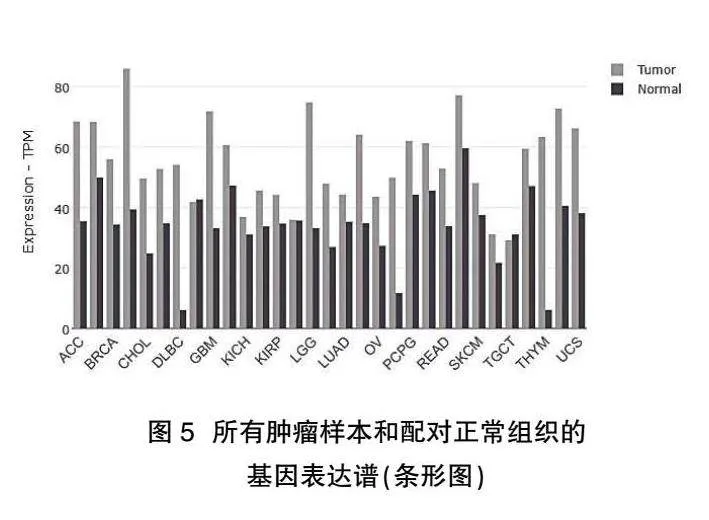
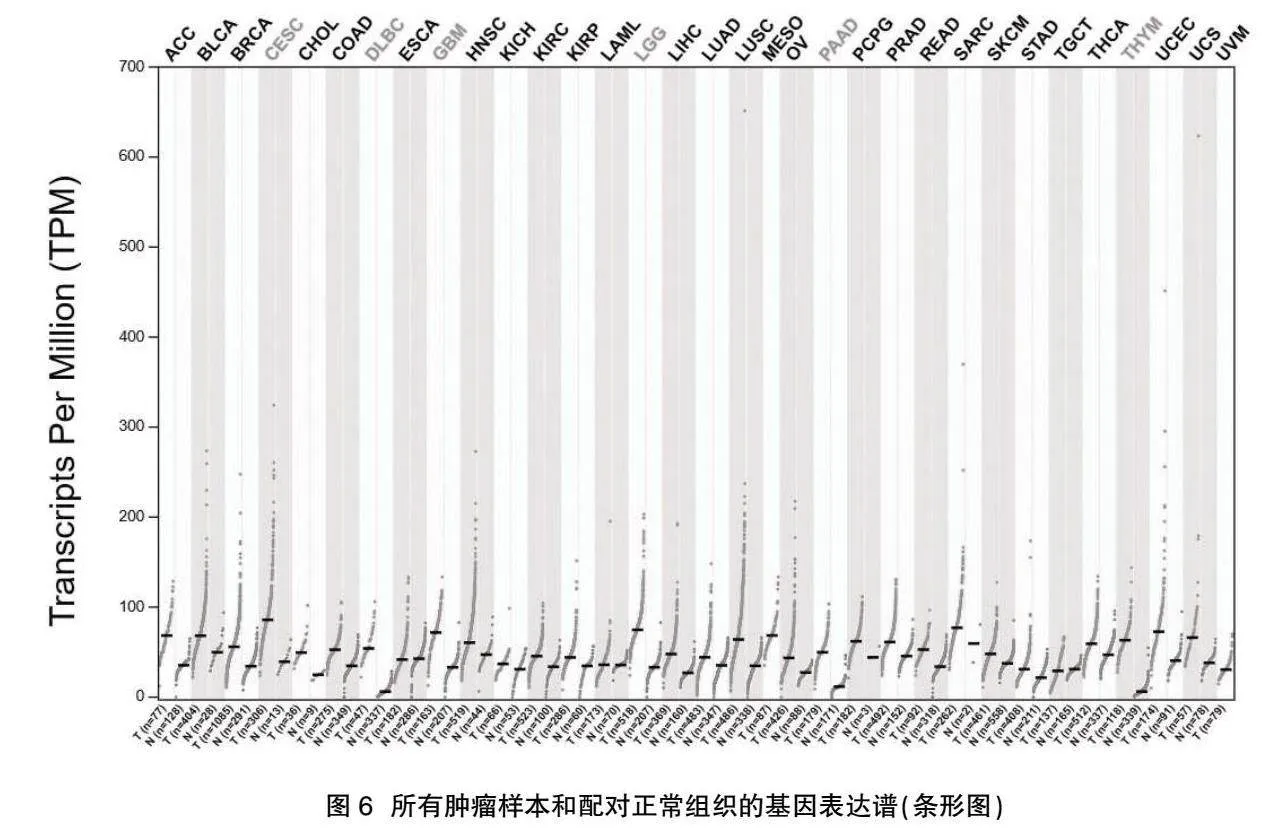
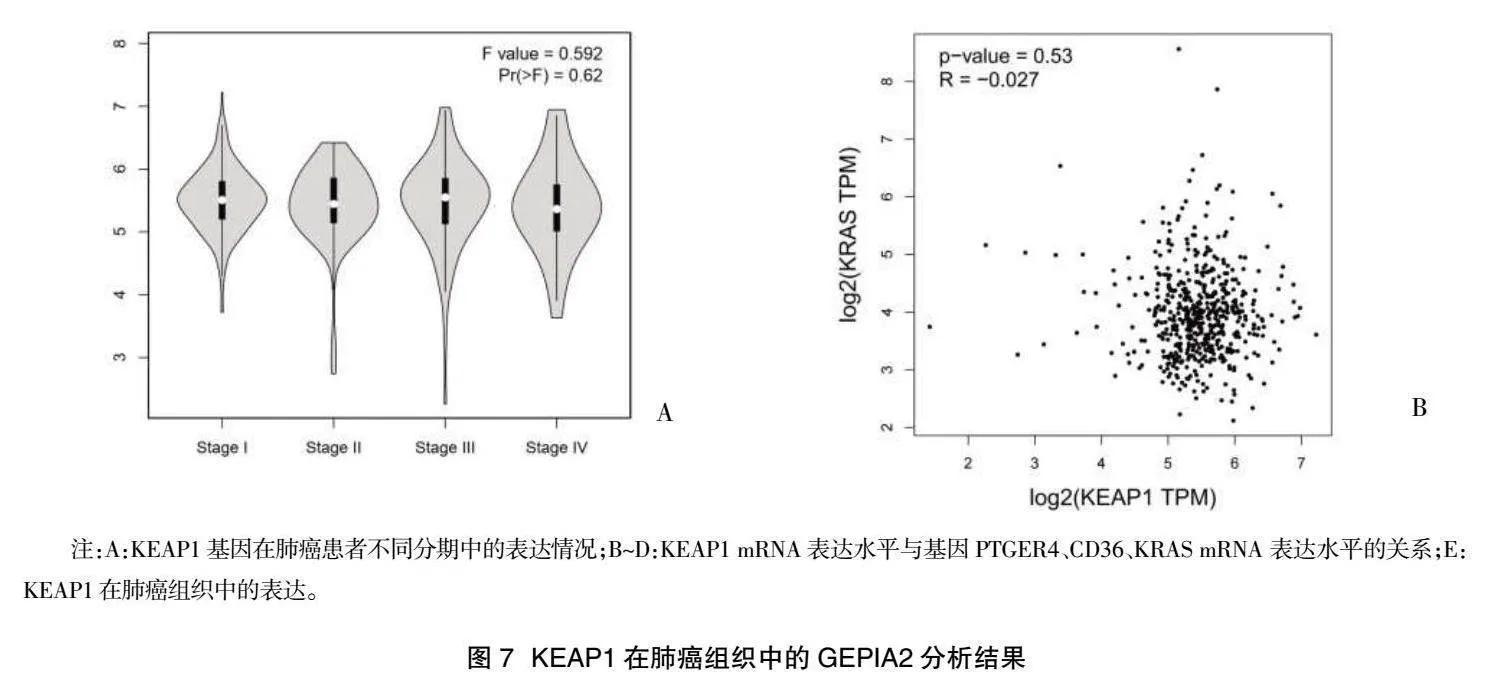
2.5 KEAP1在肺癌組織中的GEPIA2分析結果" KEAP1基因在大多數腫瘤組織中均呈高表達,但在胰腺癌(PAAD)、胸腺瘤(THYM)、淋巴樣腫瘤彌漫性大B細胞淋巴瘤(DLBC)及腦低級膠質瘤(LGG)中KEAP1表達水平比其相應的正常組織中的表達水平顯著增高,見圖5、圖6。為明確KEAP1基因表達與肺癌患者病理分期是否存在統計學意義,在GEPIA2數據庫輸入KEAP1基因并進行數據挖掘,結果顯示:KEAP1mRNA在不同病理分期肺癌患者中的表達存差異(F=0.592,P=0.62),見圖7A。基于GEPIA2數據庫挖掘與KEAP1相關基因分析結果顯示,肺癌組織KEAP1的mRNA表達水平與基因PTGER4、CD36、KRASmRNA 表達水平呈負相關(P<0.05),見圖7B~圖7D。通過GEPIA2分析KEAP1肺癌數據集,該數據集中包括例肺癌組織樣本和例正常肺組織樣本。結果顯示,與正常肺組織相比,KEAP1基因突變后在肺癌組織中表達異常,差異具有統計學意義(P<0.05),見圖7E。
2.6 KEAP1在正常肺臟與肺癌的免疫組化" 在The Human Protein Atlas 數據庫中得到KEAP1蛋白在1例正常肺組織和12例肺癌組織中的表達情況,其中2例肺癌組織蛋白表達及抗體染色程度為“高度”水平,4例為“中度”水平,5例為“低度”水平,1例未檢測到染色結果,見圖8。
3討論
在全球范圍內,肺癌在過去幾十年中一直是最常見的癌癥,是北美和其他發達國家癌癥相關死亡的主要原因[8]。肺癌通常直到晚期才被診斷出來,其死亡率較高。因而,肺癌的早期診斷至關重要,特別是在篩查高危人群時,如吸煙者、暴露于煙霧、油田、有毒職業場所時,需要確定用于早期診斷的敏感和特異性分子標志物[9]。2004年,肺癌遺傳流行病學聯盟首次發現6q23-25區域存在影響肺癌風險的主要易感位點[10]。隨著全基因組學的發展,現在有可能更全面地探索人類基因組,以了解遺傳單核苷酸多態性(snp)與人類疾病之間的關聯。有研究已經成功地確定了與肺癌易感性顯著相關的遺傳因素,相關證據的強度各不相同,一些基因座已被細化為特定的亞組,包括性別、種族、吸煙狀況和組織學亞型[11]。
KEAP1/Nrf2通路被認為是氧化還原穩態的關鍵調節因子,在生理環境下保護細胞免受氧化應激和外源物的影響。癌細胞在起始和進展過程中經常經歷這一途徑,異常的KEAP1-Nrf2活性主要在非小細胞肺癌(NSCLC)中被發現,這表明細胞/起源組織可能會影響惡性轉化過程中的遺傳選擇[12]。KEAP1在約16%的NSCLC中發生突變,包括12%的肺鱗狀細胞癌(LUSCs)和20%的肺腺癌(LUADs)[13]。KEAP1突變的肺癌患者總生存期較短,預后較差[14-16]。有研究報告了Nrf2介導的轉錄在預防癌癥方面的潛在作用是由涉及異生物質和氧化應激的機制引起的。這表明 KEAP1-Nrf2系統是通過Nrf2激活的調節劑(抑制劑/激活劑)來對抗癌癥的潛在治療靶點[6]。
KEAP1基因是人類基因組中的一種重要基因,位于染色體19p13.2位置[17]。本研究顯示,KEAP1 mRNA表達量與肺癌患者的OS和PPS均有相關性,與KEAP1mRNA低表達組相比,KEAP1 mRNA高表達組肺癌患者總體生存時間和進展生存時間縮短。通過Linkedomics數據庫對KEAP1基因進行分析,確定KEAP1基因在反應性氧化物種的清除過程中扮演著重要角色,揭示了與KEAP1相互作用的蛋白質,此外,還確定了KEAP1與其他信號通路蛋白的關聯,如Nrf2、PI3K/AKT等。KEAP1是調控氧化應激反應的關鍵基因,它通過調控Nrf2的穩定性和轉錄活性來調節細胞對氧化應激的反應[18]。根據GEPIA2的分析結果,可以看到KEAP1基因在正常組織和肺癌組織中的表達差異。在某些肺癌類型中,KEAP1基因的表達水平下降;同時,一些突變體和功能失活的KEAP1也被發現[19]。基于GEPIA2數據庫基因相關性分析結果顯示,在肺癌中過表達的PTGER4[20]、CD36[21]、KRAS[22]等基因與KEAP1基因表達呈負相關。這意味著KEAP1基因的降低與肺癌的發展和進展有關。需要注意的是,GEPIA2的分析結果雖然提供了有關KEAP1在肺癌組織中的表達變化的信息,但仍需要進一步的實驗驗證和研究來確定其確切的功能和調控機制。因此,這些結果僅作為初步的參考,需要與其他研究結果相結合來全面理解KEAP1在肺癌中的作用。利用THPA數據庫進行KEAP1免疫組化分析,發現KEAP1在正常生理狀態和疾病中的表達模式和定位信息存在差異。
綜上所述,KEAP1突變與肺癌患者更短的生存期相關,該基因的降低與肺癌的發生和進展有關。在肺癌的驅動基因中,KEAP1在肺癌患者中屬于突變風險較高的基因,有望成為肺癌預后評估和治療的有效靶點。
參考文獻:
[1]Bade BC,Dela Cruz CS.Lung Cancer 2020: Epidemiology, Etiology, and Prevention[J].Clin Chest Med,2020,41(1):1-24.
[2]Nasim F,Sabath BF,Eapen GA.Lung Cancer[J].Med Clin North Am,2019,103(3):463-473.
[3]彭旭紅,蔣靜,屈佳肴,等.基于數據挖掘分析Elafin在肺癌中的表達及意義[J].天津醫藥,2021,49(2):169-174.
[4]Baird L,Yamamoto M.The Molecular Mechanisms Regulating the KEAP1-NRF2 Pathway[J].Mol Cell Biol,2020,40(13):e00099-20.
[5]Ulasov AV,Rosenkranz AA,Georgiev GP,et al.Nrf2/Keap1/ARE signaling: Towards specific regulation[J].Life Sci,2022,291:120111.
[6]Deshmukh P,Unni S,Krishnappa G,et al.The Keap1-Nrf2 pathway: promising therapeutic target to counteract ROS-mediated damage in cancers and neurodegenerative diseases[J].Biophys Rev,2017,9(1):41-56.
[7]Dinkova-Kostova AT,Kostov RV,Canning P.Keap1, the cysteine-based mammalian intracellular sensor for electrophiles and oxidants[J].Arch Biochem Biophys,2017,617:84-93.
[8]Bray F,Ferlay J,Soerjomataram I,et al.Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA Cancer J Clin,2018,68(6):394-424.
[9]Nooreldeen R,Bach H.Current and Future Development in Lung Cancer Diagnosis[J].Int J Mol Sci,2021,22(16):8661.
[10]Bailey-Wilson JE,Amos CI,Pinney SM,et al.A major lung cancer susceptibility locus maps to chromosome 6q23-25[J].Am J Hum Genet,2004,75(3):460-474.
[11]Bossé Y,Amos CI.A Decade of GWAS Results in Lung Cancer[J].Cancer Epidemiol Biomarkers Prev,2018,27(4):363-379.
[12]Xu K,Ma J,Hall SRR,et al.Battles against aberrant KEAP1-NRF2 signaling in lung cancer: intertwined metabolic and immune networks[J].Theranostics,2023,13(2):704-723.
[13]Cancer Genome Atlas Research Network.Comprehensive molecular profiling of lung adenocarcinoma[J].Nature,2014,511(7511):543-550.
[14]Jeong Y,Hoang NT,Lovejoy A,et al.Role of KEAP1/NRF2 and TP53 Mutations in Lung Squamous Cell Carcinoma Development and Radiation Resistance[J].Cancer Discov,2017,7(1):86-101.
[15]Arbour KC,Jordan E,Kim HR,et al.Effects of Co-occurring Genomic Alterations on Outcomes in Patients with KRAS-Mutant Non-Small Cell Lung Cancer[J].Clin Cancer Res,2018,24(2):334-440.
[16]Chen X,Su C,Ren S,et al.Pan-cancer analysis of KEAP1 mutations as biomarkers for immunotherapy outcomes[J].Ann Transl Med,2020,8(4):141.
[17]Goel MK,Khanna P,Kishore J.Understanding survival analysis: Kaplan-Meier estimate[J].Int J Ayurveda Res,2010,1(4):274-278.
[18]Szklarczyk D,Gable AL,Nastou KC,et al.The STRING database in 2021: customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets[J].Nucleic Acids Res,2021,49(D1):D605-D612.
[19]Tang Z,Kang B,Li C,et al.GEPIA2: an enhanced web server for large-scale expression profiling and interactive analysis[J].Nucleic Acids Res,2019,47(W1):W556-W60.
[20]Zhang Y,Huang J,Zou Q,et al.Methylated PTGER4 is better than CA125, CEA, Cyfra211 and NSE as a therapeutic response assessment marker in stage IV lung cancer[J].Oncol Lett,2020,19(4):3229-3238.
[21]Liu LZ,Wang B,Zhang R,et al.The activated CD36-Src axis promotes lung adenocarcinoma cell proliferation and actin remodeling-involved metastasis in high-fat environment[J].Cell Death Dis,2023,14(8):548.
[22]Ricciuti B,Son J,Okoro JJ,et al.Comparative Analysis and Isoform-Specific Therapeutic Vulnerabilities of KRAS Mutations in Non-Small Cell Lung Cancer[J].Clin Cancer Res,2022,28(8):1640-1650.
收稿日期:2023-09-26;修回日期:2023-10-09
編輯/成森
基金項目:國家自然科學基金項目(編號:81760236)
作者簡介:張馨文(1999.7-),女,山西侯馬人,碩士研究生,主要從事糖尿病神經科學研究
通訊作者:趙貝(1983.1-),男,山東棗莊人,博士,副教授,主要從事糖尿病神經科學研究

