數據倉庫技術在生物信息學中的應用研究
潘 偉
(西華師范大學計算機學院,四川南充637002)
1 引言
生物信息學是在生命科學的研究中,生物學與計算機科學及應用數學等多學科相互交叉而形成的一門新興的綜合性學科[1]。它以海量生物學實驗數據為基本研究對象,進行數據的獲取、加工、存儲、檢索與分析,從而實現揭示相關生物學意義的目的。在人類基因組計劃的推動下,各種類型的生物數據,如核酸序列、蛋白質序列和蛋白質結構的生物信息數據,呈現指數增長的迅猛趨勢。為了對這些規模龐大、結構復雜的生物數據進行有效的管理和使用,早在七、八十年代,世界各地的科研人員就建立了大量的生物信息數據庫,現在它們已經成為生物信息學工作的基礎和重要內容。
目前,廣泛使用的生物信息數據庫主要有美國國家生物信息中心(NCBI)管理維護的GeneBank、歐洲生物信息研究所(EBI)管理的EMBL數據庫、以及日本的國家級核酸數據庫DDBJ,以及共同組成的GeneBank/EMBI/DDBJ國際核酸序列數據庫,定時保持同步更新。然而,在實際使用中,傳統的數據庫技術已經不能很好的勝任,將數據倉庫思想引進生物信息數據的管理則應運而生。
基于生物信息數據的特點,針對目前各大基因數據庫之間互聯、互通和相互利用中存在的一些問題,設計了基于數據挖掘技術的異地異構生物信息共享的數據倉庫解決方案,并給出了該方案的實現框架——基于Web的生物信息數據倉庫(Data Warehouse of Bioinformation based on Web,DWBW)。
2 生物信息數據庫的現狀及存在的問題
生物信息學是一門生命科學與數學、計算機科學和信息科學等交匯融合所形成的交叉學科[1]。在生物信息學的發展過程中,逐步建立起了大量基于網絡的生物數據庫,如PDB生物大分子結構數據庫、SWISS-PROT蛋白質序列數據庫和GenBank核酸序列數據庫等;開發了眾多檢索工具,如SRS、CLUSTALW、PROSITESEARCH等強有力的搜索工具;從而實現了生物信息數據的智能處理和綜合分析。目前,生物信息數據庫中主要包括核酸數據、蛋白質數據以及功能數據等內容,來自于世界各地不同的實驗室。有些是從實驗獲得的未經任何處理的原始數據,有些經過簡單的歸類整理和注釋,有些則是針對特定目標通過理論分析和復雜處理得到的。因此,生物信息數據在具有增長迅猛、更新及時、種類繁多等特點的同時,更表現出高度的復雜性、多樣性和不一致性。
隨著生物信息數據量的激增以及數據處理能力的復雜程度不斷提高,現有的生物信息數據庫已經逐漸暴露出許多問題。三大核酸數據庫依靠傳統方式交換數據,無法及時反應出其他專用生物信息數據庫(特別是三大核酸數據庫以外的其它生物信息數據庫)的變化情況以及非核酸類數據信息。網上所提供的大部分數據分析工具采用面向問題的搜索方法,搜索效率隨著問題規模的擴大而降低,并且無法智能地對數據中存在的未知知識進行發掘。為此,在后基因組時代,需要將數據倉庫思想及智能數據挖掘技術運用到生物信息學領域中。
3 基于生物信息數據倉庫的數據挖掘技術
3.1 生物信息數據庫中的算法工具
生物信息學是一門內涵非常豐富的學科,是生物、數學、計算機等多領域專家的共同協作的成果。在生物信息學中通常需要進行基因比對、基因預測和功能基因組信息分析等工作,主要涉及了大規模基因表達譜分析的相關算法、基因表達調控網絡的研究、軟件研究、核酸、蛋白質空間結構的預測和模擬、以及蛋白質功能預測等方面。目前,在生物信息學中,基因比對(Alignment)是最常用和最經典的研究手段。在核酸序列或蛋白質序列之間進行兩兩比對,比較兩個序列之間的相似區域和保守性位點,尋找二者的相似形、同源性,進而探尋可能的分子進化關系,揭示序列中蘊涵的結構、功能等信息。
數據挖掘主要存在以下幾種經典分析模式[2]:關聯模式分析、序列模式分析、分類分析、聚類分析,其中關聯模式分析應用最為廣泛。關聯模式分析通過對一組給定的Item和一個記錄集合進行分析,推導出Item間的相關性,進而挖掘隱藏的關系。可見,通過數據挖掘技術,開發面向功能解釋和功能預測的工具,能夠發現不同基因、蛋白質序列中的相關性,從而進一步比較分析其功能。
3.2 OLAP和數據挖掘技術
傳統的針對數據庫的開發工具多為聯機事物處理(Online Transaction Process,OLTP)模式,它主要是面向具體的查詢和統計,有著較為具體的應用目的。然而,隨著越來越多數據庫的出現,數據量的迅猛增長,OLTP在數據資源的充分利用、為用戶提供有效支持和幫助等方面,則顯得力不從心[2]。因此,基于數據倉庫的聯機分析處理(Online Analysis Process,OLAP)以及數據挖掘(Data Mining,DM)引起了日益廣泛的關注和應用。
OLAP是一種自上而下、不斷深入的分析工具。用戶提出問題或假設,OLAP則負責從上而下深入地提取出關于該問題的詳細信息,并以可視化的方式呈現給用戶。DM是一種決策支持過程和挖掘性工具,它主要基于人工智能、機器學習、統計學等技術,高度自動化地對原始數據進行分析,發現隱藏在數據中的模式,做出歸納性和預測性的推理。
3.3 基于生物信息數據倉庫的數據挖掘技術
目前生物信息數據庫的特點以其所存在的問題,都為數據倉庫的使用提供廣闊的活動空間,但同時也對數據挖掘提出了新的問題和難點。生物信息學的進一步發展需要數據倉庫的支撐。
實現數據挖掘的一個前提條件是必須具有海量數據[2],而這恰恰是數據倉庫的基本特點之一,二者的緊密結合可以有效地解決大量實際應用中出現的問題。在生物信息學領域,使用數據挖掘技術可以大幅提高研究人員的工作效率,改變原有的利用傳統工具人為的或機械的逐項比較功能的預測法;數據挖掘算法還可以結合生物信息專業領域的公式算法,根據研究人員的設想,對數據進行全面的、高效率的分析。
基于數據挖掘等技術發展起來的智能決策技術[3]具備了在生物信息學領域中大展身手的“天賦”,特別是基于海量數據的數據挖掘和開采技術更具有廣泛的需求和應用背景。另一方面,目前生物信息學的研究方法和發展情況,已經形成了一整套較為完善的智能決策系統,成為數據倉庫和數據開采的一個較為成功的應用案例。另外,現有的各種生物信息應用分析軟件,雖然其基本出發點仍然是傳統的數據庫技術,但是從一定程度上講,已經反映并且實現了數據倉庫及數據挖掘的思想和技術。
4 實現生物信息異地多源數據庫的綜合利用
4.1 建立基于Web的生物信息數據倉庫
已有的生物信息數據庫都已經提供基于互聯網的數據提交、序列查詢、基因比對等功能,且幾大數據庫之間也實現了準實時的互聯互通。但是,基于這些生物信息數據的分析比較工具,都不能從面向主題的角度對全部數據進行分析以找出具有價值的規律,更不具有決策支持的能力。基于現有的生物信息數據庫,運用數據倉庫思想,利用OLAP和數據挖掘技術,建立生物信息數據倉庫,是一種不需要大幅增加硬件設備以及物理裝置的前提下,實現基于Web平臺的生物信息集成與處理平臺的可行方案。
設計一個基于Web的生物信息數據倉庫(Data Warehouse of Bioinformation based on Web DWBW),其主要結構由5個主要部分組成:外部數據源,包括各大綜合生物信息數據庫以及各類專用生物信息數據庫系統;基于Web的數據調度,傳輸;數據的“變形”(抽取、變換、裝載、刷新等)整合;元數據規則,以及元數據的管理;基于Web的綜合管理平臺(包括請求分類服務器、各種分析工具服務器)。
DWBW以DDBJ/EMBI/GeneBank作為主要數據源,同時以其他主要的生物信息數據庫作為整個數據倉庫的基本數據源。通過分析數據庫結構和數據結構類型,建立DWBW的元數據規則,即為來源不同的數據建立統一的數據結構、字段內容、存儲結構等基本項目。這樣,基于上述各種數據庫就可以建立起一個基于Web的虛擬數據倉庫。由于不存在一個物理上位于某地的存儲中心來保存這些數據,用戶面對的只是一個基于網絡的分布式虛擬的數據倉庫。其模型、算法的運行由專用的分析工具服務器來完成,而對于數據的調用和整理都是在源數據庫上進行的,根據不同用戶提交問題的不同,具體的分析過程則由分配服務器交給相關的服務器去完成分析工作,只是最終將結果返回請求分類服務器,顯示給用戶。DWBW的原理框圖見圖1所示。
DWBW將通過一個統一的基于Web的頁面與用戶進行交互,在后臺則主要由請求分類服務器、Web服務器以及專用分析工具服務器共同完成對用戶問題的提交和分析,實現對數據倉庫元數據規則的理解,然后根據問題分類的結果,與相關的分析工具進行匹配,將問題提交到相應的專用分析工具服務器進行處理。處理完成后返回給請求分類服務器,進而返回給提交問題的用戶。
4.2 DWBW關鍵技術
4.2.1 生物信息學元數據的抽取
數據的整理和抽取,是開發所有數據倉庫時所遇到的最難解決也是最具挑戰性的問題之一。將不同時期生成的大量歷史數據中的數據結構、字段的定義以及對象之間的關系等一系列的描述信息整理出來,并制定出一套通用可行的規范,本身就是一個工作量巨大、難度極高的問題;特別針對來自于世界各地、不同門類、不同背景、不同應用目的的專業生物信息數據庫,就更加復雜了。因此,數據倉庫元數據的抽取整理,成為建立DWBW的關鍵問題。數據抽取、清洗、轉換和裝載過程與一般數據倉庫建立過程中的數據整理不完全相同,其數據具有規范性強,存儲結構相對簡單,轉換、裝載容易等特點。
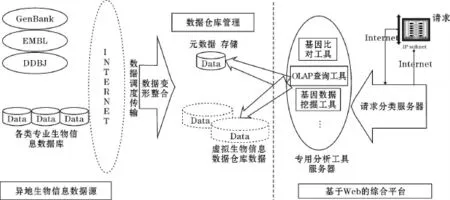
圖1 基于Web的生物信息數據倉庫系統(DWBW)
4.2.2 虛擬生物信息數據倉庫的建立
虛擬生物信息數據倉庫實現對生物信息平臺相關數據的組織和集成,并且將不同主題的數據對象分別存儲到各個數據集市中,同時還將建立起部分有價值數據的在線OLAP數據庫。與傳統的數據倉庫不同,虛擬數據倉庫采用中間件充當數據中心,提供信息的訪問接口,對存貯在不同數據源的生物信息數據進行存取操作。虛擬數據倉庫的中間件對各個數據源(各生物信息數據庫)的請求采用的是標準的XML,最終都將轉換為各數據源的標準查詢語言,提交給相應的數據庫服務器進行處理。數據庫處理完成后,中間件又將返回結果重新封裝成為XML格式的數據,并進行發送。由于虛擬生物信息數據倉庫一般不是針對實時數據進行分析統計,所以對于時間效率的要求不如一般查詢統計那么高,這就確保了開發處于不同物理位置數據源的虛擬DWBW具有可行性。同時,對于一般性的查詢比對等基本操作,不會影響其工作效率和準確性。
4.2.3 基于Web面向用戶的綜合平臺的開發設計
基于Web的面向用戶的綜合平臺主要提供一個用戶操作的平臺,除了集成基因比對、功能預測、序列分析、基因提交等傳統的操作以外,還允許用戶對虛擬數據倉庫中的數據運用數據挖掘技術,提供更多的分析支持工具。
5 DWBW適用性分析
DWBW與傳統的數據倉庫概念特征相比,兩者都是對海量的、復雜數據進行處理;能在解決“不明確問題”過程中顯示很強的能力;面向數據分析,把輔助決策作為一個重要的最終目標。DWBW還具有幾個特性:實時性要求較強;與一般的數據倉庫相比對單個記錄的準確性要求更高;數據倉庫不是物理存在的,而是通過互聯網絡將多個數據庫連接在一起,共同發揮作用的虛擬數據倉庫。
DWBW在解決目前互聯網中許多具有相似特性領域中的問題時具有很多的借鑒意義[4]。例如在基于網絡的B2B、B2C的商業網站的客戶資料分析、商品(貨品)銷售分析等方面都可以采用相同的思想。擴展開來,這一設計方法在大型的全球連鎖超市、各地的家電分銷中心以及股票市場的評估分析等等方面均有指導意義。在這方面成功的應用主要集中在大型連鎖超市的銷售記錄分析,股票、金融市場分析方面,它提供的輔助決策內容有貨物配售方案的選擇,超市選址,股票分類投資,防止金融詐騙等。
感謝西華師范大學科研啟動基金(05B061)
[1]鐘揚,張亮.簡明生物信息[M].北京:高等教育出版社,2001.
[2]王珊.數據倉庫技術與聯機分析處理[M].北京:科學出版社,1998.
[3]陳文偉.智能決策技術[M].北京:電子工業出版社,1998.
[4]劉智琚.數據挖掘在生物信息學中的應用[J].軟件導刊,2009,(7).
[6]楊文,韓濤,孫志茹.生物信息學序列庫與文獻庫的整合模式淺[J].實踐研究,2008,(1).

