海蜇養殖群體及自然捕獲群體ITS序列遺傳分析
孫國華, 劉相全, 楊建敏, 張錫佳, 劉愛英, 譚福奕
(1. 山東省海洋水產研究所, 山東 煙臺 264006; 2. 山東好當家海洋發展股份有限公司, 山東 威海264305)
海蜇養殖群體及自然捕獲群體ITS序列遺傳分析
孫國華1,2, 劉相全1, 楊建敏1, 張錫佳1, 劉愛英1, 譚福奕2
(1. 山東省海洋水產研究所, 山東 煙臺 264006; 2. 山東好當家海洋發展股份有限公司, 山東 威海264305)
為調查海蜇( Rhopilema esculentum)自然海區群體和養殖群體遺傳多樣性, 對煙臺萊州灣和江蘇海州灣自然海區捕獲群體及威海養殖群體 24個個體的 ITS序列進行了 PCR擴增和序列分析, 獲得
DNA片段長度在1 080~1 096 bp之間, 包括完整的ITS1, 5.8S和完整ITS2序列。結果表明, 在所測堿基序列中共發現26個變異位點, 4處多堿基插入位置, 29個插入缺失位點。群體內平均核苷酸差異數K和平均核苷酸多樣性指數Pi為2.179~2.750和0.002 02~0.002 54, 群體間平均核苷酸差異數K值波動在2.797~3.031之間, 遺傳距離在0.002 30~0.002 81之間, 遺傳分化指數(Fst)值在0.032 50~0.730 8之間, 群體內及群體間遺傳多樣性指標數值比較相近, 群體間的遺傳距離比較小, 群體遺傳結構相似,群體間沒有明顯的遺傳分化。
海蜇( Rhopilema esculentum); 轉錄間隔區(ITS); 遺傳多樣性; 遺傳結構
海蜇(Rhopilema esculentum)是腔腸動物門、缽水母綱、根口水母目、根口水母科、海蜇屬的大型食用水母, 為暖水性河口種類, 分布于北自遼寧南至海南島近岸海域, 資源豐富, 經濟價值大, 是我國重要漁業種類之一[1]。海蜇營養價值高, 含有豐富的蛋白質和脂肪酸[2,3], 是深受人民喜愛的水產品; 并且海蜇頭、皮、腹面黑膜等部位均可入藥, 具有清熱解毒、化痰軟堅、降壓消腫等藥用價值[4,5]。海蜇以浮游動物為食, 食物鏈短, 種群生命周期短, 資源數量年間波動大, 加之近幾年來海蜇價格逐年增高, 捕撈過度, 資源受到嚴重破壞[6,7]。海蜇池塘養殖業及增殖放流工作近幾年得到重視和發展, 但是海蜇分子生物學方面的基礎研究甚少, 有關群體遺傳多樣性研究尚比較欠缺。
核糖體基因轉錄間隔區(internal transcribed spacer ITS), 是位于核糖體DNA(rDNA)上18S和28S基因之間的區域片段, 主要包括內轉錄間隔區 1(ITS1)、5.8S rDNA、內轉錄間隔區2(ITS2) , 其兩側分別是18 S RNA基因和28 S RNA基因。與核糖體DNA中的 18S、5.8S和 28S的基因組序列相比較,ITS1和ITS2作為非編碼區, 承受的進化選擇壓力較小, 相對變化較大[8], ITS區段的擴增已普遍用來作為分類鑒定及分析近緣種和種群的系統發育關系的方法[9,10], 在水產動物中也有廣泛的應用[11~13]。
本研究利用 ITS序列多態性對兩個自然海區捕獲海蜇群體和一個人工養殖海蜇群體進行了遺傳結構分析, 以期從分子水平上了解海蜇資源的遺傳背景, 為海蜇種質資源保護及海蜇遺傳育種提供依據。
1 材料和方法
1.1 實驗材料
海蜇養殖群體于2008年7月取自山東好當家集團有限公司, 平均傘徑 420 mm; 江蘇野生群體于2008年8月取自海洲灣海域, 平均傘徑330 mm; 煙臺野生群體于2008年8月取自萊州灣海域, 平均傘徑360 mm。所有樣品取傘緣部組織凍存于-20℃。
1.2 DNA提取
樣品100 mg機械剪碎, 加入700 mL CTAB提取緩沖液(100 mmol/L Tris-HCl pH8.0, 20 mmol/L EDTA-Na2, 1.4 mol/LNaCl, 2% CTAB, 0.1%β-巰基乙醇)和終濃度為100 μg/mL蛋白酶K, 55℃消化3 h或37℃過夜, 等體積酚氯仿、氯仿抽提, 2倍體積乙醇沉淀, TE溶解, 4℃保存。
1.3 ITS區片段擴增、純化與克隆測序
ITS序列擴增使用 Heath等[14]設計的通用引物,引 物 序 列 為 : ITSF1(5′-GGTTTCCGTAGGTGAAC-CTGCGGAAGGATC-3′)和 ITSR1(5′-GCTTTGGGCT GCAGT CCCAA GCAA CCCACTC-3′)。一次擴增整個ITS區, 包括ITS1、5.8S rRNA和ITS2。PCR反應條件如下: 25 μL PCR反應體系: 包含約80 ng的基因組DNA, 10 pmol的引物, 2.5 μmolMgCl2, 0.2 μmol dNTP,Taq DNA聚合酶1 U。PCR反應條件為: 94℃ 4 min;94℃40 s, 退火溫度55℃4 0 s, 72℃延長40 s, 30個循環;72℃ 10 min; 4℃保溫, 每次均設空白對照。PCR產物1%瓊脂糖凝膠電泳檢測, 凝膠回收, PCR產物連接到pMD18T, 然后轉化到E.coliDH5α細胞, 37℃培養, 涂平板, 挑克隆, 載體引物檢測, 雙向測序。
1.4 序列分析用MEGA 4.0進行同源排序比對, 確定序列長度;計算不同序列間的堿基組成、變異位點、簡約信息位點和不同地理種群的 Kimura 2-paramter遺傳距離。采用Kimura 2-paramter距離矩陣采用鄰接法(NJ)構建單倍型分子系統樹, 系統樹中節點的自舉置信水平應用自引導(bootstrap)估計, 共 1 000次循環。用DNASP 4.50軟件計算各個群體的單倍型, 單倍型多態性, 多態位點數, 平均核苷酸差異數, 核苷酸多樣性指數, 基因流Nm等。用ARLEQUIN 3.11中的分子變異分析(AMOVA)分析方法估算遺傳變異在群體內和群體間的分布及遺傳分化系數(F-statistics,Fst), 計算并用排列測驗法(permutation test)檢驗Fst的顯著性(重復次數為1000)。
2 結果
2.1 擴增序列長度、結構及堿基組成
PCR擴增3個群體各8個個體共24條序列, 所得測序序列去載體及引物序列, 經Clustal對位排序,ITS序列長度在 1 080~1 096 bp之間, 包括完整的ITS1, 5.8S和完整ITS2序列, 圖1表示單倍型1堿基序列, 堿基 A、G、T、C和(A+T)含量分別 23.97%,25.56%, 25.98%, 24.42%和49.95%。同源片段序列對位排序和分析后, 在所測堿基序列中共發現26個變異位點, 這些變異位點包括19個轉換位點(其中T/C 12處, A/G 7處)和7個顛換位點(其中A/T 4處, G/T 2處, A/C1處, G/C1處), 其中簡約信息位點2個, 4處多堿基插入位置29個插入缺失位點, 插入序列分別為GTCGTTCG, AACAA, GAG, TGTC/TGTCTGTC。

圖1 海蜇轉錄間隔區(ITS)核苷酸序列Fig. 1 ITS nucleotide sequences of Rhopilema esculentum
2.2 三群體ITS序列的多態性分析
不計入插入缺失位點, 26個變異位點定義了17個單倍型, 在 17種單倍型中, 群體間共享單倍型有2個, 占單倍型總數的11.8%。單倍型2分別為煙臺,江蘇和養殖群體共有, 單倍型 12分別為煙臺, 江蘇群體所共有, 其他單倍型僅為某一個體獨有(表 1)。僅以29個插入缺失位點計算, 24條序列歸為9個單倍型, 共享單倍型5個, 其中單倍型Ⅱ和單倍型Ⅳ為 煙臺、江蘇和養殖3個群體所共有(表2)。

表1 海蜇3個群體ITS序列變異位點及定義單倍型Tab. 1 Variable nucleotide positions in ITS region of 17 haplotypes and number of individuals of each haplotype found in each locality

表2 海蜇3個群體ITS插入缺失位點及定義單倍型Tab. 2 Insertion-Deletion polymorphysm and the number of each haplotype in Rhopilema esculentum populations
2.3 海蜇各群體內及群體間遺傳多樣性分析
用DnaSP4.0軟件對海蜇各群體的遺傳多樣性參數進行計算, 結果列入表3。數據顯示, 在3個群體中, 煙臺和江蘇自然捕獲群體的單倍型(Nhap)要多于養殖群體, 3個群體中江蘇自然捕獲群體的堿基多態位點(S)比例是最高的。在平均核苷酸差異數K和平均核苷酸多樣性指數Pi這兩個指標上由ITS基因片段核苷酸序列反映出來的群體遺傳多樣性指數的絕對值水平不高, 3個群體數值比較相近, 江蘇自然捕獲群體遺傳多樣性相對而言比較高, 平均核苷酸差異數K和平均核苷酸多樣性指數Pi分別為 3.214和 0.004 29。
各群體之間的相關遺傳參數進行計算結果列入表 4。群體間平均核苷酸差異數K值波動在2.797~3.031之間, 群體間平均每位點核苷酸替代數(Dxy)與居群間每位點凈核苷酸替代數(Da)的值相應也不是很高。K值、Dxy值和Da值的數據顯示海蜇3個群體間遺傳多樣性指標的差異不大。
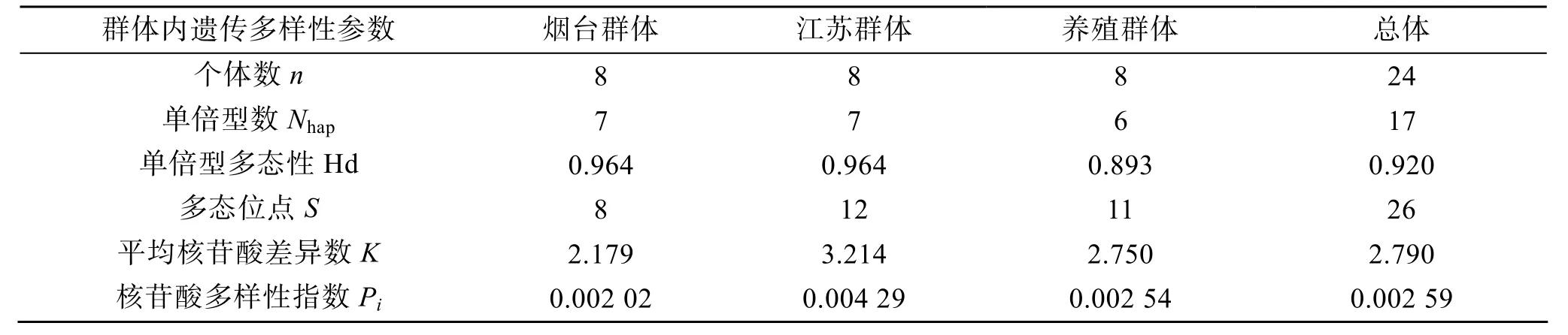
表3 海蜇各群體內的遺傳多樣性參數Tab. 3 Summary of genetic diversity of different Rhopilema esculentum populations

表4 海蜇各群體間遺傳多樣性參數及遺傳分化系數Tab. 4 Summary of genetic diversity between different Rhopilema esculentum populations and Fst
2.4 海蜇 3個群體間遺傳距離及群體遺傳分化
海蜇3個群體間遺傳距離在0.002 30~0.002 81之間, 遺傳差異AMOVA分析結果顯示, 3個群體間兩兩比較的遺傳分化指數(Fst)值在 0.032 50~0.730 8之間(表4), 三群體間遺傳分化系數Fst=0.058 02, 表明在整個遺傳變異中群體間占 5.802%, 其余的遺傳變異來源于群體內, 群體內具有程度較高的遺傳分化(表5)。
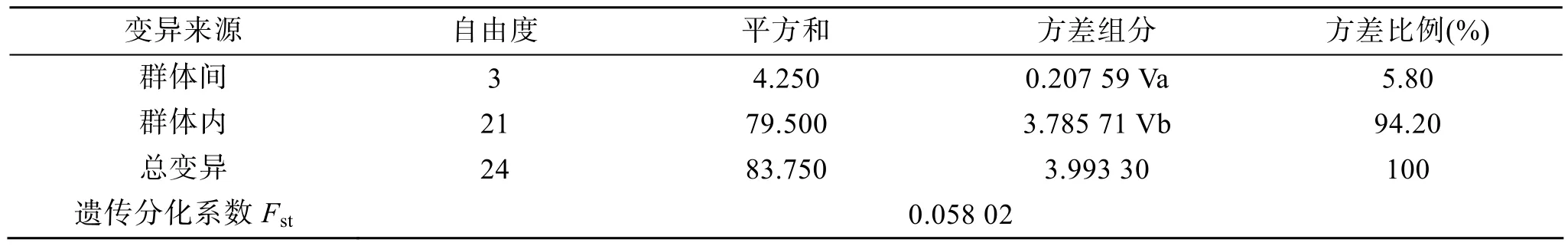
表5 海蜇各群體間遺傳差異的分子方差分析表(AMOVA)Tab. 5 Analysis of molecular variance (AMOVA) among populations of Rhopilema esculentum
2.5 分子系統樹的構建
根據所得ITS序列, 以Nemopilema nomurai作為外群, 利用MEGA4.0用NJ法構建17個單倍型個體間的分子系統樹, 自展一致樹顯示作為外群的Nemopilema nomurai獨立于海蜇 17個單倍型之外,但是一個群體的不同個體并沒有先聚為一起, 分屬于3個不同群體的17個單倍型沒有明顯的聚類, 不同群體之個體間的親緣關系沒有明顯分群(圖2)。
3 討論
作者對兩個自然捕獲群體和一個養殖群體24個海蜇個體的核糖體轉錄間隔區進行了擴增、測序, 得到長度為1 080~1 096 bp的序列, 其中包括完整的ITS1, 5.8S和ITS2序列。在所獲得序列結構中值得注意的一點是, 序列中簡單重復單元較多, 序列中存在的插入缺失位點均由簡單重復單元數目改變引起, 如在第一個位點(C/TTCG)重復數目為 5~7個。簡單重復單元數目改變的原因推測是在復制時發生滑動突變, 使其拷貝數發生改變, 進而引起序列的變異。在海蜇的ITS序列中, 共發現4處簡單重復序列的插入和缺失, 至于什么原因促成這種簡單重復序列的插入和缺失, 以及這種插入和缺失的調控機理, 目前尚未見相關研究報道。其形成是否與適應生長環境及表型性狀相關, 此方面還需更深入的研究探討。

圖2 海蜇ITS序列17個單倍型NJ聚類圖Fig. 2 Phylogenic tree of 17 haplotypes based on ITS sequences of Rhopilema esculentumYT. 煙臺自然捕獲群體; JS. 江蘇自然捕獲群體; YZ. 威海養殖群體YT. wild population of Yantai; JS. wild population of Jiangsu; YZ.cultured population
許多海洋經濟種類養殖基本是依靠采捕野生親本進行大量繁殖, 采用這種方式繁育, 由于從自然群體中選用親本數目有限, 勢必造成一定程度的隨機漂變, 引起養殖群體的遺傳結構發生改變, 總體遺傳多樣性下降[15,16], 多年的累代養殖將造成不同程度的近交繁殖, 引起種質退化。同時因過度捕撈使野生資源逐漸減少, 而養殖個體不斷逃逸到自然水體, 也將對野生群體的遺傳本質、種質資源和遺傳多樣性產生不可低估的影響[17]。海蜇發展養殖開始的比較晚, 發展十幾年來也是依靠從自然海域捕獲親本進行育苗, 因此, 隨著養殖規模的不斷擴大, 研究海蜇野生種群和養殖種群的遺傳結構以及遺傳多樣性對開展海蜇自然資源調查與保護使之可持續利用;開發種質資源, 進行優良品種培育, 最大限度地保持養殖群體的遺傳多樣性具有重要意義。
本研究中兩個自然捕獲群體和一個養殖群體由分析 ITS序列相關參數所反映出來的三群體群體內遺傳多樣性參數差異不大, 江蘇群體略高; 兩兩群體間遺傳多樣性指標數值也比較相近, 群體間遺傳距離在0.002 30~0.002 81之間, 遺傳分化指數(Fst)值在0.032 50~0.730 8之間。根據Wright[18]關于遺傳分化指數的大小和分化程度的解釋, 當Fst接近于 0時, 說明群體間的沒有發生遺傳分化,Fst值在0.05~0.15之間, 遺傳分化達到中等水平。而本研究中 3個群體間遺傳分化指數均比較小, 說明大群體之間幾乎沒有遺傳分化。相應的, 分屬 3個群體的17單倍型在分子系統樹中也沒有明顯的聚類。目前,山東沿海地區海蜇的育苗捕獲親本基本是來自于江蘇附近海域, 而同時, 由于海蜇自然野生資源的衰退, 山東省近幾年一直在開展海蜇的增殖放流工作,這種情況, 從海蜇苗種溯源上解釋了本研究的結果。
作者分析得到海蜇 ITS片段序列多態數據, 群體遺傳結構分析表明, 群體遺傳多樣性比較低, 群體間的遺傳距離比較小, 群體遺傳結構相似, 群體間沒有明顯的遺傳分化, 說明養殖群體和兩個自然捕獲群體尚沒有形成自己獨立的遺傳結構。從海蜇遺傳育種種質資源角度來看, 再捕獲不同煙臺和江蘇自然群體作為親本對繁育后代影響不大; 養殖群體由于并不是累代繁育, 其遺傳結構與自然捕獲群體也無很大區別。在未來養殖過程中, 為了防止人工選育中的近交衰退的問題, 除了注意保持選育親本的數量外, 還應注意利用分子生物學手段對選育群體的遺傳多樣性的檢測分析, 在保持選育性狀穩定性的同時, 最大限度地保留群體的遺傳多樣性, 使養殖產業健康、高效地持續發展。本研究結果同時也說明, 自然捕獲群體遺傳結構相似, 黃渤海海蜇種質資源遺傳多樣性比較低, 應加大對海蜇自然資源的保護力度, 再繼續開展海蜇的增殖放流工作的同時, 更重要的是控制自然海區海蜇資源的捕撈量,保護海蜇自然種質資源, 維護黃渤海生態平衡, 保證其自然資源可持續利用。
[1] 高尚武, 洪慧馨, 張士美.中國動物志: 無脊椎動物刺細胞動物門(第二十七卷): 水螅蟲綱缽水母綱[M].北京: 科學出版社, 2003.222-225.
[2] Hsieh Y H P, Leong F M, Rudloe J. Jellyfish as food [J].Hydrobiogia, 2001, 451: 11-17.
[3] 劉希光, 于華華, 趙增芹, 等.海蜇不同部位脂肪酸的組成研究[J].分析化學, 2004, 32(12): 1 635-1 638.
[4] 羅元凱.食用藥物和藥膳[J].新中醫, 1996, 28(12):8-9.
[5] 計光輔.海蜇的藥用功效[J].家庭中醫藥, 2007, 14(7):68.
[6] 王明德, 謝勇.中國渤海海蜇資源的保護和利用[J].自然資源, 1996, 1: 64-66.
[7] 關松, 張鵬剛, 劉春洋, 等.我省海蜇池塘養殖現狀與存在問題[J].水產科學, 2004, 23(8): 30-31.
[8] Hillis D M, Dixon M T. Ribosomal DNA: molecular evolution and phylogenetic inference [J]. Q Rev Biol,1991, 66: 411-453.
[9] 周伯平, 周開亞, 宋大祥.核r DNA ITS區序列在無脊椎動物分子系統學研究中的應用[J].動物學雜志,2002, 37(4): 67-73 .
[10] 汪文俊, 王飛久, 陳松林, 等.滸苔 ITS區的擴增和分析[J].海洋水產研究, 2008, 29(5): 124-129.
[11] Coleman A W, Vacquier V D. Exploring the phylogenetic utility of ITS sequences for animals: A test case for abalone (Haliotis) [J]. J Mol Evol, 2002, 54:246-257.
[12] 丁小雷, 何毛賢, 鄧鳳姣, 等.雙殼綱動物核糖體RNA 18S -ITS1序列及其在分子系統發育研究中的應用[J].遺傳, 2004, 26(3): 319-324.
[13] 陳琳琳, 孔曉瑜, 周立石.魁蚶核糖體 DNA基因轉錄間隔區的序列特征魁蚶核糖體DNA基因轉錄間隔區的序列特征[J].中國水產科學, 2005, 12(1):104-108.
[14] Heath D D, Rawson P D, Hilbish T J. PCR-based nuclear markers identify alien blue mussel (Mytilus spp.)genotypes on the west coast of Canada [J]. Can J Fish Aquat Sci, 1995, 52 (12): 2621-2627.
[15] Sunden S L F, Davis S K. Evaluation of genetic variation in a domestic population of Penaeus vannamei(Bonne): A comparison whit three natural population[J]. Aquaculture, 1991, 151:131-142.
[16] Wolfus G M, Garcia D K, Alcivar-Warren A. Application of the microsatellite technique for analysing genetic diversity breeding programs [J]. Aquaculture,1997, 152:35-47.
[17] Allendorf F W, Phelps S R. Loss of genetic variation in a hatchery stock of cutthroat trout [J]. Trans Am Fish Soc, 1980, 109: 537-543.
[18] Wright S. The genetical structure of population [J].Annals of Eugenics, 1951, 15: 323-334.
Genetic diversity of ITS sequences in farmed and natural Rhopilema esculentum populations
SUN Guo-hua1,2, LIU Xiang-quan1, YANG Jian-min1, ZHANG Xi-jia1, LIU Ai-ying1,TAN Fu-yi2
(1. Marine Fisheries Research Institute of Shandong Provience, Yantai 264006, China; 2. Shandong Homey Aquatic Development Co.LTD, Weihai 264305, China)
Feb., 25, 2010
Rhopilema esculentum, internal transcribed spacers(ITS), population diversity, genetic structure
For the purpose of investigating genetic diverstiy in wild and cultured populitions of Rhopilema esculentum, ITS regions of 24 individuals within Yantai and Jiangsu wild populitions and Weihai cultured population were amplified and analyzed. The whole sequences were between 1 080~1 096 bp, including ITS1-5.8S-ITS2. The result showed that 26 variable nucleotide positions were detected and four nucleotide sites had 29 insertion-deletion positions. Indexes of Piand K among three populations were 2.179~2.750 and 0.002 02~0.002 54, respectively. Indexes of K, genetic distance and fixation indices (Fst) between populations were 2.797~3.031, 0.002 30~0.002 81 and 0.032 50~0.730 8, respectively. The indexs of genetic diversity among and between populations were closed and genetic distances between populations were small, which indicated that the genetic structure of populations were smilar and there were no distinct genetic differentiation between populations.
Q347
A
1000-3096(2010)10-0090-06
2010-02-25;
2010-05-12
國家海洋局項目(海科字[2006]16號)
孫國華(1979-), 女, 山東萊西人, 博士, 助理研究員,主要從事海洋生物分子遺傳研究, E-mail: sghqingdao@hotmail.com;楊建敏, 通信作者, E-mail: ladderp@126.com
(本文編輯:梁德海)

