具有除草活性的大環內酯類衍生物的定量構效關系
段紅霞 王瑞剛 張建軍 董燕紅 梁曉梅 吳景平 王道全
(中國農業大學應用化學系,農業部農藥化學與應用技術重點開放實驗室,北京 100193)
具有除草活性的大環內酯類衍生物的定量構效關系
段紅霞 王瑞剛 張建軍 董燕紅 梁曉梅*吳景平 王道全*
(中國農業大學應用化學系,農業部農藥化學與應用技術重點開放實驗室,北京 100193)
研究了一系列結構新穎的具有除草活性的大環內酯衍生物的定量構效關系(QSAR).構建的比較分子力場分析(CoMFA)、比較分子近似指數分析(CoMSIA)和全息定量構效關系(HQSAR)分子模型的交叉驗證系數均大于0.5,非交叉驗證系數r2都超過0.8,表明獲取的QSAR模型具有可信的預測能力.對CoMFA、CoMSIA模型的三維(3D)等勢圖分析,發現除了立體場和靜電場外,疏水場和氫鍵受體場也是影響大環內酯類化合物除草活性的重要因素.構建的HQSAR模型的原子貢獻圖提示的結構改造信息與三維QSAR的結果基本一致.利用CoMFA、CoMSIA模型提供的信息,對目前已合成的活性最高化合物B1-3進行分子結構改造,預測結果發現部分化合物可能具有更好的除草活性.
比較分子力場分析;比較分子近似指數分析;全息定量構效關系;大環內酯;除草活性
苯氧羧酸類除草劑的研究與開發始于上世紀40年代,2,4-二氯苯氧乙酸(2,4-D)是其成功的例子[1].其后,圍繞2,4-D的分子結構開展的研究工作,促使更多具有更高除草活性或具有更廣除草譜的化合物商品化[2].我們在新農藥的創制研究中,模仿具有廣譜生物活性的大環內酯類化合物的結構,將大環內酯環引入到芳氧羧酸類除草劑的分子結構中,合成了一系列結構新穎的含十六元環內酯的芳氧羧酸酯類化合物[2].生物測試結果表明,它們對單子葉植物和雙子葉植物均具有較好的除草活性.為了探討其進一步結構改造的可能性,研究其構效關系是必要的.在分子結構與生物活性的定量構效關系(QSAR)研究中,以80年代末發展起來的比較分子力場分析方法(CoMFA)[3]及在CoMFA方法基礎上擴展起來的比較分子近似指數分析方法(CoMSIA)[4]最為有效,已成為藥物和農藥領域重要的研究工具.國內外一些研究小組在新農藥的創制,特別是在除草劑的研究中,采用上述兩種方法研究其構效關系,為進一步的分子設計提供參考,加速了創制的進程.研究的化合物類型主要包括:磺酰脲類[5],三唑并嘧啶磺酰胺類[6],氰基丙烯酸酯類[7],嘧啶苯甲酸類[8],咪唑啉酮類[9],異吲哚啉酮類[10],膦酸酯類[11],N-取代芐胺類[12],二苯醚類[13],單脒類[14].近年來,分子全息定量構效關系(HQSAR)法由于具有模型建立快速、預測能力高且能直接給出分子中對活性起主要作用的原子等優點成為一種新型高效的定量構效關系研究方法[15],目前HQSAR技術已經成功地應用到許多不同類化合物的構效關系研究當中[16-18].本文采用CoMFA、CoMSIA和HQSAR法研究前面提到的大環內酯類化合物的結構與對莧菜除草活性之間的定量構效關系,以便為進一步的結構改造提供參考.
1 研究方法
本文所有計算均使用SGI分子設計工作站上安裝的Sybyl 7.3軟件完成[19],計算中所選用的參數均為默認值.
1.1 目標化合物
本文研究的化合物及其對莧菜的除草活性均來源于文獻[2],化合物的結構通式見圖1,結構及除草活性如表1所示.

圖1 研究化合物A,B及預測化合物P的結構通式Fig.1 Structural formula of the studied compounds A,B and predicted compounds P

表1 研究化合物的結構和對莧菜的除草活性(PLC50)Table 1 Structure and herbicidal activity(PLC50)to Amaranthus tricolor L.of the studied compounds
所研究化合物在結構上有三處變化:大環內酯環上C15上的取代基(R),側鏈上的芳基(Ar),和羧酸部分α-C上的取代基(R1,R2),除草活性采用致死中濃度的對數(PLC50)表示.
所有化合物因含肟醚結構而存在順反異構.文獻[2]研究表明,對應的順式異構體和反式異構體對莧菜的除草活性基本無差異,順反異構體的除草活性等同于單一異構體的除草活性.基于上述事實,本文采用順反異構體的生物測試數據表征順式和反式構型化合物的各自除草活性,進行QSAR建模.
1.2 分子構象優化
分子構象的確定是建立有效3D-QSAR模型的首要前提.最好能獲取各個活性化合物的藥效構象,但由于通常化合物的靶標結構未知,很難確定它們真正的藥效構象,通常采用分子的最低能量構象作為可能的藥效構象進行理論研究.
大環內酯類化合物因含大環骨架結構具有較大的柔性,因此,我們選取已經報道的化合物A1-6的Z型異構體的晶體結構[2]為分子模板,利用單晶數據中大環母體骨架數據矯正并優化其它分子結構,進而使用Sybyl 7.3程序包做進一步結構優化.通過對計算方法、分子力場、電荷參數的系統考察,確定適用于大環內酯類化合物的最佳優化參數包括:方法, Broyden-Fletcher-Goldfarb-Shanno(BFGS);收斂梯度,0.21 kJ·mol-1·nm-1(即為0.005 kcal·(mol·?)-1);力場,MMFF94;電荷,MMFF94,其它參數選取默認值.在上述優化參數的條件下,獲得Z型A1-6分子構象較接近其單晶結構,如圖2所示.
1.3 分子疊加
CoMFA和CoMSIA方法均是在分子周圍三維網格上建立相關分子場,分析其與生物活性的相關性,網格點上能量的大小與分子的相對位置密切相關.因此,確定分子的取向是CoMFA和CoMSIA研究的關鍵步驟之一.所有分子需要選取一定的定位規則在空間疊合,才能保證分子場取向的一致性.目前廣泛采用的定位方式是fit atoms,即以較高活性的分子為模板,選定基本骨架相似的原子,使其它要定位分子的相應原子與模板中原子的距離均方根偏差(RMSD)最小,此方法又稱atoms-by-atom[20].

圖2 A1-6的Z型晶體結構[2](a)與優化后的構象(b)Fig.2 Crystal structure[2](a)and optimizedconformation(b)of Z-A1-6

圖3 E-A1(a)與Z-A1(b)的大環骨架Fig.3 Skeletons of macrocycle of E-A1(a)and Z-A1(b)
我們分別選取活性較好、具有一定代表性的A1-11的Z型和E型異構體為模板,以大環骨架及肟酯結構為疊合基本骨架(圖3),運用Sybyl 7.3中的alignment database模塊進行自動疊合,以保證所有分子在三維網格中的取向具有一致性,使分子間相互重疊時均方根偏差最小.疊合結果如圖4所示,所有分子的疊合效果是令人滿意的.
1.4 CoMFA和CoMSIA模型建立
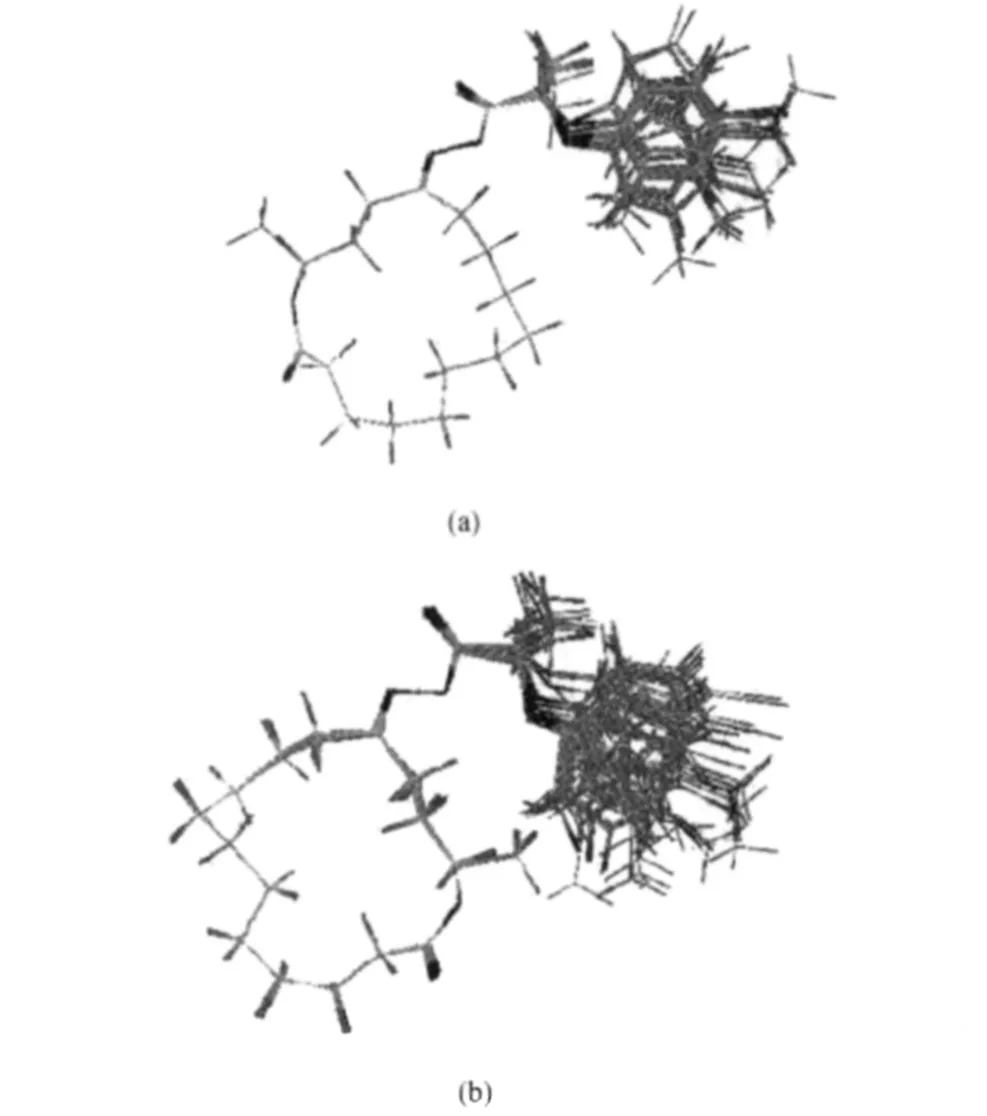
圖4 研究化合物的E-系列(a)與Z-系列(b)分子疊合圖Fig.4 Maps of superimposition of E-series(a)and Z-series(b)
所有CoMFA和CoMSIA研究均在Sybyl 7.3的QSAR模塊中進行.選取sp3雜化的C+作為分子探針,對疊合分子外圍網格點上的各種場能大小和分布進行計算.網格點步長設置為0.2 nm,場能的截斷值設置為125.4 kJ·mol-1,柱過濾值設置為8.36 kJ·mol-1.在CoMFA研究中考慮立體場(steric)和靜電場(electrostatic);在CoMSIA計算中引入立體場、靜電場、疏水場(hydrophobic)、氫鍵受體場(acceptor).在PLS分析時,首先采用抽一法(LOO)進行交叉驗證,求得交叉驗證系數用來衡量模型的預測能力,一般認為,當時模型具有可信的預測能力,可繼續進行非交叉驗證),然后通過確定的最優主成分數進行非交叉驗證回歸分析,得到非交叉驗證系數r2,建立相應的3D-QSAR模型[21].最后,采用Stedev coeff方法[22]顯示所構建的3D-QSAR模型的三維等勢圖,直觀地反映各個分子場對化合物生物活性的貢獻.通過對訓練集分子和新設計的化合物的活性預測,評價所建3D-QSAR模型的預測能力.
1.5 HQSAR模型的建立
分子全息結構-活性關系技術(HQSAR)是將系列化合物的分子結構轉換為分子碎片數目,然后用偏最小二乘回歸方法將分子碎片數目的類型即分子全息與生物活性之間建立相關關系.HQSAR方法只要求輸入分子的二維結構和生物活性數據,分子碎片和全息的產生完全是自動的,非常迅速.HQSAR方法不但避免了經典QSAR方法的共線性和參數計算與選擇的問題,而且不需要面對3D-QSAR中難以確定的疊合規則困難,具有極大的優點和廣泛的應用前景.
采用Sybyl 7.3軟件在SGI工作站上進行分子模擬,HQSAR模塊產生分子全息.首先,每個分子的結構被劃分為包含一定原子個數的分子碎片,其中,碎片中包含的原子數目被定義為碎片大小參數;碎片中所包含的結構類型有原子、鍵、連接方式、氫和手性等.根據碎片中包含原子數目的多少,分為較小原子碎片(1-3個原子),中等原子碎片(4-7個原子)和較大原子碎片(3-10個原子)三種情況.然后,對應于一個分子的所有亞結構碎片按照CYC算法[23]映射成一定長度的整數串,這即分子全息.整數串的長度稱為分子全息長度參數,在HQSAR模塊中,提供12個缺省的質數(53,59,61,71,83,97,151,199, 257,307,353和401)作為全息長度.通過調整碎片類型、碎片長度以及全息長度等參數,改善模型的統計能力.
采用PLS進行建模和交叉驗證.分別以“逐一剔除”和“分組剔除”的交叉驗證相關系數平方值來反映模型的穩健性和預測能力,用非交叉驗證相關系數平方值r2和標準估計誤差SEE表征模型的擬合程度.采用不同顏色對分子的各個原子進行編碼,以反映各原子對生物活性貢獻的大小,從而快速指導新配體的合成.
2 結果與討論
2.1 CoMFA模型分析與討論
我們對所選取的29個化合物進行CoMFA模型構建,計算結果如表2所示.獲得CoMFA模型的統計學參數為:對于E系列化合物,其交叉驗證相關系數,對Z系列化合物,得到的交叉驗證系數,表明模型具有較好的可靠性和預測能力;由最佳主成分數確定的CoMFA模型的非交叉驗證系數分別為r2=0.948(E系列),r2=0.944(Z系列);對于E系列化合物,立體場和靜電場對模型的貢獻分別為62.7%和37.3%,Z系列化合物的立體場和靜電場對模型的貢獻分別為63.9%和36.1%,可見,無論是E系列還是Z系列,立體場的貢獻明顯大于靜電場,說明立體場對大環內酯類化合物與其靶標的相互作用影響更為顯著.通過上述一系列的研究結果表明,盡管E系列與Z系列化合物在結構上有一定差異,但其生物活性具有很大的相似性,這與實測結果是一致的[2].
利用建立的CoMFA模型對部分訓練集分子進行除草活性預測,評價模型的實際預測能力,計算結果如表3所示.可見,無論是E構型還是Z構型, CoMFA模型對大環內酯類化合物除草活性的預測結果與實驗值吻合度都較高,差值在誤差允許范圍內,這表明構建的3D-QSAR模型具有很好的重現性和預測能力,能夠進一步對同類其它大環內酯類化合物的除草活性進行預測.
CoMFA方法由于使用的自變量遠大于因變量,故難以用數學函數表示,多采用直觀的三維圖形表示3D-QSAR模型的計算結果,從中分析化合物各個取代基性質及位置變化對活性的影響,獲取系列化合物結構改造的信息.為了作出更好說明,選擇具有最高生物活性的化合物B1-3作為參考分子,采取Stedev coeff方法獲得CoMFA模型分子場的三維等勢圖(圖5).在立體場等勢圖中,綠色區域表示在該區域存在體積大的基團有利于活性的提高,黃色區域表示在該區域不宜引入位阻大的基團;在靜電場等勢圖中,紅色區域表示引入電負性基團有利于提高生物活性,藍色區域表明引入正電性基團有利于生物活性的提高.
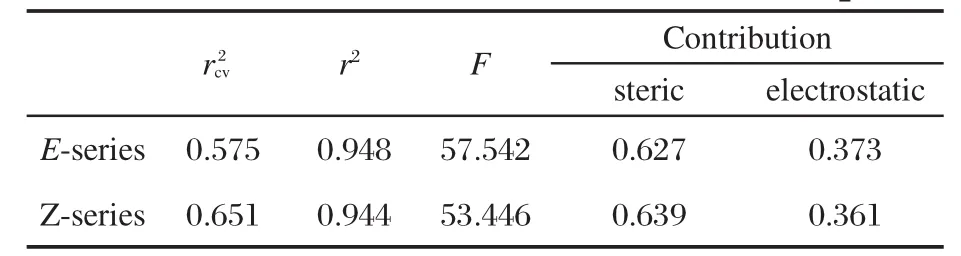
表2 E、Z兩個系列化合物CoMFA建模結果Table 2 CoMFA models of E-and Z-series compounds
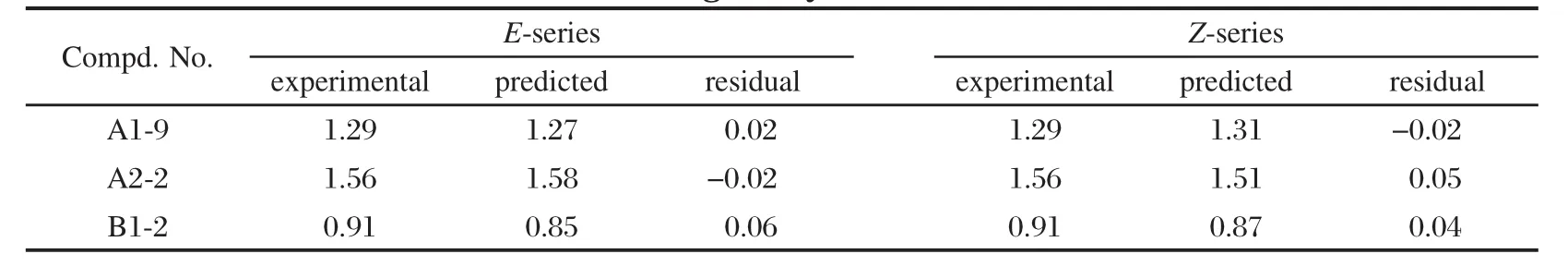
表3 部分訓練集分子的除草活性(PLC50(mg·L-1))及采用CoMFA模型預測結果Table 3 Experimental and predicted herbicidal activity results(PLC50(mg·L-1))for some compounds in the training set by CoMFA model
如圖5所示,就E系列化合物而言,在大環內酯骨架的C15位上,覆蓋有較大范圍的黃色區域,表明此位置不宜引入空間位阻較大的基團.我們正是在大環內酯C15位引入了較小的甲基(R=Me)合成了系列化合物,有效地增加了化合物的除草活性.如表1所示,B系列化合物的活性明顯高于A系列化合物.同樣,在側鏈羧酸的α-位上也有黃色區域分布,說明這里也不宜引入體積過大的基團,如化合物A2-1(PLC50=1.35 mg·L-1)和化合物A3-1(PLC50= 1.54 mg·L-1),在該C原子上由一個甲基取代變成兩個甲基后,化合物活性明顯降低;此外,在苯環的對位覆蓋有明顯的綠色區域,說明在該位置引入空間位阻較大的基團有利于大環內酯類化合物活性的提高.如化合物A1-2(PLC50=1.74 mg·L-1)和化合物A1-3 (PLC50=1.69 mg·L-1)對比發現,增大苯環對位取代基的體積,即甲基替換為甲氧基后,化合物的活性有一定提高.可見,在苯環對位較大的取代基對生物活性是有利的.
圖5還顯示,在苯環4位上覆蓋較大范圍的藍色區域,說明在該位置引入正電基團有利于大環內酯類化合物除草活性的提高.如對比化合物A1-1 (PLC50=1.86 mg·L-1)和A1-2(PLC50=1.74 mg·L-1)發現,苯環對位為帶正電的甲基取代的化合物A1-2的活性要高些.此外,在苯環平面的上、下方覆蓋大面積的紅色區域,說明增加苯環的電負性會顯著提高化合物的生物活性.如化合物A1-1(PLC50=1.86 mg·L-1)和化合物A1-11(PLC50=1.23 mg·L-1),由于吸電子基團—Cl,—Br的存在使得化合物A1-11的活性更高.對比圖5中Z系列的CoMFA模型與E系列的CoMFA模型,發現兩個系列的CoMFA模型圖像提供的結構改造信息基本一致,這進一步論證了該類化合物的順反異構對除草活性的影響很小.
2.2 CoMSIA模型分析與討論
對疊加好的E、Z兩個系列的化合物進行CoMSIA建模.在構建CoMSIA模型過程中,考慮了立體場、靜電場、疏水場和氫鍵受體場,經過參數不斷調整與優化,確定最佳的3D-QSAR模型.所有計算結果顯示,構建的CoMSIA模型,說明具有較高的可信度.
為了作更好的解釋說明,選擇具有最高生物活性的化合物B1-3為參考分子,采取Stedev coeff方法獲得CoMSIA模型各個分子場的三維等勢圖.所構建的CoMSIA模型的立體場和靜電場等勢圖(略)與前述分析討論的CoMFA模型的立體場和靜電場所提供的化合物結構改造信息基本一致,這里不再累述.如圖6所示,在疏水場等勢圖中,黃色區域表示該區域取代基疏水性增強有利于增加化合物的生物活性,灰色區域表示該區域疏水性減弱將提高化合物的生物活性.對E系列化合物,芳香環上方覆蓋較大范圍的黃色區域,這說明芳香苯環的疏水作用對大環內酯類化合物的除草活性極其重要,增加苯環的疏水性質,尤其是在2,3,5位增加取代基的疏水特征,可以有效的提高生物活性.在苯環4位分布有灰色區域,如若降低其取代基的疏水性質有可能增加化合物的生物活性.Z系列化合物疏水場提供的結構信息與E系列基本一致.

圖5 E系列(a)和Z系列(b)CoMFA模型的立體場和靜電場等勢圖Fig.5 Contour maps of steric and electrostatic fields for CoMFA models of E-series(a)and Z-series(b)
在氫鍵受體場等勢圖中,紅色區域表示增大氫鍵受體場有利于提高活性,紫色區域表示減小氫鍵受體場有利于化合物活性的提高.如圖7所示,無論是E系列化合物還是Z系列化合物,在大環內酯羰基鄰位和側鏈肟酯羰基附近均分布有較大范圍的紅色區域,如若在該位置引入具有氫鍵受體特征的基團將有利于增加大環內酯類化合物的生物活性;而在大環內酯羰基部位、側鏈肟酯的N原子上覆蓋有一定范圍的紫色區域,此處不宜引入氫鍵受體基團.

圖6 E系列(a)和Z系列(b)CoMSIA模型的疏水場等勢圖Fig.6 Contour maps of hydrophobic field for CoMSIA models of E-series(a)and Z-series(b)
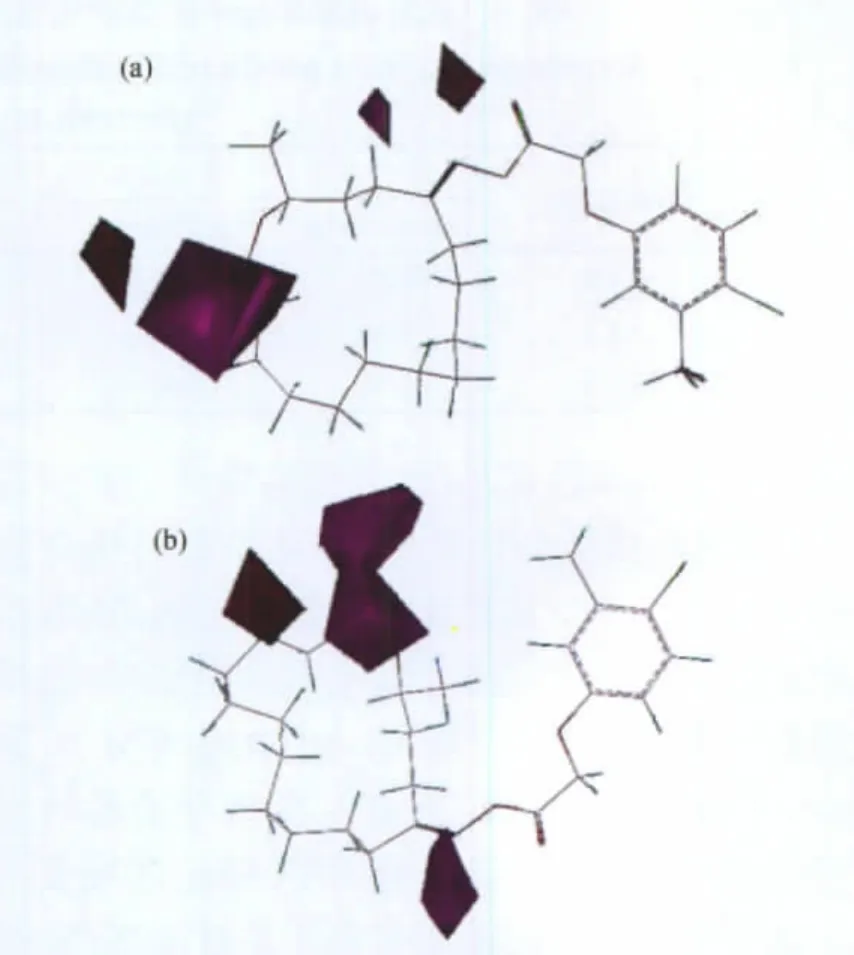
圖7 E系列(a)和Z系列(b)CoMSIA模型的氫鍵受體場等勢圖Fig.7 Contour maps of hydrogen-bond acceptor field for CoMSIA models of E-series(a)and Z-series(b)
基于上述一系列的CoMFA和CoMSIA模型的研究結果,結合大環內酯類化合物的結構特征,我們發現除了立體場、靜電場以外,疏水場和氫鍵受體場也是影響大環內酯類化合物除草活性的重要因素.
2.3 HQSAR模型分析與討論

圖8 A2-6的HQSAR模型的色碼圖Fig.8 Color coding figure of HQSAR model of A2-6

表4 不同碎片長度得到的HQSAR模型aTable 4 Influences of various fragment size parameters on HQSAR modela
分別在碎片長度為1-3、4-7和8-10范圍內產生分子全息,得到一系列QSAR模型,列于表4.從模型“逐一剔除”的交叉驗證結果來看,最佳模型處于分子碎片長度為8-10范圍內,對應分子全息長度為97,交叉驗證相關系數r2cv為0.680,模型的最佳主成分數為4.這是因為較大的分子碎片長度才能更詳盡地區分大環內酯類化合物分子的不同結構特征.為了考察不同類型的碎片區分因素對模型質量的影響,在分子碎片長度為8-10、分子全息長度為97的條件下,改變所用的分子碎片區分參數例如原子類型A、化學鍵類型B、連接性Con、手性Ch、氫鍵給體和受體DA、氫原子H等,得到一系列的分子全息模型列于表5中.研究結果發現當考慮上述所有分子碎片區分參數組合時獲得的模型質量最好,該HQSAR模型的統計學參數為0.713,最小標準預測偏差SEP為0.204;非交叉驗證相關系數r2為0.870,標準估計誤差SEE為0.137,最佳主成分數為4.一般認為,模型具有顯著的預測能力.可見,所有分子碎片區分參數都對大環內酯類衍生物的除草活性有影響,尤其是氫鍵特征對生物活性的貢獻最大,它的引入可以較大程度的提高HQSAR模型質量.
HQSAR的最大優點是可以采用不同顏色對分子中的各原子進行編碼,以反映各原子對生物活性貢獻的大小,從而快速直接地指導新配體的合成.處于光譜中紅色系的顏色反映的是不利貢獻,從橙色、橙紅色到紅色這種不利影響依次增大,合成時,應盡量避免引入這些基團.處于光譜藍色系的顏色反映的是有利貢獻,從黃色、藍綠色到綠色表示對活性的貢獻依次增大,在合成時這些基團應盡量保留.而白色,則表示對活性沒有影響.
以化合物A2-6為例對HQSAR模型進行色碼解釋,如圖8所示.圖中青色為該系列化合物的共同結構.A2-6化合物R1位甲基上的氫原子呈現綠色或黃色,表明它們對化合物的活性有正性貢獻,而R2位置的氫原子呈現白色對活性沒有影響.所以在化學合成過程中應該盡可能保留R1位甲基取代,這一研究結果與之前CoMFA和CoMSIA模型中所指出的R1取代基體積不能過大的結論是一致的.其原因可能是只有合適大小的取代基如一個CH3,才能與靶標有較好的結合作用;若R1和R2同時取代,會導致空間位阻過大,不利于與受體很好的作用.取代基R位置上的氫原子呈現橙紅色,表明該位置原子對活性的貢獻是不利的,應該考慮引入新的取代基團.我們實驗結果已經證實,R位置被甲基取代后化合物活性有顯著提高(如化合物B1-2的活性為PLC50=0.90 mg·L-1,化合物A1-9的活性為PLC50=1.29 mg·L-1).苯環3位被取代的原子呈現黃色或綠色,表明該位置取代對活性是有利的,在合成過程中應盡量保留3位的取代,實驗結果已經充分證實了這一點(如化合物B1-2的活性為PLC50=0.90 mg·L-1,化合物B1-3的活性為PLC50=0.53 mg·L-1). 4位氫原子呈現紅色,說明對活性是不利的,因此在具體的合成過程中應該考慮設計其它基團取代,這與CoMFA和CoMSIA模型中指出的在苯環對位引入具有特性的取代基團對該系列化合物的除草活性是非常有利的結論相吻合,我們在實驗過程中已經充分考慮到這一點,基本上設計的都是在4位引入不同取代基團來提高系列化合物活性.與肟酯中N相鄰的大環內酯上的氫原子呈現綠色,對活性正性貢獻,其它距離較遠的氫原子為紅色或橙色對活性負性貢獻.

表5 碎片區分因素對HQSAR模型質量的影響aTable 5 Influences of various fragment type parameters on HQSAR modela
2.4 新分子活性預測
結合上述構建的CoMFA、CoMSIA模型的研究結果,以目前合成的大環內酯類化合物中活性最好的B1-3(PLC50=0.53 mg·L-1)為母體,分別對大環母體、側鏈及側鏈苯環上取代基進行結構改造,運用CoMFA模型對其除草活性進行預測,期望能獲取具有更高除草活性的新型分子.設計的部分新化合物結構及其預測的PLC50值如表6所示.
結合CoMFA模型的立體場等勢圖信息,對大環內酯骨架部分的羰基和甲基進行結構改造,將B1-3大環內酯骨架上的—CH3換成—F、—CF3、—CH2CH3、—OCH3、—CN后,預測活性有所降低(如P1),這進一步證明保留大環內酯骨架上的—CH3對活性是有利的;考慮CoMSIA模型的氫鍵受體場等勢圖結果,大環部分的羰基作為氫鍵受體不應削弱,當羰基變為硫羰基后,預測活性明顯降低(如P2).
根據CoMFA模型的立體場和靜電場信息,對B1-3側鏈羰基上的α-CH2進行結構改造.將兩個—H中的一個用強電負性的—F取代,活性有所提高(如P3);但將兩個—H均由—F取代后,可能由于空間位阻的原因,活性下降(如P4).
對B1-3苯環部分的改造:根據CoMFA模型的立體場和靜電場等勢圖結果,結合CoMSIA模型的疏水場等勢圖信息,保持對位Cl原子不變,在鄰、間位上引入空間位阻大、或電負性強、或疏水性強的基團,如—CN、—CF3、苯基、環己基、環丙基等,結果發現只有在間位上單一引入這些取代基團,生物活性才有望提高(如P7、P8、P9、P10、P11).可見,在以后的結構改造中應著重針對苯環間位進行結構改造.此外,在保留間位CH3的情況下,將對位上的Cl原子替換為Br原子后預測活性有所下降(P5);但將其替換為電負性更強的NO2后,預測活性顯著提高(P6),這可能是因為—NO2疏水性弱的緣故.同時將側鏈羧酸α-C上的—H替換為—F及苯環對位Cl替換成NO2,同樣可以獲取良好的效果,活性有很大的提高(如P13、P14).因此,在后續的結構改造過程中,建議在側鏈羧酸α-C上引入F,在苯環對位引入NO2,而間位則引入體積更大、給電子和疏水性均強的烴基等基團,有望獲得除草活性更好的新型大環內酯類化合物.

表6 部分設計化合物的結構及除草活性的預測Table 6 Structure and predicted herbicidal activity of designed compounds
3 結 論
運用比較分子力場分析(CoMFA)、比較分子相似性指數分析(CoMSIA)和全息定量構效關系(HQSAR)方法構建了具有除草活性的大環內酯類系列化合物的定量構效關系模型.對部分已知化合物進行除草活性的預測結果與實驗值相當,說明所構建的模型具有良好的預測能力.研究結果表明,由CoMFA和CoMSIA模型獲得的結構改造信息基本一致,除了立體場和靜電場外,疏水場和氫鍵受體場也是影響上述大環內酯類衍生物除草活性的重要因素.通過構建最優HQSAR模型給出的原子貢獻圖提示的化合物結構改造信息與三維QSAR提供的結果相吻合.根據構建的CoMFA和CoMSIA模型,以B1-3為母體,分別對大環內酯環,側鏈及側鏈苯環上的取代基進行結構改造,并對其除草活性進行預測,結果獲得數個可能具有更高活性的化合物,為大環內酯類化合物通過結構改造獲得活性更高的化合物奠定了基礎,也為其它類型大環化合物的構效關系研究提供了借鑒.
Supporting Information Available: CCDC 689231 contains the supplementary crystallographic data for this paper. These data can be obtained free of charge from the CCDC,12 Union Road,Cambridge CB2 1EZ,UK;Tel:(44)01223 762910; E-mail:deposit@ccdc.cam.ac.uk.
1 Fletcher,W.W.;Kirkwood,R.C.Herbicides and plant growth regulators.London:Granada Publishing,1982:21-27
2 Meng,X.Q.;Zhang,J.J.;Liang,X.M.;Dong,Y.H.;Huang,J.X.; Rui,C.H.;Fan,X.L.;Chen,F.H.;Wang,D.Q.J.Agric.Food Chem.,2009,57(2):610
3 Cramer,R.D.;Patterson,D.E.;Bunce,J.D.J.Am.Chem.Soc., 1988,110:5959
4 (a)Klebe,G.;Abraham,U.;Mietzner,T.J.Med.Chem.,1994,37: 4130 (b)Klebe,G.;Abraham,U.J.Comput.-Aided Mol.Des., 1999,13:1 (c)B?hm,M.;Stürzebecher,J.;Klebe,G.J.Med.Chem.,1999, 42:458 (d)Bord?s,B.;K?mives,T.;Lopata,A.Pest Manag.Sci.,2003, 59:393
5 (a)Liu,J.;Li,Z.M.;Wang,X.;Ma,Y.;Lai,C.M.;Jia,G.F.; Wang,L.X.Sci.China Ser.B-Chem.,1998,41(1):50 (b)Hou,T.J.;Li,Z.M.;Li,Z.;Liu,J.;Xu,X.J.J.Chem.Inf. Comput.Sci.,2000,40(4):1002 (c)Wang,B.L.;Ma,N.;Wang,J.G.;Ma,Y.;Li,Z.M.;Li,Y.H. Acta Phys.-Chim.Sin.,2004,20(6):577 [王寶雷,馬 寧,王建國,馬 翼,李正名,李永紅.物理化學學報,2004,20(6):577] (d)Ma,Y.;Jiang,L.;Li,Z.M.;Lai,C.M.Chem.J.Chin.Univ., 2004,25(11):2031 [馬 翼,姜 林,李正名,賴城明.高等學校化學學報,2004,25(11):2031] (e)Ban,S.R.;Niu,C.W.;Chen,W.B.;Ren,X.B.;Yu,Z.H.;Xi, Z.Chem.J.Chin.Univ.,2007,28(3):543 [斑樹榮,牛聰偉,陳文彬,任曉白,余志紅,席 真.高等學校化學學報,2007,28(3): 543] (f)Yu,Z.H.;Niu,C.W.;Ban,S.R.;Wen,X.;Xi,Z.Chin.Sci. Bull.,2007,52(14):1929 (g)Guo,W.C.;Ma,Y.;Li,Y.H.;Wang,S.H.;Li,Z.M.Acta Chim.Sin.,2009,67(6):569 [郭萬成,馬 翼,李永紅,王素華,李正名.化學學報,2009,67(6):569]
6 (a)Ren,T.R.;Chen,H.M.;Xie,G.R.;Zhou,J.J.;Chen,F.H. Chem.J.Chin.Univ.,1998,19(12):1950 [任天瑞,陳紅明,謝桂榮,周家駒,陳馥衡.高等學校化學學報,1998,19(12):1950] (b)Yang,G.F.;Liu,H.Y.;Yang,X.F.;Yang,H.Z.Acta Chim. Sin.,1999,57(7):706 [楊光富,劉華銀,楊秀風,楊華錚.化學學報,1999,57(7):706] (c)Yang,G.F.;Liu,H.Y.;Yang,X.F.;Yang,H.Z.Acta Phys.-Chim.Sin.,1999,15(2):190 [楊光富,劉華銀,楊秀風,楊華錚.物理化學學報,1999,15(2):190]
7 (a)Liu,X.L.;Sun,M.;Wen,X.;Qi,L.N.;Miao,F.M.Comput. Appl.Chem.,2000,17(1-2):11 [劉小蘭,孫 命,文 欣,齊麗寧,繆方明.計算機與應用化學,2000,17(1-2):11] (b)Han,X.F.;Liu,Y.X.;Liu,Y.;Lai,L.H.;Huang,R.Q.;Wang, Q.M.Chin.J.Chem.,2007,25(8):1135 (c)Liu,Y.X.;Wei,D.G.;Zhu,Y.R.;Liu,S.H.;Zhang,Y.L.; Zhao,Q.Q.;Cai,B.L.;Li,Y.H.;Song,H.B.;Liu,Y.;Wang,Y.; Huang,R.Q.;Wang,Q.M.J.Agric.Food Chem.,2008,56(1): 204
8 (a)Li,A.X.;Wang,J.L.;Su,H.Q.;Sun,M.;Yu,M.;Miao,F.M. Comput.Appl.Chem.,2000,17(1-2):27 [李愛秀,王謹玲,蘇華慶,孫 命,郁 銘,繆方明.計算機與應用化學,2000,17(1-2): 27] (b)Su,H.Q.;Wang,J.L.;Li,A.X.;Sun,M.;Miao,F.M.Chem. Res.Appl.,1999,11(6):626 [蘇華慶,王謹玲,李愛秀,孫 命,繆方明.化學研究與應用,1999,11(6):626]
9 Wang,J.L.;Li,A.X.;Su,H.Q.;Sun,M.;Miao,F.M.Acta Chim. Sin.,1999,57(12):1291 [王謹玲,李愛秀,蘇華慶,孫 命,繆方明.化學學報,1999,57(12):1291]
10 Soung,M.G.;Lee,Y.J.;Sung,N.D.Bull.Korean Chem.Soc., 2009,30(3):613
11 Peng,H.;Wang,T.;Xie,P.;Chen,T.;He,H.W.;Wan,J.J.Agric. Food Chem.,2007,55(5):1871
12 Feng,X.;Yao,J.H.;Lü,L.;Tang,Q.H.;Fan,B.T.Acta Chim. Sin.,2006,64(11):1097 [馮 驍,姚建華,呂 龍,唐慶紅,范波濤.化學學報,2006,64(11):1097]
13 Wang,J.L.;Li,A.X.;Yu,M.;Zhai,X.H.;Miao,F.M.Comput. Appl.Chem.,2000,17(1-2):25 [王謹玲,李愛秀,郁 銘,翟秀紅,繆方明.計算機與應用化學,2000,17(1-2):25]
14 Wang,B.L.;Wang,J.G.;Ma,Y.;Li,Z.M.;Li,Y.H.;Wang,S.H. Acta Chim.Sin.,2006,64(13):1373 [王寶雷,王建國,馬 翼,李正名,李永紅,王素華.化學學報,2006,64(13):1373]
15 HQSAR is the product of Tripos Inc.,St.Louis,MO,1997
16 Avery,M.A.;Alvim-Gaston,M.;Woolfrey,J.R.J.Med.Chem., 2002,45:292
17 Li,H.;Zhang,H.B.Acta Chim.Sin.,2005,63(11):1018 [李 華,張華北.化學學報,2005,63(11):1018]
18 Wang,X.D.;Xiao,Q.F.;Cui,S.H.;Liu,S.S.;Yin,D.Q.;Wang, L.S.Sci.China Ser.B-Chem.,2005,35(1):58 [王曉棟,肖乾芬,崔世海,劉樹深,尹大強,王連生.中國科學B輯:化學,2005,35 (1):58]
19 SYBYL 7.3 is available from Tripos Associates Inc.,1699 S. Hanley Rd.,St.Louis,MO631444,USA
20 Xu,X.J.;Hou,T.J.;Qiao,X.B.;Zhang,W.Computer-aided drug design.Beijing:Chemical Industry Press,2004 [徐筱杰,侯廷軍,喬學斌,章 威.計算機輔助藥物分子設計.北京:化學工業出版社,2004]
21 Luo,X.Chemometrics.Beijing:Science Press,2001 [羅 旭.化學統計學.北京:科學出版社,2001]
22 Jiang,Y.R.;Qin,W.Acta Phys.-Chim.Sin.,2008,24(10):1859 [蔣玉仁,秦 偉.物理化學學報,2008,24(10):1859]
23 Masahiro,N.Toxicol.Lett.,1998,103:627
November 27,2009;Revised:January 25,2010;Published on Web:March 9,2010.
QSAR of Macrolactone Derivatives with Herbicidal Activity
DUAN Hong-Xia WANG Rui-Gang ZHANG Jian-Jun DONG Yan-Hong LIANG Xiao-Mei*WU Jing-Ping WANG Dao-Quan*
(Key Laboratory of Pesticide Chemistry and Application Technology,Ministry of Agriculture, Department of Applied Chemistry,China Agricultural University,Beijing 100193)
Westudiedthequantitativestructure-activityrelationships(QSAR)ofmacrolactone derivatives.Statistical results fromthe established comparative molecularfield analysis(CoMFA),the comparative molecular similarity indices analysis(CoMSIA),and the hologram quantitative structure-activity relationship(HQSAR)models showed believable predictability based on the cross-validated value(r2cv>0.5)and the non cross-validated value(r2>0.8).A contour maps analysis of the CoMFA and CoMSIA models showed that the hydrophobic and hydrogen-bond acceptor fields are important factors that affect the herbicidal activity of macrolactone compounds except for the steric and electrostatic fields.The structural modification information from the different atom contributions in the HQSAR model is in agreement with those of the 3D-QSAR models.According to the results from the CoMFA and CoMSIA models,the structure of compound B1-3 with the best herbicidal activity was modified and some designed compounds were predicted to have higher activity.
CoMFA;CoMSIA;HQSAR;Macrolactone;Herbicidal activity
O641
*Corresponding authors.Email:wangdq@cau.edu.cn,liangxiaomeim@yahoo.com.cn;Tel:+86-10-62732219.
The project was supported by the National Key Basic Research Program of China(973)(2003CB114407).
國家重點基礎研究發展計劃(973)(2003CB114407)資助項目
王道全,1978年10月考取北京大學分子工程與化學學院(原化學系)研究生,師從張滂院士,從事有機合成研究,1984年3月獲理學博士學位.

