不同國家菜豆核DNA ITS序列分析
歐立軍 ,張人文 ,譚智文 ,蔣向輝 ,佘朝文
(1.民族藥用植物資源研究與利用湖南省重點實驗室,湖南懷化,41800;
2.湘西藥用植物與民族植物學湖南省高校重點實驗室;3.懷化學院生命科學系)
菜豆(Phaseolus vulgarisL.)又名四季豆、梅豆、蕓豆,因其色澤嫩綠、肉莢肥厚、味道鮮美、極富營養價值而深受廣大消費者的喜愛,是一種鮮嫩可口,色、香、味俱全,營養豐富的優質蔬菜。19世紀末期之前,人們認為普通菜豆起源于亞洲[1]。根據形態學與生物化學的證據顯示,秘魯南部、玻利維亞和阿根廷地區是野生菜豆的一個起源中心,即安第斯中心;墨西哥、危地馬拉、巴拿馬、洪都拉斯、尼瓜拉加、哥斯達黎加和哥倫比亞等地區是另一個起源中心,即中美洲中心[2]。野生菜豆在這2個中心分別被馴化成栽培植物,然后才逐漸被引入世界各地,包括南美、北美、非洲、歐洲和亞洲的一些地區[3]。普通菜豆于16世紀初傳到歐洲,16世紀和17世紀在歐洲迅速傳播,并且在西班牙和葡萄牙北部地區形成了大粒白蕓豆的次級多樣性中心[4~5]。隨后,普通菜豆由這一地區傳播到歐洲其他地區,再由歐洲傳入中東、西亞以及世界上其他國家和地區[6]。普通菜豆傳入非洲的具體時間不太清楚,但是2個起源中心的材料在非洲都存在并且主要以安第斯中心的材料為主[4]。鄭卓杰等[7]認為,中國栽培的菜豆是15世紀直接從美洲引入的,但尚缺乏直接有力的證據。
高等植物中的rRNA基因是高度重復的串聯序列,18S、5.8S和26S rDNA連結在一起作為一個轉錄單位,拷貝數可達500~40 000,ITS序列包括被5.8S隔開的ITS1和ITS2。編碼18S、5.8S和26S的序列為高度保守區,ITS序列為進化速度較快的中度保守序列,且各重復單元間具有同步進化的特點。因此,ITS作為基因標記很快成為在序列水平上探討系統發育和進化研究的有效手段[8~11]。
本文基于ITS序列對來自不同國家的菜豆進行聚類分析,從分子水平探討菜豆種質的親緣關系,以期為菜豆核心種質篩選和優異種質培養提供理論依據,同時為確定菜豆的起源及次級起源中心提供參考。
1 材料與方法
1.1 試驗材料
16份菜豆種質來自亞洲、歐洲、美洲和非洲,每份材料取3個樣本(表1),由美國農業部種子庫和民族藥用植物資源研究與利用湖南省重點實驗室提供。
1.2 試驗方法
采取CTAB法提取DNA。ITS引物采用參考White 等[12],5'-TCCTCCGCTTATTGATATGC-3',5'-GGAAGGTAAAAGTCAAGG-3',反應體系包括10×PCR buffer 5 μL,10 mmol/L dNTP 1 μL,50 mmol/L 引物各 1 μL,DNA 40 ng,Taq DNApolymerase 0.4 μL,補充雙蒸水至50 μL。反應程序為:95℃預變性4 min;94℃變性 45 s,56℃退火 45 s,72℃延伸 45 s,35 個循環;72℃后延伸10 min。PCR產物純化用購自上海生工的DNA純化試劑盒。純化產物鑒定后送往上海生工雙向測序。所得序列采用 DNAMAN和MEGA.4軟件進行分析并提交Genebank得到登錄號。
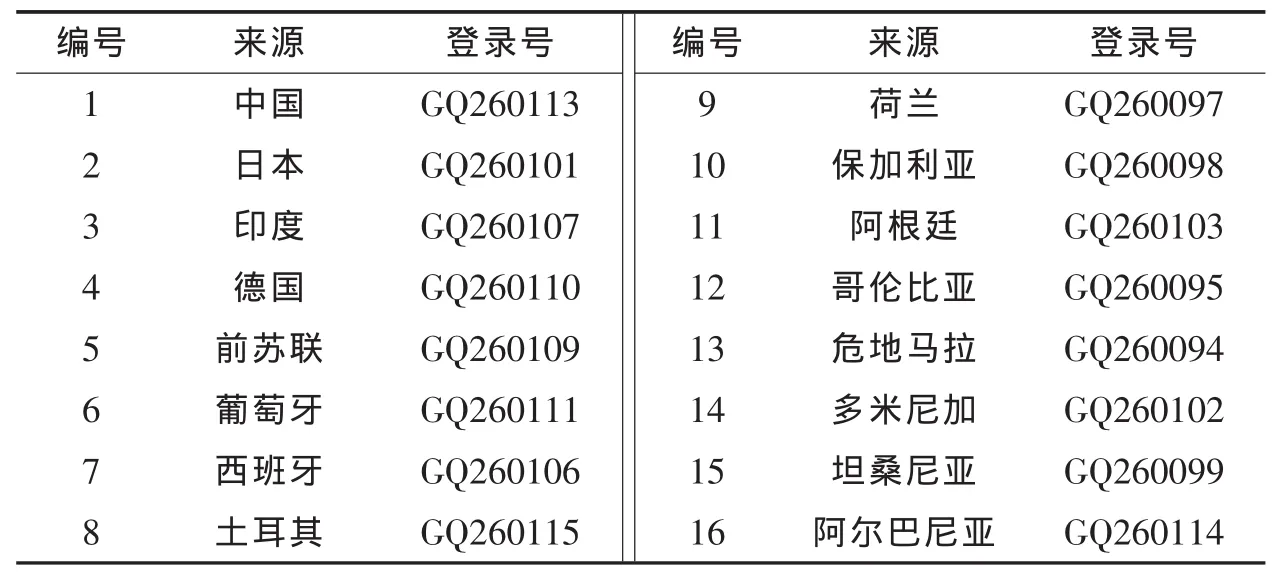
表1 材料及來源
2 結果與分析
2.1 ITS序列分析
PCR擴增結果顯示,ITS片段大小在700 bp左右(圖1a),純化后條帶清晰,達到測序要求(圖1b)。測序分析發現,ITS序列長度很保守,為645 bp或635 bp,片段大小相差10 bp,變異為1.5%;GC含量變化較大,在52.8%~57.1%波動;ITS1片段為 250 bp或259 bp,GC含量較高,變化范圍不大,在58.7%~59.9%波動;ITS2片段長度為222 bp或223 bp,相差僅1個bp,GC含量在57.9%~60.3%波動(表2)。
2.2 遺傳相似度分析
16份種質的ITS1相似度為88.13%,ITS2相似度為 85.14%,ITS1的相似度高于ITS2。除種質14和16外,其他14份種質的相似度很高,其中ITS1的相似度達到99.35%,ITS2則為93.56%,以上結果說明ITS2片段的遺傳多態性高于ITS1片段。
2.3 同源樹分析
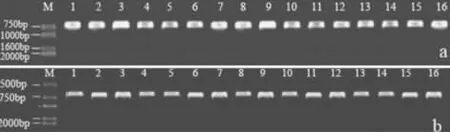
圖1 菜豆ITS PCR擴增(a)和純化(b)
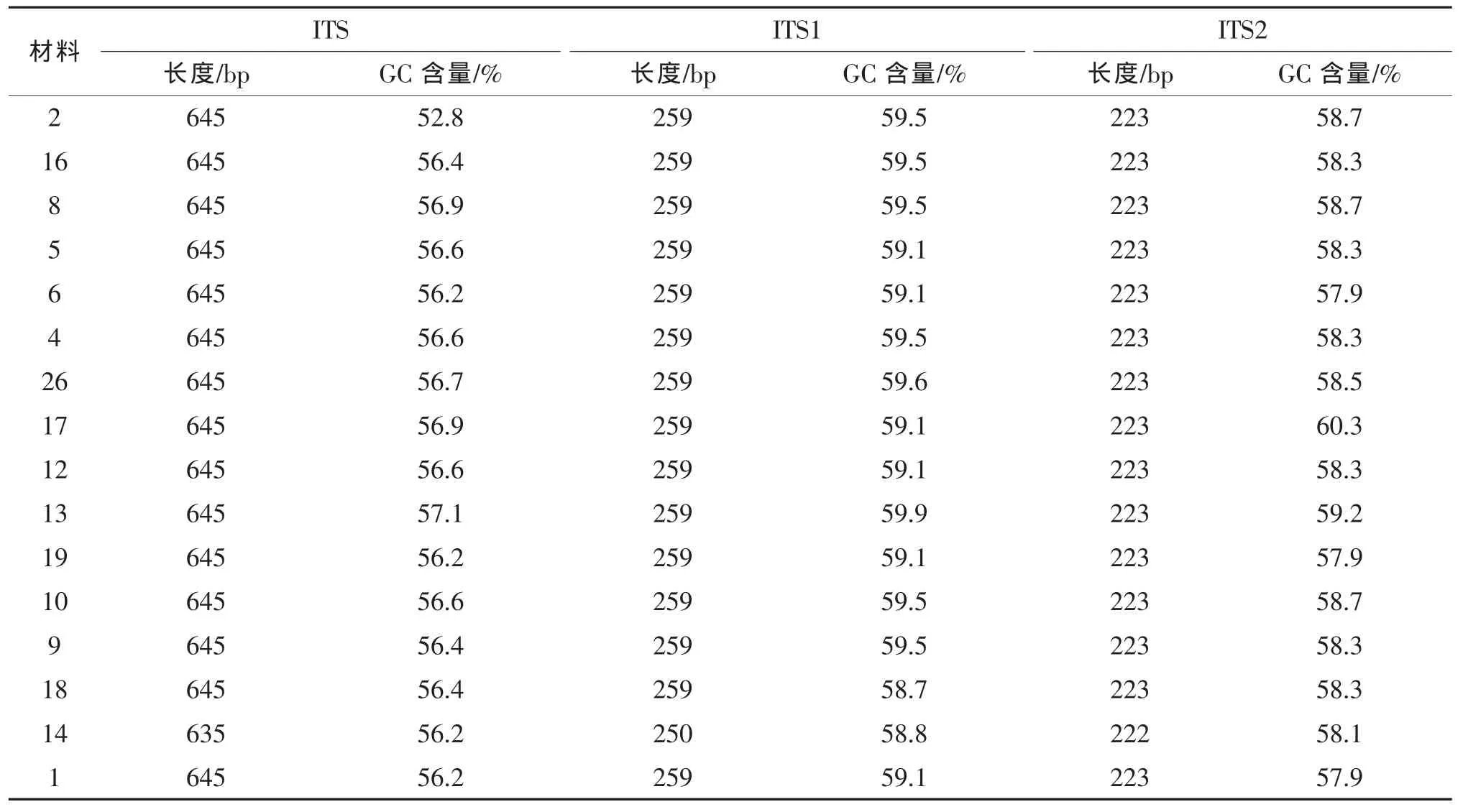
表2 菜豆ITS序列長度和GC含量
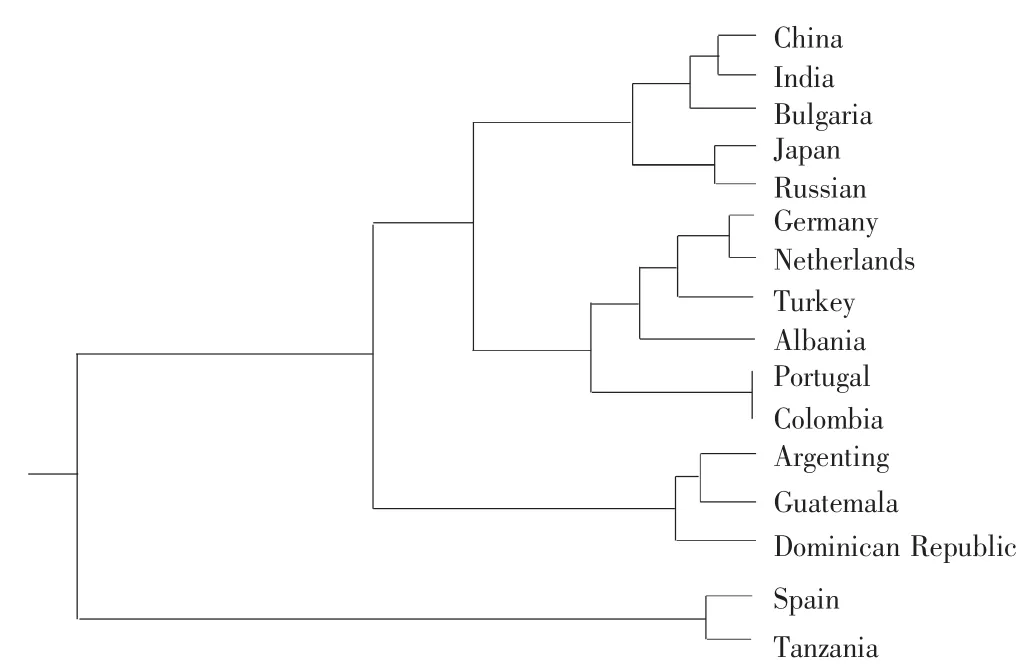
圖2 基于ITS序列構建的同源樹
基于ITS序列構建同源樹,不同菜豆種質被分為2大支,來自西班牙的菜豆與來自坦桑尼亞的菜豆為1大支,其他種質為另1大支。第2大支中,來自中國的菜豆優先與來自印度的菜豆聚類,然后與來自保加利亞的菜豆聚類;來自日本的菜豆與來自俄羅斯的菜豆優先聚類,其他來自歐洲的菜豆優先聚類后與來自亞洲的菜豆聚類,然后與來自美洲的菜豆聚類(圖2)。
3 小結與討論
來自葡萄牙和哥倫比亞菜豆種質ITS序列完全相同,這與Santalla等[5]提出的普通菜豆于16世紀初由美洲傳到歐洲,然后16世紀和17世紀在歐洲迅速傳播,并且與在西班牙和葡萄牙北部地區形成了大粒白蕓豆的次級多樣性中心的說法一致。來自坦桑尼亞和西班牙的菜豆的ITS序列特點相近,序列長度均為635 bp(其他種質都為645 bp),在進化樹上聚為一類,表明坦桑尼亞的菜豆可能直接來自于歐洲的西班牙。來自非洲阿爾巴尼亞的菜豆則與來自歐洲的菜豆優先聚類,說明非洲的菜豆可能直接來自歐洲,這與Schoonhoven等[13]提出非洲是菜豆第2遺傳中心的說法不相一致,因此非洲是否為菜豆的次級起源中心有待深入研究。
來自中國的菜豆優先與來自歐洲的菜豆聚類后,再與來自美洲的菜豆聚類,表明中國的菜豆與歐洲的菜豆之間的親緣關系更為密切,與鄭卓杰[7]通過形態學比較提出中國的菜豆是15世紀直接從美洲引進的說法不一致。考證歷史發現,1497年,葡萄牙航海家達·伽馬經過加那利群島,繞好望角,經莫桑比克等地,于1498年到達印度西南部卡利卡特,1869年蘇伊士運河通航前,歐洲對印度洋沿岸各國和中國的貿易,主要通過這條航路,因此中國的菜豆可能是通過這條航路傳播過來的。通過基因組得到的結果比形態上得到的結果更可靠,雖然本研究僅僅分析了1個DNA片斷,但這個片段是常用來進行物種內遺傳進化的分子標記,因此本研究認為中國的菜豆是否直接從美洲引進值得商榷,并且更有可能是來自于歐洲。
[1]Linnaeus C.Species plantarum[M].London:England,Ray Society,1753.
[2]Gepts P,Bliss F A.Dissemination pathways of common bean(Phaseolus vulgrisFabaceae)deduced from phaseolin electrophoretic variability.II.Europe and Africa[J].Economic Botany,1988,42(1):86-104.
[3]Broughton W J,Hernández G,Blair M,et al.Vanderleyden Beans(Phaseolusspp.)-model food legumes[J].Plant and Soil,2003,252:55-128.
[4]Singh S P.Patterns of variation in cultivated common bean(Phaseolus vulgarisFabaceae)[J].Economic Botany,1989,43:39-57.
[5]Santalla M,Rodino A,De Ron A.Allozyme evidence supporting southwestern Europe as a secondary center of genetic diversity for common bean[J].Theoretical and Applied Genetics,2002,104:934-944.
[6]Smartt J,Simmonds N W.Evolution of Crop Plants[M].UK:Longman Scientific and Technical,1995:287-296.
[7]鄭卓杰.中國食用豆類學[M].北京:中國農業出版社,1997.
[8]Mao S,Kenji K,Eiji M,et al.Identification of medicinal A-tractylodes based on ITS sequences of nrDNA [J].Biol Pharm Bull,2006,29(2):315-320.
[9]Christopher H J,Kenneth J S,Harvey E B J.Evolutionary relationships,interisland biogeography,and molecular evolution in the Hawaiian violets(Viola:Violaceae)[J].American Journal of Botany,2009,96:2 087-2 099.
[10]Vijaykumar A,Saini A,Jawali N.Phylogenetic analysis of subgenus vigna species using nuclear ribosomal RNA ITS:evidence of hybridization among vigna unguiculata subspecies[J].Journal of Heredity,2010,101:177-188.
[11]Yan J,Deng J,Zhou C J,et al.Phenotypic and molecular characterization ofMadurella pseudomycetomatissp.nov.,a novel opportunistic fungus possibly causing black-grain mycetoma[J].Journal of Clinical Microbiology,2010,48:251-257.
[12]White T J,Bruns T,Lee S,et al.Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[M]//In:Innes M,Gelfand D,Sninsky J,et al.eds.PCR protocols:a guide to methods and applications,1990,315-322.
[13]Schoonhoven A V,Voyest O.Common beans:Research for Crop Improvement[M].CABI,1991.

