基于Cyt b基因序列變異探討鯡形目系統發育關系
王丹生,程遠志,徐田軍,王日昕
(1.遼東學院農學院,遼寧丹東 118001;2.浙江海洋學院海洋科學學院,浙江舟山 316004)
鯡形目Clupeiformes魚類共約401種,全世界范圍內均有分布,主要集中于印度洋和西太平洋,絕大多數種類棲息于熱帶亞熱帶海洋沿岸水域,少數則適應于淡水生態環境。鯡形目魚類包括眾多高經濟價值種類,如鯡魚Clupea pallasi、鰣魚Tenualosa reevesii、斑鰶Clupanodom punchtatus、寶刀魚Chirocentrus dorab和鳳鱭Coilia mystus等[1-2],是全球商業漁業資源的重要組成部分,2003年,鯡形目魚類捕獲量達到1 900×104t,占當年全球所有魚類捕獲量的25%(FAO,Fishery statistics 2003,http://www.fao.org/fi/statist/statist.asp)。黃鯽Setipinna taty、鳳鱭隸屬于鯡形目、鳀科Engraulidae,是我國長江、河口區和海洋近岸水域重要的經濟魚類[4-5]。
鯡形目劃歸為鯡形總目Clupeomorphe,該總目包括許多白堊紀的化石類群,而鯡形目則包括了全部的現生種類[5]。有關鯡形目的系統分類,已有較為廣泛的形態學研究,在鯡形目魚類單系起源以及與骨鰾類為姐妹群關系方面觀點一致[1,6-8]。然而鯡形目科間與科內系統分類關系目前爭議頗多,不同的學者由于個人觀點的不同,所編分類系統存在較大差異,如國內孟慶聞等[9]所編分類系統,鯡形目分為4科:齒鯡科Denticepitidae、鯡科CIupeidae、鳀科以及寶刀魚科Chirocentridae;而NELSON[10]則將其分為5科,隸屬于孟慶聞等劃定的分類系統中的鯡科鰳屬Ilisha、后鰭魚屬Opisthopterus、多齒鰳屬Pellona以及鋸腹鰳屬Pristigaster被劃歸為鋸腹鰳科Pristigasteridae。另外,GRANDE[5]和PATTERSON等[11]依據肋骨同肋椎骨比例以及椎體同肋骨連接部位2個共同衍征將寶刀魚科與鯡科劃入鯡超科,相反,DI[12]依據形態學分析后指出,寶刀魚科實際上與鳀科親緣關系更近。由于形態特征容易受到環境因素的影響,不同魚類長期在相似的生境中發展可能會使外部及骨骼形態表現出趨同性,僅利用形態特征對其分類會存在一定的偏差。分類上的混亂與爭議不僅影響鯡形目魚類系統進化研究,也會在一定程度上對養殖生產中苗種及親本鑒定造成困難,尤其是在人工繁育過程中,物種鑒定錯誤有可能在繁育過程中出現雜交現象而使人工繁育歸于失敗[13],因而有必要對鯡形目魚類系統發育關系尤其是爭議問題進行更為深入的研究以明確其分類關系。
物種進化的歷程被DNA所忠實地記錄,通過研究物種DNA序列變異信息,可追溯物種進化歷史,為重建系統發育關系提供重要線索[14-15]。隨著近年來DNA分子標記技術與分子系統學的快速發展,基于基因序列和分子數據對魚類親緣關系的分析以及種屬鑒定得到廣泛應用,較好地確定了許多形態上難以界定的分類關系。Cyt b基因進化速度適中,易用通用引物擴增和測序,且是線粒體基因組中唯一的結構與功能了解較為清楚的蛋白質編碼基因,在魚類分子系統學研究中應用較為廣泛。區又君等[16]通過測定4種石斑魚的Cyt b基因全長序列,并結合Genbank中7種石斑魚同源序列進行系統發育分析,支持將鞍帶石斑魚Epinephelus lanceolatus歸入石斑魚屬Epinephelus。章群等[17]利用Cyt b基因序列分析了鱖類的系統發育關系后認為長體鱖Siniperca roulei應從長體鱖屬Siniperca劃入到鱖屬Siniperca中。莊軒等[13]通過分析石斑魚亞科Epinephlinal 6屬28種Cyt b基因片段序列,從分子系統學角度研究了其分化順序以及親緣關系等。但是應用線粒體序列變異研究鯡形目魚類系統進化則鮮有報道。
本研究以我國東海舟山海域黃鯽、鳳鱭為研究對象,測定了其Cyt b基因全長序列,并結合所有已發布的鯡形目魚類Cyt b基因序列,進行分子系統學分析,探討鯡形目魚類系統發育關系,與形態學研究相互印證補充,以期從DNA水平為其系統進化提供依據。
1 材料與方法
1.1 樣品采集與總DNA提取
黃鯽、鳳鱭個體采集于浙江省舟山市沈家門碼頭,經形態學鑒定確認后剪取部分尾鰭用于總DNA提取。參照薩姆布魯克等[18]方法,抽提總DNA,瓊脂糖凝膠電泳檢測DNA的質量和濃度,將所得DNA溶于TE溶液中-20℃保存備用。
1.2 Cyt b基因擴增
Cyt b-F:5’-GTG ACT TGA AAA ACC ACC GTT-3’和 Cyt b-R:5’-CTC CAT CTC CGG TTT ACA A-GA C-3’,用以擴增黃鯽、鳳鱭的 Cyt b 基因全長[19]。PCR 擴增體系為 50 μL,包括:模板 2 μL,10×Reaction Buffer 5 μL,dNTP 各 0.2 mmol/L,上、下游引物各 0.2 μmol/L,Taq DNA 聚合酶 2 U,MgCl21.5 mmol/L。反應在Bio-Rad Mycycler PCR儀上進行。循環參數設置為:94℃預變性4 min,94℃變性30 s,55℃退火45 s,72℃延伸60 s,循環30次,最后于72℃延伸10 min。每組反應均設不含模板的空白對照。1.5%的瓊脂糖凝膠電泳檢測擴增產物。
1.3 純化回收,TA克隆與測序
擴增產物經1.5%的瓊脂糖凝膠電泳后,割取目的帶,利用TIANGEN瓊脂糖凝膠DNA回收試劑盒純化回收目的產物。將純化后的Cyt b的PCR產物與PMD-19T載體16℃連接過夜,轉化進DH5α感受態細胞。轉化子于涂有IPTG和X-gal含氨芐青霉素的LB平板上37℃培養15 h,藍白斑篩選,挑取單克隆用M13F/M13R引物雙向測序。
1.4 數據分析
用DNAstar程序包中的SeqMan進行序列拼接,獲得黃鯽、鳳鱭的Cyt b基因全長序列。從Genbank數據庫中下載所有已發布的鯡形目魚類Cyt b基因全長序列(表1)。利用MEGA4軟件進行所有Cyt b基因序列比對,并計算變異位點數、簡約信息位點數、堿基替換百分比、轉換顛換比率、相對遺傳距離、堿基組成與密碼子使用頻率。采用鄰接法(NJ),以南美肺魚Lepidosiren paradoxus為外群建立鯡形目系統發生樹,自舉檢驗1 000次評估系統樹各分枝的置信度。
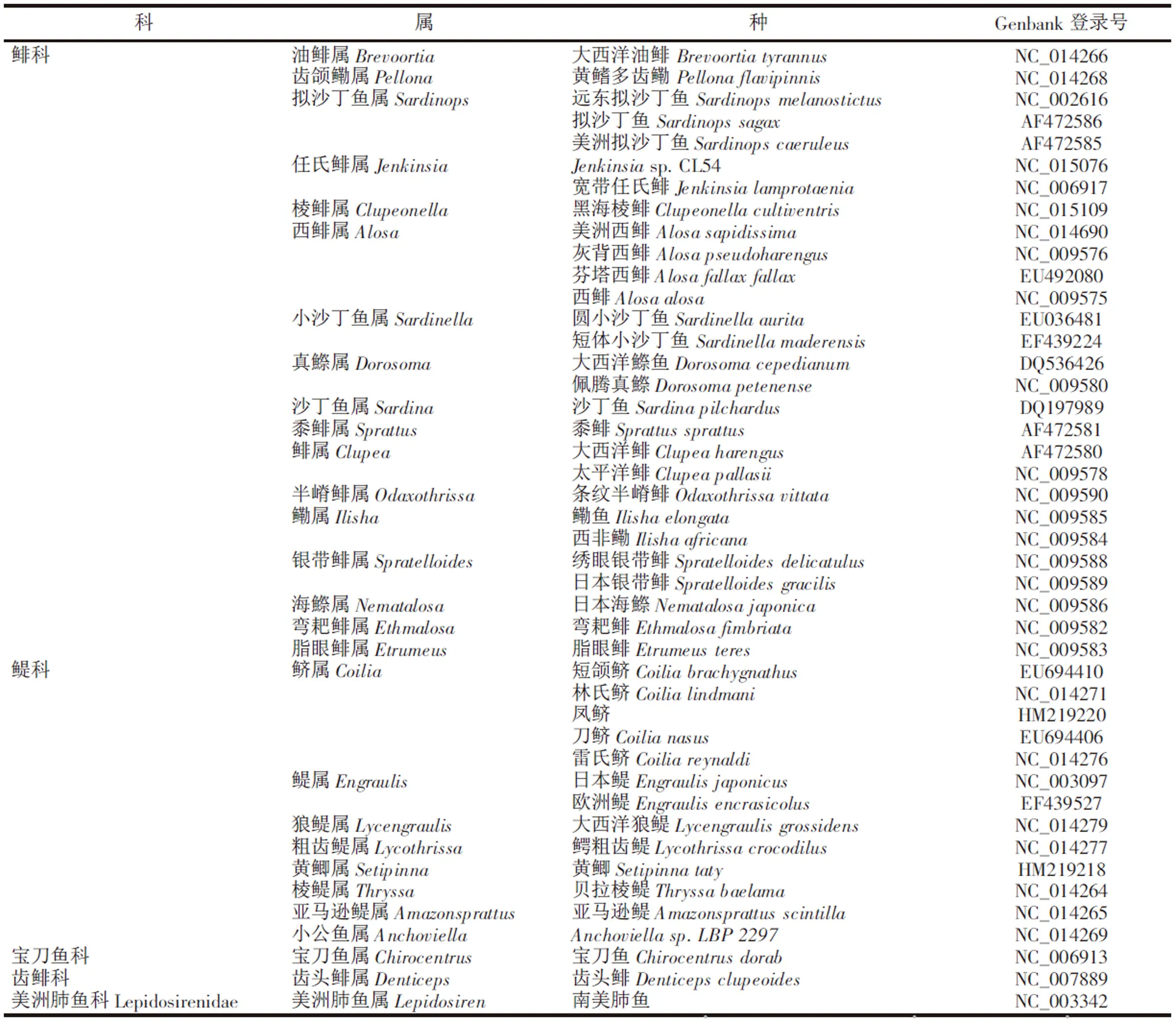
表1 本研究所用物種,Cyt b基因登錄號Tab.1 The list of species,Cyt b gene sequences accession numbers used in this study
2 結果與討論
2.1 Cyt b基因序列變異與堿基組成特征
經克隆測序所得黃鯽、鳳鱭的Cyt b基因序列全長為1 141 bp,編碼380個氨基酸,序列長度與編碼氨基酸個數同已報道的絕大多數魚類一致,如圖1所示。Blast比較分析也顯示兩者同其他鯡形目魚類Cyt b基因具極高的相似性[19-21]。序列提交至Genbank,獲得序列號,見表1。經比對分析所有43個鯡形目魚類Cyt b基因全長后,共檢測出變異位點553個,占全部序列的48.5%,其中504個簡約信息位點。總體而言,序列中轉換多于顛換,Ts/Tv為2.448,明顯大于2,表明所分析的Cyt b基因的突變并未達到飽和狀態,其中T-C轉換多于A-G,A-T和A-C的顛換多于G-T和G-C。T、C、A和G堿基的平均含量分別為28.8%、29.0%、25.2%和17.0%,A+T平均含量為54%,大于G+C的含量,同已報道的脊椎動物Cyt b基因以及整個線粒體基因組的堿基組成特征一致[22]。密碼子第三位G堿基(G-3)的平均含量為9.5%,明顯低于其余3種堿基含量,表現出反G偏倚,見表2。但是,不同種類Cyt b基因G-3含量存在較大個體差異性,例如沙丁魚G-3含量為20.8%,大于A-3的含量(20.3%),而在鱷粗齒鳀中,卻表現出極強的反G偏倚,G-3含量僅為1.8%。另外,一部分鯡形目魚類,如遠東擬沙丁魚、擬沙丁魚以及美洲擬沙丁的Cyt b基因G-3盡管表現出反G偏倚,但仍具有較高含量。在高級脊椎動物中,如鳥類和哺乳類中則有發現Cyt b基因密碼子第三位表現出反A和C堿基[23-24]。目前尚不清楚這些差異是否表明各個主要動物類群適應于不同的進化道路從而定向地累積了不同的變異,值得進一步的研究。

圖1 黃鯽與鳳鱭Cyt b基因序列比較Fig.1 Comparison of complete sequence of Cyt b gene in S.tay and C.mystus
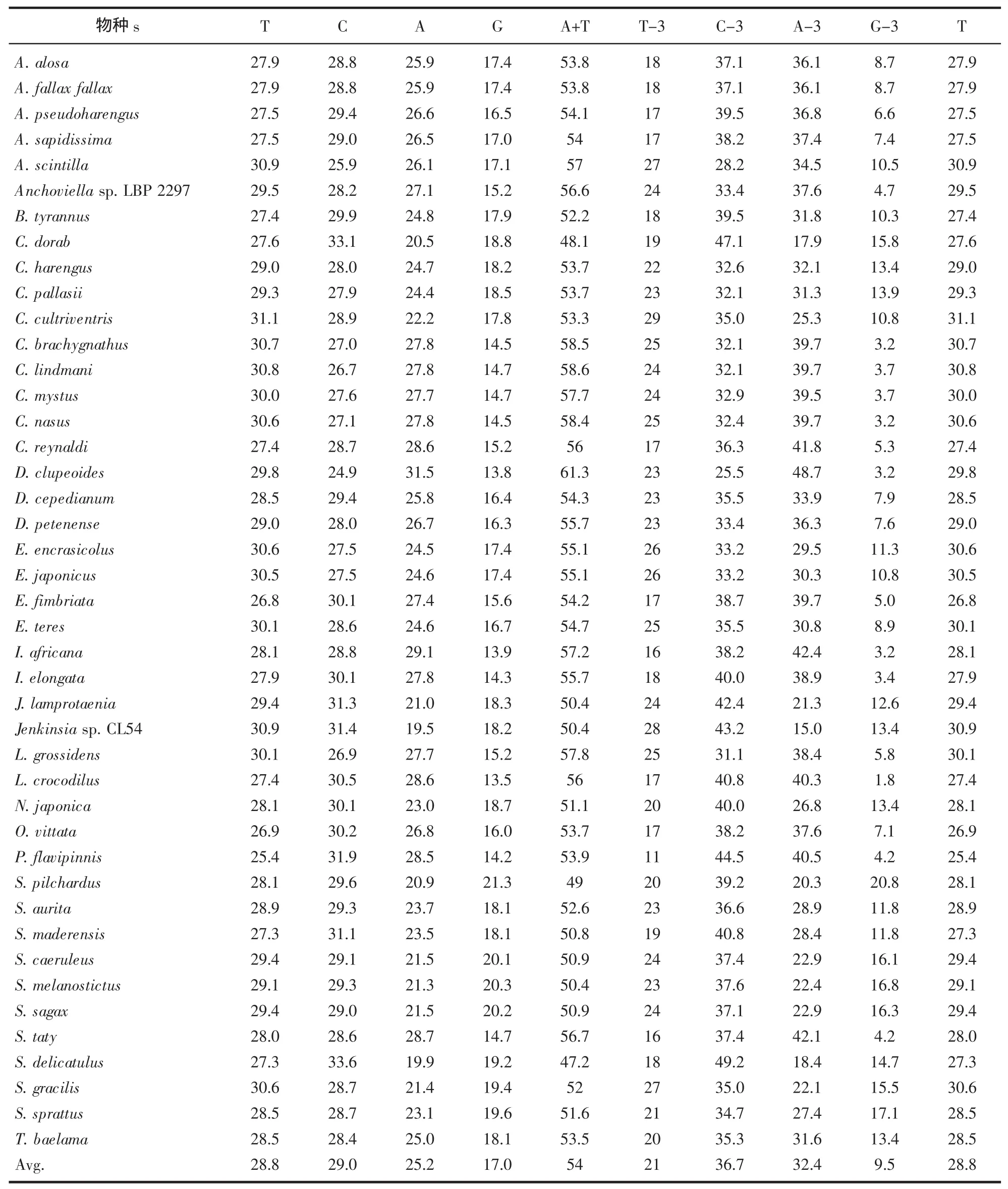
表2 鯡形目魚類Cyt b基因堿基組成Tab.2 Base composition of Cyt b gene of Clupeiformes fishes
2.2 鯡形目Cyt b基因密碼子分布
利用MEGA4軟件分析Cyt b基因密碼子分布情況,結果表明密碼子分布存在偏性。使用最多的密碼子為亮氨酸(CUA),平均為1.89%,其次是Cyt(GGA),平均為1.86%;使用最少的密碼為絲氨酸(AGU),為0.05%。在具有4個或2個同義密碼子的氨基酸中,以C堿基結尾的密碼子占最多數,其次為A堿基結尾的密碼子,見表3。研究發現,雖然脊椎動物mtDNA編碼的蛋白基因在序列上存在很大差異,但其中絕大部分為同義替代,且主要發生在密碼子第3位,該位置存在顯著的核苷酸偏性選擇,由于mtDNA進化速率較快,因此,同義取代可被認為是線粒體系統在進化速度較快的情況下保持自身蛋白質結構與功能穩定的一種方式[25]。另外,所有分析的Cyt b基因終止子均為不完全終止子T--,其在轉錄翻譯過程中的多聚腺苷酸化過程中,補齊成為完整的TAA終止密碼子[26]

表3 密碼子平均使用頻率Tab.3 Average frequence of codon usage
2.3 Cyt b基因相對遺傳距離
利用MEGA4軟件計算了鯡形目魚類相對遺傳距離(表4)。從表4可以看出,各物種之間的遺傳距離差異較大,在0.143~0.027之間。其中齒頭鯡與西鯡的遺傳距離最大(0.143),同時齒頭鯡與其他種類的遺傳距離也較大,約為0.1,表明齒頭鯡與其他物種之間親緣關系較遠,支持齒頭鯡科在鯡形目中處于分化較早的地位。鯡科內黍鯡與大西洋鯡遺傳距離最小(0.019),另外遠東擬沙丁同大西洋鰶魚與日本銀帶鯡間遺傳距離也較小,分別為0.024和0.027。寶刀魚科同鯡科、鳀科的科間遺傳距離同鯡科內屬間遺傳距離相當,甚至低于個別屬間遺傳距離,并未顯示出分化差異。

表4 鯡形目魚類Cyt b基因序列遺傳距離Tab.4 Genetic distance of Clupeiformes fishes Cyt b gene
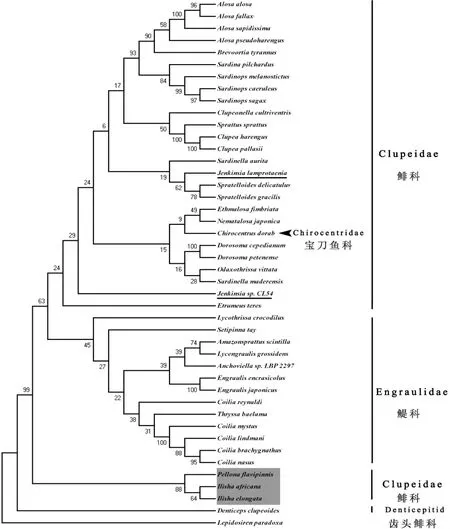
圖2 鯡形目NJ系統發育樹Fig.2 NJ phylogenetic tree of Clupeiformes
2.4 鯡形目系統發育關系
基于Cyt b基因變異構建的NJ系統樹,將43個鯡形目魚類分為4個大支(圖2)。第一支為齒鯡科,第二支由鯡科多齒鰳屬和鰳屬構成,寶刀魚科并入到鯡科中聚為第三支,最后一支為鳀科魚類組成。齒鯡科最先分化出來,為鯡形目中較為原始的類群。黃鯽、鳳鱭所在的鳀科獨立聚為一支,為單系發生,且黃鯽較鳳鱭分化更早,支持傳統形態學分類結果。在鯡形目科內分類關系層次上,孟慶聞等認為多齒鰳屬和鰳屬魚類隸屬于鯡科鋸腹鰳亞科[9],而NELSON則將鋸腹鰳亞科歸為獨立的一科。NJ系統樹表明鯡科并未能形成單系,黃鰭多齒鰳、鰳以及西非鰳,獨立于鯡科單獨聚為一支,且具較高的置信度,與LAVOUé等利用線粒體基因全序列構建鯡形目系統樹所得結論一致,支持鋸腹鰳亞科提升為鋸腹鰳科[27]。在LI等[28]利用線粒體12S rRNA,16S rRNA以及RAG1與RAG2核基因進行系統發育分析,以及LAVOUé所進行的鯡形目系統發育分析中,寶刀魚科寶刀魚同鯡科任氏鯡屬及銀帶鯡屬魚類親緣關系最近,與鳀科關系較遠,而我們的結果則表明寶刀魚科同鯡科海鰶屬和彎耙鯡屬關系最近,但都支持寶刀魚科同鯡科較近的親緣關系,與DI[12]的形態分類學結果不一致。另外,我們在Genbank中檢索到1條只具屬名的任氏鯡屬魚類Cyt b基因序列,Jenkinsia sp.CL54,但在NJ系統樹中,其并未與同屬的寬帶任氏鯡聚為一支,而是獨立形成一支,提示該物種可能并非任氏鯡屬魚類。
我們的研究結果與傳統分類不太相符,主要為鯡科單系地位以及寶刀魚科同鯡科及鳀科親緣關系,然而鯡形目魚類眾多,只有采集更多的樣品,開發并采用多個分子遺傳標記,基于不同的進化模型構建系統樹,并結合傳統形態分類結果進行比較分析,方能得出更接近真實的鯡形目物種系統進化歷史。
[1]WHITEHEAD P J P.FAO species catalogue.Vol.7.Clupeoid fishes of the world (suborder Clupeoidei)An annotated and illustrated catalogue of the herrings,pilchards,sparats,shads,anchovies and wold-herrings.Part1-Chirocentridae,Clupeidae and pristigasteridase.FAO-Fisheries-Synopsis.1985.
[2]FROESE R,PAULY D.FishBase.World Wide Web electronic publication[DB/OL].2004[2010-12-20].http://www.fishbase.org,version.
[3]丘書院.福建魚類志鯡形目[M].福州:福建科學技術出版社,1984.
[4]中國水產科學研究院東海水產研究所,上海市水產研究所.上海魚類志[M].上海:上海科學技術出版社,1990.
[5]GRAND L.Recent and fossil Clupeomorph fishes with materials for revision of the subgroups of clupeoids[J].Bull Am Mus Nat Hist,1985,181:231-372.
[6]GREENWOOD P H.The osteology and relationships of the Denticipitidae a family of Clupemorph Wshes denticeps-clupeoides palaeodenticeps-tanganikae[J].Bull Br Mus(Nat.-Hist.)Zool,1968,16:273.
[7]WHITEHEAD P J P.A synopsis of the clupeoid Wshes of India[J].J Mar Biol Assn India,1972,14:160-256.
[8]WONGRATANA T.Four new species of clupeoid Wshes Clupeidae and Engraulidae from Australian waters[J].Proc Biol Soc Wash,1987,100:104-111.
[9]孟慶聞,蘇錦祥,繆學祖,等.魚類分類學[M].北京:中國農業出版社,1995:606-622.
[10]NELSON J S.Fishes of the World[M].New York:J Wiley,1994.
[11]PATTERSON C,JOHNSON G D.The intermuscular bones and ligaments of teleostean Wshes[J].Smiths Contrib Zool,1995,559:1-85.
[12]DI D F.Rela??es Wlogenéticas entre os grandes grupos de Clupeomorpha e suas possíveis rela??es com Ostariophysi(Actinopterygii,Teleostei)[Z].Universidade de Sáo Paulo,2005:629.
[13]莊 軒,丁少雄,郭 豐,等.基于細胞色素b基因片段序列研究中國近海石斑魚類系統進化關系[J].中國科學:C輯,2006,36(1):27-34.
[14]RASMUSSEN A S,ARNASON U.Phylogenetic studies of complete mitochondrial DNA molecules place cartilaginous fishes within the tree of bony fishes[J].Mol Evol,1999,48:118-123.
[15]SACCONE C.The evolution of mitochondrial DNA[J].Curr Opin Genet Dev,1994,4:875-881.
[16]區又君,吳 勇,劉楚吾.從細胞色素b基因全序列研究鞍帶石斑魚的分類學地位[J].熱帶海洋學報,2008,27(3):45-49.
[17]章 群,任 崗,錢開誠,等.鱖類系統發育的線粒體Cyt b基因全序列分析[J].生態科學,2006,25(5):430-432.
[18]薩姆布魯克J,拉塞爾D W.分子克隆實驗指南[M].第3版.北京:科學出版社,2002.
[19]徐田軍,王日昕,王健鑫.舟山海域4種鯛科魚類線粒體Cyt b基因全序列克隆分析[J].南方水產,2010,6(1):30-36.
[20]田蘭香,梁 冰,張樹義,等.細胞色素b基因序列與7種石首魚類的系統進化[J].臺灣海峽,2004,23(4):436-443.
[21]牟希東,白俊杰,葉 星,等.金魚線粒體Cyt b基因序列分析及與鯽魚親緣關系探討[J].南方水產,2007,3(1):26-30.
[22]MIYA M,TAKESHIMA H,ENDO H,et al.Major patterns of higher teleostean phylogenies:a new perspective based on 100 complete mitochondrial DNA sequences[J].Mol Phylogenet Evol,2003,26:121-138.
[23]IRWIN D M,KOCHER T D,WILSON A C.Evolution ofthe cytochrome b of mammals[J].Mol Evol,1991,32:128-144.
[24]EDWARDS S V,ARCTANDER P,WILSON A C.Mitochondrial resolution of a deep branch in the genealogical tree for perching birds[J].Proceedings:Biol Sci,1991,B243:99-107.
[25]郭新紅,劉少軍,劉 巧,等.魚類線粒體研究新進展[J].遺傳學報,2004,31(9):983-1 000.
[26]OJALA D,MONTOYA J,ATTARDI G.tRNA punctuation model of RNA processing in human mitochondria[J].Nature,290:470-474.
[27]LAVOUé S,MIYA M,SAITOH K,et al.Phylogenetic relationships among anchovies,sardines,herrings and their relatives(Clupeiformes),inferred from whole mitogenmoe sequences[J].Molecular phylogenetics and evolution,2007,43:1 096-1 105.
[28]LI C H,ORTí G.Molecular phylogeny of Clupeiformes (Actinopterygii)inferred from nuclear and mitochondrial DNA sequences[J].Molecular phylogenetics and Evolution,2007,44:386-398.

