豬瘟病毒廣東流行毒株遺傳分型及序列分析
李儒曙 ,蘇惠龍 ,楊傲冰 ,禹思宇 ,黃 巖 ,向 華 ,張健騑
(1.珠海市動植物防疫監督檢驗中心,廣東 珠海 519000;2.珠海出入境檢驗檢疫局,廣東珠海 519000;3.廣東省農業科學院獸醫研究所廣東省獸醫公共衛生公共實驗室,廣東 廣州 510000)
豬瘟是由豬瘟病毒(CSFV)引起的一種高度接觸性傳染病,豬瘟對養豬業危害嚴重,可造成巨大損失,世界動物衛生組織(OIE)列為法定報告傳染病。本病在國內大規模暴發基本停止,但是在全國各地仍有不間斷的散發。豬瘟的流行和發病特點近年來也已發生了很大變化,其流行形式已從頻發的大流行轉變為周期性、波浪形的地區性散發性流行,通常3~4年一個周期,疫點顯著減少,多局限于所謂“豬瘟不穩定地區”散發性流行造成了巨大的經濟損失[1]。因此,豬瘟仍然是危害養豬業的頭號大敵,在近年“高熱病”肆虐的背景下,研究豬瘟的防制以及豬瘟病毒的遺傳進化具有重大的經濟和社會意義。
豬瘟病毒遺傳分型是追蹤CSFV傳播、變異以及持續性感染等極為有效的方法,CSFV的E2基因是國際上通用的用來進行豬瘟病毒遺傳分型的區域之一[2-3]。對現存市面上的豬瘟活疫苗毒株和流行于廣東省內的豬瘟野毒株E2基因進行序列測定、遺傳分型和比較分析,進而找出各毒株之間主要抗原結構差異與免疫保護的相關性,為控制豬瘟提供理論基礎。
1 材料與方法
1.1 病料樣品 豬瘟陽性可疑病料(組織21份、血清50份)于2006—2009年采自廣東省內,由廣東永順生物制藥有限公司技術服務部、佛山科技學院、廣東省農業科學院獸醫研究所豬病研究室和華南農業大學獸醫學院傳染病實驗室提供。疫苗毒株為市面上購買的不同廠家產品。
1.2 主要試劑、質粒載體及引物 提取總RNA所用裂解液Trizol為Invitrogen公司產品;cDNA第一鏈合成試劑盒、PCR試劑盒、質粒載體pMD18-T、DNA Marker DL2000均為寶生物工程(大連)有限公司產品;E.coli DH5α工程菌為北京鼎國生物科技公司。根據GenBank收錄的豬瘟病毒全基因組序列,通過軟件評價,采用中國獸藥監察所公開發表的一對 引 物[4],P :5'-CTGGTAACTGGGGCACAAG-3'、5'-GCAGTAGTATCCATTTCTTTAT-3'擴增區域包括E2基因ABCD編碼區、E2基因5' 端的部分信號肽序列和3' 端的跨膜區(TMR)序列。
1.3 樣品處理 組織樣品:無菌取待檢病料(脾臟、淋巴結或扁桃體)100 mg,剪成碎塊,制成組織勻漿,低速離心后取上清。血液樣品:4 000 r/min以下冷凍離心取上清,或常溫靜置讓血清自然析出。疫苗毒樣品:用PBS溶液稀釋后取上清。
1.4 豬瘟病毒E2全基因的克隆 取處理好的樣品200 μL 加入 800 μL的 Trizol?LS Reagent裂解液,總RNA的抽提以及RT-PCR按照試劑說明書操作,PCR反應結束后取反應產物以1%瓊脂糖凝膠進行電泳,凝膠成像系統檢測擴增結果。回收目的條帶,將純化的PCR產物與載體pMD18-T-載體進行連接反應,CaCl2法將連接產物轉化E.coli DH5α。經PCR菌液鑒定為陽性的克隆,送上海生工生物工程公司測序。
1.5 CSFV廣東流行株遺傳分型及生物信息學分析
測序結果經MegAlign軟件分析,確定本研究所測野毒株的遺傳分型,并參考Genbank中已發表的相關序列繪制系統進化發生樹。運用DNAStar中相關軟件模塊對本研究中所測野毒株序列和疫苗株序列作核苷酸和氨基酸的相似性比對,以及蛋白在分子親水性、分子表面氨基酸殘基的分布特征、柔性、抗原性差異等方面比較。
2 結果
2.1 豬瘟病毒E2全基因的RT-PCR結果
從三個疫苗廠家4個批次的豬瘟病毒兔化弱毒疫苗以及來自茂名、佛山、珠海、惠州、廣州的6份組織樣品中分別擴增出約1.3 kb大小的特異性條帶,與預計產物大小相符。經菌液PCR鑒定、測序確證后提交GenBank,將本研究中這6個野毒株擴增的E2基因片段按分離地分別編號為“Huizhou06 (GenBank登錄號:HQ697222)”、“Foshan08 (GenBank登錄號 :HQ697224) ”、“Guangzhou06(GenBank 登錄 號 :HQ697225)”、“Guangzhou09(GenBank 登錄 號 :HQ697223)”、“Maoming07(GenBank 登錄 號 :HQ697227)”、“Zhuhai07(GenBank 登 錄號:HQ697228)”。將不同廠家的疫苗毒分別編號為“VYS”、、“VDHN”、“VZM”、“VZM1”。
2.2 豬瘟病毒E2全基因的生物信息學分析
2.2.1 豬瘟病毒的遺傳分型(Genetic Typing)
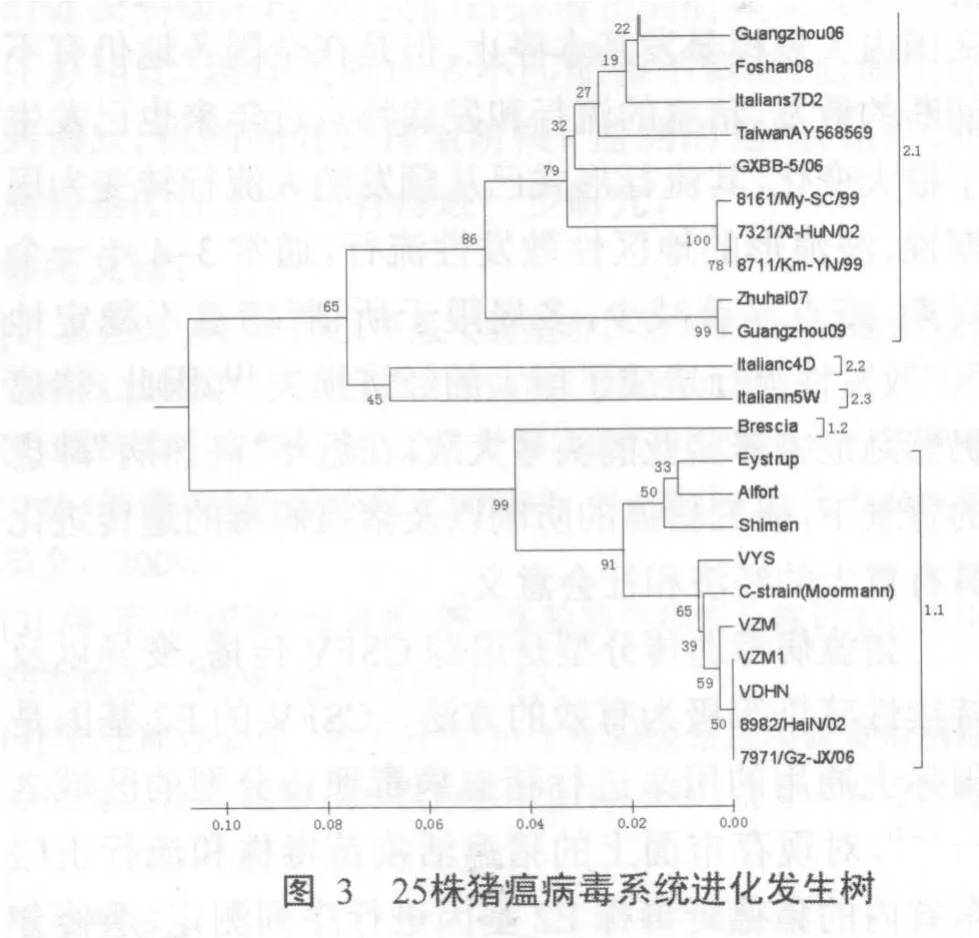
對25株豬瘟病毒E2基因的完全或部分序列進行序列比對,這25個病毒序列分別是本研究測得的10個病毒基因序列,以及GenBank中調用的15個國內外公開發表的毒株序列,其中引用的國內發表序列主要是廣東周邊省份上世紀末和本世紀初分離的毒株序列,并參照5個國外參考毒株序列Alfort187(subgroup1.1)、Brescia(subgruop1.2)、s7D2(subgroup2.1)、c4D(subgroup2.2)、n5W(subgroup2.3),繪制進化發生樹。系統進化發生樹顯示本研究中病毒基因分型結果顯示所測豬瘟病毒廣東野毒株全部分布在2.1亞群;所測疫苗毒株位全部屬于1.1亞群。
2.2.2 8株CSFV核苷酸和氨基酸的相似性、趨異性比對
運用DNAStar MegAlign對5個豬瘟病毒廣東野毒株基因序列、2個疫苗毒株基因序列和Shimen經典強毒株基因序列進行核苷酸和氨基酸的相似性比較。其中豬瘟疫苗株序列VYS與國外所測的C株序列C-strain(Moormann)核苷酸同源性為98.7%、氨基酸同源性為99.1%。本試驗所測的四個國內豬瘟疫苗株的核苷酸同源性均為在99.0%以上,氨基酸的同源性均在98.8%以上;其中來源于同一個廠家的 “VZM”、“VZM1”兩個毒株的核苷酸的同源性為99.6%,氨基酸的同源性為99.5%。
6個廣東野毒株與VYS株相比, 核苷酸的相似性從高到低為:Maoming07(82.5%)>Foshan08(82.2%)>Zhuhai07(82.0)> Huizhou06(81.8%)=Guangzhou09(81.8%)>Guangzhou06(80.9%);氨基酸的相似性從高到低為:Maoming07(89.6%)=Zhuhai06(89.6%)>Guangzhou09(89.4%)>Huizhou06(88.9%)>Foshan(88.4%)>Guangzhou06(83.3%)。
6個野毒株與Shimen株相比,核苷酸的相似性 從 高 到 低 為 :Maoming07(83.7%)>Foshan08(83.3%)>Zhuhai07(82.7)= Huizhou06 (82.7%)>Guangzhou09(82.5%)>Guangzhou06(81.2%);氨基酸的相似性從高到低為:Zhuhai07(91.0%)>Maoming07(90.8%)=Guangzhou09(90.8%)>Huizhou06(90.1%)>Foshan08(89.4%)>Guangzhou06(85.6%)
2.2.3 分子親水性、蛋白質骨架柔韌性、抗原性的差異
通過生物信息學軟件DNAStar Protean分析比較5個豬瘟病毒廣東野毒株和疫苗株E2蛋白在分子親水性、蛋白質骨架柔韌性、抗原性的差異。E2蛋白在分子親水性、抗原性具有較高的相似性;在蛋白質骨架的柔韌性方面有一定的相似性,同時也存在著一定的差異性。
2.2.4 跨膜疏水區(TMR)的差異性分析
在TMR序列中,6個野毒株在圖中標尺中382、400、412、413四處氨基酸對應均為Q、N、I、I,而疫苗株和Shimen強毒株在此四處對應氨基酸均為P、I、V、M。這四處是不是2.1亞群和1.1亞群之間的遺傳標記有待研究并值得所有研究者關注。

3 討論
豬瘟病毒毒株間的差異取決于所比較的基因組靶區域的長度和變異性。關于豬瘟病毒基因組的哪一區域用來進行遺傳比較更科學,哪一種比較方法最有意義目前尚有爭議。5'NTR、E2和NS5B基因是CSFV遺傳分型時應用最廣泛的基因組區域,對這三種分型方法結果進行分析比較,證實利用NS5B進行遺傳分型的可信度最高,5'NTR的可信度最低[5]。雖然普遍認為采用NS5B基因序列進行豬瘟病毒的遺傳分型具有較高的置信度,但由于E2片段的易變性,采用E2基因序列進行遺傳分型在保證一定的置信度的同時卻有更好的區分度。為了保持世界各地序列數據的一致性,國外以E2基因對豬瘟病毒分型通常采用德國漢諾威豬瘟參考實驗室公開發表的一對引物:5'-TCRWCAACCAAYGAGATAGGG-3'(Alfort株 基因組的2467-2487位)、5'-CACAGYCCRAAYCCRA AGTCATC-3'(Alfort株基因組的2738-2716位株其因組的),擴增片段長度為190 bp[3]。筆者認為190 bp的片段長度相對于完整E2片段長度來說,擴增的區域較短,而采用序列比對的方法對病毒進行遺傳分型時,比對的序列越長越有利于不同毒株的區分。本研究擴增的6個野毒株和4個疫苗毒株序列均為1 273 bp,包括了豬瘟病毒完整的E2基因序列,這些序列的獲得使不同毒株的進化分析具有較高的置信度和區分度。由于病毒的變異性,采用簡并引物更能有效地進行PCR擴增,本研究所采用的引物為非簡并引物,這大概是本實驗在RT-PCR檢測豬瘟可疑病料過程中有效擴增率較低的原因。毒株的數量越大,得到的序列越多,越能有效說明問題[5]。但從大量“高熱病”和“疑似豬瘟”的病料中,我們只有效擴增了6個CSFV野毒序列。雖有效擴增數目偏少,對反映各毒株之間的關系以及病毒流行等相關問題有一定的局限性,但另一方面可以說明“高熱病”的復雜性,此結果對我們探討“高熱病”與豬瘟的關系有一定的參考價值。
本研究所測6株豬瘟野毒均為2.1亞群,可以推測近年廣東省流行的豬瘟病毒主要以2.1亞群為主。廣東省目前是我國養豬業規模化、集約化程度最高的省份之一,與外省在種豬、生豬、豬苗等方面的交易都非常活躍,從而造成廣東省豬瘟病毒毒株的復雜性和多樣性,從這個意義上說,廣東省流行的病毒毒株具有一定的代表性,廣東省流行毒株可以間接反映全國特別是華南地區毒株的流行情況。本研究的結果與Tu等的研究結果一致[6],即豬瘟病毒2.1亞群基因型在我國流行毒株中占有最大的比重。
本研究測得的適應于牛睪丸細胞培養的豬瘟病毒疫苗株VYS與國外早年測得的豬瘟病毒C株相比,核苷酸的同源性為98.7%,氨基酸的同源性為99.1%;國內四個不同廠家的豬瘟病毒疫苗株之間無論是核苷酸還是氨基酸也具有極高的相似性,說明C株細胞疫苗毒的主要保護性抗原蛋白(gp55)基因遺傳穩定,微小的趨異性可能是由于病毒傳代的代次不同造成的。豬瘟病毒疫苗株VYS與6株豬瘟病毒廣東野毒株相比,核苷酸的相似性為82.5%~80.9%,氨基酸的相似性為89.6%~83.3%;豬瘟病毒Shimen經典強毒株與這6個野毒株相比,核苷酸的相似性為83.7%~81.2%,氨基酸的相似性為91.0%~85.6%。中國獸藥監察所對22株不同年代CSFV E2基因部分編碼序列作同源性比較,發現其中13株CSFV流行毒株的核苷酸和氨基酸的同源性范圍分別為78.1%~100%、78.4%~100%,4株70~80年代分離的毒株的核苷酸和氨基酸的同源性范圍分別為79.0%~88.3%、81.1%~87.8%,9株90年代分離的毒株的核苷酸及氨基酸同源性范圍分別為80.8%~100%、83.8%~100%[7]。其結果與本研究的結果比較接近,說明CSFV流行株的變異呈現一定的多樣性,隨著時間的推移,不是只向遠離疫苗株的方向變異,也不是向經典強毒株的方向變異。李紅衛[8]、韓雪清[9]等關于我國豬瘟流行毒株已向遠離疫苗毒株的方向變化的結論與本研究的試驗結果不符。
在這6個廣東野毒株、4個疫苗株和Shimen株的序列中也都含有一段非常保守的氨基酸序列ALNVVSRRYLASLHK,11株病毒在此處都沒有發生氨基酸變異,對這段保守氨基酸進行抗原性及疏水性分析,均為親水性,且抗原性指數在整個E2抗原譜中最高,其位置正好位于E2基因最具抗原性的B區和C區,而B、C區是誘導產生中和抗體最為重要的部分。Lowings等[3]發現細胞受體序列中也有極為類似的保守區RYLAIVH,因此推測該段保守性氨基酸除了與豬瘟病毒和宿主細胞的親嗜性以及宿主范圍有關外,還對E2糖蛋白抗原性起決定作用[4]。如果這種推測是正確的,那么疫苗的穩定性和有效性將得到解釋。
分析比較6個豬瘟病毒廣東野毒株和疫苗株E2蛋白在分子親水性、抗原性的差異,發現它們之間保持較高的相似性。通過軟件分析可以比較直觀地看出所測毒株在某些相同的肽段親水性指數和抗原性指數均較高,具有較高的相似性。而6個野毒株與疫苗株E2蛋白在蛋白質骨架的柔韌性方面有一定的相似性,也存在著一定的差異性,表明E2肽段的二級結構有較大的相似性,局部的差異可能導致高級結構的折疊有部分差異。這種高級結構的部分差異會不會影響疫苗免疫對野毒攻擊的保護效果值得研究。當然相似性略有差異的片段也有可能在功能上不會有差別,它們的差異可能來自非抗原決定簇的位點變異。我們假設認為E2肽段在抗原性和疏水性上與疫苗毒沒有顯著區別,影響抗原性的抗原決定簇的結構不會發生顯著變化,那么疫苗毒產生的中和抗體對豬群的保護是相當有效的。
位于C端TMR(1030-1063)是一段保守序列,這段跨膜疏水序列對誘發高效價中和抗體和產生完全免疫保護非常重要[10],有研究證實,用含有完整E2基因重組疫苗免疫豬所產生的中和抗體效價比不含TMR的重組疫苗產生的抗體效價高很多,并且前者抗體產生的速度也較快。所測的10株病毒的E2糖蛋白的TMR是完整的,且均形成4個疏水區,該疏水性結構對E2蛋白的穿膜作用十分重要,且使得E2糖蛋白鑲嵌在囊膜上十分牢固。野毒株和疫苗株的TMR具有一定的穩定性,從一方面保證了誘發中和抗體的穩定性。
軍事醫學科學院軍事獸醫研究所做了豬瘟兔化弱毒疫苗對動物性保護性試驗,試驗結果表明疫苗對感染豬瘟病毒2.1亞群、2.2亞群的豬具有完全的保護性[6]。而廣東省主要以豬瘟病毒2.1亞群流行為主,因此推測疫苗對目前廣東省的豬瘟防控是有效的。
[1] 杜念興.豬瘟的回顧與展望[J].中國畜禽傳染病,1998,20(5):317-319.
[2] Lowings J P, Paton D J,Sands J J,et al. Classical swine fever: genetic detection and analysis of differences between isolates. J Gen Virol,1994,75(12):3461-3468.
[3] Lowings P, Ibata G, Needham J,et al. Classical swine fever diversity and evolution[J]. J Gen Virol,1996,77(6),1311-1321.
[4] 王琴,李博,王在時,等.豬瘟病毒強弱毒株和野毒株E2全基因序列測定及比較分析[J].微生物學報,2001,41(3):320-327.
[5] Paton D J, McGoldrick A, Greiser-Wilke I, et al. Genetic typing of classic swine fever virus[J]. Vet Microbiol,2000,73(2/3):135-157.
[6] Tu C C, Lu Z J, Li H W, et al. Phylogenetic comparison of classical swine fever virus in China[J]. Virus Res2001,81(1):29-37.
[7] 趙耘,王在時,王琴等.22株豬瘟病毒E2基因部分編碼序列的序列分析[J].微生物學通報,2001,28(5):42-48.
[8] 李紅衛,涂長春,呂宗吉等.兩個豬瘟病毒野毒株gp55基因抗原編碼區序列的分析及比較[J].中國病毒學,1997,12(4) :354-357.
[9] 韓雪清,劉湘濤,趙啟祖,等.豬瘟病毒遺傳發生關系分析[J].中國獸醫科技,1999,29(6):3-7.
[10] Van Zijl M,Wensvoort G,De KluyverE,et al.Live attenuated pseudorabies virus expres sing envelope glycoprotein EI of hog cholera virus protects swine against both pseudorabies and hog cholera[J].J Virol,1991,65(5):2761-2765.

