單增李斯特氏菌MALDI-TOF-MS鑒定與分型研究
王 耀,曹際娟*,趙 昕,鄭秋月,王 剛,田 卓,史媛媛,曹遠(yuǎn)銀
(1. 沈陽農(nóng)業(yè)大學(xué)植物保護學(xué)院植物免疫研究所,遼寧 沈陽 110161;2. 遼寧出入境檢驗檢疫局,遼寧 大連 116001;3. 莊河出入境檢驗檢疫局,遼寧 莊河 121013;4. 營口出入境檢驗檢疫局,遼寧 營口 115000)
單增李斯特氏菌MALDI-TOF-MS鑒定與分型研究
王 耀1,2,曹際娟2,*,趙 昕2,鄭秋月2,王 剛2,田 卓3,史媛媛4,曹遠(yuǎn)銀1
(1. 沈陽農(nóng)業(yè)大學(xué)植物保護學(xué)院植物免疫研究所,遼寧 沈陽 110161;2. 遼寧出入境檢驗檢疫局,遼寧 大連 116001;3. 莊河出入境檢驗檢疫局,遼寧 莊河 121013;4. 營口出入境檢驗檢疫局,遼寧 營口 115000)
為建立單增李斯特氏菌的基質(zhì)輔助激光解吸電離飛行時間質(zhì)譜(MALDI-TOF-MS)快速鑒定與分型方法,實驗收集37株單增李斯特氏菌分離株,應(yīng)用MALDI-TOF-MS采集圖譜,獲取獨特的蛋白質(zhì)指紋圖譜,匯總成標(biāo)準(zhǔn)圖譜,建立單增李斯特氏菌鑒定數(shù)據(jù)庫。采用單增李斯特氏菌標(biāo)準(zhǔn)菌株進行驗證,表明鑒定結(jié)果的可信度很高。在數(shù)據(jù)庫信息的基礎(chǔ)上,對37株單增李斯特氏菌分離株進行聚類分型。分型結(jié)果表明,在蛋白質(zhì)水平上,MALDITOF-MS可把37株單增李斯特氏菌分成9個型別。
單增李斯特氏菌;基質(zhì)輔助激光解吸電離飛行時間質(zhì)譜(MALDI-TOF-MS);鑒定;分型
單增李斯特氏菌為典型的胞內(nèi)寄生菌,能在巨噬細(xì)胞和上皮細(xì)胞、內(nèi)皮細(xì)胞和肝細(xì)胞內(nèi)增殖,是一種人畜共患病病原菌[1-2]。該菌可引起人和動物患腦膜炎、腦炎、敗血癥及造成孕婦流產(chǎn)、死胎等疾病[3]。孕婦、新生兒、老年人和免疫缺陷者是易感人群[4]。食源性李斯特氏菌發(fā)病率雖不高,但死亡率可高達20%~50%[5]。WHO在20世紀(jì)90年代已將其列為食品四大致病菌之一[6]。該菌在自然界分布廣泛,以家畜、家禽為主要宿主,很容易污染食品而引起食物中毒和李斯特病暴發(fā)[7]。肉類、蛋類、禽類、海產(chǎn)品、乳制品、蔬菜等都已被證實是李斯特氏菌的感染源[8]。
經(jīng)典的單增李斯特氏菌檢測方法是進行增菌、選擇性分離培養(yǎng)、初篩、鑒定等步驟。這種方法耗時長,程序繁瑣,試劑和人力成本均較高。此外,還有免疫磁性分析法[9]、PCR法[10]、實時PCR法[11]等許多快速檢測方法。基質(zhì)輔助激光解析電離飛行時間質(zhì)譜(matrix-assisted laser desorption ionisation time-of-flight mass spectrometry,MALDI-TOF-MS)是近幾年發(fā)展起來的一種新型軟電離生物質(zhì)譜。它能完成細(xì)菌多種成分的分析,包括蛋白質(zhì)、脂類、脂多糖和脂寡糖、DNA、多肽及其他能被離子化的分子。細(xì)菌含有一些成分,能給出唯一的質(zhì)荷比,因此可作為生物標(biāo)志分子進行細(xì)菌的特異性鑒定。由于其具有快速、準(zhǔn)確、靈敏、分辨率高和高質(zhì)量檢測范圍等優(yōu)點,MALDI-TOF-MS已經(jīng)逐漸成為臨床診斷、食品生產(chǎn)、環(huán)境檢測以及軍事領(lǐng)域研究的一種有力手段[12]。但是細(xì)菌比對標(biāo)準(zhǔn)數(shù)據(jù)庫的資源不足是其局限性之一[13]。目前,Biotyper數(shù)據(jù)庫仍需要不斷補充單增李斯特氏菌的特征譜圖。因此,本研究利用MALDI-TOF-MS軟件支持用戶自定義數(shù)據(jù)庫的特點,按統(tǒng)一的建庫標(biāo)準(zhǔn),采集37株單增李斯特氏菌分離株的數(shù)據(jù)并獲得特征指紋圖譜,添加創(chuàng)建質(zhì)譜圖數(shù)據(jù)庫,并進一步開展單增李斯特氏菌鑒定和分子分型研究。
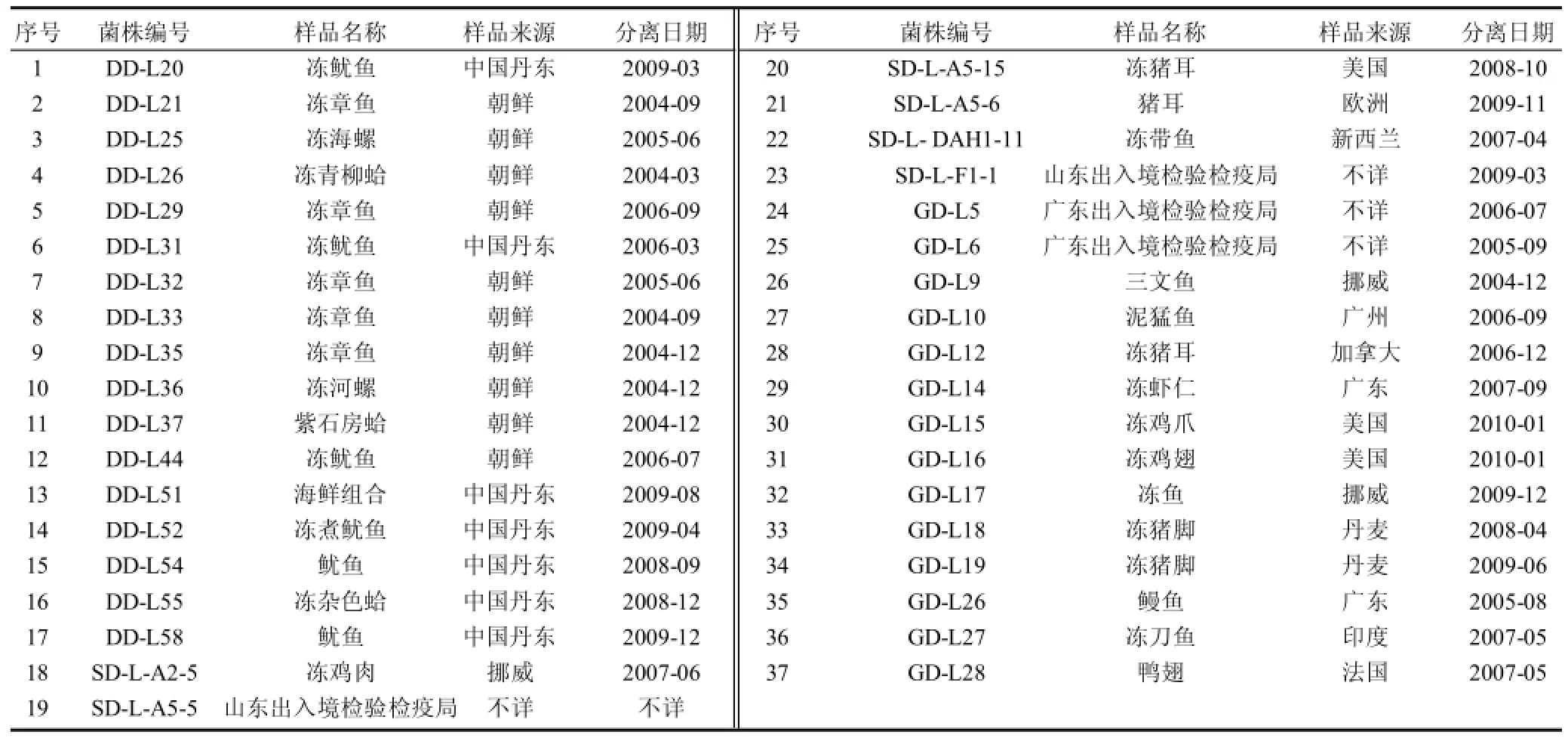
表1 單增李斯特氏菌分離株信息一覽表Table 1 Details of 37 Listeria monocytogenes isolates tested in this study
1 材料與方法
1.1 材料
37株單增李斯特氏菌分離株用于建立MALDI-TOF-MS鑒定數(shù)據(jù)庫,具體信息見表1。標(biāo)準(zhǔn)菌株單增李斯特氏菌1a血清型ATCC 19111、2a血清型ATCC 19112、4a血清型ATCC 19114、4b血清型ATCC 19115、4e血清型ATCC 19118、不溶血型ATCC 15313、ATCC BAA-751、ATCC 7644購自美國典型菌種保藏中心(ATCC);不溶血型NCTC 10890購自中國國家標(biāo)準(zhǔn)菌庫(NCTC),用于驗證所建立數(shù)據(jù)庫的鑒定可信度。
1.2 試劑與儀器
α-氰-4-羥基肉桂酸(α-cyano-4-hydroxy-cinnamic acid,CHCA)、乙腈(acetonitrile,ACN)、三氟乙酸(trifluoroacetic acid,TFA)、肽標(biāo)準(zhǔn)品、蛋白標(biāo)準(zhǔn)品 德國Bruker Daltonik GmbH公司;營養(yǎng)瓊脂(nutrient agar,NA)、檢測樣品配制基質(zhì)的溶劑為ACN、TFA、水(體積比50:2.5:47.5)、標(biāo)準(zhǔn)肽溶液配制基質(zhì)的溶劑為ACN、TFA、水(體積比30:0.1:69.9)、MALDI-TOF-MS 基質(zhì)溶液(用溶劑將基質(zhì)配成飽和溶液,基質(zhì)溶液現(xiàn)用現(xiàn)配)美國BD公司;API-20E生化鑒定試劑盒 法國梅里埃公司。
Microflex LRF20基質(zhì)輔助激光解吸電離飛行時間質(zhì)譜儀 德國Bruker Daltonik GmbH公司;PHOENIX-100全自動細(xì)菌生化分析儀 美國BD公司。
1.3 方法
1.3.1 分離菌株的鑒定
本研究對收集到的單增李斯特氏菌分離株,經(jīng)API-20E生化鑒定和全自動細(xì)菌生化分析儀鑒定,37株分離菌株均具有單增李斯特氏菌典型的生化特性,生長旺盛,可用于建立MALDI-TOF MS鑒定數(shù)據(jù)庫。對這37株單增李斯特氏菌進行唯一性編號,用于建立MALDITOF MS鑒定數(shù)據(jù)庫。
1.3.2MALDI-TOF-MS檢測
1.3.2.1 樣品處理與點樣
根據(jù)GB4789.30—2010《食品安全國家標(biāo)準(zhǔn) 食品微生物學(xué)檢驗 單核細(xì)胞增生李斯特氏菌檢驗》取單增李斯特氏菌新鮮培養(yǎng)物,在Eppendorf管中加入300μL純凈水,挑取適量(5~10mg)菌體,混勻,再加入900μL無水乙醇,混勻后以13000r/min離心2min,棄去上清液,加入50μL 70%甲酸,混勻,再加入50μL乙腈,混勻,同樣以13000r/min離心2min,吸取上清液,涂布于96 孔樣品板上,自然晾干1h 后,用基質(zhì)溶液(CHCA)覆蓋菌苔,用標(biāo)準(zhǔn)肽溶液覆蓋校準(zhǔn)孔,每孔1μL。晾干5min 后進樣。
MALDI-TOF-MS儀器參數(shù)為:線性操作模式,正離子模式;檢測分子質(zhì)量范圍:3000~13000D;激光點擊數(shù):每個圖譜50;激光頻率:30.0Hz;離子源加速電壓:20kV。采用氮激電源,質(zhì)荷比(m/z)數(shù)據(jù)采集范圍為800~17378D。
1.3.2.2 數(shù)據(jù)采集
將上樣的樣品板小心置于板孔中,加有樣品一面朝上。蓋上蓋子,抽真空。打開儀器控制軟件FlexControl,調(diào)好儀器參數(shù),校準(zhǔn)儀器。采集標(biāo)準(zhǔn)品及樣品的質(zhì)譜圖。對采集的數(shù)據(jù)進行保存,每次實驗前都要在采集數(shù)據(jù)的質(zhì)量范圍內(nèi)使用肽標(biāo)準(zhǔn)品進行校準(zhǔn),校正后進行質(zhì)譜數(shù)據(jù)采集,通過Biotyper軟件進行分析鑒定。
1.3.2.3 建立數(shù)據(jù)庫
將單增李斯特氏菌分離株分別接種于NA培養(yǎng)基上,37℃培養(yǎng)24h,得到新鮮菌株培養(yǎng)物。取每個菌株培養(yǎng)物再分別平行接種5個NA斜面,37℃培養(yǎng)24h,得到5個平行樣品。按1.3.2.1節(jié)的方法進行樣品處理與點樣,每個平行樣品重復(fù)點兩個樣品孔。每株菌共點10個樣品孔。按1.3.2.2節(jié)的方法校準(zhǔn)儀器后,點擊樣品孔采集數(shù)據(jù),每個樣品孔點擊10次,采集2個質(zhì)譜圖。每株菌可以采集20個質(zhì)譜圖。使用BioTyper軟件,分別調(diào)入所采集的每株單增李斯特氏菌的20個質(zhì)譜圖,將此20個質(zhì)譜圖匯總生成整合圖譜,對所得的圖譜進行分析統(tǒng)一化,該圖譜便作為單增李斯特氏菌質(zhì)譜圖數(shù)據(jù)庫的標(biāo)準(zhǔn)圖譜。
1.3.3 結(jié)果判斷
鑒定結(jié)果給出數(shù)據(jù)庫中與鑒定菌種最為匹配的10個菌株種屬,并給出相對應(yīng)的分?jǐn)?shù)。匹配分?jǐn)?shù)在2.300~3.000之間,標(biāo)記為(+++),表示菌種鑒定的可信度很高;在2.000~2.299之間,標(biāo)記為(++),表示保守的菌屬鑒定或可能的菌種鑒定;在1.700~1.999之間,標(biāo)記為(+),表示可能的菌屬鑒定;在0.000~1.699之間,標(biāo)記為(ˉ),表示不可信的鑒定。
2 結(jié)果與分析
2.1 單增李斯特氏菌MALDI-TOF-MS鑒定數(shù)據(jù)庫的建立
本研究對供試37株單增李斯特氏菌分離株全部進行了采集與圖譜匯總,建立標(biāo)準(zhǔn)圖譜,最終建成包含37株單增李斯特氏菌信息的獨立的鑒定數(shù)據(jù)庫,將其標(biāo)注編號為No.LMLNCJJ20100503。這37株單增李斯特氏菌分離株的主要離子峰基本一致,非常穩(wěn)定。以單增李斯特氏菌(SD-L-A5-6)為例,如圖1所示,經(jīng)MALDI-TOFMS分析,單增李斯特氏菌(SD-L-A5-6)主要離子峰為:m/z 3252.30、3702.88、4325.83、4878.28、6365.26、7405.70、9757.97。
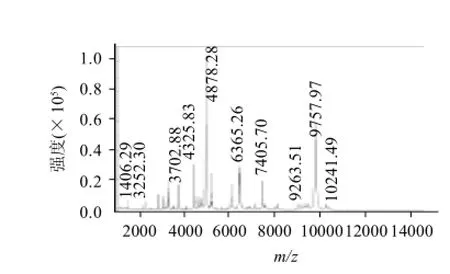
圖1 MALDI-TOF-MS分析單增李斯特氏菌(SD-L-A5-6)的主要離子峰圖譜Fig.1 MALDI-TOF-MS spectrum of Listeria monocytogenes showing major ion peaks
為驗證單增李斯特氏菌數(shù)據(jù)庫的鑒定效果,將9株單增李斯特氏菌標(biāo)準(zhǔn)菌株(ATCC 19111、ATCC 19112、ATCC 19114、ATCC 19115、ATCC 19118、ATCC 15313、ATCC BAA-751、ATCC 7644、NCTC 10890)分別進行MALDI-TOF-MS鑒定,分析在數(shù)據(jù)庫中的匹配結(jié)果。經(jīng)鑒定,9株標(biāo)準(zhǔn)菌株的匹配分?jǐn)?shù)全部大于2.300,均報告為單增李斯特氏菌,表明鑒定結(jié)果的可信度很高。
2.237 株單增李斯特氏菌MALDI-TOF-MS聚類分型鑒定結(jié)果
通過模式匹配計算新數(shù)據(jù)庫(No.LMLNCJJ20100503)中37株單增李斯特氏菌主要譜圖的相似性,采用相似分值構(gòu)建系統(tǒng)樹,對37株菌進行聚類分型分析。37株單增李斯特氏菌聚類分型樹狀圖如圖2所示。差異水平代表彼此間的親緣關(guān)系,其值在0到1.6之間。差異為0表示兩個MALDI-TOF-MS 質(zhì)譜圖完全相同,可歸屬為同一類;差異為1.6時表示兩株菌之間的親緣關(guān)系最遠(yuǎn)。所選擇的差異水平不同,則單增李斯特氏菌被分型的數(shù)目不一。在差異水平為0.7~0.8之間時,MALDI-TOF-MS分型結(jié)果將37株菌分成4個型;在差異水平為0.5時,將37株菌分成7個型;在差異水平為0.3~0.4之間時,將37株菌分成9個型。
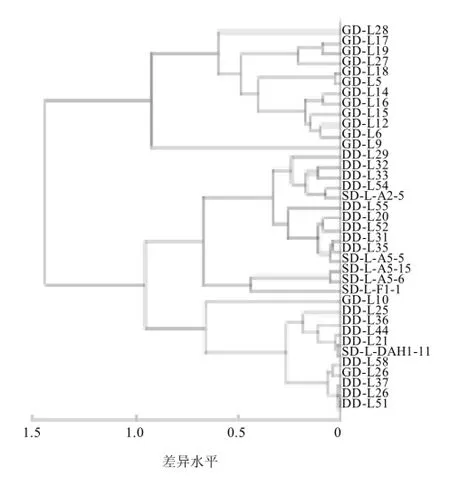
圖2 37株單增李斯特氏菌MALDI-TOF MS聚類分型樹狀圖Fig.2 Clustering dendrogram of 37 Listeria monocytogenes isolates
由圖2可見,從朝鮮進口的凍章魚、海螺、青柳蛤、河螺、紫石房蛤、魷魚中分離出來的單增李斯特氏菌(編號為DD-L21、DD-L25、DD-L26、DD-L36、DD-L37、DD-L44)與從中國丹東的海鮮組合、魷魚中分離出來的單增李斯特氏菌(編號為DD-L51、DD-L58)屬于同一型,親緣關(guān)系非常近,表明這些菌株可能為同一個污染源,都來源于中國丹東和朝鮮邊境的鴨綠江水域。而從新西蘭進口的凍帶魚(SD-DAH1-11)、中國廣東的鰻魚(GD-L26)中分離出來的單增李斯特氏菌,從分型結(jié)果來看,也與上述菌株具有比較近的親緣關(guān)系。
從中國丹東的魷魚、雜色蛤等5個樣品中分離出來的單增李斯特氏菌(編號為DD-L20、DD-L31、DD-L52、DD-L54、DD-L55),與從朝鮮進口的4個凍章魚樣品中分離出來的單增李斯特氏菌(編號為DD-L29、DD-L32、DD-L33、DD-L35),屬于同一型,親緣關(guān)系非常近,表明這些菌株可能為同一個污染源,都來源于中國丹東和朝鮮邊境的鴨綠江水域。而山東出入境檢驗檢疫局提供的從挪威進口的凍雞肉中分離的單增李斯特氏菌(SD-L-A2-5)和來源信息不詳?shù)木?SD-L-A5-5),從分型結(jié)果來看,也與上述菌株具有比較近的親緣關(guān)系。
廣東出入境檢驗檢疫局從加拿大、美國、挪威、丹麥、印度、法國進口的凍豬耳、凍雞爪、凍雞翅、凍魚、凍豬腳、帶魚、鴨翅中分離出來的單增李斯特氏菌(編號為GD-L12、GD-L15、GD-L16、GD-L17、GDL18、GD-L19、GD-L27、GD-L28)屬于同一型,親緣關(guān)系比較近,表明這些菌株可能為同一個污染源。而從廣東凍蝦仁中分離出來的單增李斯特氏菌(GD-L14)以及廣東出入境檢驗檢疫局提供的來源信息不詳?shù)膬芍昃?GD-L5、GD-L6),從分型結(jié)果來看也與上述菌株具有比較近的親緣關(guān)系。
此外,從挪威進口的三文魚和中國廣州泥猛魚中分離出來的單增李斯特氏菌(編號分別為GD-L9、GD-L10),卻與其他35株單增李斯特氏菌分離株都不屬于同一型,與它們的親緣關(guān)系最遠(yuǎn)。
3 討 論
利用質(zhì)譜進行細(xì)菌鑒定的研究始于30多年前[14],但直到1988年Tanaka和Hillenkamp兩個研究組分別提出基質(zhì)輔助激光解吸電離這種“軟電離”方式,才使得質(zhì)譜技術(shù)應(yīng)用于生物大分子的分析得到飛躍性進步[15]。其具有的高靈敏度、高準(zhǔn)確度及高分辨率等特點已使之成為生命科學(xué)研究領(lǐng)域的一種強有力的分析測試手段。曾有科學(xué)家預(yù)言,21 世紀(jì)是MALDI-TOF-MS 的世紀(jì)[16]。因為蛋白質(zhì)在微生物體內(nèi)的含量較高,所以常被用于細(xì)菌屬、種和株的鑒定。MALDI-TOF-MS被用于分析蛋白質(zhì)及酶消化產(chǎn)物的研究也較多。如Dieckmann等[17]利用蛋白質(zhì)圖譜分析,建立了沙門氏菌在種和亞種水品的分類鑒定方法。徐昕榮等[18]利用MALDI-TOF-MS對生物脫氮廢水處理系統(tǒng)中分離得到的兩株細(xì)菌進行蛋白質(zhì)質(zhì)量指紋圖譜分析,建立了生物脫氮細(xì)菌的快速鑒定方法。本研究通過采集37株單增李斯特氏菌分離株的數(shù)據(jù)并獲得特征指紋圖譜,創(chuàng)建了單增李斯特氏菌鑒定質(zhì)譜圖數(shù)據(jù)庫,可以實現(xiàn)對單增李斯特氏菌的準(zhǔn)確鑒定。
本研究在建立單增李斯特氏菌數(shù)據(jù)庫的同時,還通過模式匹配計算數(shù)據(jù)庫中單增李斯特氏菌主要譜圖的相似性,構(gòu)建系統(tǒng)樹進行分型,可以分析菌株之間的親緣關(guān)系,追溯可能的污染源。在以往的分型方法中,脈沖場電泳(pulsed field gel electrophoresis,PFGE)分型方法被譽為分子流行病學(xué)中的“金標(biāo)準(zhǔn)”[19],其分辨率高,可區(qū)分不同菌株間的細(xì)微差別,其不足之處是實驗技術(shù)較難掌握,且實驗耗時,相應(yīng)的試劑費用比較昂貴。MALDI-TOF-MS 和PFGE 分型分別代表了同一菌株基于蛋白質(zhì)和DNA 分型的兩個方面,二者結(jié)合使用可擴大基因型對亞種水平的分型能力,是PFGE 方法的補充。
參考文獻:
[1]AWAISHEH S S. Survey of Listeria monocytogenes and other Listeria sp. contamination in different common ready-to-eat food products in Jordan[J]. Pak J Biol Sci, 2009, 12(23): 1491-1497.
[2]GUILLET C, JOIN-LAMBERT O, le MONNIER A, et al. Human listeriosis caused by Listeria ivanovii[J]. Emerg Infect Dis, 2010, 16(1): 136-138.
[3]JACKSON K A, IWAMOTO M, SWERDLOW D. Pregnancy-associated listeriosis[J]. Epidemiol Infect, 2010, 17: 1-7.
[4]曹際娟. 食品微生物學(xué)與現(xiàn)代檢測技術(shù)[M]. 大連: 遼寧師范大學(xué)出版社, 2006: 150-187.
[5]董麗麗, 王春生, 安笑秋, 等. 單核細(xì)胞增生李斯特菌簡介[J]. 中國鄉(xiāng)村醫(yī)藥, 2000, 7(12): 24-25.
[6]RYSER T, ELMER H M. Listeriosis and food safety[J]. Marcel Dekkeinc, 1991, 6(2): 214-216.
[7]GILMOUR M W, GRAHAM M, van DOMSELAAR G, et al. Highthroughput genome sequencing of two Listeria monocytogenes clinical isolates during a large foodborne outbreak[J]. BMC Genomics, 2010, 11: 120.
[8]WALECKA E, MOLENDA J, BANIA J. The impact of environmental stress on Listeria monocytogenes virulence[J]. Pol J Vet Sci, 2009, 12 (4): 575-579.
[9]寇運同, 雷質(zhì)文, 林修光, 等. 用免疫磁珠捕集法快速檢測食品中的單核細(xì)胞增生李斯特菌[J]. 檢驗檢疫學(xué), 2001, 11(6): 12-13.
[10]KEROUANTON A, MARAULT M, PETIT L, et al. Evaluation of a multiplex PCR assay as an alternative method for Listeria monocytogenes serotyping[J]. J Microbiol Methods, 2010, 80(2): 134-137.
[11]O'G RADY J, RUTTLEDGE M, SEDANO-BALBAS S, et al. Rapid detection of Listeria monocytogenes in food using culture enrichment combined with real-time PCR[J]. Food Microbiology, 2009, 26(1): 4-7.
[12]劉海洪. MALDI-TOF-MS在細(xì)菌檢測和鑒定中的應(yīng)用[J]. 微生物免疫學(xué)進展, 2003, 31(2): 47-53.
[13]BRIGHT J J,CLAYDON M A, SOUFIAN M, et al. Rapid typing of bacteria using matrix-assisted laser desorption ionisation time-of-flight mass spectrometry and pattern recognition software[J]. J Microbiol Methods, 2002, 48: 127-138.
[14]ANHALT J P, FENSELAU C. Identification of bacteria using mass spectrometry[J]. Anal Chem, 1975, 47(2): 219-225.
[15]張永樂, 婁國強. 生物質(zhì)譜分析的研究進展及臨床應(yīng)用[J]. 生物醫(yī)學(xué)工程學(xué)進展, 2009, 30(2): 103-106.
[16]MILLER G M, BYRD S E, KUZNICKY R I. Nature insight: functional genomics[J]. Nature, 2000, 405: 819-846.
[17]DIECKMANN R, HELMUTH R, ERHARD M, et al. Rapid classification and identification of Salmonellae at the species and sub species levels by whole cell matrix-assisted laser desorption ionization-time of flight mass spectrometry[J]. Appl Environ Microbiol, 2008, 74(24): 7767-7778.
[18]徐昕榮, 朱斌, 馬瑜璐, 等. MALDI-TOF-MS鑒定生物脫氮細(xì)菌的方法研究[J]. 環(huán)境科學(xué)與技術(shù), 2010, 33(10): 148-161.
[19]王麗麗, 徐建國. 脈沖場凝膠電泳技術(shù)(PFGE)在分子分型中的應(yīng)用現(xiàn)狀[J]. 疾病監(jiān)測, 2006, 21(5): 276-279.
Identification and Subtyping of Listeria monocytogenes by MALDI-TOF-MS
WANG Yao1,2,CAO Ji-juan2,*,ZHAO Xin2,ZHENG Qiu-yue2,WANG Gang2,TIAN Zhuo3,SHI Yuan-yuan4,CAO Yuan-yin1
(1. Institute of Plant Immunology, College of Plant Protection, Shenyang Agricultural University, Shenyang 110161, China;2. Liaoning Entry-Exit Inspection and Quarantine Bureau, Dalian 116001, China;3. Zhuanghe Entry-Exit Inspection and Quarantine Bureau, Zhuanghe 121013, China;4. Yingkou Entry-Exit Inspection and Quarantine Bureau, Yingkou 115000, China)
In order to establish a matrix-assisted laser desorption ionisation time-of-flight mass spectrometry (MALDI-TOFMS) method for the rapid identification and subtyping of Listeria monocytogenes, 37 Listeria monocytogenes isolates were tested using MALDI-TOF-MS and characteristic protein fingerprints from their spectra were acquired and summarized into standard spectra to construct a database for the identification of Listeria monocytogenes. The method was highly reliable as demonstrated by the results obtained by the method for standard Listeria monocytogenes strains. Based on the identification database constructed, the Listeria monocytogenes isolates were classed into 9 subtypes at the protein level.
Listeria monocytogenes;matrix-assisted laser desorption ionisation time-of-flight mass spectrometry(MALDITOF-MS);identification;subtyping
Q503
A
1002-6630(2012)03-0194-05
2011-03-11
國家質(zhì)檢總局科研項目(2010IK175);國家質(zhì)檢公益性行業(yè)科研項目(200910270)
王耀(1982—),男,博士,研究方向為有害生物與環(huán)境安全。E-mail:wangyao_00@126.com
*通信作者:曹際娟(1968—),女,研究員,博士,研究方向為食品安全檢驗。E-mail:cjj0909@163.com

