乳酸菌與酵母菌共生機理綜述
閆 彬,賀銀鳳*
(內蒙古農業大學 食品科學與工程學院,內蒙古 呼和浩特 010018)
乳酸菌和酵母菌共同發酵制品例如酸馬奶酒、牛奶酒、葡萄酒、發酵乳飲料、酸奶油、奶酪[1]、黃油[2]、酥油[3]、面包等深受世界各地人們喜愛,其中酸馬奶酒在治療肺結核和胃腸疾病以及降血壓、降血脂等多種醫療功能方面得到了廣泛的應用。乳酸菌與酵母菌對成品的質地、風味、生物活性和醫療功能方面起著不可替代的作用,但是國內外學者對研究二者的共生機制卻鮮有報道。因此,研究發酵制品中乳酸菌與酵母菌的協同生長機理,對更廣泛的挖掘益生菌的功能具有重要的理論意義和實際應用價值,并對發酵制品行業的發展有著深遠的指導意義。
1 共生機理概述
普通酸奶發酵劑是由保加利亞乳桿菌與嗜熱鏈球菌共同制備的,二者之間的共生關系是保加利亞乳桿菌在乳中代謝生成的氨基酸可以刺激嗜熱鏈球菌的生長,而嗜熱鏈球菌在代謝過程中產生的甲酸又可以促進保加利亞乳桿菌的生長,兩種菌之間的共生作用已被廣泛認可。但是目前很少有人研究乳酸菌與酵母菌之間的共生關系。
開菲爾被譽為神奇的發酵乳[4],以特殊營養和保健功能卓越而著稱,它是由一種復雜菌系—開菲爾粒子直接發酵的酒精性發泡飲料,開菲爾粒與普通發酵乳制品的發酵劑有明顯區別,它由多種酵母菌和乳酸菌混合發酵而成,菌群組成十分復雜,發酵后的乳制品中含有大量黏性多糖、少量蛋白質、脂質等成分[5]。大多數酵母菌能夠利用乳中蛋白質、脂肪、乳糖和檸檬酸鹽[1,6],賦予開菲爾特殊的酒香味和二氧化碳氣體,使其具有一般飲料所沒有的爽口感和風味;乳酸菌在發酵過程中水解鮮乳中的乳糖、蛋白質及脂肪,產生易于吸收的單糖、游離氨基酸和揮發性脂肪酸,以及多種維生素(VB1、VB6、VB12、葉酸等)[7]。
由于乳酸菌是營養缺陷型菌株,有學者認為,乳酸菌與酵母菌之所以能夠在一個體系中共存,是因為酵母菌在發酵過程中為乳酸菌提供了許多營養因子例如氨基酸、維生素和丙酮酸鹽等其他物質[1],而乳酸菌的代謝產物又為酵母菌提供了能量來源[8]。在乳酪的生產中,酵母菌不僅分泌出蛋白酶和脂肪酶,分解基質產生營養物質促進乳酸菌的生長,而且還將乳酸菌代謝出的乳酸鹽作為能量物質進行代謝,產生出芳香物質[9-11],促進了乳酪的后熟。在自然發酵乳中,大部分酵母菌不能利用乳糖,但是可以利用半乳糖作為碳源進行發酵。Cheirsilp等[12]研究發現,將乳酸菌與酵母菌混合培養在牛乳中,乳酸菌將乳糖分解為半乳糖和葡萄糖為酵母菌提供碳源,同時酵母菌通過利用乳酸鹽促進了乳酸菌的生長,但是乳酸菌也有可能會因為酵母菌分解脂肪產生出的游離脂肪酸而抑制其生長[13-14]。
賦予酸奶風味的主要物質乙醛,是由乳酸菌利用蘇氨酸轉化而來的,但是乳酸菌的水解蛋白和脂肪能力較弱,更傾向于從牛乳蛋白中獲取必需氨基酸[15-16]。Rysstad等[17]假設,如果酵母菌分泌蘇氨酸,則會使乙醛在乳酸菌與酵母菌混合培養時的量有所升高。
Tamime等[18]發現乳酸菌與酵母菌之間存在代謝產物互補機制,也就是說一種菌產生的物質會被另一種菌代謝掉。一個著名的例子就是乳球菌屬新陳代謝產生的乙醛能夠被明串珠菌屬所利用。
釀酒酵母和乳酸菌都是葡萄酒釀造中的重要微生物,釀酒酵母主要進行酒精發酵,而乳酸菌進行蘋果酸-乳酸發酵[19]。葡萄酒在酵母泥上成熟期間,酵母的自溶能力明顯影響含氮化合物的濃度,包括氨基酸、縮氨酸、蛋白質以及其他大分子物質如葡聚糖、吡喃甘露糖等,其釋放濃度與酵母菌種類及釀酒工藝有關[20-21]。將乳酸菌在合成培養基上培養, 發現縮氨酸的分子質量<1000D時對乳酸菌的增殖有一定的促進作用;當蛋白質分子質量>5000D時對乳酸菌生長的影響沒有縮氨酸明顯。酵母泥中的大分子物質通過乙醇提取后,添加在合成培養基上,可以縮短乳酸菌的遲滯期,增加乳酸菌的數量,原因可能是:酵母大分子物質能誘導乳酸菌中氨基肽酶的合成,使氨基酸及小分子縮氨酸含量增加,從而促進乳酸菌的生長;另外酒精發酵或酵母自溶產生的吡喃甘露糖可以吸收中鏈脂肪酸,解除其對乳酸菌的毒害作用,還可能增加乳酸菌的α-葡萄糖苷酶、β-葡萄糖苷酶、N-乙酰基β-葡萄糖脫水酶及肽酶的活性,使培養基的營養豐富,間接地促進乳酸菌的生長[22]。
釀酒酵母的生理特性如蛋白酶活性、大分子物質生成及自溶量等,都可能對乳酸菌的生長及蘋果酸-乳酸發酵起促進作用,因此添加酵母提取物對乳酸菌的生長和代謝活性有一定的促進作用,但關于這方面研究的資料很少。
2 實驗研究概況
Alvarez-Martin等[23]將屬于8個不同種的12株酵母菌和屬于3個不同種的4株乳酸菌單獨和混合培養在UHT高溫滅菌乳中研究了乳酸菌與酵母菌的相互作用關系。結果顯示,4株乳酸菌與12株酵母菌混菌培養的36個組合中,有15個組合的乳酸菌活菌數顯著高于單菌培養的活菌數(P≤0.01),其中,大部分酵母菌都促進了乳酸菌L2BA1的生長;相反,乳酸菌LVI-19和LWg2在與酵母菌Candida famata、D. hansenii、Kluyveromyces lactis和Pichia membranifaciens培養時,兩種乳酸菌的生長受到了抑制。同時,在此36個混菌培養的組合中,3株酵母菌G. candidum 3AM4、G. candidum 3AM9和K. lactis 3AD10的活菌數顯著高于單菌培養的活菌數(P≤0.01);酵母菌C. pararugosa 3AD19的生長略有促進;酵母菌C. famata 2BD10、 G. candidum 3AM9、P.fermentans 3AD16和 P. membranifaciens 1AD8在與乳酸菌L2BC7混合培養時其活菌數與單菌培養比較略有抑制,而與乳酸菌L2BC7 和LVI-19混合培養時,其生長受到了顯著的抑制(P≤0.01)。
有研究顯示,乳酸菌與酵母菌混合發酵制品與純乳酸菌發酵的制品相比,某些特定代謝產物產生的量有所不同[24]。例如,乙醛、麥芽風味物質(2-甲基正丁醛、3-甲基正丁醛、2-甲基丙醇、2-甲基丁醇)在酵母菌的存在下,高濃度的麥芽風味物質的存在意味著有更多的乙醛轉化為乙醇。Gadaga等[25]利用從津巴布韋自然發酵乳中分離出的9株酵母菌和4株乳酸菌進行研究,結果發現乳酸菌與酵母菌混合培養在UHT乳中與單獨培養在UHT乳中相比,前者產生出的乙醛、麥芽芳香物質(3-甲基正丁醛等)、乙醇明顯比后者多,暗示了兩種菌之間的相互作用關系。乳酸菌可以通過丙酮酸鹽裂解酶將丙酮酸鹽轉化為甲酸鹽,或者將檸檬酸鹽分解為甲酸鹽、乙酸鹽、乳酸鹽、雙乙酰、乙酰甲基原醇、2,3-丁二醇等芳香物質[26];同時酵母菌負責產生大量的麥芽風味物質[23]。
Gobbetti等[27]在MRS培養基中通過添加4種不同碳源(麥芽糖、蔗糖、葡萄糖、果糖)來研究兩株酵母菌與兩株乳酸菌之間的相互作用。結果顯示,在添加了蔗糖的MRS培養基中,雙菌培養的乳酸菌活菌數明顯高于單菌培養的活菌數,作者通過蔗糖分解的動力學曲線分析其原因是酵母菌可以將蔗糖分解為葡萄糖和果糖為乳酸菌提供穩定的碳源。
Graves等[28]在玉米糊培養基中按梯度添加乳酸和乙酸,設置3個溫度(30、34、37℃),結果顯示溫度越高,對酵母菌產乙醇的抑制作用越強。可能是由于酵母菌體在高溫環境中由于細胞膜中磷脂流動性降低而不能維持最佳的細胞活性而導致發酵效率降低[29-31]。Abbott等[32]同樣在玉米糊培養基中培養酵母菌,由于玉米糊這類培養基的基質物質較多,所以其緩沖能力較強,會促進酵母菌體的生長,尤其是當酵母菌暴露在一個特定的酸性和高溫環境下,存在于培養基中的各種復雜成分例如酵母菌的代謝產物和蛋白胨等會降低外界高滲透壓和高溫對酵母菌造成的不利影響[33],而且一種適應反應機制被激活,促使酵母菌增強對外界壓力的防御能力,這種現象被稱為交叉保護[34]。因此對于暴露在一定量的乳酸與乙酸環境中的酵母菌來講,某種特殊的物質被引導合成用來保護酵母菌體。而且,若添加乙酸的量適宜,則會促進酵母菌發酵產醇,Graves等[35]玉米糊中添加了少量的乙酸(0.2g/100mL),結果顯示酵母菌產乙醇的效率提高了,產量也有所增加。
3 信號分子水平研究概況
群體感應(Quorum sensing,QS)也稱為自誘導,最初是指細菌調節自身菌體密度的一種環境感應系統[36]。通過擴散性信號小分子(又稱為自誘導物) 與轉錄活化蛋白的相互作用而打開與細胞群體密度有關的基因表達。這些信號分子從細菌細胞擴散到環境中,一旦達到一個臨界濃度(或者說達到某一特定的群體密度),這些信號分子就可誘導調節一系列目標基因的轉錄[37]。群體感應可以使細菌在其生長的特定環境中調節其生命活動,每種群體感應系統都能使細菌對其他細菌的存在做出反應,從而調整自身的生命活動。群體感應調節能使一個群體中每個細胞協同作用,成為一個多細胞整體[38]。
已經有多種微生物相關的化學信號分子被發現,這些物質大體可分為兩類:一類是氨基酸和短肽類,主要作用于革蘭氏陽性細菌[39-41];另一類是脂肪類的衍生物,主要作用于革蘭氏陰性細菌。 信號分子(自誘導物)的產生是一個和環境因素直接相關的過程,如溫度和碳源種類對信號分子產生的水平有動態的影響。群體感應和營養饑餓顯然是相關的,饑餓和細胞密度決定了細菌是否進入穩定生長期[42]。植物根際細菌Ralstonia solanacearum 產生AHL 需要一種σs因子,該因子在饑餓條件下活性最高[43]。不少研究者都采用最低營養水平的基質來促使QS信號分子的產生。QS分子的產生大多出現在對數生長后期或穩定生長初期[44-45]。
Guerzoni等[46]通過改變培養環境來研究乳酸菌與酵母菌之間的互作關系。他們在小麥粉水解液培養基中分別用乳酸調成酸度為pH 3.6的高酸環境,添加5mmol/L H2O2作為高氧環境,以及質量濃度40g/100mL的蔗糖調節成高滲透壓環境,以純小麥水解液為對照,來探討乳酸菌與酵母菌單獨培養和混合培養時產生乙醇及芳香物質的差異。結果顯示,乳酸菌在高氧環境產出了較多的γ-癸內酯,2(5H)-呋喃酮和乙醛;在高酸環境下,不論是在乳酸菌與酵母菌單獨培養基中還是混合培養的培養基中都發現了乙酸、異戊酸的積累以及較多的乙醇,而且還發現酵母菌在高酸、高氧這種壓力環境下會分泌出長鏈不飽和脂肪酸酯,這種現象可以看作是酵母菌體的自我保護機制,而有的學者則認為酵母菌分泌的這種不飽和脂肪酸酯可以看作是其在特定環境下釋放的信號分子[47]。那么如此說來,在研究乳酸菌與酵母菌之間的作用機制時,就可以以這種信號分子為標志,通過測量其代謝產物(乙醇等)與對照樣品相比較來觀察二者之間的促生作用。同樣,乳酸菌在高氧環境下分泌的2(5H)-呋喃酮,酵母菌和乳酸菌在高酸環境下分泌的異戊酸,都可以將其看成為信號分子。Losel等[48]報道了在研究雙孢蘑菇休眠時,其釋放的異戊酸就起著信號分子的作用。盡管這類種內和種間信號分子釋放的分析尚不全面,但通過研究在特定壓力環境下菌體分泌的代謝產物,有助于更好的了解乳酸菌與酵母菌之間的共生機制。
4 基因水平研究概況
Erasmus等[49]曾認為微生物菌體若在發酵過程中處于某種壓力環境下,那么其在轉錄時成百個隱性基因會被誘導為顯性基因表達出來。隨著分子生物學技術的革新,生物芯片技術為我們提供了強有力的工具,Maligoy等[50]就在轉錄水平上研究了乳酸菌與酵母菌混合培養時乳酸菌菌體中表達基因的變化。盡管在乳酸菌單獨培養和與酵母菌混合培養的生長動力學曲線上未發現明顯變化,但是在混合培養中乳酸菌的信使RNA水平上的158個重要基因發生了明顯的調整,和乳酸菌單獨培養相比,乳酸菌與酵母菌混合培養時信使RNA水平上有54%的基因消失,同時增加了46%的基因,這些重要的基因負責多種新陳代謝途徑,例如氨基酸的生物合成,能量的新陳代謝,嘌呤嘧啶的新陳代謝、調節、翻譯功能,運輸攜帶蛋白功能等。像調節能量新陳代謝的基因和調節嘌呤、嘧啶、核苷酸新陳代謝的基因變化較大,尤其是負責代謝嘧啶基因的再定位,其生物合成途徑的8個隱性基因控制6個酶促反應,其中6個基因(carA、carB、pydB、pyrB、pyrC、pyrE)負責嘧啶的生物合成,兩個基因(pyrZ、rmlB)負責嘧啶的新陳代謝,且這8個基因比例較低。而顯性基因pyrG、purL和deoD的比例要高于單獨培養時的基因比例,其中pyrG負責三磷酸尿苷轉化為三磷酸胞苷的酶催化反應。由此可以看出,混合培養時由于負責代謝嘧啶的隱性基因比例很低,所以導致三磷酸胞苷合成減少,因此促使pyrG基因的表達比例提高。究其原因,根據DNA芯片技術的結果可以推斷出可能是由于在混合培養時酵母菌產生的乙醇導致carB、pydB、pyrE和pyrR基因表達水平的降低,暗示出在混合培養時乙醇的積累是導致乳酸菌mRNA水平上負責各種新陳代謝途徑(像糖代謝、氨基酸合成等)的功能基因變化的主要原因。因此,酵母菌產乙醇的量可以作為研究乳酸菌與酵母菌種間關系的主要指標(表1)。
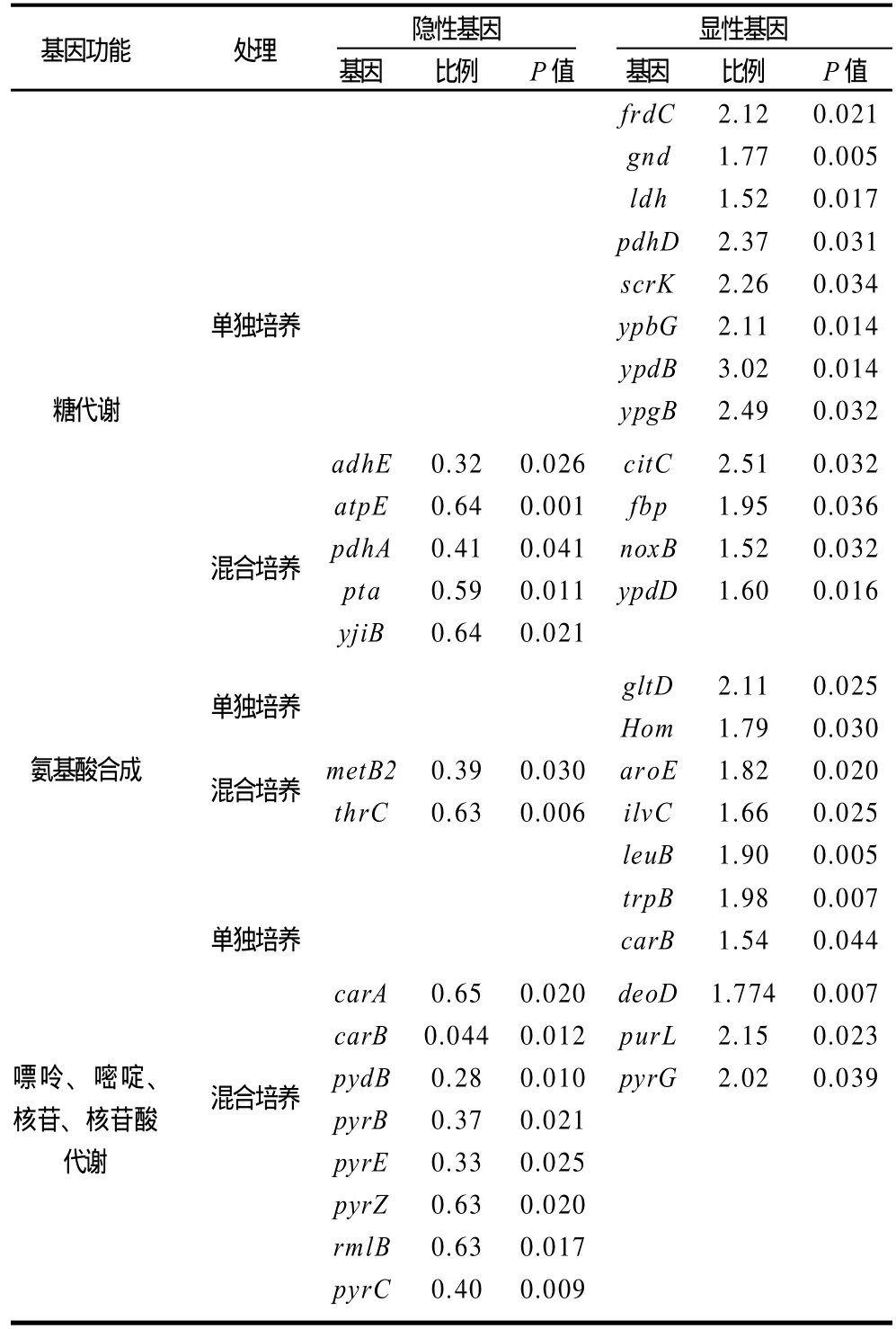
表1 乳酸菌單獨培養與乳酸菌和酵母菌混合培養時mRNA水平上的比較Table 1 Comparison between single culture and co-culture of lactic acid bacteria and yeasts at mRNA level
5 展 望
乳酸菌和酵母菌共同發酵制品是國內學者研究的一個熱點。在乳制品、飲料、面食品等發酵制品中得到了很好的應用,顯現出神奇的作用。在產品的風味、口感、營養價值及生理功能等方面優于任何單一種類菌的發酵產品[51-57]。
通過以內蒙古傳統乳制品中特定微生物為基點,利用PCR-DGGE、FISH技術研究民族乳制品中微生物群落結構和多態性,探討乳酸菌和酵母菌共生過程中生物信號系統,摸清乳酸菌與酵母菌之間互生關系及其調控機理,挖掘千百年來自然形成的微生物“團隊”的耐粗放、抗雜菌及醫療保健功效的作用,具有非常深遠的意義;提出個性互生機理與共性互生機理的應用前景,將為內蒙古乳制品中菌種資源庫的開發應用提供理論依據。
盡管國內外關于乳酸菌與酵母菌共生機理的相關報道還很少,但是相信隨著分子生物學和分子遺傳學的飛速發展,16S rRNA基因序列分析,限制性片段長度多態性(RFLP)、熒光原位雜交技術(FISH)等一些技術的應用將會在研究環境中微生物生態系統組成結構、功能的分子機理以及微生物之間相互關系等方面顯示出巨大的潛力,也將對我們深入研究二者的共生機理提供出新的思路和方法,從而為今后開發和研制出更多發酵制品提供重要的科學依據,并給發酵制品工業化的生產與發展打下堅實的基礎。
[1] ROOSTITA R, FLEET G H. The occurrence and growth of yeast in Camembert and blue-veined cheese[J]. International Journal of Food Microbiology, 1996, 28(3): 293-404.
[2] BEYENE F. Present situation and future aspects of milk production,milk handling and processing of dairy products in southern Ethiopia[D].Norway: Agricultural University of Norway, 1994: 218-227.
[3] SSERUNJOGI M L, ABRAHAMSEN R K, NARVHUS J. A review paper: current knowledge of ghee and related products[J]. International Journal of Dairy, 1998, 8(8): 677-688.
[4] 張曉琳, 王煒. 類開菲爾粒的發酵特性極強產酸乳酸菌的分離鑒定[J]. 新疆農業科學, 2009, 46(4): 856-860.
[5] MOTAGHI M, MARAHERI M, MOAZAMI N, et al. Kefir production in Iran[J]. World Journal of Microbiology and Biotechnology, 1997, 13(1): 579-581.
[6] FLEET G H. Yeasts in dairy products: a review[J]. Journal of Applied Bacteriology, 1990, 68: 199-211.
[7] 王和平, 田瑞華. 類開菲爾粒發酵特性的研究[J]. 乳業科學與技術,2004, 4(1): 153-155.
[8] LORETAN T. The diversity and technological properties of yeasts from indigenous traditional South African fermented milks[D]. South Africa:University Orange Free State, Bloemfontein, 1999.
[9] WELTHAGEN J J, VILJOEN B C. Yeast profile in Gouda cheese during processing and ripening[J]. International Journal of Food Microbiology,1998, 41(3): 185-194.
[10] VILJOEN B C, GREYLING T. Yeasts associated with Cheddar and Gouda making[J]. International Journal of Food Microbiology, 1995, 28(1): 79-88.
[11] LAUBSCHER P J. The occurrence, growth and survival of yeasts in matured Cheddar[D]. South Africa: University Orange Free State,Bloemfontein, 1999.
[12] CHEIRSILP B, SHIMIZU H, SHIOYA S. Enhanced kefiran production by mixed culture of Lactobacillus kefiranofaciens and Saccharomyces cerevisiae[J]. Journal of Biotechnology, 2003, 100(1): 43-53.
[13] BROOME M C, THOMAS M P, HILLIER A J, et al. The effect of linoleic acid on the growth and metabolisms of Streptococcus lactis[J].Australian Journal of Dairy Technology, 1979, 34(4): 163-168.
[14] NIELSEN M S, FRISVAD J C, NIELSEN P V. Protection by fungal starters against growth and secondary metabolite production of fungal spoilers of cheese[J]. International Journal of Food Microbiology, 1998,42(1/2): 91-99.
[15] NARVHUS J A,STERAASA K, MUTUKUMIRA T, et al. Production of fermented milk using a malty compound- producing strain of Lactococcus lactis subsp. lactis biovar. diacetylactis, isolated from Zimbabwean fermented milk[J]. International Journal of Food Microbiology, 1998, 14(1): 73-80.
[16] AYAD E H E, VERHEUL A, de JONG C, et al. Flavour forming abilities and amino acid requirements of Lactococcus lactis strains isolated from artisanal and non-dairy origin[J]. International Journal of Dairy, 1999, 9(10): 725-735.
[17] RYSSTAD G, KNUTSEN W J, ABRAHAMSEN R K. Effect of threo-nine and glycine on acetaldehyde formation in goats,milk yogurt[J].Journal of Dairy Research, 1990, 57(3): 401-411.
[18] TAMIME A Y, MARSHALL V M E. Microbiology and technology of fermented milks[M]// LAW B A. Microbiology and biochemistry of cheese and fermented milk. London, UK, Blackie Academic & Professional,1997.
[19] 屈慧鴿, 程顯好. 葡萄酒生境對乳酸菌代謝的影響[J]. 微生物學通報, 2008, 35(11): 1797-1801.
[20] MARTINEZ-RODRIGUEZ A J, CARRASCOSA A V, POLO M C.Release of nitrogen compounds to the extracellular medium by three strains of Saccharomyces cerevisiae during induced autolysis in a model wine system[J]. International Journal of Food Microbiology, 2001, 68(1/2): 155-160.~
[21] CAPUCHO I, SAN- ROMAO M V. Effect of ethanol and fatty acids on malolactic activity of Leuconostoc oenos[J]. Applied Microbiology and Biotechnology, 1994, 42(2/3): 391-395.
[22] GUILLOUX- BENATIER M, CHASSAGNE D. Comparison of components released by fermented or active dried yeasts after aging on lees in a model wine[J]. Journal of Agricultural and Food Chemistry, 2003, 51(3): 746-751.
[23] ALVAREZ-MARTIN P, FLOREZ A B, HERNANDEZ-BARRANCO A, et al. Interaction between dairy yeasts and lactic acid bacteria strains during milk fermentation[J]. Food Control, 2008, 19(1): 62-70.
[24] GADAGA T H, VILJOEN B C, NARVHUS J A. Volatile organic compounds in naturally fermented milk and milk fermented using yeasts,lactic acid bacteria and their combinations as starter cultures[J]. Food Technology and Biotechnology, 2007, 45(2): 195-200.
[25] GADAGA T H, MUTUKUMIRA A N, NARVHUS J A. The growth and interaction of yeasts and lactic acid bacteria isolated from Zimbabwean naturally fermented milk in UHT milk[J]. International Journal of Food Microbiology, 2001, 68(1/2): 21-32.
[26] HUGENHOLTZ J. Citrate metabolism in lactic acid bacteria[J]. FEMS Microbiology Review, 1993, 12(1/3): 165-178.
[27] GOBBETTI M, CORSETTI A, ROSSI J. The sourdough microflora.Interactions between lactic acid bacteria and yeasts: metabolism of carbohydrates[J]. Applied Microbiology and Biotechnology, 1994, 41(4): 456-460.
[28] GRAVES T, NARENDRANATH N V, DAWSON K, et al. Interaction effects of lactic acid and acetic acid at different temperatures on ethanol production by Saccharomyces cerevisiae in corn mash[J]. Applied Microbiology and Biotechnology, 2007, 73(5): 1190-1196.
[29] CARMELO V, SANTOS R, VIEGAS C A, et al. Modification of Saccharomyces cerevisiae thermotolerance following rapid exposure to acid stress[J]. International Journal of Food Microbiology, 1998, 42(3):225-230.
[30] ROSE A H. Composition of the envelope layers of Saccharomyces cerevisiae in relation to flocculation and ethanol tolerance[J]. Journal of Applied Bacteriology, 1993, 74: 110S-118S.
[31] LLOYD D, MORELL S, CARLSEN H N, et al. Effects of growth with ethanol on fermentation and membrane fluidity of Saccharomyces cerevisiae[J]. Yeast, 1993, 9(8): 825-833.
[32] ABBOTT D A, INGLEDEW W M. Buffering capacity of whole corn mash alters concentrations of organic acids required to inhibit growth of Saccharomyces cerevisiae and ethanol production[J]. Biotechnology Letters, 2004, 26(16): 1313-1316.
[33] STEWART G G, DAMORE T, PANCH,AL C J, et al. Factors that influence the ethanol tolerance of brewer s yeast strains during high gravity wort fermentations[J]. MBAA Technical Quarterly, 1988, 25(2):47-53.
[34] CARMELO V, SANTOS R, VIEGAS C A,et al. Modification of Saccharomyces cerevisiae thermotolerance following rapid exposure to acid stress[J]. International Journal of Food Microbiology, 1998, 42(3): 225-230.
[35] GRAVES T, NARENDRANATH N, DAWSON K, et al. Effect of pH and lactic or acetic acid on ethanol productivity by Saccharomyces cerevisiae in corn mash[J]. Journal of Microbiology and Biotechnology,2006, 36(6): 91-97.
[36] FUQUA W C, WINANS S C, GREENBERG E P. Quorum sensing in bacteria: the LuxR-LuxI family of cell density-responsive transcriptional regulators[J]. Journal of Bacteriology, 1994, 176(2): 269-275.
[37] 陳峰. 微生物的群體感應信號分子(綜述) [J]. 上海農業學報, 2005,21(1): 98-102.
[38] 鐘增濤, 鄭會明, 高軼靜. 細菌中的群體感應[J]. 生命的化學, 2003,23(6): 421-424.
[39] KLEEREBEZEM M , QUADRI L E N , KUIPERS O P, et al. Quorum sensing by peptide pheromones and two component signal transduction systems in Gram positive bacteria[J]. Molecular Microbiology, 1997,24(5): 895-904.
[40] LAZAZZERA B, GROSSMAN A D. The ins and outs of peptide signaling[J]. Trends Microbiology, 1998, 6(7): 288-294.
[41] SHAPIRO J A. Thinking about bacterial populations as multicellular organisms[J]. Annual Reviews of Microbiology, 1998, 52(1): 81-104.
[42] LAZAZZERA B. Quorum sensing and starvation: signals for entry into stationary phase[J]. Current Opinion in Microbiology, 2000, 3(2): 177-182.
[43] FLAVIER A B , GANOVA R L M, SCHELL M A, et al. Hierarchical autoinduction in Ralstonia solanacearum: control of acyl homoserine lactone production by a novel autoregulatory system responsive to hydroxypaImitic acid methyl ester[J]. Journal of Bacteriology, 1997,179(22): 7089-7097.
[44] SHAW P D, PING G, DALY S, et al. Detecting and characterizing acyl homoserine lactone signal molecules by thin layer chromatography[J].Proceedings of the National Academy of Science, 1997, 94(12): 6036-6041.
[45] BRELLES M G, BEDMAR E J. Bradyrhizobium japonicum produces quorum sensing signal molecules[C]//The 4th, European Nitrogen Fixation Conference. Seville Spain, 2000.
[46] GUERZONI M E, VERNOCCHI P, NDAGIJIMANA M , et al.Generation of aroma compounds in sourdough: effects of stress exposure and lactobacilli-yeasts interactions[J]. Food Microbiology, 2007, 24(2):139-148.
[47] THOMA I, LOEFFLER C, SINHA A K, et al. Cyclopentenone isoprostanes induced by reactive oxygen species trigger defense gene activation and phytoalexin accumulation in plants[J]. Plant Journal,2003, 34(3): 363-375.
[48] LOSEL D M. Microbial lipids[M]. London: Academic Press, 1989:399.
[49] ERASMUS D J, van der MERWE G K, van VUUREN H J .Genomewide expression analyses: metabolic adaptation of Saccharomyces cerevisiae to high sugar stress[J]. FEMS Yeast Research, 2003, 3(4):375-399.
[50] MALIGOY M, MERCADE M, COCAIGN-BOUSQUET M, et al.Transcriptome analysis of Lactococcus lactis in coculture with Saccharomyces cerevisiae[J]. Applied and Environmental Microbiology, 2008,74(2): 485-494.
[51] 王洪志. 奶啤生產中乳酸菌對酵母菌發酵作用的研究[J]. 中國釀造,2009(6): 134-136.
[52] 匡逢春, 劉惠知. 飼用微生態制劑生產用酵母菌與乳酸菌的特性研究[J]. 湖南農業科學, 2009(3): 140-141; 144.
[53] 蘇東民, 胡麗花. 饅頭發酵過程中酵母菌和乳酸菌的代謝作用[J]. 食品科學, 2010, 31(13): 200-204.
[54] 李紀岳, 張愛民. 純種發酵早秈米生產新式扎粉的研究[J]. 安徽農業科學, 2009, 37(3): 1325-1326.
[55] 劉貞, 劉小翠. 發酵米漿中高發酵性能酵母菌和乳酸菌的篩選和鑒定[J]. 食品科學, 2010, 31(7): 232-235.
[56] 焦宇知. 無糖花生香蕉開菲爾飲料生產工藝研究[J]. 食品科學, 2009,30(2): 286-288.
[57] 胡麗紅, 傅力. 紅棗醋生產中酒精發酵階段最佳工藝條件的研究[J].食品科學, 2008, 29(10): 418-422.

