正交設(shè)計(jì)優(yōu)化山野豌豆SRAP-PCR反應(yīng)體系與引物篩選
劉穎,王顯國,張巨明*,劉芳
(1.華南農(nóng)業(yè)大學(xué)農(nóng)學(xué)院,廣東 廣州510642;2.中國農(nóng)業(yè)大學(xué)動(dòng)物科技學(xué)院,北京100193;3.全國畜牧總站,北京100193)
山野豌豆(Vicia amoena)又叫落豆秧、高蔓草藤、宿根苕子,為豆科巢菜屬多年生草本植物[1]。它表現(xiàn)出耐寒、抗旱、葉量大、粗蛋白及動(dòng)物必需氨基酸含量高、適應(yīng)性廣、再生力強(qiáng)、容易繁殖、產(chǎn)量高、品質(zhì)好等優(yōu)點(diǎn)[2-5]。另外,山野豌豆不僅是一種優(yōu)良牧草,而且還是很好的防風(fēng)固沙水保植物[6]、改土肥田綠肥作物[7]和草坪及藥用植物[8],具有推廣和應(yīng)用價(jià)值。目前SRAP(sequence related amplified polymorphism)標(biāo)記已在農(nóng)作物和園藝作物上有了較多成功的應(yīng)用,但SRAP在豆科牧草種質(zhì)資源上的應(yīng)用還非常少,僅在苜蓿(Medicago)[9-15]、大豆(Glycine)[16-18]、三葉草(Trifolium)[19,20]、山羊豆(Galege)[21]、小扁豆(Lens)[22,23]等屬有相關(guān)報(bào)道。本研究主要采用正交設(shè)計(jì)法對SRAP-PCR反應(yīng)條件中主要因子:Mg2+、dNTP、引物、Taq酶進(jìn)行優(yōu)化分析并確定了模板DNA濃度,旨在篩選出一個(gè)適用于野豌豆屬的穩(wěn)定性及重復(fù)性好,多態(tài)性高的最佳反應(yīng)條件。此外,本研究還對部分SRAP引物組合進(jìn)行了多態(tài)性篩選,希望獲得一些多態(tài)性豐富的SRAP引物組合,為開展SRAP標(biāo)記技術(shù)在山野豌豆種質(zhì)鑒定、遺傳多樣性等方面的分子生物學(xué)研究提供有利依據(jù)。
1 材料與方法
1.1 供試材料
試驗(yàn)所用5份山野豌豆材料采自全國3個(gè)不同的地域,優(yōu)化SRAP-PCR體系的DNA模板來自內(nèi)蒙古呼倫貝爾的9號、山西五臺(tái)山的6號及北京百花山的14號DNA;篩選引物所用DNA模板來自山西沁源的11號、北京靈山的5號及內(nèi)蒙古呼倫貝爾的9號DNA。
1.2 試驗(yàn)設(shè)計(jì)與方法
1.2.1 PCR反應(yīng)體系設(shè)計(jì)與引物組合 采用L9(34)正交試驗(yàn)設(shè)計(jì),對 Mg2+、dNTP、引物、Taq DNA 聚合酶進(jìn)行4因素3水平篩選(表1)。體系優(yōu)化分別采用3份不同DNA進(jìn)行,每份DNA做2次重復(fù),取其平均值作為統(tǒng)計(jì)數(shù)據(jù)。體系總體積為25μL,除表中變化的因素外,每管中還加入2μL 10×PCR buffer和50ng的模板DNA,所用引物組合為Me2+Em4。
1.2.2 SRAP-PCR擴(kuò)增程序及擴(kuò)增產(chǎn)物的檢測SRAP-PCR擴(kuò)增程序?yàn)椋?4℃預(yù)變性4min;94℃變性1min,37℃退火45s,72℃延伸1min,5個(gè)循環(huán);94℃變性1min,50℃退火45s,72℃延伸1min,35個(gè)循環(huán);循環(huán)結(jié)束后,72℃延伸7min,4℃保存。擴(kuò)增結(jié)束后,在擴(kuò)增產(chǎn)物中加入5μL 6×loading Buffer緩沖液混勻,取8μL上樣于2%的瓊脂糖凝膠中進(jìn)行分離。
1.2.3 模板DNA濃度優(yōu)化 應(yīng)用上述體系最佳組合,引物組合為 Me2+Em4,DNA模板為5號DNA。將25μL擴(kuò)增體系中模板DNA用量分別設(shè)置6個(gè)梯度處理,即10,20,30,40,50和60ng,對反應(yīng)體系中的模板DNA濃度進(jìn)行優(yōu)化。
1.2.4 多態(tài)性SRAP引物組合的篩選 根據(jù)上述試驗(yàn)結(jié)果確定的SRAP-PCR最佳反應(yīng)體系,選取Li和Quiros[24]及Riaz等[25]發(fā)表的引物序列,包括7條正向引物和14條反向引物兩兩組合成98個(gè)SRAP引物組合(表2),對3份山野豌豆種源材料進(jìn)行多態(tài)性SRAP引物組合的篩選。

表1 SRAP-PCR[L9(34)]正交試驗(yàn)設(shè)計(jì)Table 1 Orthogonal design for SRAP-PCR [L9(34)]
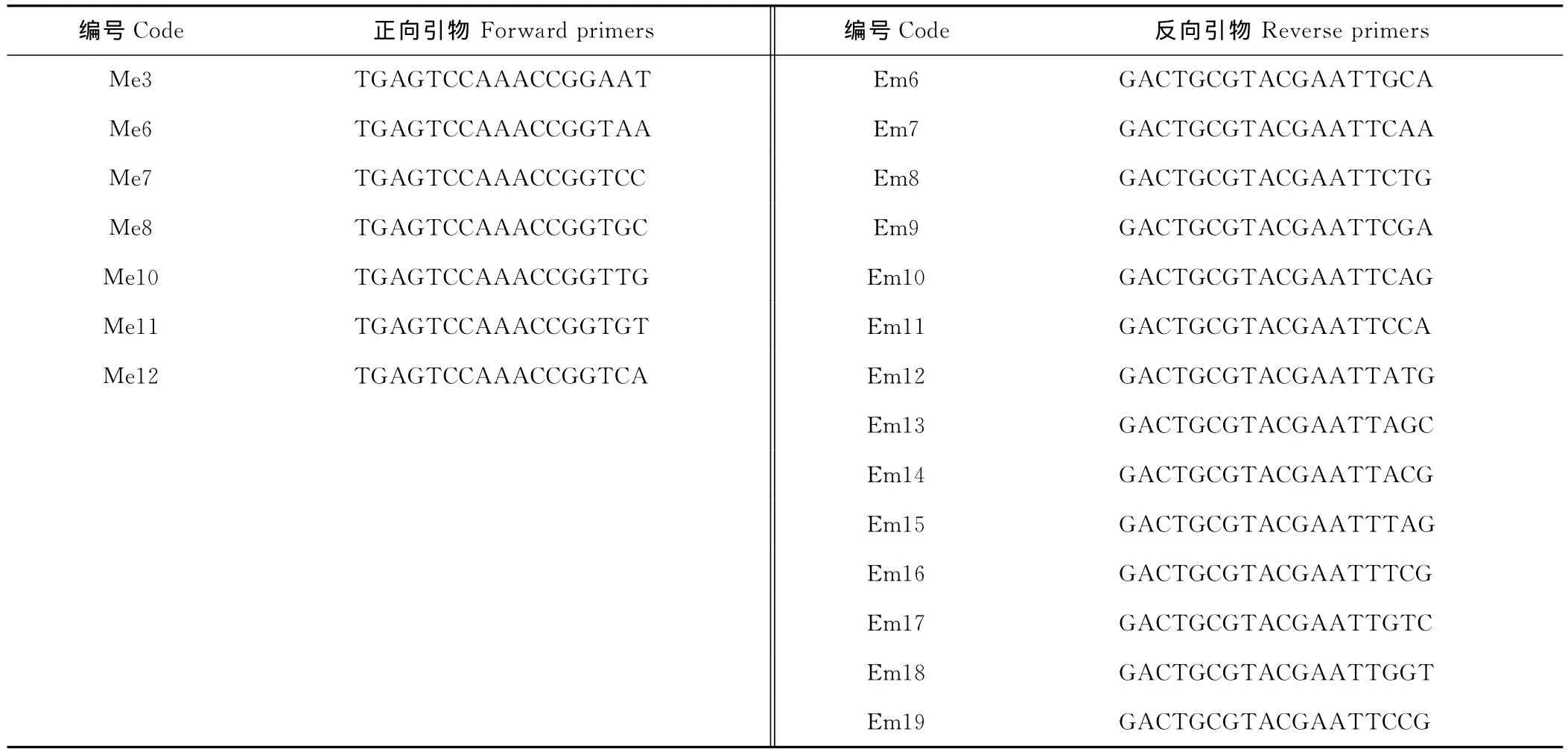
表2 SRAP引物序列Table 2 Primer sequences used for SRAP analysis
2 結(jié)果與分析
2.1 SRAP-PCR正交試驗(yàn)設(shè)計(jì)的擴(kuò)增結(jié)果分析
山野豌豆SRAP-PCR體系優(yōu)化的正交試驗(yàn)結(jié)果如圖1所示。參照何正文等[26]的方法,對擴(kuò)增結(jié)果進(jìn)行直觀分析,依據(jù)譜帶的亮度、清晰度以及條帶數(shù),將9個(gè)處理結(jié)果劃分為9個(gè)評分等級以便統(tǒng)計(jì)分析,內(nèi)蒙古呼倫貝爾的DNA擴(kuò)增結(jié)果得分為1,3,2,5,6,7,9,4和8。山西五臺(tái)山與北京百花山的DNA擴(kuò)增結(jié)果得分分別為1,6,7,8,5,2,9,3和4及6,7,4,3,5,1,9,2和8。3份不同種源的9個(gè)處理結(jié)果的總平均得分為2.67,5.33,4.33,5.33,5.33,3.33,9.00,2.67,6.67。得分最高為組合7帶型清晰,亮度高,且雜帶少,擴(kuò)增效果最好,選為山野豌豆基因組SRAP-PCR的最佳擴(kuò)增反應(yīng)體系。
參照張麗等[27]的方法,對各組分濃度組合的正交試驗(yàn)結(jié)果進(jìn)行了統(tǒng)計(jì)分析(表3),其中T值代表某水平下某因子參與反應(yīng)所產(chǎn)生的擴(kuò)增條帶的總和;K代表某因子在某水平參與反應(yīng)所產(chǎn)生的擴(kuò)增條帶的平均值;R為某因子的極差,即某因子在不同水平下最大平均值與最小平均值之差。R值的大小反映了該因子對試驗(yàn)結(jié)果影響的大小,R值越大,影響越顯著。從R值可看出,在選定的3個(gè)水平范圍內(nèi),4個(gè)因素對結(jié)果的影響由大到小依次為:引物> Mg2+>Taq DNA聚合酶>dNTP。K值反映了影響因素各水平對反應(yīng)體系的影響情況,K值越大,反應(yīng)水平越好。從K值結(jié)果來看,引物以水平3最好,Mg2+以水平2最好,Taq DNA聚合酶水平3最好,dNTP以水平1最好。綜上,各因素最佳的水平具體為引物2.0μmol/L、Mg2+2.0mmol/L、Taq DNA 聚合酶1.5U、dNTP 0.2mmol/L,該組合與直觀分析得出的最佳組合7一致,進(jìn)一步確定組合7為最優(yōu)組合。
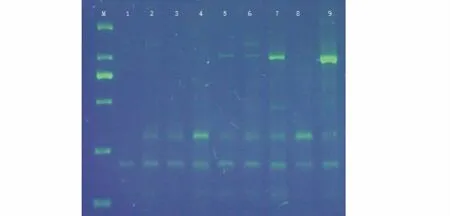
圖1 SRAP-PCR正交試驗(yàn)設(shè)計(jì)結(jié)果Fig.1 Electrophoresis of SRAP-PCR orthogonal design
2.2 模板DNA濃度的確定
應(yīng)用上述最佳反應(yīng)體系,對模板DNA設(shè)置了10,20,30,40,50和60ng 6個(gè)濃度梯度進(jìn)行優(yōu)化。擴(kuò)增結(jié)果表明,在25μL反應(yīng)體系中6種模板DNA用量均能擴(kuò)增出譜帶,且?guī)鸵恢隆50錎NA濃度不同,譜帶的清晰度存在一定差異。模板DNA用量為10和20ng時(shí)條帶清晰度不佳,30~60ng條帶清晰度稍強(qiáng)且表現(xiàn)一致。考慮到節(jié)省模板用量,最終選定在25μL反應(yīng)體系中加30ng模板DNA為宜。
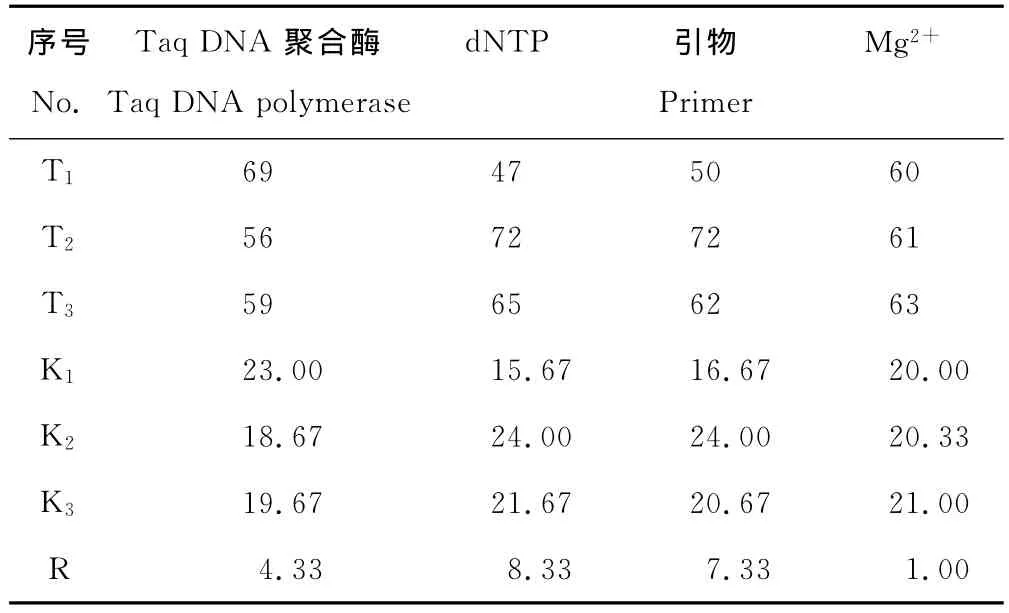
表3 正交試驗(yàn)結(jié)果的統(tǒng)計(jì)分析Table 3 Statistic result of the orthogonal design
2.3 反應(yīng)體系的驗(yàn)證及多態(tài)性引物組合的篩選
應(yīng)用上述最佳反應(yīng)體系,隨機(jī)組合了98對SRAP引物組合對3份山野豌豆種源進(jìn)行了SRAP擴(kuò)增(圖2)。結(jié)果表明,1)在篩選的98對引物組合中,94對引物組合擴(kuò)增得到清晰穩(wěn)定的譜帶,占總數(shù)的95.9%,表明該反應(yīng)體系具有較好的穩(wěn)定性;在有擴(kuò)增產(chǎn)物的94對引物組合中有20對引物組合擴(kuò)增出多態(tài)性條帶,占總數(shù)21.3%。2)98對引物共擴(kuò)增出983條帶,其中20對具有多態(tài)性的引物組合擴(kuò)增出的條帶總數(shù)為164條,占總條帶數(shù)的16.7%,具有多態(tài)性的條帶有30條,平均每對引物組合擴(kuò)增出1.5條。3)不同引物組合檢測到的多態(tài)性位點(diǎn)數(shù)目存在差異,變異幅度為1~7。
3 討論與結(jié)論
3.1 正交試驗(yàn)設(shè)計(jì)優(yōu)化SRAP-PCR反應(yīng)體系
PCR擴(kuò)增體系主要影響因素包括 Mg2+、dNTP、引物、Taq DNA聚合酶以及模板DNA。本研究優(yōu)化出的體系表明引物濃度對擴(kuò)增結(jié)果影響最大,這與黃春瓊等[28]、周良彬等[29]的結(jié)果不一致,這主要是因?yàn)楸狙芯吭谝锾荻仍O(shè)計(jì)時(shí)加入了以前文獻(xiàn)中不常見到的高濃度——2.0μmol/L,僅在昝逢剛等[30]對荔枝(Litchichinensis)和許永華等[31]對人參(Panaxginseng)的研究中分別出現(xiàn)過5 mmol/L和2.0μmol/L的高引物濃度。雖說文獻(xiàn)中常見引物濃度要以最低引物量產(chǎn)生所需要的結(jié)果為好,引物濃度偏高會(huì)引起錯(cuò)配和非特異性擴(kuò)增,但本研究結(jié)果表明高濃度的引物濃度不但沒有引起錯(cuò)配反而得到更清晰的條帶,從圖1可看到7道與9道均得到1kb處的條帶,而9道的引物濃度僅為0.2μmol/L,這說明此帶并非非特異性條帶。此后以6號與14號為DNA模板進(jìn)行的重復(fù)試驗(yàn),也證實(shí)了2.0μmol/L的引物濃度并無非特異性擴(kuò)增條帶,反而條帶清晰這一論斷。其次,影響較大的是Mg2+濃度。這與王芳[18]、王俊娥[21]、艾鵬飛等[32]、仲叢來等[33]在大豆、山羊豆、小麥(Triticumaestivum)和麻瘋樹(Jatrophacurcas)上的試驗(yàn)結(jié)果一致,這說明在SRAP-PCR反應(yīng)體系中Mg2+濃度的影響是很大的,在以后的優(yōu)化試驗(yàn)中要重點(diǎn)關(guān)注。再次,影響因子較小的是Taq DNA聚合酶用量和dNTP濃度,其中不同dNTP濃度的擴(kuò)增效果差異很小。在正交試驗(yàn)之后,本研究又對DNA模板用量進(jìn)行了單因素優(yōu)化。雖然DNA模板用量對擴(kuò)增的影響不大[34,35],但本研究證實(shí)用量過少會(huì)明顯影響條帶的數(shù)量與清晰度,DNA模板用量為10~20ng明顯沒有30~60ng的條帶清晰度高,所以本實(shí)驗(yàn)選擇30ng的DNA模板用量,此結(jié)果與孫佩光等[36]、陳慶榆等[37]的研究結(jié)果一致。

圖2 優(yōu)化的SRAP-PCR反應(yīng)體系在部分引物組合中的擴(kuò)增結(jié)果Fig.2 Results amplified by optimized system using some primer combinations
3.2 SRAP-PCR反應(yīng)優(yōu)化體系的驗(yàn)證
本研究不僅通過98對引物對優(yōu)化出的SRAP-PCR反應(yīng)體系進(jìn)行了驗(yàn)證,而且在優(yōu)化過程中選用了3種來自北京、山西、內(nèi)蒙古不同地域的模板DNA取得了二次驗(yàn)證,以此保證了優(yōu)化體系的穩(wěn)定性與良好的適用性。與此同時(shí),本研究亦篩選出20對多態(tài)性良好的引物,對以后山野豌豆的分子生物學(xué)研究打下了良好的基礎(chǔ)。
3.3 結(jié)論
本研究利用正交試驗(yàn)設(shè)計(jì)對山野豌豆SRAP-PCR反應(yīng)體系進(jìn)行優(yōu)化,得到的最佳反應(yīng)體系為:2μL 10×PCR buffer(不含 Mg2+)、30ng的模板 DNA、引物2.0μmol/L、Mg2+2.0mmol/L、Taq DNA 聚合酶1.5U、dNTP 0.2mmol/L,總體積為25μL。
[1] 陳默君,賈慎修.中國飼用植物[M].北京:中國農(nóng)業(yè)出版社,2002.
[2] 陳默君.五種野豌豆飼用價(jià)值的初步探討[J].中國草地學(xué)報(bào),1986,3:43-46.
[3] 馬章全,馮忠義.三個(gè)適合農(nóng)牧區(qū)種植的優(yōu)質(zhì)牧草品種[J].農(nóng)村百事通,2008,(19):40-41.
[4] 郜玉田,張雪艷.有發(fā)展前途的新草種——山野豌豆[J].吉林畜牧獸醫(yī),1991,(2):39-40.
[5] 洪銳民,肖文一.黑龍江省野豌豆屬野生牧草的特征、習(xí)性及其利用[J].黑龍江畜牧獸醫(yī),1992,(7):14-15.
[6] 吳學(xué)敏.一種優(yōu)良野生牧草——山野豌豆[J].山西水土保持科技,1983,(3):93.
[7] 王鶴橋.野生牧草綠肥植物——山野豌豆[J].中國草原與牧草,1986,3(6):49.
[8] 楊巧荷,羅素琴,劉樂樂,等.蒙藥材山野豌豆中的化學(xué)成分定性分析[J].內(nèi)蒙古醫(yī)學(xué)院學(xué)報(bào),2010,32(5):475-477.
[9] 周良彬,盧欣石,王鐵梅,等.雜花苜蓿種質(zhì)SRAP標(biāo)記遺傳多樣性研究[J].草地學(xué)報(bào),2010,18(4):544-549.
[10] 何慶元,吳萍,張曉紅,等.不同秋眠性苜蓿SRAP體系優(yōu)化及遺傳多樣性分析[J].草業(yè)學(xué)報(bào),2011,20(2):201-209.
[11] 張宇.利用SRAP分子標(biāo)記研究苜蓿雜種優(yōu)勢[D].呼和浩特:內(nèi)蒙古農(nóng)業(yè)大學(xué),2010.
[12] Ariss J J,Vandemark G J.Assessment of genetic diversity among nondormant and semidormant alfalfa populations using sequence-related amplified polymorphisms[J].Crop Science,2007,47(6):2274-2284.
[13] Vandemark G J,Ariss J J,Bauchan G A,et al.Estimating genetic relationships among historical sources of alfalfa germplasm and selected cultivars with sequence related amplified polymorphisms[J].Euphytica,2006,152(1):9-16.
[14] Castonguay Y,Cloutier J,Bertrand A,et al.SRAP polymorphisms associated with superior freezing tolerance in alfalfa(Medicago sativa spp.sativa)[J].Theoretical and Applied Genetics,2010,120(8):1611-1619.
[15] 關(guān)瀟.野生紫花苜蓿種質(zhì)資源遺傳多樣性研究[D].北京:北京林業(yè)大學(xué),2009.
[16] Baloch F S,Kurt C,Arioglu H.Assaying of diversity among soybean(Glycin max (L.)Merr.)and peanut(Arachis hypogaea L.)genotypes at DNA level[J].Turkish Journal of Agriculture and Forestry,2010,34(4):285-301.
[17] 王賢智.大豆產(chǎn)量相關(guān)性狀的遺傳與穩(wěn)定性分析及QTL定位研究[D].武漢:中國農(nóng)業(yè)科學(xué)院,2008.
[18] 王芳.大豆的SRAP遺傳多樣性及蛋白含量多樣性研究[D].新鄉(xiāng):河南師范大學(xué),2009.
[19] 張婧源,彭燕,羅燕,等.不同產(chǎn)地白三葉種質(zhì)遺傳多樣性的SRAP分析[J].草業(yè)學(xué)報(bào),2010,19(5):130-138.
[20] 李潤芳.三葉草的細(xì)胞、分子標(biāo)記及多倍體誘導(dǎo)研究[D].武漢:華中農(nóng)業(yè)大學(xué),2007.
[21] 王俊娥.山羊豆屬植物遺傳多樣性研究[D].北京:中國農(nóng)業(yè)科學(xué)院北京畜牧獸醫(yī)研究所,2008.
[22] Saha G C,Sarker A,Chen W D,et al.Identification of markers associated with genes for rust resistance in Lens culinaris Medik[J].Euphytica,2010,175(2):261-265.
[23] Saha G C,Sarker A,Chen W D.Inheritance and linkage map positions of genes conferring resistance to stemphylium blight in lentil[J].Crop Science,2010,50(5):1831-1839.
[24] Li G,Quiros C F.Sequence related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:its application to mapping and gene tagging in Brassica[J].Theoretical and Applied Genetics,2001,103(2-3):455-461.
[25] Riaz A,Li G,Quresh Z,et al.Genetic diversity of oil seed Brassica napus inbred lines based on sequence-related amplified polymorphism and its relation to hybrid performance[J].Plant Breeding,2001,120(5):411-415.
[26] 何正文,劉運(yùn)生,陳立華,等.正交設(shè)計(jì)直觀分析法優(yōu)化PCR條件[J].湖南醫(yī)科大學(xué)學(xué)報(bào),1998,23(4):403-404.
[27] 張麗,周蘭英,肖千文,等.正交試驗(yàn)設(shè)計(jì)在建立杜鵑花RAPD-PCR反應(yīng)體系中的應(yīng)用[J].北方園藝,2007,(5):124-126.
[28] 黃春瓊,周少云,劉國道,等.狗牙根SRAP-PCR反應(yīng)體系的優(yōu)化[J].基因組學(xué)與應(yīng)用生物學(xué),2009,28(5):981-989.
[29] 周良彬,盧欣石,賴?yán)棼?雜花苜蓿SRAP-PCR反應(yīng)體系的正交優(yōu)化[J].草地學(xué)報(bào),2010,18(3):345-351.
[30] 昝逢剛,吳轉(zhuǎn)娣,曾淇,等.荔枝SRAP-PCR反應(yīng)體系的優(yōu)化[J].基因組學(xué)與應(yīng)用生物學(xué),2009,28(1):132-136.
[31] 許永華,王士杰,陳曉林,等.人參SRAP-PCR體系優(yōu)化條件的建立(英文)[J].農(nóng)業(yè)科學(xué)與技術(shù),2010,11(4):56-58.
[32] 艾鵬飛,武玉芬,魏景芳.小麥SRAP-PCR體系的正交設(shè)計(jì)優(yōu)化(英文)[J].農(nóng)業(yè)科學(xué)與技術(shù),2011,12(3):366-368,374.
[33] 仲叢來,丁貴杰,沈凌麻.麻瘋樹SRAP-PCR反應(yīng)體系的優(yōu)化[J].貴州農(nóng)業(yè)科學(xué),2010,38(4):19-22.
[34] Guo D L,Luo Z R.Genetic relationships of some PCNA persimmons(Diospyros kaki Thunb.)from China and Japan revealed by SRAP analysis[J].Genetic Resources and Crop Evolution,2006,53(8):1597-1603.
[35] 郭大龍,羅正榮.部分柿屬植物SRAP-PCR反應(yīng)體系的優(yōu)化[J].果樹學(xué)報(bào),2006,23(1):138-141.
[36] 孫佩光,奚如春,鈕世輝,等.油茶SRAP-PCR反應(yīng)體系的建立和優(yōu)化[J].基因組學(xué)與應(yīng)用生物學(xué),2010,29(6):1192-1199.
[37] 陳慶榆,繆成貴,劉曉鋒.棉花高質(zhì)量DNA的提取及SRAP反應(yīng)體系的優(yōu)化[J].生物學(xué)雜志,2010,27(5):31-34.

