DNA條形碼技術在魚肉及其制品鑒別中的應用
李新光,王 璐,趙 峰,馬麗萍,孫 永,周德慶,*
(1.中國水產科學研究院黃海水產研究所,山東 青島 266071;2.上海海洋大學食品學院,上海 201306;3.中國海洋大學食品科學與工程學院,山東 青島 266003)
DNA條形碼技術在魚肉及其制品鑒別中的應用
李新光1,2,王 璐1,3,趙 峰1,馬麗萍1,2,孫 永1,周德慶1,*
(1.中國水產科學研究院黃海水產研究所,山東 青島 266071;2.上海海洋大學食品學院,上海 201306;3.中國海洋大學食品科學與工程學院,山東 青島 266003)
為鑒別冷凍魚、凍魚片和烤魚片中魚肉成分的來源,用以判別其與食品標簽是否相符,以COI基因為靶點,利用基因克隆和生物信息學方法建立魚肉制品DNA條形碼鑒別技術。對20種冷凍魚、10種凍鱈魚片和15種烤魚片樣品進行鑒定,結果表明:20種冷凍魚鑒定的結果與形態學鑒定的結果一致;10種凍鱈魚片樣品主要以“狹鱈”為主(6/10),存在將“白鱈魚”標識為“銀鱈魚”的現象;烤魚片樣品與其標簽上所標識的原料多數不符,一些烤魚片還檢有月尾兔頭鲀。DNA條形碼技術是一種簡單、有效的分子鑒別技術,可用于冷凍魚、凍魚片和烤魚片中魚肉成分的鑒定。
DNA條形碼;物種鑒別;凍鱈魚片;烤魚片
在生物物種鑒定領域,DNA條形碼技術是發展最為迅速的一種新技術,其理論依據是使用一段標準DNA片段,對物種進行準確、快速的鑒定[1-3]。通常采用680bp長的COI序列,97%以上的魚類可以鑒定到種的水平。DNA條形碼鑒別技術要有專門的數據庫作為支撐,含有較全面的生物學信息。DNA條形碼技術能避免形態學分類的缺陷,對鑒定者的經驗和專業知識背景要求較低,只需測序獲得樣品的線粒體COI序列,并與DNA條形碼數據庫(http://www. barcodinglife.org)或GenBank數據庫(http:// blast.ncbi.nlm.nih.gov/Blast.cgi)中該物種及其近似種序列進行比對,根據相互間的遺傳距離或相似度即可確定待檢樣品種屬。
目前,國外已將DNA條形碼技術應用于食品分析。Haye等[4]利用DNA條形碼技術對智利市場上7種螃蟹制品進行鑒別,發現一種螃蟹制品與其商品標簽不符;Wong等[5]利用DNA條形碼技術對市場上的91個海產品樣品進行分析,推斷出可能有25%的海產品的標簽與實物不符,認為DNA條形碼技術是一種經濟、有效的可將海產品鑒定到種的分子鑒別技術。在我國,DNA條形碼技術用于海洋生物的分類鑒定和動物性珍貴藥材的鑒定,而用于食品鑒別的文獻報道較少[6-9]。消費者在購買鮮活或冷凍魚時,多是通過魚的形態特征來判斷魚的種類,對于一些魚肉制品(如魚片、魚丸等),不易通過形態特征來判斷其所用的原料。據報道,近年來我國魚肉制品的標簽存在標識錯誤的問題,如油魚冒充鱈魚[10],嚴重損害了消費者的利益。
本研究參照Ivanova[11]和柳淑芳[12]等的文獻,篩選了COI基因的通用引物,采用基因克隆技術和生物信息學技術用于市場上購買的冷凍魚、凍魚片和烤魚片的鑒別,旨在建立準確、高效的魚肉制品的鑒定方法,為魚肉制品的溯源和安全監管提供技術支持。
1 材料與方法
1.1 材料與試劑
2012年5月,從市場上隨機抽取45個樣品。其中,20種冷凍魚樣品、10種凍鱈魚片樣品、15種烤魚片樣品(7種烤鱈魚片、8種其他標識的烤魚片),冷凍魚及凍鱈魚片置于-30℃冰箱保存備用,烤魚片置于常溫條件下備用。
無水乙醇、氯仿、異戊醇和異丙醇(分析純) 上海國藥集團化學試劑有限公司;Tris-平衡酚(pH8.0) 北京索萊寶科技有限公司;10×Buffer緩沖液、Taq DNA聚合酶、Top10感受態細胞、瓊脂糖凝膠DNA回收試劑盒天根生化科技(北京)有限公司;dNTPs(2.5mmol/L)、DNA Marker(DL2000)、克隆試劑盒 寶生物工程(大連)有限公司;蛋白酶K 德國Merck公司;Gene Finder核酸染料 廈門百維信生物科技有限公司。
1.2 儀器與設備
Sigma 1-14冷凍離心機 德國Sigma公司;PCR儀德國Biometra公司;DYCP-31E電泳儀 北京六一儀器廠;凝膠成像系統 法國Vilber Lourmat公司;NanoPhotometerTM微量核酸蛋白測定儀 德國Implen公司。
1.3 方法
1.3.1 冷凍魚片DNA提取
采用改良后的高鹽法[13]。用滅菌的手術刀片取凍魚片150mg于1.5mL的離心管中,再分別加入400μL的TE(含10mmol/L Tris-HCl和1mmol/L EDTA)、40μL 10% SDS以及8μL 20mg/mL的蛋白酶K,渦旋30s,充分混勻;將離心管置于55~65℃的恒溫水浴槽中消化4h,加入300μL 6mol/L飽和的NaCl溶液,高速渦旋30s,12000r/min離心30min;取上清液至新的1.5mL離心管中,加入等體積的酚-氯仿-異戊醇(25:24:1,V/V),緩慢振蕩1min,12000r/min離心10min;取上清液至新的離心管中,加氯仿-異戊醇(24:1,V/V)緩慢振蕩至乳白色,12000r/min離心10min;取上清液至新的離心管中,加入1/10體積3mol/L NaAc溶液,等體積的異丙醇,冰浴30min,12000r/min離心10min;然后加入800μL 70%乙醇進行洗滌,棄去乙醇,在56℃的烘箱中烘干,然后加入50μL雙蒸水,-20℃條件下保存。
1.3.2 烤魚片DNA提取
1.3.2.1 脫脂前處理
取3g烤魚片置于50mL的離心管中,用消毒過的剪刀剪碎,然后加入5mL正己烷(氯仿-甲醇、無水乙醇作為對比),渦旋1min;將離心管置于45℃的恒溫水浴槽2~3h,然后8000r/min離心5min;棄油脂層,加入10~15mL 70%乙醇溶液,渦旋1~2min,8000r/min離心5min(可視情況重復幾次);加入無水乙醇10mL,渦旋1~2min,8000r/min離心10min;棄乙醇溶液,將處理后的魚肉制品置于60℃熱風干燥箱中烘干,研磨成細小顆粒即可用于DNA提取。
1.3.2.2 DNA提取
稱取經研磨的鱈魚片粉末30mg于1.5mL的離心管中,分別加入400μL TE、50μL 10% SDS以及15μL 20mg/mL蛋白酶K,渦旋30s,充分混勻;將離心管置于55~65℃的恒溫水浴槽中消化過夜,隨后步驟同1.3.1節中所述,結果分析采用SPSS 17軟件。
1.3.3 樣品DNA 純度及質量濃度的測定
吸取稀釋后的DNA模板2μL,以不含DNA的去離子水作為空白對照,使用微量核酸蛋白測定儀測定DNA模板的質量濃度及A260nm/A280nm。其中A260nm/A280nm用于估測DNA的純度,而DNA的質量濃度根據以下公式計算:
DNA的質量濃度/(ng/μL)= A260nm×50×稀釋倍數
1.3.4 通用引物設計及PCR擴增
COI序列的通用引物設計參照Ivanova[11]和柳淑芳[12]等的方法,目的片斷是COI基因靠近5’末端長度為680bp左右的序列,其通用引物由華大基因合成。上游引物FishF1:5’ TCA ACC AAC CAC AAA GAC ATT GGC AC 3’,上游引物FishF2:5’ TCG ACT AAT CAT AAA GAT ATC GGC AC 3’;下游引物FishR1:5’ TAG ACT TCT GGG TGG CCA AAG AAT CA 3’,下游引物FishR2:5'ACT TCA GGG TGA CCG AAG AAT CAG AA 3’。將引物分別混合均勻,終濃度為25μmol/L,-20℃冰箱保存備用。聚合酶鏈式反應(polymerase chain reaction,PCR)采用50μL的體系,DNA模板100~500ng,10×緩沖液(含Mg2+)5μL,dNTPs(各2.5mol/L)4μL,混合的上游引物與下游引物(25μmol/L)各0.6μL,Taq DNA聚合酶(2.5U/mL)0.6μL,加去離子水至50μL。PCR擴增采用升式PCR,反應程序為:94℃、4min;94℃、30s,53℃、40s,72℃、1min,8個循環;94℃、30s,54℃、40s,72℃、1min,8個循環;94℃、30s,55℃、40s,72℃、1min,16個循環;72℃、10min。PCR擴增完成后,經1%瓊脂糖凝膠進行電泳分離后,使用數碼凝膠成像系統分析擴增產物。
1.3.5 克隆、測序及序列比對
挑選陽性擴增的PCR產物,按照瓊脂糖凝膠DNA回收試劑盒說明書的操作步驟,對PCR產物進行純化,隨后與pMD T-18載體連接,導入Top10感受態細胞中,36℃培養過夜,經過藍-白斑篩選后,挑取3個不同的白色單菌落于LB液體培養基(含有100μg/mL氨芐青霉素)中,在36℃、180r/min的搖床上培養6~8h,經PCR檢測后,吸取陽性克隆菌液1mL,送到華大基因進行測序。然后,應用DNAMAN 5.2.2進行多序列比對,繪制同源樹。同時,使用BOLD(http://www.boldsystems.org)以及NCBI(http://blast.ncbi.nlm.nih.gov)網站的數據庫對樣品的COI序列進行鑒定以及相似度分析。
2 結果與分析
2.1 冷凍魚鑒定分析
2.1.1 冷凍魚COI序列的擴增
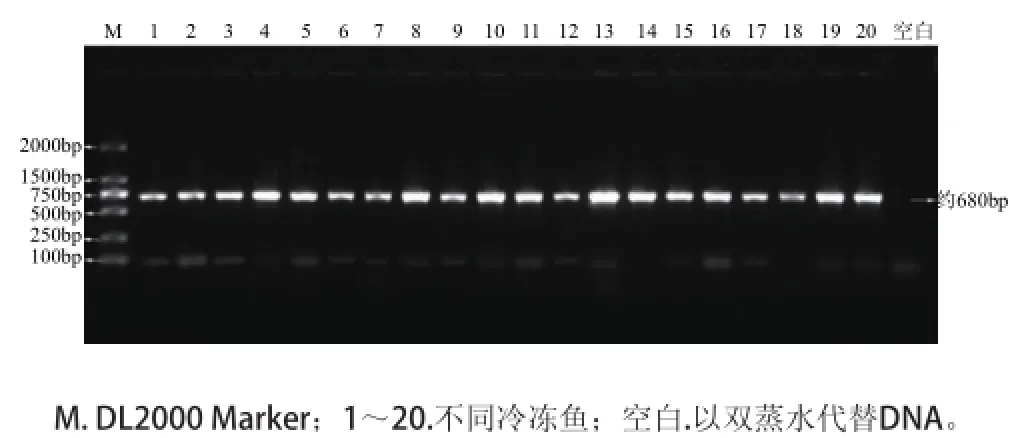
圖1 不同冷凍魚COICOI基因的擴增條帶Fig.1 COI gene amplification from different commercial fish
由圖1可知,冷凍魚COI序列均可以用通用引物擴增出來,說明通用引物的通用性高,可用于魚肉中COI基因的擴增。這可能是由于線粒體DNA在細胞中比細胞核DNA拷貝量大[3]。
2.1.2 冷凍魚COI測序結果分析
在分析種內種間的遺傳距離時,生物條形碼聯盟推薦使用K-2-P(Kimura-2-parameter)模型計算。同時指出,基于K-2-P模型構建的系統發育樹,不是用來做系統發育的研究,僅僅是為了驗證物種的單源性和聚類關系[3]。對于物種序列同源性較高的COI序列,DNA條形碼技術多采用基于遺傳距離的Neighbor Joining法構建進化樹[2-3]。雖然這種方法處理較多的COI序列時,速度快又能反映出物種間的親緣關系,但繪制的進化樹難以理解又不能讀出物種COI序列間的相似度。為了克服這些缺點,又能驗證物種的單源性和聚類關系,本實驗采用同源樹對樣品的COI序列進行聚類分析。
對市場上常見的20不同種屬的冷凍魚,先進行形態學鑒定,然后利用DNA條形碼技術進行驗證,結果顯示:DNA條形碼技術鑒定的結果和魚類形態學鑒定的結果完全一致,且COI序列鑒定后的相似度均大于97.7%(表1)。說明DNA條形碼技術很容易將市面上常見的20種魚類區分開來,可用于冷凍魚和冷凍魚肉制品魚肉成分的種屬鑒定。

表1 不同冷凍魚在BOLD數據庫的鑒定結果Table1 Identification of different commercial fish in the BOLD dataabbaassee
2.2 凍鱈魚片鑒定分析
2.2.1 凍鱈魚片COI測序分析
對鱈魚片的COI序列和GenBank數據庫收錄的常見鱈魚的COI序列進行聚類分析(圖2),結果顯示各種鱈魚均形成獨立的分支,其中,鱈魚與其易混淆魚類COI序列的相似度為79%,在種的水平上同一種鱈魚COI序列的相似度為99%或100%,說明在種的水平上狹鱈、太平洋鱈魚、大西洋鱈魚、裸蓋魚、小鱗犬牙南極魚與異鱗蛇鯖及棘鱗蛇鯖很容易區分開來。樣品D10、D1可以初步鑒定為小鱗犬牙南極魚;樣品D2、D4、D5、D6、D8和D9可以明確地鑒定為狹鱈;樣品D3可以明確地鑒定為大西洋鱈魚。
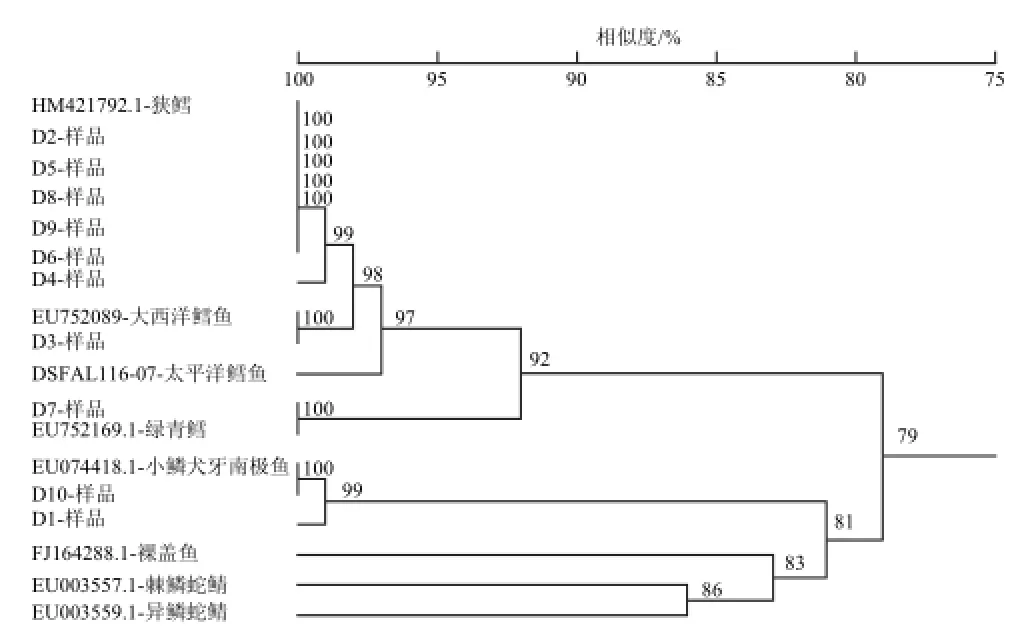
圖2 食品標簽為凍鱈魚片樣品的CCOOII序列與GenBank或BOLLDD數據庫收錄的CCOOII序列的聚類同源樹Fig.2 Homology tree based on COI sequences of commercial frozen cod fillets and its related sequences from GenBank or BOLD
2.2.2 凍鱈魚片鑒定結果
10個凍鱈魚片樣品的鑒定結果均為鱈魚,存在將“白鱈魚”標識為“銀鱈魚”的現象,其他凍鱈魚片樣品的鑒定主要以“狹鱈”為主(6/10),只有1個樣品為大西洋鱈魚,說明凍鱈魚片主要是以價格便宜的狹鱈為原料加工而成的。
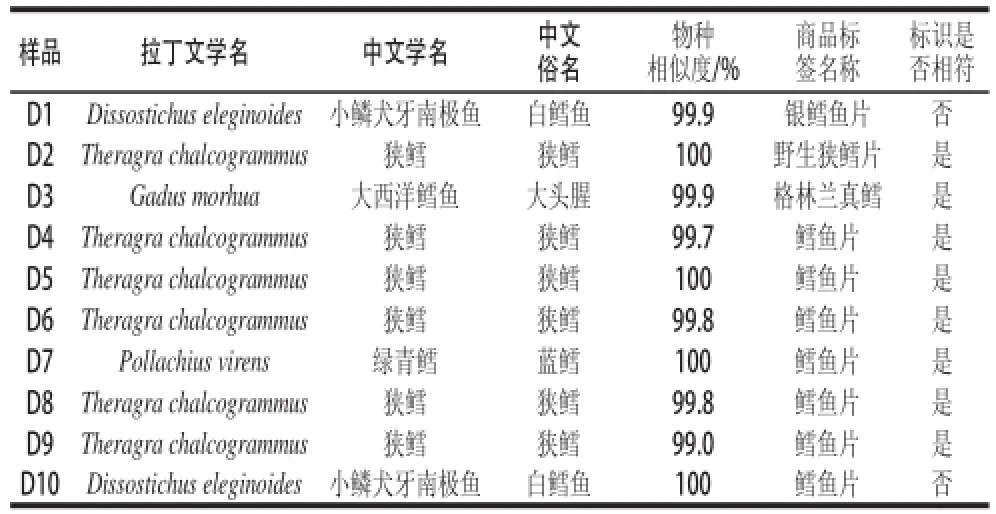
表2 凍鱈魚片在BOLD數據庫的鑒定結果Table2 Identification of commercial frozen cod fillets in the BOLD dataabbaassee
2.3 烤魚片鑒定分析
2.3.1 前處理試劑對烤魚片DNA質量濃度及純度的影響
為了提取高質量濃度和高純度的DNA,用3種不同的有機試劑(氯仿-甲醇、正己烷、無水乙醇)對烤魚片脫脂處理。通過對烤魚片DNA質量濃度和純度進行單因素方差分析(表3),發現3種不同的脫脂溶液對DNA質量濃度具有極其顯著的差異(P=0.003<0.01),而對烤魚片DNA的純度沒有顯著性差異(P=0.093>0.05)。采用Duncan法對烤魚片DNA質量濃度和純度進行分析[14],發現使用氯仿-甲醇(2:1,V/V)和正己烷作為樣品的前處理試劑所獲得的DNA質量濃度較高,且純度間沒有顯著性差異(P=0.058>0.05)。雖然使用氯仿-甲醇作為前處理試劑可獲得較高的DNA質量濃度(表4),但氯仿-甲醇混合液具有致癌作用,處理樣品后油層不易分離且殘留多;而正己烷低毒,通常作為食用植物油的浸提劑,處理樣品后油層容易分離且殘留少,故選正己烷作為烤魚片的前處理試劑。
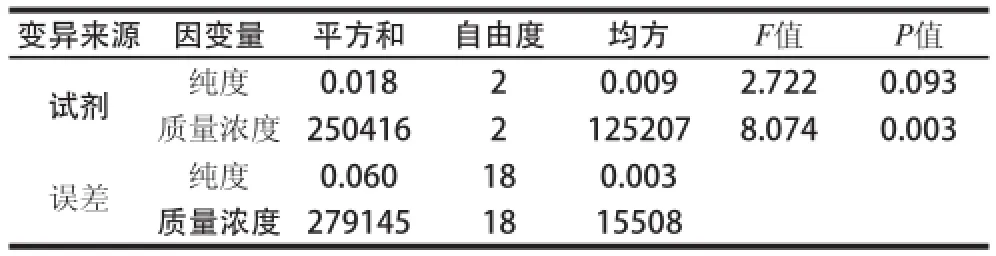
表3 烤魚片DNA質量濃度和純度的方差分析表Table3 Variance analysis table for DNA concentration and purity from grilled fillets
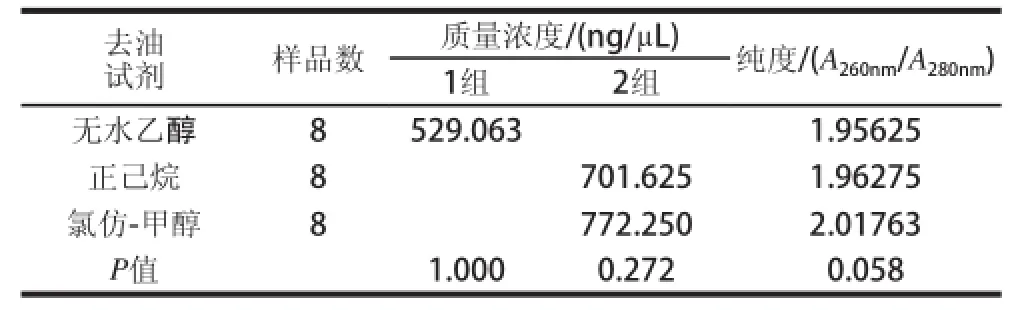
表4 烤魚片DNA質量濃度和純度的Duncan分析表Table4 Duncan analysis table for DNA concentration and purity of grilled fish fillets
2.3.2 烤魚片COI序列的擴增
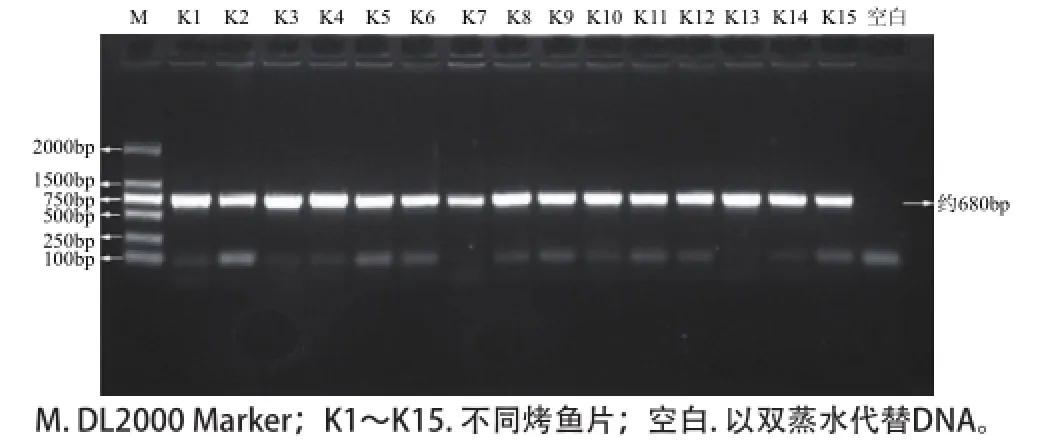
圖3 不同烤魚片COICOI基因的擴增條帶Fig.3 COI gene amplification from different commercial baked fish fillets
據相關文獻報道,加熱和氧化是導致DNA降解的兩個重要因素[15-16]。烤魚片在加工時,經過烘烤處理(150~250℃,3~7min)可能會導致DNA嚴重的降解。但由圖3可以看出,COI基因的通用引物均可將烤魚片的目的基因擴增出來,說明短時間高溫處理不足以破壞烤魚片全部的線粒體DNA。
2.3.3 烤魚片鑒定結果
在BOLD和GenBank數據庫對15個不同烤魚片的COI序列進行鑒定(表5),結果表明,絕大多數的烤魚片與其食品標簽不符。其中,7個食品標簽為“烤鱈魚片”的樣品均未檢出鱈魚的成分,其鑒定結果共涉及3種魚類,分別是細紋獅子魚、花斑蛇鯔、月尾兔頭鲀;其他標識的烤魚片也存在類似的情況,只有“針良魚片”的鑒定結果與其食品標簽相符,其余的都不相符,鑒定結果涉及的魚類更多,分別是花斑蛇鯔、多齒蛇鯔、龍頭魚、針良魚、多鱗鱚和黑鰓兔頭鲀。由于黑鰓兔頭鲀(南方產)和月尾兔頭鲀的魚肉和內臟含有毒素[17],人食用了以黑鰓兔頭鲀或月尾兔頭鲀為原料生產的烤魚片,可能會出現中毒癥狀。
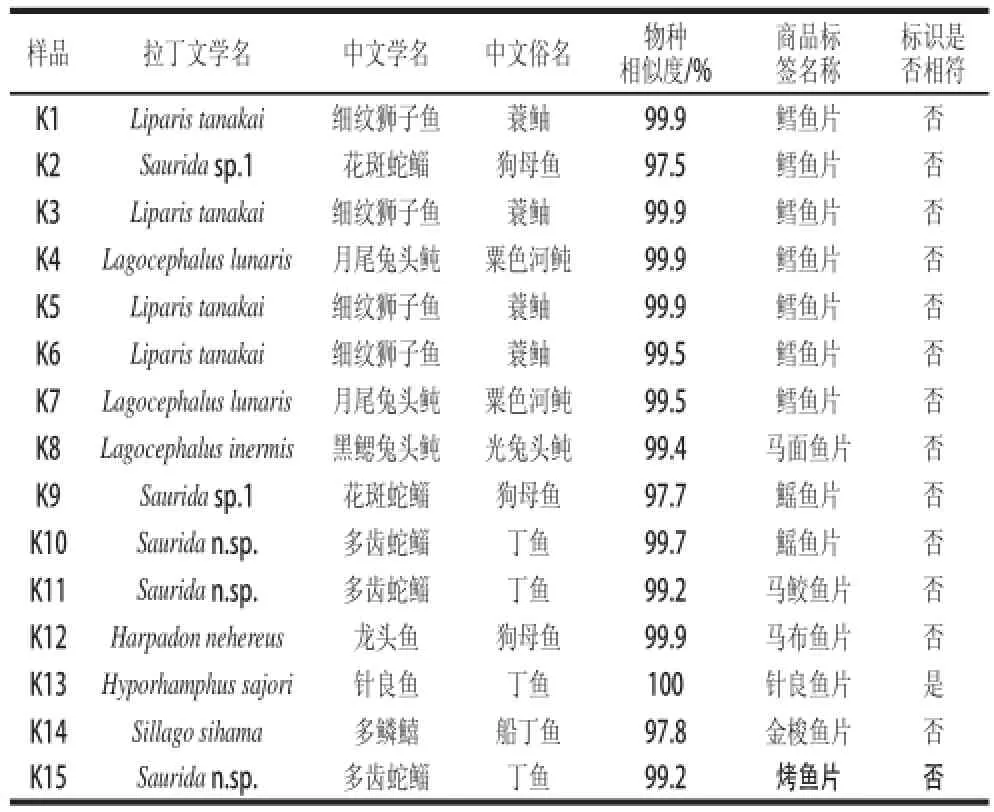
表5 烤魚片在BOLD數據庫的鑒定結果Table5 Identification of commercial frozen cod fillets and grilled fillets in the BOLD database
3 討 論
3.1 鱈魚與易混淆魚類的鑒別
魚類分類學上,“鱈魚”泛指鱈科魚類及屬于鱈形目相關的品種。根據糧農組織和FishBase兩個機構的資料,鱈科約60個品種,鱈形目約500個品種。市面場上常見的鱈魚有狹鱈、綠青鱈、黑線鱈、大西洋鱈魚和太平洋鱈魚等。通常冒充鱈魚的油魚為鱸形目蛇鯖科魚類,其學名是異鱗蛇鯖或棘鱗蛇鯖,俗名為龍鱈魚、水鱈魚、玉梭魚。市場上,鱈魚還包含一些俗名中帶有“鱈”字但親緣關系較遠的名貴魚類,如裸蓋魚(銀鱈魚)、小鱗犬牙南極魚(白鱈魚)和鱗頭犬牙南極魚(白鱈魚)[18-19]。其中,狹鱈較為常見且價格最為便宜;大西洋鱈魚、太平洋鱈魚及裸蓋魚常見價格處于中檔水平;小鱗犬牙南極魚和鱗頭犬牙南極魚稀少價格最為昂貴。根據COI序列的相似度區分,不同品種的鱈魚與其易混淆魚COI序列的相似度在78.1%~80.6%之間。
3.2 加工原料和加工條件對DNA條形碼技術的影響
通過研究發現,DNA條形碼技術適用于含有單一肉類成分魚肉制品的鑒別,如冷凍魚片、烤魚片、罐頭制品中魚肉成分的鑒別。若魚肉制品含有多種肉類成分,從理論上可以挑選多個單克隆菌體去測序,以達到鑒定多種肉類成分的目的,但這樣目的性不強,還會增加檢測的費用,如魚丸(含有多種雜魚或摻入雞肉、豬肉)中肉類成分的鑒別。此外,樣品DNA模板的獲取也是影響魚肉制品鑒別的另一個重要因素,尤其是一些經過高溫高壓或深度油炸加工后的魚肉制品,線粒體DNA出現嚴重的降解,如一些罐頭制品在加工過程中,要經過高溫高壓滅菌工序,可能使DNA斷鏈或降解,導致DNA提取率降低。此外,罐頭制品中還加入了糖、植物油以及各種食品添加劑,這些物質也可能降低DNA的提取率或抑制PCR反應。因此,DNA條形碼技術存在局限性,適用于加工條件比較溫和、肉類成分單一的魚肉制品的鑒別。3.3 DNA條形碼技術的優勢
目前,魚肉制品的鑒定技術主要有近紅外光譜技術、DNA條形碼技術、AFLP(擴增片段長度多態性)技術、SSR標記(微衛星標記)技術等[20-21]。通常近紅外光譜技術鑒別魚肉制品的優點是較為快速,缺點是需要建模、適用范圍窄;AFLP 優點是比較可靠、操作簡便,但AFLP的譜帶可能發生錯配或缺失、成本高等[21];SSR標記具有呈共顯性遺傳,可區分雜合子和純合子,但在采用SSR 技術時必須知道重復序列兩端的DNA 序列的信息,而SSR 標記的等位基因數目多,帶型復雜,難以判型,給DNA 指紋識別自動化和規模化帶來困難[20-21]。而DNA條形碼鑒別技術是以COI基因作為鑒定物種的靶基因,能避免形態學分類的缺陷,對鑒定者的經驗和專業知識背景要求較低,此外,DNA條形碼鑒別技術還有專門的數據庫作為支撐,含有的較全面的生物學信息,截止到2012年6月23日,魚類條形碼的序列庫已有49條公開的COI擴增引物,75249條COI序列,8795種被編碼的魚類[19]。隨著研究的深入,DNA條形碼鑒別技術還可以與基因芯片技術、RFLP(限制性片段長度多態性)、TTGE(時間溫度梯度電泳)相結合,達到對肉類成分單一的魚肉制品更準確、更快速的鑒別[22-24]。
[1] ZHANG J B, HANNER R. DNA barcoding is a useful tool for the identification of marine fishes from Japan[J]. Biochemical Systematics and Ecology, 2011, 39(1): 31-42.
[2] 鄭海輝, 張守純, 郭冬. 動物DNA條形碼研究進展及應用前景[J]. 黑龍江畜牧獸醫, 2011(1): 28-30.
[3] 武宇鵬, 丁亮, 李捷, 等. DNA條形碼的應用進展及討論[J]. 環境昆蟲學報, 2011(1): 99-106.
[4] HAYE P A, SEGOVIA N I, VERA R, et al. Authentication of commercialized crab-meat in Chile using DNA barcoding[J]. Food Control, 2011, 25(1): 1-25.
[5] WONG E H K, HANNER R H. DNA barcoding detects market substitution in North American seafood[J]. Food Research International, 2008, 41(8): 828-837.
[6] 彭居俐. 東亞特有鯉科類群的DNA條形碼研究及其系統發育分析[D]. 北京: 中國科學院研究生院, 2007.
[7] 呂國慶, 牛憲立, 姬可平. 動物性中藥材地龍DNA條形碼初步研究[J]. 廣東農業科學, 2011, 38(17): 114-116.
[8] 李力, 顧海豐, 夏云, 等. 蛤蚧及其偽品微型DNA條形碼的引物篩選[J]. 時珍國醫國藥, 2011, 22(1): 202-205.
[9] 杜鶴, 崔麗娜, 姚輝, 等. 基于COI條形碼序列的珍珠母及其混偽品的DNA分子鑒定[J]. 中國現代中藥, 2012, 13(11): 12-14.
[10] 張敬偉. 油魚鱈魚疑云折射監管問題[EB/OL]. (2012-04-21) [2012-05-01]. http://www.ycwb.com/ePaper/ycwb/html/2012-04/21/ content_1374516.htm.
[11] IVANOVA N V, ZEMLAK T S, HANNER R H, et al. Universal primer cocktails for fish DNA barcoding[J]. Molecular Ecology Notes, 2007, 7(4): 544-548.
[12] 柳淑芳, 陳亮亮, 戴芳群, 等. 基于線粒體CO1基因的DNA條形碼在石首魚科(Sciaenidae)魚類系統分類中的應用[J]. 海洋與湖沼, 2010(2): 223-232.
[13] CHAPELA M J, SOTELO C G, REREZMART R I, et al. Comparison of DNA extraction methods from muscle of canned tuna for species identification[J]. Food Control, 2007, 18(10): 1211-1215.
[14] 朱紅兵, 盧紋岱. 應用統計與SPSS應用[M]. 北京: 電子工業出版社, 2011: 402-419.
[15] ASLAN O, HAMILL R M, SWEENEY T, et al. Integrity of nuclear genomic deoxyribonucleic acid in cooked meat: implications for food traceability[J]. Journal of Animal Science, 2009, 87(1): 57-61.
[16] ZHANG L, WU Q. Single gene retrieval from thermally degraded DNA[J]. Journal of Biosciences, 2005, 30(5): 599-604.
[17] Fishbase [EB/OL]. (1984) [2012-06-30]. http://www.fishbase.org/ summary/Lagocephalus-inermis.html.
[18] 香港政府食物安全中心. 有關識別及標簽油魚/鱈魚的指引[EB/ OL]. (2007-08-16) [2012-06-30]. http://www.boldsystems.org/index. php//TaxBrowser Home.
[19] DNA條形碼官方網站[EB/OL]. (2003) [2012-06-30]. http://www. boldsystems.org/index.php/TaxBrowser_Home.
[20] TELETCHEA F. Molecular identification methods of fish species: reassessment and possible applications[J]. Reviews in Fish Biology and Fisheries, 2009, 19(3): 265-293.
[21] 吳瀟, 潘玉春, 唐雪明. 肉制品的DNA溯源技術[J]. 豬業科學, 2009, 26(3): 105-106.
[22] COLOMBO F, CHESSA S, CATTANEO P. Polymerase chain reaction products (PCR) on “DNA barcode zone” resolved by temporal temperature gradient electophoresis: a tool for species identification[J]. Food Control, 2011, 22(8): 1471-1472.
[23] HAIDER N, NABULS I, SAFADI B. Identification of meat species by PCR-RFLP of the mitochondrial COI gene[J]. Meat Science, 2012, 90(2): 490-493.
[24] 律迎春, 左濤, 唐慶娟, 等. 海參DNA條形碼的構建及應用[J]. 中國水產科學, 2011(4): 782-789.
Application of DNA Barcoding to Identify Commercial Fish and Fish Products
LI Xin-guang1,2,WANG Lu1,3,ZHAO Feng1,MA Li-ping1,2,SUN Yong1,ZHOU De-qing1,*
(1. Yellow Sea Fisheries Research Institute, Chinese Academy of Fishery Sciences, Qingdao 266071, China;2. College of Food Science and Technology, Shanghai Ocean University, Shanghai 201306, China;3. College of Food Science and Engineering, Ocean University of China, Qingdao 266003, China)
A DNA barcoding technique for species identification of fish ingredients in frozen whole fish, frozen fish fillets and grilled fish fillets by using COI gen as the target was established for use in examining whether the identified species are in accordance with those indicated on the label or not. Twenty fresh fishes, ten frozen cod fillets and fifty grilled fish fillets were identified using the DNA Barcoding technique. The results showed that DNA barcoding identification of the twenty frozen fishes was identical to morphological identification. Out of the ten frozen cod fillets, six samples were identified as Theragra chalcogrammus and some samples of Merluccius productus were labeled as Anoplopoma fimbria. Most of the grilled fish fillets were identified to be not the same as indicated on the label and Lagocephalus lunaris ingredients were detected in some of them. DNA barcoding as a simple and effective molecular identification approach is useful for the identification of fish ingredients in frozen whole fish, frozen fillets and grilled fish fillets.
DNA barcoding;species identification;frozen cod fillets;grilled fish fillets
TS254.7
B
1002-6630(2013)18-0337-06
10.7506/spkx1002-6630-201318069
2012-07-30
中央級公益性科研院所基本科研業務費專項(20603022013018)
李新光(1985—),男,碩士研究生,研究方向為水產品質量與安全。E-mail:lixinguang0102@163.com
*通信作者:周德慶(1962—),男,研究員,博士,研究方向為水產品加工與質量安全。E-mail:zhoudq@ysfri.ac.cn

