matK序列作為DNA條形碼在蒼耳屬中的應用
胡偉毅, 邵套喜
(1.連云港出入境檢驗檢疫局,江蘇連云港 222042; 2.江蘇省贛榆縣塔山鎮農業技術服務中心,江蘇連云港 222123)
matK序列作為DNA條形碼在蒼耳屬中的應用
胡偉毅1, 邵套喜2
(1.連云港出入境檢驗檢疫局,江蘇連云港 222042; 2.江蘇省贛榆縣塔山鎮農業技術服務中心,江蘇連云港 222123)
以matK序列作為DNA條形碼,對從國外截獲的7種蒼耳屬植物進行物種區分鑒定研究,采用DNeasy? Plant Mini Kit試劑盒進行總DNA的提取,應用通用引物對其matK基因進行擴增,測序得到7種蒼耳屬植物的matK序列,利用MEGA 5.1軟件對這7種蒼耳屬植物的matK序列進行比對和分析并構建系統樹,結果顯示:matK序列能從基因位點層面對蒼耳屬植物進行區分鑒定。
matK序列;DNA條形碼;蒼耳屬
蒼耳屬(Xanthium)植物為農田中的主要雜草,且可黏附于動物毛皮上,從而降低毛皮的品質,影響動物的健康,因此在2006年發布的農業部與國家質量監督檢驗檢疫總局共同制定的《中華人民共和國進境植物檢疫性有害生物名錄》中,將所有的蒼耳屬(非中國種)植物都列為檢疫性有害生物,防止其進入我國,危害我國農業生態安全。近年來,隨著我國檢驗檢疫鑒定技術的提高,許多蒼耳屬(非中國種)植物都被鑒定到種,但是蒼耳屬植物的形態鑒定手段對實驗室鑒定經驗有著較高的要求,而且蒼耳屬的果實在糧谷的長期運輸中難免會受到磨損,因此檢驗檢疫工作的一線工作人員有必要尋求新技術、新方法的支持。
植物DNA條形碼技術是針對植物基因組中的特定基因進行片段擴增、測序并發現其堿基變化規律的技術手段,其概念由加拿大科學家Paul Hebert于2003年正式提出[1],該技術在動物的COI基因中應用較為成熟[2-4],但由于COI基因在植物中變化保守,不能起到很好的區分作用,使得植物DNA條形碼技術的研究重點仍在通用基因片段的應用、篩選、搭配上[5]。盡管如此,植物DNA條形碼技術仍在保護瀕危物種、藥品食品監督、中草藥資源鑒定及生態學調查等諸多領域中起到了突破性的作用,發揮了以前形態學研究所無法產生的威力[6-8]。matK序列是編碼成熟酶K蛋白的基因,是國際生物條形碼聯盟(CBOL)推薦的植物DNA條形碼核心序列[9]。本試驗以2012年連云港口岸截獲的7種蒼耳屬植物為研究對象,探討其matK序列變化規律。
1 材料與方法
1.1 試驗材料
7種蒼耳屬植物:北美蒼耳(Xanthiumchinese)、蒼耳(Xanthiumsibiricum)、刺蒼耳(Xanthiumspinosum)、西方蒼耳(Xanthiumoccidentale)、巴西蒼耳(Xanthiumbrasilicum)、美麗蒼耳(Xanthiumspeciosum)、賓州蒼耳(Xanthiumpensylvanicum)。以上外來蒼耳均為2012年在進口大豆中截獲,并由中國檢驗檢疫科學院鑒定的樣本;DNeasy? Plant Mini Kit、2×Taqmaster mix、瓊脂糖、Golden View Ⅰ(GV Ⅰ)等試劑耗材。
matK引物[10]:matKf:5′-C G A T C T A T T C A T T C A A T A T T T C-3′;matKr:5′-T C T A G C A C A C G A A A G T C G A A G T-3′。
1.2 試驗方法
1.2.1 DNA的提取 采用DNeasy? Plant Mini Kit試劑盒法提取DNA,按照說明書逐步操作,可得到200 μL DNA樣品,-20 ℃冰箱保存。
1.2.2 PCR反應體系 PCR反應使用中美泰和公司2×Taq master mix進行擴增,反應采用25μL體系:DNA模板,3μL;引物1,1μL;引物2,1μL;2×Taq master mix,12.5 μL;ddH2O,7.5μL。
1.2.3 PCR擴增反應條件 95 ℃,4 min;94 ℃、45 s,52 ℃、1.5 min,72 ℃、1.5 min,35個循環;72 ℃、10 min;4 ℃保存。
1.2.4 瓊脂糖凝膠置備 稱取1.5 g 瓊脂糖 G-10于錐形瓶中,分別加入100 mL ddH2O、2 mL 50×TAE buffer,高火微波 4 min,將錐形瓶置于75 ℃水浴中5 min;再加入 10 μL GV Ⅰ,搖勻后靜置10 min,倒入凝膠模具中,放置 30 min 后使用。
1.2.5 電泳 取4 μL PCR產物點樣電泳,100 V,45 min。將凝膠放入凝膠成像系統拍照,得到電泳結果圖(圖1)。與Marker對比可知,改擴增產物大小接近 750 bp,符合預期。
1.2.6 測序及拼接 將PCR產物送往中美泰和生物技術(北京)有限公司進行雙向測序,將測序結果用CodenCode Aligner軟件對其剪切和拼接,得到完整序列。

2 結果分析
2.1 BLAST檢測
登陸NCBI,將拼接后的序列進行BLAST檢測,證明7種蒼耳屬植物的matK序列擴增及測序成功。
2.2 ClustalW校對
利用MEGA5.1軟件[11]對7種蒼耳屬植物進行ClustalW校對,校對后的序列繼續用MEGA5.1進行分子進化遺傳分析。
2.3 堿基構成分析
從表1可以看出,蒼耳屬植物matK序列的T、A、G、C堿基的變化范圍依次減小,分別為8.6%、7.9%、1.5%、0.3%。其中T堿基含量最高的是刺蒼耳,為37.8%,最低的是西方蒼耳,為29.2%;A堿基含量最高的是西方蒼耳,為37.2%,最低的是為巴西蒼耳、北美蒼耳,均為29.3%;G堿基含量最高的是蒼耳,為17.5%,最低的是賓州蒼耳,為16.0%;C堿基含量最高的是賓州蒼耳,為16.9%,最低的是西方蒼耳、蒼耳,均為16.6%。
2.4 基因位點分析
由MEGA5.1測算可知,蒼耳屬植物matK序列共有927個位點,其中保守位點有422個、變異位點505個,變異位點中單突變位點有19個(表2)。
2.5 遺傳距離分析
利用MEGA5.1軟件,采用Tajima-Nei模型計算遺傳距離,得到表3,可以看出,蒼耳屬植物matK序列的遺傳距離最高值為2.553 3,為蒼耳與美麗蒼耳的遺傳距離;最小值為0.000 0,為巴西蒼耳與北美蒼耳的距離。這說明雖然巴西蒼耳與北美蒼耳單從序列上看,于33、904、916位點存在有位點的插入缺失差異,但在Tajima-Nei模型中被忽略不計,所從遺傳距離上無法區分。
2.6 系統進化發育樹
利用MEGA5.1軟件,采用Neighbor-Joining方法構建系統發育樹(圖2),可以看出:蒼耳、西方蒼耳被分為一支;賓州蒼耳被分為一支;巴西蒼耳、北美蒼耳、刺蒼耳、美麗蒼耳被分為一支;對其進行bootstrap檢驗,其分組支持率為100%,說明這3支蒼耳屬植物在系統進化發育中是相互獨立的。巴西蒼耳、北美蒼耳、刺蒼耳、美麗蒼耳這一支又被劃分為不同級別的分支,但是其bootstrap檢驗支持率最高僅為47%,說明從植物matK序列進化層面講,這4種蒼耳屬植物在蒼耳屬系統進化發育中的獨立性相對較弱。

表1 蒼耳屬植物matK序列堿基構成Table 1 The matK sequence base composition of Xanthium plants
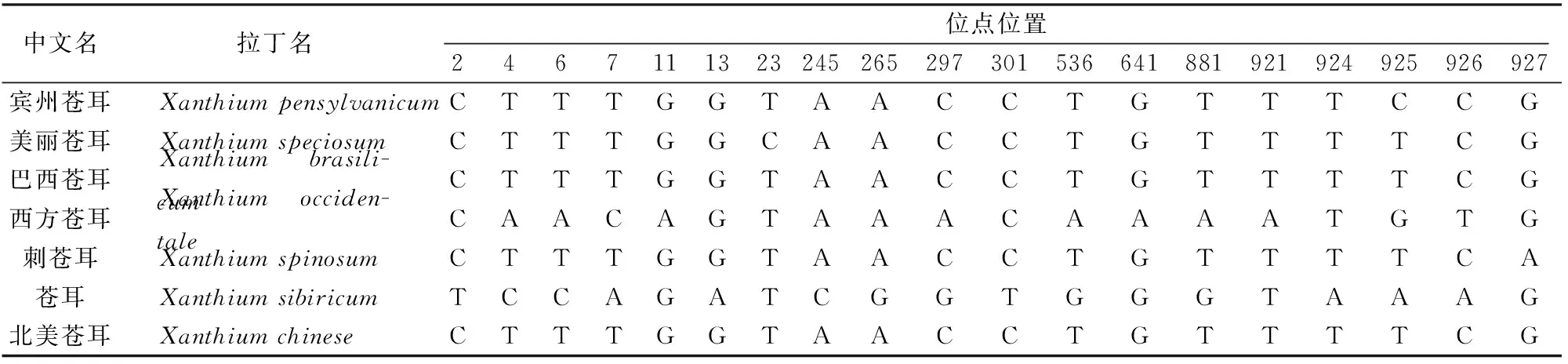
表2 蒼耳屬植物matK序列單突變位點Table 2 The matK sequence single mutations of Xanthium plants
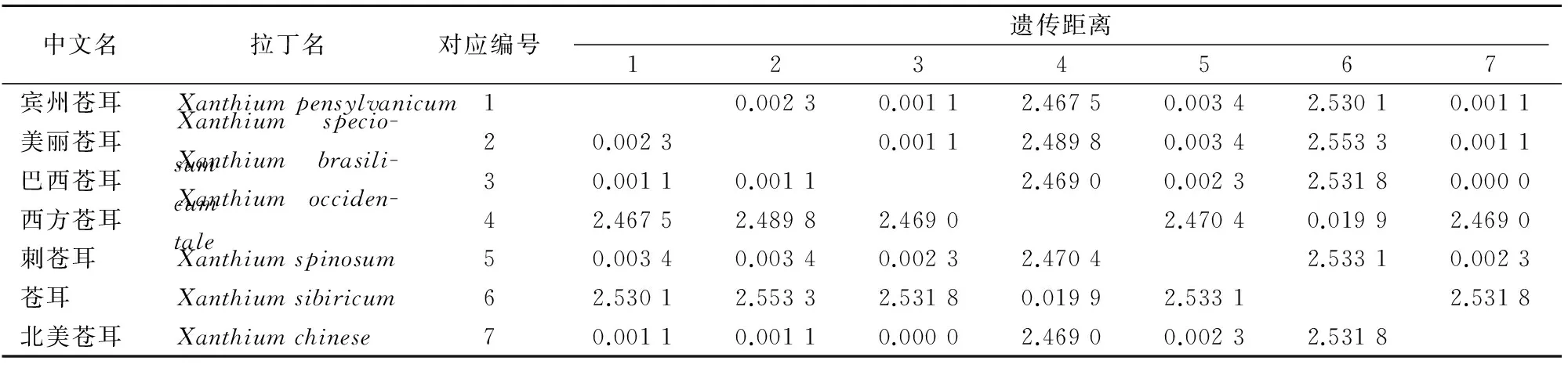
表3 蒼耳屬植物matK序列遺傳距離Table 3 The matK sequence genetic distance of Xanthium plant

3 討論
外來雜草對我國產生了嚴重危害[12],對外來雜草種子的鑒定工作一直以來都是進境糧谷檢驗檢疫工作的重點。但是由于雜草種子在糧谷的長期運輸中難免會受到磨損,從而不具備鑒定到物種的足夠信息,而進口貨物的檢驗檢疫周期又有一定的時間要求,這就使得外來雜草種子的鑒定工作成為糧谷檢驗檢疫工作的難點。解決這個問題亟需加強檢驗檢疫系統與各個科研院所的合作,引進新的科技手段。本試驗就是將DNA條形碼鑒定手段應用到來自世界各地的蒼耳屬植物中,從試驗結果來看,matK序列能從基因位點層面對蒼耳屬植物進行區分,但是也存在有以下不足:(1)巴西蒼耳與北美蒼耳在matK序列僅存在基因位點插入缺失的差異;(2)matK序列對巴西蒼耳、北美蒼耳、刺蒼耳、美麗蒼耳這一系統發育支系的繼續分化沒有提供較高的支持值。在植物DNA條形碼的研究中,trnH-psbA、rbcL、ITS、ITS2等基因序列也是研究工作的重點,或許對matK序列可以起到補充作用[13-14]。
[1]Tautz D,Arctander P,Minelli A,et al. A plea for DNA taxonomy[J]. Trends in Ecology and Evolution,2003,18(2):70-74.
[2]郭 銳,張守純,宋艷珠. 9種鶴類DNA條形碼的研究[J]. 中國農學通報,2012,28(14):50-55.
[3]姜 帆,劉佳琪,李志紅,等. 基于DNA條形碼的廣西苦瓜中實蠅幼蟲分子鑒定研究[J]. 植物保護,2011,37(4):150-153.
[4]李鵬飛,朱文斌,賀舟挺,等. 東海帶魚DNA條形碼的建立及COⅠ序列變異分析[J]. 浙江海洋學院學報:自然科學版,2013,32(1):6-9.
[5]Chase M W,Salamin N,Wilkinson M,et al. Land plants and DNA barcodes:short-term and long-term goals[J]. Philosophical Transactions of the Royal Society of London. Series B,Biological Sciences,2005,360(1462):1889-1895.
[6]任保青,陳之端. 植物DNA條形碼技術[J]. 植物學報,2010,45(1):1-12.
[7]張 欣,于瑞祥,方曉明,等. 橄欖油摻假檢測技術的研究進展[J]. 中國油脂,2013,38(3):67-71.
[8]龐曉慧,宋經元,陳士林. 應用DNA條形碼技術鑒定中藥材燈心草[J]. 中國中藥雜志,2012,37(8):1097-1099.
[9]Hollingsworth P M,Forrest L L,Spouge J L,et al. A DNA barcode for land plants[J]. Proceedings of the National Academy of Sciences of the United States of America,2009,106(31):12794-12797.
[10]高連明,劉 杰,蔡 杰,等. 關于植物DNA條形碼技術研究規范[J]. 植物分類與資源學報,2012,34(6):592-606.
[11]Tamura K,Peterson D,Peterson N,et al. MEGA5:molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods[J]. Molecular Biology and Evolution,2011,28(10):2731-2739.
[12]強 勝,曹學章. 外來雜草在我國的危害性及其管理對策[J]. 生物多樣性,2001,9(2):188-195.
[13]劉宇婧,劉 越,黃耀江,等. 植物DNA條形碼技術的發展及應用[J]. 植物資源與環境學報,2011,20(1):74-82,93.
[14]Kress W J,Erickson D L. DNA barcodes:genes,genomics,and bioinformatics[J]. Proceedings of the National Academy of Sciences of the United States of America,2008,105(8):2761-2762.
TheapplicationofmatKsequenceasDNAbarcodeinXanthium
HU Wei-yi1,SHAO Tao-xi2
(1.Lianyungang Entry-Exit Inspection and Quarantine Bureau,Lianyungang 222042,China;2.Ganyu Tashan Agricultural Technology Service Center,Ganyu222042,China)
MatK sequence was used as DNA barcode to distinguish and indentify seven plant species in the genusXanthiumof foreign origin. DNeasy? Plant Mini Kit was used to extract the total DNA. A universal primer was used to amplify matK genes for sequencing. The matK sequences of the sevenXanthiumspecies were contrasted and analyzed by MEGA 5.1 software,and a phylogenetic tree was constructed. The matK sequences were able to distinguish and indentifyXanthiumplants at the gene level.
matK sequence;DNA barcode;Xanthium
S41-30
A
1003-935X(2013)04-0013-04
胡偉毅,邵套喜. matK序列作為DNA條形碼在蒼耳屬中的應用[J]. 雜草科學,2013,31(4):13-16.
2013-09-13
江蘇出入境檢驗檢疫局科技項目(編號:2012KJ54)。
胡偉毅(1984—),男,碩士,主要從事港口外來有害生物截獲工作。E-mail:94087540@qq.com。

