菲律賓蛤仔脂多糖誘導的腫瘤壞死因子α VpLITAF基因克隆及其對微生物侵染的應答研究*
張林寶 孫 偉 寧璇璇 蔡文貴 張 喆 陳海剛 馬勝偉 賈曉平①
(1.中國水產科學研究院南海水產研究所 農業部南海漁業資源開發利用重點實驗室 廣東省漁業生態環境重點實驗室廣州 510300;2.國家海洋局煙臺海洋環境監測中心站 煙臺 264003)
細胞因子(Cytokine)是多種細胞分泌的能調節細胞生長分化、調節免疫功能、參與炎癥反應和創傷愈合的小分子多肽,它們是細胞和體液免疫反應不可缺少的介質(Kuby,1997)。無脊椎動物中關于細胞因子樣物質的研究主要集中在白介素和腫瘤壞死因子TNF-α(Tumor necrosis factor alpha)上。TNF-α 作為前炎性細胞因子介導炎癥反應和免疫調節反應(Bazzoniet al,1996),能直接導致腫瘤細胞死亡并參與機體內炎癥和免疫應答調節(García-Castilloet al,2002)。脂多糖(Lipopolysaccharide,LPS)誘導的腫瘤壞死因子α LITAF(LPS-induced TNF a factor)是 TNF-α 重要的轉錄因子,在調節 LPS誘導 TNF-α的表達過程中起著非常重要的作用(Tanget al,2003)。LITAF在脊椎動物和無脊椎動物中均被分離出來(Bolcato-Belleminet al,2004;Honget al,2006;Yuet al,2007;Parket al,2008;Zhanget al,2009),并證實其可以特異結合人TNF-α啟動子上位于-515到-511位的 CTCCC序列(Tanget al,2003),在受到LPS刺激后與STAT6B蛋白形成復合體進入細胞核調控TNF-α的表達(Tanget al,2005)。
迄今為止,在扇貝、牡蠣等貝類中相繼報道了LITAF的基因克隆與功能研究(Yuet al,2007;Parket al,2008;Zhanget al,2009;赫崇波等,2012;Liet al,2012;Yanget al,2012;Yuet al,2012)。然而,有關菲律賓蛤仔(Venerupis philippinarum)LITAF基因的功能研究則未見報道。菲律賓蛤仔作為我國海水養殖業的重要經濟貝類,年產量高達180萬噸,是沿海居民最常食用的海產品之一(Zhanget al,2006)。但是,近年來頻發的病害問題嚴重制約了蛤仔養殖業的健康發展,因此開展菲律賓蛤仔抗病及免疫相關基因的研究是非常重要和十分緊迫的。本研究在已構建的cDNA文庫和大規模EST(expressed sequence tags,表達序列標簽)分析的基礎上,采用RACE(Rapid Amplification of cDNA Ends,cDNA末端快速擴增技術)等方法克隆菲律賓蛤仔LITAF基因序列,用生物信息學方法對基因和推測的蛋白進行結構分析和功能預測,同時通過實時熒光定量PCR技術分析該基因在蛤仔不同組織以及受到不同微生物刺激后的表達情況。研究旨在深入了解菲律賓蛤仔的免疫抗病機制,為其遺傳改良和抗病品系選育工作提供參考資料。
1 材料與方法
1.1 實驗樣品及處理
實驗所用菲律賓蛤仔購自廣州市黃沙水產市場,選取殼長為3—4cm個體進行實驗。正式實驗開始前,在過濾海水中馴養一周,每日定時投喂小球藻,水溫控制在20—22°C。微生物侵染實驗中,設置對照組和鰻弧菌(Vibrio anguillarum)、藤黃微球菌(Micrococcus luteus)刺激組。鰻弧菌作為本研究中革蘭氏陰性菌的代表,廣泛存在于養殖環境,是海水魚、蝦、貝類的常見細菌性病原(Egidius,1987)。藤黃微球菌是一種典型的革蘭氏陽性菌,屬條件致病菌,目前有文獻報道其在水體以及水生生物體內都有分布(Spanggaardet al,2001;彭彬等,2011)。對照組不做任何處理,隨機取6只蛤仔,采集閉殼肌、外套膜、肝胰腺、鰓和血淋巴細胞五種組織。侵染組菲律賓蛤仔分別浸泡于含有 1×107CFU/mL鰻弧菌和藤黃微球菌的海水中,于6、12、24、48 h每組隨機采集6只蛤仔血淋巴樣品。血淋巴樣品于4°C,3000r/min離心5 min收集血淋巴細胞,加入1mL Trizol(Invitrogen公司)后置于超低溫冰箱,其余組織放入預先裝有1mL Trizol的離心管中充分研磨,在4°C,5000r/min離心5min,取上清置于超低溫冰箱中備用。
1.2 方法
1.2.1 菲律賓蛤仔LITAF基因的克隆 采用Trizol提取菲律賓蛤仔各組織以及鰻弧菌和藤黃微球菌侵染后的血淋巴細胞總RNA,用Dnase I(Promega公司)消化殘留DNA,然后用M-MLV(Promega公司)反轉錄合成第一鏈cDNA。根據本實驗室已構建的菲律賓蛤仔cDNA文庫獲得LITAFEST序列,并分別設計3′和5′ Race特異性引物(表1),以反轉錄的cDNA為模板,按 Race試劑盒(Clonetech公司)說明步驟分別擴增該基因的 3′和 5′端。PCR 產物經純化后連接到pMD18-T載體上(TaKaRa公司),轉化大腸桿菌TOP10F感受態,PCR檢測陽性克隆后測序。
1.2.2 菲律賓蛤仔VpLITAF基因生物信息學分析序列同源性比對和相似性搜索利用 NCBI在線軟件BLAST進行分析(http://www.ncbi.nlm.nih.gov/blast),多序列比對采用 CLUSTAL W 多重比對程序(http://www.ebi.ac.uk/clustalw/);信號肽查找用 SingalP 3.0 Server 軟件(http://www.cbs.dtu.dk/services/ SignalP/);同時運用 MEGA 5.0軟件包(Tamuraet al,2011)的Neighbor-joining法構建進化樹(Saitouet al,1987)。
1.2.3 菲律賓蛤仔VpLITAF基因表達分析 采用PCR Sybrgreen Master Mix(TaKaRa)試劑盒,使用ABI公司7500Fast型熒光定量 PCR儀分析VpLITAF基因的組織分布特征以及在鰻弧菌和藤黃微球菌感染下的時序表達規律。根據擴增獲得的VpLITAF基因序列,設計熒光定量引物VpLITAF-QF和VpLITAF-QR,以β-actin為內參照(引物信息見表1),采用相對定量方法檢測VpLITAF基因的表達特征。PCR反應條件:第一步:50°C 2min,第二步:95°C 10min,第三步:95°C 15s,60°C 1min,共40個循環,反應完成后進行熔解曲線分析:95°C 15s,60°C 20s,然后緩慢升溫至95°C,連續記錄熒光信號的變化,將溫度的變化與熒光信號的變化求負倒數后對溫度作圖,可得到產物的Tm值。實驗過程中,每個反應均設3個重復,并在反應結束后進行熔解曲線分析確保產物的特異性。數據處理首先用β-actin作為內標基因對VpLITAF基因表達量進行標準化,計算ΔCT值。組織分布研究將閉殼肌組織的樣品作為參照因子(calibrator),其倍數變化為1,對于其它組織樣本,目標基因表達差異相對于參照因子基因表達的倍數為2–ΔΔCT。菌刺激樣品VpLITAF基因相對表達水平用微生物處理組的表達量與海水對照組表達量的比值來計算(Zhanget al,2011)。所得數據采用SPSS 13.0軟件包進行t-test統計分析。
2 結果
2.1 菲律賓蛤仔VpLITAF基因序列分析
通過5′和3′ RACE方法獲得菲律賓蛤仔VpLITAF基因 cDNA序列(GenBank注冊號 HQ174259),如圖1 所示。該cDNA序列長為725 bp,含有393 bp的開放閱讀框序列(Open Reading Frame,ORF),編碼130個氨基酸殘基,該多肽的理論分子量為 14.39 kDa,等電點為 7.47。VpLITAFcDNA 序列 5′非編碼區(5′-UTR)為 23 bp,3′非編碼區(3′-UTR)為 309 bp,包含一個加尾信號(AATAAA)和典型的 polyA尾巴。利用SignalP軟件分析發現VpLITAF不具有信號肽序列。
2.2 VpLITAF同源性分析與進化分析
經BLASTP比對發現VpLITAF基因編碼的蛋白與文蛤(Meretrix meretrix)和竹蟶(Solen grandis)LITAF蛋白同源性最高,分別為96%和84%,與櫛孔扇貝(Chlamys farreri)、太平洋牡蠣(Crassostrea gigas)、中華圓田螺(Cipangopaludina chinensis)和鮑(Haliotis discus discus)等貝類 LITAF也有 55%—67%的一致性。利用SMART軟件(http://cn.expasy.org)分析發現,VpLITAF蛋白C端含有一個保守的LITAF結構域和兩個保守的 CXXC基序。多序列比對發現在該保守結構域內含有 8個保守的半胱氨酸殘基(圖1,圖2),推測這些保守的半胱氨酸殘基可能在維持蛋白的高級結構中發揮著重要作用。采用 Neighbor-joining方法構建系統進化樹,發現 LITAF蛋白首先分為脊椎動物和無脊椎動物兩大類,菲律賓蛤仔 VpLITAF與海洋軟體動物LITAF聚類在一起,然后再與魚類、鳥類及哺乳動物的 LITAF蛋白聚在一起,這與生物進化的遠近關系基本一致(圖3)。
2.3 VpLITAF基因在菲律賓蛤仔組織中的分布特征
以β-actin為對照,利用實時熒光定量PCR檢測VpLITAFmRNA在菲律賓蛤仔鰓、肝胰腺、外套膜、血淋巴細胞和閉殼肌 5種正常組織中的表達情況(圖4);結果顯示,VpLITAFmRNA在5種組織中均可見不同豐度的表達。VpLITAF在肌肉中的表達量最低,血淋巴細胞、外套膜和鰓中VpLITAF的表達量相對較高,分別是閉殼肌組織中表達量的19.89、29.56和82.28倍。VpLITAF基因在菲律賓蛤仔肝胰腺中的表達量最高,為閉殼肌組織中表達量的315.90倍。

圖1 菲律賓蛤仔VpLITAF基因核苷酸及推導的氨基酸序列Fig.1 The complete nucleotide and deduced amino acid sequence of VpLITAF
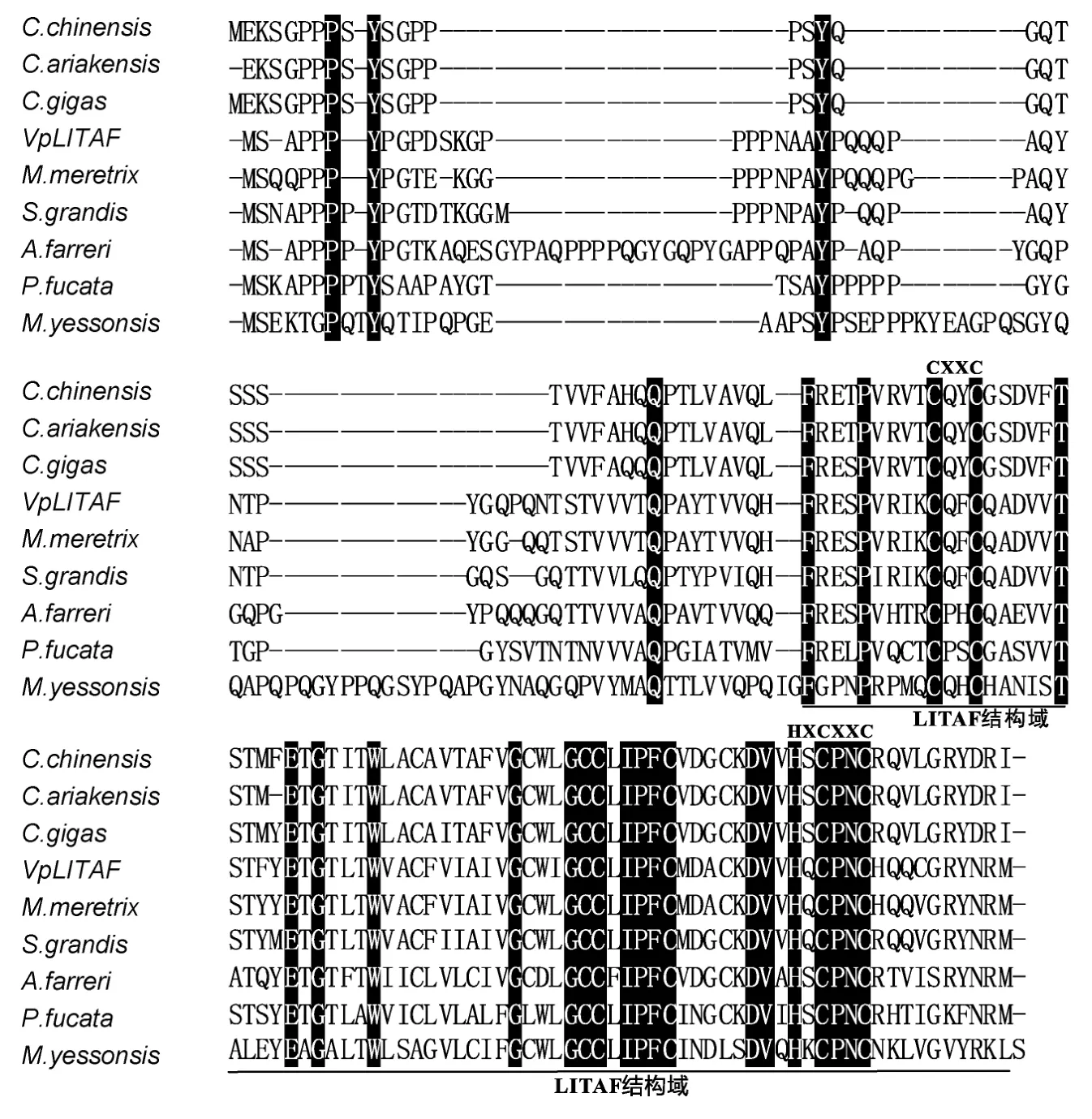
圖2 VpLITAF與其它物種LITAF氨基酸序列的多序列比對Fig.2 Multiple sequence alignment of VpLITAF with LITAFs from other organisms

圖3 不同物種LITAF進化樹Fig.3 Phylogenetic tree of LITAF sequences from different organisms

圖4 VpLITAF在菲律賓蛤仔各組織中的相對表達情況Fig.4 Distribution of VpLITAF mRNA in different tissues of V.philippinarum
2.4 菌刺激后菲律賓蛤仔VpLITAF基因的時序表達特征
利用熒光定量 PCR方法,分析菲律賓蛤仔VpLITAF基因 mRNA在鰻弧菌和藤黃微球菌刺激后的時序表達情況(圖5)。其中,對照組樣品VpLITAF表達量在所檢測的時間范圍內未出現明顯變化。鰻弧菌感染組菲律賓蛤仔VpLITAF基因表達量不斷變化,12h和48h的表達量有著顯著增加,分別為對照組的2.1倍和3.5倍。藤黃微球菌感染24h以內,VpLITAF基因 mRNA表達量有所升高,但與對照組表達水平并無顯著差異。隨著感染時間的延長,VpLITAF基因表達水平在48h被鰻弧菌顯著誘導,表達量為對照組的1.9倍。
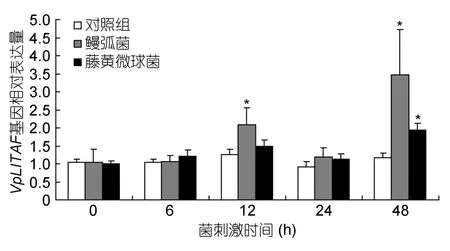
圖5 菌刺激后菲律賓蛤仔血淋巴細胞中VpLITAF的時序表達特征Fig.5 Temporal expression of VpLITAF in haemocytes of V.philippinarum after bacterial infection
3 討論
LITAF是一種重要的轉錄因子,在調節LPS誘導炎癥因子TNF-α的表達過程中起著非常重要的作用。迄今為止,在扇貝、牡蠣、文蛤和中華圓田螺等貝類中相繼報道了LITAF基因(Yuet al,2007;Liet al,2012;Yuet al,2012)。然而有關菲律賓蛤仔LITAF基因的相關研究則未見報道。本研究首次克隆了蛤仔的LITAF基因cDNA序列,并對其免疫調節作用進行了初步研究。
比對分析發現,VpLITAF氨基酸序列與其它貝類LITAF具有較高的相似性(Yuet al,2007,2012;Liet al,2012),都具有高度保守的LITAF結構域。LITAF結構域由N端的CXXC區、25個氨基酸長的疏水區和C端的(H)XCXXC區組成,N端和C端的CXXC基序結合在一起,形成緊密結合的 Zn2+結構(Pontinget al,2001),介導LITAF與DNA之間的結合啟動相關基因轉錄(Yuet al,2012)。系統進化分析發現,VpLITAF首先與貝類 LITAF聚為一簇,然后與各類脊椎動物 LITAF聚在一起,這與物種進化的發生順序基本一致。綜上所述,同源比對、結構域和進化分析均表明菲律賓蛤仔VpLITAF為LITAF家族一員。
研究表明LITAF基因主要表達于脊椎動物外周血細胞、淋巴結和脾等淋巴組織中,在非淋巴器官中也有不同程度的表達(Bolcato-Belleminet al,2004;Tanget al,2005;Honget al,2006)。本研究中VpLITAF在菲律賓蛤仔所檢測組織中均有表達,其中肝胰腺的表達量最高(圖4),這與LITAF基因在中華圓田螺和太平洋牡蠣中的組織分布情況類似(Yanget al,2012;Yuet al,2012)。牡蠣全基因組研究揭示許多免疫相關基因高度表達于消化腺中(Zhanget al,2012),這表明消化系統與血淋巴細胞一樣是軟體動物對抗、清除病原體的重要一線防御器官(江靜波,1982;Parket al,2008;Zhanget al,2009),因此推測菲律賓蛤仔肝胰腺中高表達的 VpLITAF在機體免疫反應中發揮著重要作用。然而,櫛孔扇貝和文蛤LITAF的組織分布情況則有所不同,二者的最高表達量分別發生在性腺和鰓組織中(Yuet al,2007;Liet al,2012),表明LITAF基因組織分布特征隨物種的不同而不同。
雖然本研究中VpLITAF在蛤仔血淋巴細胞中的表達量相較于其它組織來說偏低,然而血淋巴細胞在貝類固有免疫系統中發揮著重要的作用(王信超等,2012;劉世良等,2003),經常作為研究免疫相關基因的組織材料(Zhanget al,2011;Liet al,2012),因此本研究選取血淋巴細胞研究VpLITAF基因表達與微生物感染的相關性。研究結果表明鰻弧菌和藤黃微球菌侵染在某些時段均可顯著誘導VpLITAF基因的表達水平(圖5),類似的結論在太平洋牡蠣、蝦夷扇貝、文蛤和珍珠貝中也有發現(Parket al,2008;Zhanget al,2009;赫崇波等,2012;Liet al,2012),表明LITAF參與了機體的免疫調節過程。鰻弧菌和藤黃微球菌侵染24 h,VpLITAF基因表達水平與12 h相比有所回落,這可能是因為隨著微生物侵染時間的延長,蛤仔血淋巴系統中產生 VpLITAF的血細胞數量減少或轉移至微生物增殖的其它組織中,如外套膜、消化腺和鰓等。微生物感染下,類似的誘導-降低-再誘導的表達模式在貝類其它免疫相關基因的表達分析中也有發現,如盤鮑(Haliotis discus discus)抗菌肽(De Zoysaet al,2010)和菲律賓蛤仔谷胱甘肽過氧化物酶(Zhanget al,2011)。另外,本研究中鰻弧菌對VpLITAF基因表達的誘導作用明顯強于藤黃微球菌。鰻弧菌是一種革蘭氏陰性菌,其細胞外膜的主要結構成分為LPS ,目前已有諸多研究表明LPS可顯著誘導LITAF的轉錄水平(Bolcato-Belleminet al,2004;Honget al,2006;Yuet al,2007,2012)。藤黃微球菌是一種革蘭氏陽性菌,其細胞壁主要結構成分為肽聚糖(peptidoglycan,PGN),研究表明PGN對櫛孔扇貝LITAF基因表達無顯著誘導作用(Yuet al,2007)。因此,不難理解菲律賓蛤仔VpLITAF基因表達對鰻弧菌感染更為敏感。藤黃微球菌侵染蛤仔48 h后,VpLITAF基因被顯著誘導的原因需進一步深入研究。總之,VpLITAF基因克隆與表達分析表明 VpLITAF參與了菲律賓蛤仔的先天性免疫反應,這為進一步研究蛤仔分子水平的免疫防御機制研究提供了新的基礎資料。
王信超,孫敬敬,范美華等,2012.厚殼貽貝血細胞的蛋白質組學分析.南方水產科學,8(2):7—14
劉世良,麥康森,2003.貝類免疫系統和機理的研究進展.海洋學報,25(2):95—105
江靜波,1982.無脊椎動物學.北京:高等教育出版社
彭 彬,楊光友,陳曉利等,2011.黃鱔藤黃微球菌的分離鑒定及藥敏實驗.上海海洋大學學報,20(3):405—411
赫崇波,朱 寶,劉衛東等,2012.蝦夷扇貝脂多糖誘導的腫瘤壞死因子 LITAF基因的克隆及表達分析.遺傳,34(6):736—741
Bazzoni F,Beutler B,1996.The tumor necrosis factor ligand and receptor families.N Engl J Med,334(26):1717—1725
Bolcato-Bellemin A L,Mattei M G,Fenton Met al,2004.Molecular cloning and characterization of mouse LITAF cDNA:role in the regulation of tumor necrosis factor-alpha(TNF-alpha)gene expression.J Endotoxin Res,10(1):15—23
De Zoysa M D,Whang I,Lee Yet al,2010.Defensin from disk abaloneHaliotis discus discus:Molecular cloning,sequence characterization and immune response against bacterial infection.Fish Shellfish Immunol,28(2):261—266
Egidius E,1987.Vibriosis:pathogenicity and pathology,a review.Aquaculture,67:15—28
García-Castillo J,Pelegrín P,Mulero Vet al,2002.Molecular cloning and expression analysis of tumor necrosis factor α from a marine fish reveal its constitutive expression and ubiquitous nature.Immunogenetics,54(3):200—207
Hong Y H,Lillehoj H S,Lee S Het al,2006.Molecular cloning and characterization of chicken lipopolysaccharide-induced TNF-α factor(LITAF).Dev Comp Immunol,30(10):919—929
Kuby J,1997.Immune effect mechanisms,Immunology,3rd.W.H.Freeman and Company Press
Li H J,Yang Q,Gao X Get al,2012.Identification and expression of a putative LPS-induced TNF-α factor from Asiatic hard clamMeretrix meretrix.Mol Biol Rep,39(2):865—871
Park E M,Kim Y O,Nam B Het al,2008.Cloning,characterization and expression analysis of the gene for a putative lipopolysaccharide-induced TNF-alpha factor of the Pacific oyster,Crassostrea gigas.Fish Shellfish Immunol,24(1):11—17
Ponting C P,Mott R,Bork Pet al,2001.Novel protein domains and repeats inDrosophila melanogaster:insights into structure,function,and evolution.Genome Res,11(12):1996—2008
Saitou N,Nei M,1987.The neighbor-joining method:a new method for reconstructing phylogenetic trees.Mol Bio Evol,4(4):406—425
Spanggaard B,Huber I,Nielsen E Bet al,2001.The probiotic potential against vibriosis of the indigenous microflora of rainbow trout.Environ Microbiol,3(12):755—765
Tamura K,Peterson D,Peterson Net al,2011.MEGA5:Molecular evolutionary genetics analysis using maximum likelihood,evolutionary distance,and maximum parsimony methods.Mol Biol Evol,28(10):2731—2739
Tang X R,Fenton M J,Amar S,2003.Identification and functional characterization of a novel binding site on TNF-α promoter.Proc Natl Acad Sci USA,100(7):4096—4101
Tang X R,Marciano D L,Leeman S Eet al,2005.LPS induces the interaction of a transcription factor,LPS-induced TNF alpha factor,and STAT6(B)with effects on multiple cytokines.Proc Natl Acad Sci USA,102(14):5132—5137
Yang S,Li P,Mi Z,2012.LPS-induced TNFa factor(LITAF)in the snailCipangopaludina chinensis:gene cloning and its apoptotic effect on NCI-H446 cells.Fish Shellfish Immunol,32(2):268—272
Yu F,Zhang Y,Yu Z,2012.Characteristics and expression patterns of the lipopolysaccharide-induced TNF-α factor(LITAF)gene family in the Pacific oyster,Crassostrea gigas.Fish Shellfish Immunol,33(4):899—908
Yu Y D,Qiu L M,Song L Set al,2007.Molecular cloning and characterization of a putative lipopolysaccharide-induced TNF-alpha factor(LITAF)gene homologue from Zhikong scallopChlamys farreri.Fish Shellfish Immunol,23(2):419—429
Zhang D C,Jiang J J,Jiang S Get al,2009.Molecular characterization and expression analysis of a putative LPS-induced TNF-alpha factor(LITAF)from pearl oysterPinctada fucata.Fish Shellfish Immunol,27(3):391—396
Zhang G F,Yan X W,2006.A new three-phase culture method for Manila clam,Ruditapes philippinarum,farming in northern China.Aquaculture,258(1—4):452—461
Zhang G,Fang X,Guo Xet al,2012.The oyster genome reveals stress adaptation and complexity of shell formation.Nature,490:49—54
Zhang L,Liu X,Chen Let al,2011.Transcriptional regulation of selenium-dependent glutathione peroxidase fromVenerupis philippinarumin response to pathogen and contaminants challenge.Fish Shellfish Immunol,31(6):831—837

