基因組轉(zhuǎn)錄調(diào)控元件分析方法研究進(jìn)展
李文茂賈東方許嶸,2
(1.華僑大學(xué)生物醫(yī)學(xué)學(xué)院,泉州 362021;2.泉州醫(yī)學(xué)高等專科學(xué)校,泉州 362100)
基因組轉(zhuǎn)錄調(diào)控元件分析方法研究進(jìn)展
李文茂1賈東方1許嶸1,2
(1.華僑大學(xué)生物醫(yī)學(xué)學(xué)院,泉州 362021;2.泉州醫(yī)學(xué)高等專科學(xué)校,泉州 362100)
真核生物的基因表達(dá)是一個(gè)非常復(fù)雜的過程,需要多種轉(zhuǎn)錄調(diào)控元件的協(xié)同作用精確調(diào)控。全基因組轉(zhuǎn)錄調(diào)控元件的篩選和鑒定對(duì)于基因表達(dá)調(diào)控機(jī)制和生物學(xué)功能研究具有重要意義。但轉(zhuǎn)錄調(diào)控元件的高效篩選及功能驗(yàn)證仍是研究人員面臨的主要挑戰(zhàn)。結(jié)合近年來轉(zhuǎn)錄調(diào)控元件的研究成果,對(duì)基因組中轉(zhuǎn)錄調(diào)控元件的預(yù)測(cè)、篩選及功能驗(yàn)證方法做一綜述。
轉(zhuǎn)錄調(diào)控元件 高通量篩選 標(biāo)志物
諸多生物學(xué)功能的實(shí)現(xiàn)取決于基因在時(shí)間和空間上的特異性表達(dá)。真核生物的基因表達(dá)調(diào)控是一個(gè)極其復(fù)雜的過程,需要一系列轉(zhuǎn)錄調(diào)控因子和DNA序列精確地相互作用。參與基因轉(zhuǎn)錄調(diào)控的DNA序列稱之為轉(zhuǎn)錄調(diào)控元件(Transcriptional regulatory elements,TREs),主要包括啟動(dòng)子、增強(qiáng)子、絕緣子等。據(jù)估算,哺乳動(dòng)物基因組中約有5%的DNA序列含有編碼信息,但僅有1.5%的序列能夠編碼蛋白質(zhì)[1]。最近的研究表明,許多疾病的發(fā)生與轉(zhuǎn)錄調(diào)控元件的突變相關(guān),因此,這些非編碼轉(zhuǎn)錄調(diào)控元件的篩選及功能探索對(duì)基因表達(dá)調(diào)控機(jī)制和生物學(xué)功能研究具有重要意義[2]。
如何對(duì)基因組中的TRE進(jìn)行準(zhǔn)確預(yù)測(cè),將是研究人員面臨的一個(gè)重大挑戰(zhàn)。與固定于基因5'端的啟動(dòng)子不同,一些調(diào)控元件在基因組中位置不固定,如增強(qiáng)子,可以通過遠(yuǎn)程相互作用與遠(yuǎn)端啟動(dòng)子相互作用,調(diào)控基因表達(dá)。對(duì)于轉(zhuǎn)錄調(diào)控元件的篩選,傳統(tǒng)的方法是通過比較基因組學(xué)尋找物種間同源非編碼DNA保守序列。近幾年,隨著基因芯片技術(shù)和高通量測(cè)序技術(shù)的發(fā)展,通過檢測(cè)轉(zhuǎn)錄調(diào)控元件的相關(guān)表觀修飾物,可以準(zhǔn)確、快捷的對(duì)TRE進(jìn)行分析和定位。本研究對(duì)近幾年TRE元件的表觀標(biāo)志物研究及篩選方法進(jìn)行分析、比較、探索,旨在為相關(guān)研究提供參考以及思路。
1 轉(zhuǎn)錄調(diào)控元件指示性表觀修飾物
在基因轉(zhuǎn)錄調(diào)控過程中,除了轉(zhuǎn)錄調(diào)控元件及
轉(zhuǎn)錄調(diào)控因子外,還存在許多表觀修飾信息影響基因表達(dá)調(diào)控。這些表觀調(diào)控信息主要包括:組蛋白修飾、染色質(zhì)重塑和核小體變形等。組蛋白修飾是其中表觀遺傳調(diào)控最重要的內(nèi)容。在染色質(zhì)中組蛋白N末端容易受到修飾酶的甲基化、乙酰化等共價(jià)修飾。這種組蛋白修飾往往對(duì)基因的表達(dá)具有很大影響[3]。不同的TRE元件具有不同的組蛋白修飾模式,且多種組蛋白形成修飾組合協(xié)同影響基因的表達(dá)。不同的轉(zhuǎn)錄調(diào)控元件具有不同的表觀修飾標(biāo)志物,根據(jù)這些標(biāo)志物可以對(duì)相關(guān)調(diào)控元件在基因組中的位點(diǎn)進(jìn)行預(yù)測(cè)和定位(表1)。
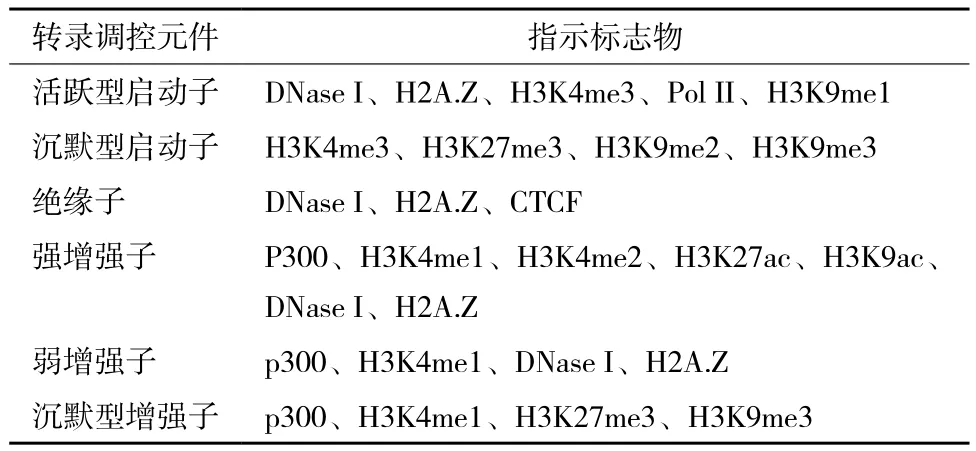
表 1 TRE相關(guān)指示標(biāo)志物
1.1 啟動(dòng)子
研究顯示組蛋白3賴氨酸4三甲基化(H3K4-me3)主要分布于啟動(dòng)子轉(zhuǎn)錄起始位點(diǎn)(Transcription strartsite,TSS)周圍,同時(shí)在增強(qiáng)子區(qū)域也存在[4]。在對(duì)胚胎干細(xì)胞(Embryonicstem cell,ES)和分化細(xì)胞的染色質(zhì)H3K4me3修飾研究時(shí)發(fā)現(xiàn),H3K4me3廣泛存在于所有類型的啟動(dòng)子中,與啟動(dòng)子元件存在線性相關(guān)性[5,6]。此外,除H3K4me3修飾外,啟動(dòng)子元件還存在一些其他表觀特征,包括組蛋白乙酰化、H3.3組蛋白變體、DNase I超敏性(DNase I hypersensitivity)及Pol II(RNA-polymerase II)聚合酶結(jié)合位點(diǎn)等[7-10]。
不同活性的啟動(dòng)子元件,其表觀修飾特征也存在一定的差異。一些處于抑制狀態(tài)的啟動(dòng)子元件也具有獨(dú)特的染色質(zhì)修飾特征,如組蛋白3賴氨酸27(H3K27)甲基化、組蛋白3賴氨酸9(H3K9)甲基化等。H3K27三甲基化(H3K27me3)是一種多梳蛋白抑制物,H3K27me3在沉默型啟動(dòng)子中分布水平要比在活躍啟動(dòng)子高得多[11-14]。但研究顯示,H3K27me1主要分別在活躍型啟動(dòng)子中[14]。H3K9的甲基化主要參與異染色質(zhì)的形成和基因表達(dá)沉默[15]。研究表明,H3K9雙甲基化和三甲基化(H3K9me2 和H3K9me3)主要分布在沉默型啟動(dòng)子的TSS周圍,而單甲基化(H3K9me1)卻分布于活躍型啟動(dòng)子TSS的周圍[14]。
1.2 絕緣子元件
絕緣子是基因組中阻止增強(qiáng)子活性和異染色質(zhì)延伸的一種DNA元件。在哺乳動(dòng)物中,轉(zhuǎn)錄抑制因子CTCF一種廣泛存在于脊椎動(dòng)物中的鋅指蛋白,對(duì)絕緣子功能發(fā)揮的關(guān)鍵調(diào)控因子,且在染色質(zhì)環(huán)狀結(jié)構(gòu)的形成和穩(wěn)定方面具有重要作用[16]。在不同的細(xì)胞系中,CTCF(CCCTC-binding factor)結(jié)合位點(diǎn)具有相同的序列,因此CTCF具有廣泛性,可用于絕緣子元件的篩選[14,17]。
1.3 增強(qiáng)子元件
1.3.1 P300 P300是一種乙酰轉(zhuǎn)移酶,它與某些特異性轉(zhuǎn)錄因子,如CREB結(jié)合,可以使組蛋白發(fā)生乙酰化進(jìn)而激活基因的轉(zhuǎn)錄[18]。Heintzman等[19]利用ChIP-chip方法對(duì)人基因組中30 Mb區(qū)域進(jìn)行組蛋白修飾等相關(guān)研究時(shí)發(fā)現(xiàn),P300結(jié)合位點(diǎn)的分布情況與增強(qiáng)子在基因組中的分布模式相吻合,且大部分P300結(jié)合位點(diǎn)與DNase I位點(diǎn)和基因序列保守區(qū)域相重疊。這些證據(jù)表明P300和增強(qiáng)子功能之間可能存在著相關(guān)性。Visel等[20]運(yùn)用ChIP-seq技術(shù)在小鼠胚胎組織中進(jìn)行P300結(jié)合位點(diǎn)的進(jìn)一步篩選,將克隆的DNA片段進(jìn)行轉(zhuǎn)基因小鼠試驗(yàn),證實(shí)P300結(jié)合位點(diǎn)可精確的鑒定出增強(qiáng)子片段。相比于比較基因組學(xué)方法,P300結(jié)合位點(diǎn)可以明顯提高增強(qiáng)子預(yù)測(cè)的精確度,且可以預(yù)測(cè)出序列保守性較弱的增強(qiáng)子元件[20,21]。因此,P300是一種有效預(yù)測(cè)增強(qiáng)子的標(biāo)志物。
1.3.2 組蛋白修飾和增強(qiáng)子的分類 早期對(duì)增強(qiáng)子的組蛋白修飾物主要集中在組蛋白3賴氨酸4單甲基化修飾(H3K4me1)上。基因組范圍內(nèi)的H3K4me1分布模式研究發(fā)現(xiàn),H3K4me1的分布具有高度的細(xì)胞特異性,且主要分布于增強(qiáng)子元件區(qū)域,可用于增強(qiáng)子元件預(yù)測(cè)[19,22,23]。但研究顯示,單獨(dú)的H3K4me1修飾并不能有效預(yù)測(cè)所有增
強(qiáng)子元件,一些H3K4me1片段在報(bào)告基因驗(yàn)證中并不具有增強(qiáng)子活性[22]。在對(duì)鼠的胚胎干細(xì)胞及其他幾種分化細(xì)胞的全基因組分析發(fā)現(xiàn),另一種組蛋白修飾H3K27乙酰化(H3K27)與增強(qiáng)子功能元件存在很大相關(guān)性[24]。Rada等[25]研究發(fā)現(xiàn),在人胚胎干細(xì)胞中也發(fā)現(xiàn)了這種相關(guān)性。相比沒有發(fā)生H3K27ac修飾的增強(qiáng)子,H3K4me1+和H3K27ac+增強(qiáng)子片段顯然具有更強(qiáng)的基因表達(dá)調(diào)控活性[24,25]。 在 令H3K27ac-狀 態(tài) 的 增 強(qiáng) 子 獲 得H3K27ac修飾后則顯著增強(qiáng)調(diào)控基因的表達(dá)活性,而增強(qiáng)子元件去H3K27ac修飾或H3K4me1和H3K27ac雙修飾后,基因表達(dá)活性顯著下降。因此,H3K27ac修飾對(duì)增強(qiáng)子元件功能具有重要影響。Rada等[25]將H3K4me1+、H3K27ac+型增強(qiáng)子歸類為活躍型增強(qiáng)子元件,認(rèn)為這類元件與細(xì)胞特異性調(diào)控功能相關(guān)。而H3K4me1+、H3K27ac-型增強(qiáng)子可能與細(xì)胞分化功能相關(guān),在分化的某個(gè)過程中處于靜止?fàn)顟B(tài)等待激活[25]。
此外,在進(jìn)一步對(duì)增強(qiáng)子相關(guān)組蛋白標(biāo)志物研究時(shí)發(fā)現(xiàn),一部分沉默型增強(qiáng)子還含有一種甲基化抑制物(H3K27 me3或H3K9me3)。Zentner等[26]根據(jù)H3K4me1、H3K27ac、H3K27me3修飾及相關(guān)基因表達(dá)活性,將增強(qiáng)子元件分為3類:(1)H3K4me1+、H3K27ac+活躍型增強(qiáng)子,參與細(xì)胞特異性表達(dá)調(diào)控;(2)H3K4me1+、H3K27ac-活性較弱型增強(qiáng)子,它參與非特異性細(xì)胞功能調(diào)控;(3)H3K4me1+、H3K27me3+(H3K9me3+)沉默型增強(qiáng)子,該型增強(qiáng)子在發(fā)育過程中起作用。
Ernst等[27]還對(duì)增強(qiáng)子相關(guān)組蛋白標(biāo)志物進(jìn)行了進(jìn)一步的研究,他們對(duì)9種細(xì)胞全基因組范圍進(jìn)行9種組蛋白修飾物的高通量定位分析發(fā)現(xiàn),活躍型增強(qiáng)子分為兩類:一部分增強(qiáng)子含有明顯的 H3K4me1、H3K4me2、H3K4me3、H3K27ac 和H3K9ac修飾。另一部分則H3K4me1 和 H3K27ac信號(hào)很強(qiáng),但是H3K4me2 和 H3K9ac修飾相對(duì)較弱。而對(duì)于較弱型和沉默型增強(qiáng)子,H3K4me1的修飾水平較弱。同時(shí),該研究也表明,活躍型增強(qiáng)子與細(xì)胞特異性功能調(diào)控相關(guān)。
以H3K4me1和p300作為單一標(biāo)志物并不能判斷增強(qiáng)子的表達(dá)活性,因此對(duì)增強(qiáng)子進(jìn)行類型劃分非常有必要。通過組蛋白修飾模式,將增強(qiáng)子進(jìn)行分類,有助于對(duì)增強(qiáng)子類型以及增強(qiáng)子活性的預(yù)測(cè)和篩選。Ernst等[27]的研究發(fā)現(xiàn)H3K27me3+沉默型增強(qiáng)子比其他增強(qiáng)子序列上更保守,這也可能解釋了為什么大部分高度保守的序列在報(bào)告基因檢測(cè)中沒有活性。
1.4 轉(zhuǎn)錄調(diào)控元件的染色質(zhì)核小體變體特征
真核生物核小體由核心組蛋白八聚體和纏繞的DNA及組蛋白H1組成。含有H3變體H3.3和H2A變體H2A.Z的核小體很不穩(wěn)定,容易在中性鹽環(huán)境中被破壞。通過這些變體可以改變?nèi)旧|(zhì)的空間構(gòu)象和穩(wěn)定性,影響基因的轉(zhuǎn)錄。染色質(zhì)中基因轉(zhuǎn)錄調(diào)控區(qū)域普遍認(rèn)為是一段核小體退化的區(qū)域,主要分布在在啟動(dòng)子、增強(qiáng)子、絕緣子等區(qū)域[14,25]。
2 轉(zhuǎn)錄調(diào)控元件篩選方法
2.1 比較基因組學(xué)序列保守性分析
比較基因組學(xué)方法主要基于這樣的理論:基因組中許多功能序列由于凈化選擇保存下來,而非功能性片段突變對(duì)機(jī)體的健康的影響微不足道,使得這種突變可以漂移累積。隨著物種間進(jìn)化發(fā)生,發(fā)揮重要功能的DNA片段比非功能性片段具有很低的突變率而有序列保守性[28]。因此,同源基因間的序列保守性與序列的功能性之間存在一定的線性相關(guān)性[29]。通過算法分析出一定進(jìn)化距離的物種間同源基因的序列保守性,可用于轉(zhuǎn)錄調(diào)控元件在基因組中分布的預(yù)測(cè)及定位。已有多種基因組瀏覽器和軟件用于物種間保守非編碼序列篩選,如表2。

表 2 常用保守非編碼序列預(yù)測(cè)軟件
此外,多個(gè)研究組已成功應(yīng)用這種方法篩選到多種轉(zhuǎn)錄調(diào)控元件[30-33]。
比較基因組學(xué)借助計(jì)算機(jī)進(jìn)行算法分析,然后對(duì)調(diào)控元件進(jìn)行活性篩選,易于進(jìn)行。且隨著多個(gè)
物種基因組測(cè)序項(xiàng)目的完成,各物種的基因序列越來越容易獲取。因此比較基因組學(xué)日益成為調(diào)控元件預(yù)測(cè)和篩選的重要方法。但由于算法等技術(shù)限制,基于比較基因組學(xué)的方法并不能篩選出所有功能性調(diào)控元件。很多功能性元件并沒有呈現(xiàn)物種間序列保守性[34,35]。根據(jù)DNA元件百科全書(Encyclopedia of DNA elements,ENCODE)項(xiàng)目組公布的信息,現(xiàn)已證實(shí)約50%的TRE序列沒有顯示序列保守性[36]。此外,研究表明一些保守的非編碼序列并沒有生物學(xué)功能[33,37]。因此,單獨(dú)根據(jù)序列保守性篩選功能性調(diào)控元件的成功率較低,并不是一種理想的方法,但可作為功能性調(diào)控元件篩選的依據(jù)。
2.2 高通量篩選實(shí)驗(yàn)方法
2.2.1 DNase I超敏性和FAIRE DNase I超敏性常用于分析染色質(zhì)中的相對(duì)開放性區(qū)域。轉(zhuǎn)錄調(diào)控因子結(jié)合到DNA序列上需要形成相對(duì)開放的染色質(zhì)構(gòu)象,使得這些位點(diǎn)對(duì)于DNase I的切割具有非常高的敏感性。這些染色體開放區(qū)域常是一些DNA調(diào)控元件位點(diǎn),包括啟動(dòng)子、增強(qiáng)子、隔離子、絕緣子等。利用DNase I超敏性結(jié)合基因芯片技術(shù)(DNase-chip)或深度測(cè)序技術(shù)(DNase-Seq)可應(yīng)用于全基因組轉(zhuǎn)錄調(diào)控元件的篩選和鑒定[38-40]。與DNaseI超敏性不 同,F(xiàn)AIRE(formaldehyde-assisted identification of regulatory elements)方法則是利用甲醛對(duì)染色質(zhì)進(jìn)行交聯(lián),通過超聲波對(duì)染色質(zhì)開放區(qū)域進(jìn)行切割[41]。雖然DNase I超敏性和FAIRE不能用于確定具體的TRE類型,但與其他方法相結(jié)合可以有效提高TRE的篩選效率。
2.2.2 染色質(zhì)免疫共沉淀技術(shù)(Chromatin Immunoprecipitation,ChIP) 染色質(zhì)免疫共沉淀技術(shù)是在活細(xì)胞狀態(tài)下,通過甲醛交聯(lián)蛋白質(zhì)和DNA,并將其隨機(jī)切割成小片段。通過某種轉(zhuǎn)錄因子的特異性抗體富集與目的蛋白結(jié)合的DNA片段。傳統(tǒng)的方法是PCR分析富集的DNA片段,該方法很難實(shí)現(xiàn)全基因組范圍的高通量研究。Ren等[42]將ChIP技術(shù)和基因芯片技術(shù)結(jié)合(ChIP-chip),實(shí)現(xiàn)全基因組范圍轉(zhuǎn)錄因子的定位和功能分析。近幾年來高通量測(cè)序技術(shù)的發(fā)展,結(jié)合ChIP技術(shù)發(fā)展的ChIP-seq技術(shù)被廣泛用于全基因組轉(zhuǎn)錄調(diào)控因子的研究。
基于ChIP-chip/seq等高通量方法可以全基因組范圍定位轉(zhuǎn)錄調(diào)控因子的結(jié)合位點(diǎn),因此被廣泛應(yīng)用于轉(zhuǎn)錄調(diào)控元件的高通量篩選。如通過ChIP-seq技術(shù)繪制全基因組轉(zhuǎn)錄因子CTCF結(jié)合位點(diǎn),用于篩選絕緣子元件[17]。但ChIP技術(shù)需要針對(duì)每種轉(zhuǎn)錄因子的特異性抗體,理論上若在基因組范圍內(nèi)篩選所有的TRE元件需要制備針對(duì)每種轉(zhuǎn)錄因子的抗體,這是幾乎不可能完成的。但是應(yīng)用ChIP-chip/seq檢測(cè)一些TRE標(biāo)志性表觀標(biāo)志物,可以為這一問題的解決提供光明前景。可以依據(jù)ChIP-chip/seq技術(shù)檢測(cè)的相關(guān)表觀標(biāo)志物在基因組中的分布對(duì)TRE元件進(jìn)行分析及預(yù)測(cè)。
3 轉(zhuǎn)錄調(diào)控元件的功能驗(yàn)證
DNA片段的轉(zhuǎn)錄調(diào)控活性一般是通過報(bào)告基因瞬時(shí)轉(zhuǎn)染的方法進(jìn)行驗(yàn)證。將預(yù)測(cè)的TRE序列插入到熒光素酶報(bào)告基因質(zhì)粒中,瞬時(shí)轉(zhuǎn)染到細(xì)胞中,通過檢測(cè)熒光素酶報(bào)告基因的發(fā)光強(qiáng)度,對(duì)TRE元件的活性進(jìn)行對(duì)比分析;對(duì)于啟動(dòng)子活性的檢測(cè),是將預(yù)測(cè)的TRE元件置于報(bào)告基因的上游;而增強(qiáng)子活性則是在活性較弱的啟動(dòng)子元件下進(jìn)行驗(yàn)證,絕緣子元件需要置于增強(qiáng)子和啟動(dòng)子元件之間進(jìn)行驗(yàn)證。通過報(bào)告基因瞬時(shí)轉(zhuǎn)染法可以高通量篩選分析TRE元件[43-45],但這種方法也存在一些缺陷。例如,瞬時(shí)轉(zhuǎn)染的質(zhì)粒不能整合到基因組中,因此DNA片段不能正確呈現(xiàn)出染色質(zhì)的構(gòu)象,可能會(huì)出現(xiàn)異常表達(dá)。其次用于體外培養(yǎng)的細(xì)胞不能真實(shí)模擬體內(nèi)環(huán)境。
動(dòng)物模型轉(zhuǎn)基因分析方法可以克服這些缺點(diǎn),該方法可以通過顯微操作將質(zhì)粒隨機(jī)整合到受精卵基因組中,通過報(bào)告基因在胚胎的表達(dá)模式可以檢測(cè)插入DNA片段的組織特異性和活性。目前已有多種模式動(dòng)物中應(yīng)用于轉(zhuǎn)基因分析篩TRE元件[46-48],Pennacchio等[49]更是通過轉(zhuǎn)基因小鼠分析對(duì)人基因組中保守非編碼序列進(jìn)行高通量分析,篩選出75種增強(qiáng)子序列。但是轉(zhuǎn)基因分析對(duì)實(shí)驗(yàn)室條件要求嚴(yán)苛,且實(shí)驗(yàn)成本相比瞬時(shí)轉(zhuǎn)染要高,限制了該方法的廣泛應(yīng)用。
4 結(jié)語(yǔ)
許多TRE在某種類型細(xì)胞或發(fā)育的某個(gè)階段
處于沉默狀態(tài),在進(jìn)一步的功能驗(yàn)證中無表達(dá)活性。因此,需要有效的手段對(duì)相關(guān)調(diào)控元件基因組中分布位置以及表達(dá)活性進(jìn)行預(yù)測(cè)分析。比較基因組學(xué)、DNaseI 超敏性、FAIRE等方法不具有特異性,不能對(duì)TRE的類型進(jìn)行預(yù)測(cè),且不能分析TRE元件的活性狀態(tài)。通過ChIP-chip/seq可以實(shí)現(xiàn)全基因組范圍篩選,并可對(duì)相關(guān)的TRE進(jìn)一步細(xì)分。但目前ChIP-chip/seq的分辨率較低,在基因組中分布范圍廣,因此很難確定篩選序列的核心區(qū)域。但是通過多種方法的綜合分析及對(duì)比驗(yàn)證,將會(huì)大大提高TRE篩選的效率和成功率。
近年來,雖然相關(guān)表觀修飾物的研究對(duì)TRE篩選具有指導(dǎo)意義,但我們對(duì)染色質(zhì)修飾機(jī)制及這些修飾對(duì)基因的調(diào)控機(jī)制仍十分缺乏。同時(shí)還有許多的新的表觀修飾物去發(fā)現(xiàn)和歸類。因此,隨著篩選技術(shù)的發(fā)展和相關(guān)機(jī)制的深入研究,基因調(diào)控元件將進(jìn)一步應(yīng)用于基因治療等領(lǐng)域的研究中。
[[1]Ward LD,Kellis M. Evidence of abundant purifying selection in humans for recently acquired regulatory functions[J]. Science, 2012, 337(6102):1675-1678.
[2]Maurano MT, Humbert R, Rynes E, et al. Systematic localization of common disease-associated variation in regulatory DNA[J]. Science, 2012, 337(6099):1190-1195.
[3]Gardner KE, Allis CD, Strahl BD. Operating on chromatin, a colorful language where context matters[J]. J Mol Biol, 2011, 409(1):36-46.
[4]Straussman R, Nejman D, Roberts D, et al. Developmental programming of CpG island methylation profiles in the human genome[J]. Nature Structural & Molecular Biology, 2009, 16(5):564-571.
[5]Mikkelsen TS, Ku M, Jaffe DB, et al. Genome-wide maps of chromatin state in pluripotent and lineage-committed cells[J]. Nature, 2007, 448(7153):553-560.
[6]Guenther MG, Levine SS, Boyer LA, et al. A chromatin landmark and transcription initiation at most promoters in human cells[J]. Cell, 2007, 130(1):77-88.
[7]Consortium EP, Bernstein BE, Birney E, et al. An integrated encyclopedia of DNA elements in the human genome[J]. Nature, 2012, 489(7414):57-74.
[8]Goldberg AD, Banaszynski LA, Noh KM, et al. Distinct factors control histone variant H3. 3 localization at specific genomic regions[J]. Cell, 2010, 140(5):678-691.
[9]Bannister AJ,Kouzarides T. Regulation of chromatin by histone modifications[J]. Cell Res, 2011, 21(3):381-395.
[10]Ernst J,Kellis M. Discovery and characterization of chromatin states for systematic annotation of the human genome[J]. Nat Biotechnol, 2010, 28(8):817-825.
[11]Sauvageau M,Sauvageau G. Polycomb group proteins:multifaceted regulators of somatic stem cells and cancer[J]. Cell Stem Cell, 2010, 7(3):299-313.
[12]Christophersen NS,Helin K. Epigenetic control of embryonic stem cell fate[J]. Journal of Experimental Medicine, 2010, 207(11):2287-2295.
[13]Hawkins RD, Hon GC, Lee LK, et al. Distinct epigenomic landscapes of pluripotent and lineage-committed human cells[J]. Cell Stem Cell, 2010, 6(5):479-491.
[14]Barski A, Cuddapah S, Cui K, et al. High-resolution profiling of histone methylations in the human genome[J]. Cell, 2007, 129(4):823-837.
[15]Yun M, Wu J, Workman JL, et al. Readers of histone modifications[J]. Cell Res, 2011, 21(4):564-578.
[16]Ohlsson R, Bartkuhn M,Renkawitz R. CTCF shapes chromatin by multiple mechanisms:the impact of 20 years of CTCF research on understanding the workings of chromatin[J]. Chromosoma, 2010, 119(4):351,360.
[17]Cuddapah S, Jothi R, Schones DE, et al. Global analysis of the insulator binding protein CTCF in chromatin barrier regions reveals demarcation of active and repressive domains[J]. Genome Res, 2009, 19(1):24-32.
[18]Bedford DC, Kasper LH, Fukuyama T, et al. Target gene context influences the transcriptional requirement for the KAT3 family of CBP and p300 histone acetyltransferases[J]. Epigenetics, 2010, 5(1):9-15.
[19]Heintzman ND, Stuart RK, Hon G, et al. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome[J]. Nat Genet, 2007, 39(3):311-318.
[20]Visel A, Blow MJ, Li Z, et al. ChIP-seq accurately predicts tissuespecific activity of enhancers[J]. Nature, 2009, 457(7231):
854-85 8.
[21]Blow MJ, McCulley DJ, Li Z, et al. ChIP-Seq identification of weakly conserved heart enhancers[J]. Nat Genet, 2010, 42(9):806-810.
[22]Heintzman ND, Hon GC, Hawkins RD, et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression[J]. Nature, 2009, 459(7243):108-112.
[23]Patel DJ,Wang Z. Readout of epigenetic modifications[J]. Annu Rev Biochem, 2013, 82:81-118.
[24]Creyghton MP, Cheng AW, Welstead GG, et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state[J]. Proceedings of the National Academy of Sciences, 2010, 107(50):21931-21936.
[25]Rada-Iglesias A, Bajpai R, Swigut T, et al. A unique chromatin signature uncovers early developmental enhancers in humans[J]. Nature, 2011, 470(7333):279-283.
[26]Zentner GE, Tesar PJ,Scacheri PC. Epigenetic signatures distinguish multiple classes of enhancers with distinct cellular functions[J]. Genome Res, 2011, 21(8):1273-1283.
[27]Ernst J, Kheradpour P, Mikkelsen TS, et al. Mapping and analysis of chromatin state dynamics in nine human cell types[J]. Nature, 2011, 473(7345):43-49.
[28]Nei M, Suzuki Y,Nozawa M. The neutral theory of molecular evolution in the genomic era[J]. Annual Review of Genomics and Human Genetics, 2010, 11:265-289.
[29]Nelson AC,Wardle FC. Conserved non-coding elements and cis regulation:actions speak louder than words[J]. Development, 2013, 140(7):1385-1395.
[30]Kague E, Bessling SL, Lee J, et al. Functionally conserved cisregulatory elements of COL18A1 identified through zebrafish transgenesis[J]. Dev Biol, 2010, 337(2):496-505.
[31]Langlais D, Couture C, Sylvain-Drolet G, et al. A pituitary-specific enhancer of the POMC gene with preferential activity in corticotrope cells[J]. Mol Endocrinol, 2011, 25(2):348-359.
[32]Doglio L, Goode DK, Pelleri MC, et al. Parallel evolution of chordate cis-regulatory code for development[J]. PLoS Genet, 2013, 9(11):e1003904.
[33]Kim MJ, Skewes-Cox P, Fukushima H, et al. Functional characterization of liver enhancers that regulate drug-associated transporters[J]. Clinical Pharmacology & Therapeutics, 2011, 89(4):571-578.
[34]Antonellis A, Huynh JL, Lee-Lin SQ, et al. Identification of neural crest and glial enhancers at the mouse Sox10 locus through transgenesis in zebrafish[J]. PLoS Genet, 2008, 4(9):e1000174.
[35]McGaughey DM, Vinton RM, Huynh J, et al. Metrics of sequence constraint overlook regulatory sequences in an exhaustive analysis at phox2b[J]. Genome Res, 2008, 18(2):252-260.
[36]Consortium EP, Birney E, Stamatoyannopoulos JA, et al. Identification and analysis of functional elements in 1% of the human genome by the ENCODE pilot project[J]. Nature, 2007, 447(7146):799-816.
[37]Ahituv N, Zhu Y, Visel A, et al. Deletion of ultraconserved elements yields viable mice[J]. PLoS Biol, 2007, 5(9):e234.
[38]Boyle AP, Song L, Lee BK, et al. High-resolution genome-wide in vivo footprinting of diverse transcription factors in human cells[J]. Genome Res, 2011, 21(3):456-464.
[39]Kim YW,Kim A. Histone acetylation contributes to chromatin looping between the locus control region and globin gene by influencing hypersensitive site formation[J]. Biochim Biophys Acta, 2013, 1829(9):963-969.
[40]Madrigal P,Krajewski P. Current bioinformatic approaches to identify DNase I hypersensitive sites and genomic footprints from DNase-seq data[J]. Frontiers in Genetics, 2012(3):230.
[41]Simon JM, Giresi PG, Davis IJ, et al. Using formaldehydeassisted isolation of regulatory elements (FAIRE) to isolate active regulatory DNA[J]. Nat Protoc, 2012, 7(2):256-267.
[42]Ren B, Robert F, Wyrick JJ, et al. Genome-wide location and function of DNA binding proteins[J]. Science, 2000, 290(5500):2306-2309.
[43]Cooper SJ, Trinklein ND, Anton ED, et al. Comprehensive analysis of transcriptional promoter structure and function in 1% of the human genome[J]. Genome Res, 2006, 16(1):1-10.
[44]Fort A, Fish RJ, Attanasio C, et al. A liver enhancer in the fibrinogen gene cluster[J]. Blood, 2011, 117(1):276-282.
[45]Groth AC,Emery DW. A functional screen for regulatory elements that improve retroviral vector gene expression[J]. Blood Cells Mol Dis, 2010, 45(4):343-350.
[46]Chatterjee S, Bourque G,Lufkin T. Conserved and non-conserved enhancers direct tissue specific transcription in ancient germ layer
specific developmental control genes[J]. BMC Dev Biol, 2011, 11:63.
[47]Narlikar L, Sakabe NJ, Blanski AA, et al. Genome-wide discovery of human heart enhancers[J]. Genome Res, 2010, 20(3):381-392.
[48]Visel A, Zhu Y, May D, et al. Targeted deletion of the 9p21 noncoding coronary artery disease risk interval in mice[J]. Nature, 2010, 464(7287):409-412.
[49]Pennacchio LA, Ahituv N, Moses AM, et al. In vivo enhancer analysis of human conserved non-coding sequences[J]. Nature, 2006, 444(7118):499-502.
(責(zé)任編輯 狄艷紅)
Research Advance in Genome-wide Identification of Transcription Regulatory Elements
Li Wenmao1Jia Dongfang1Xu Rong1,2
(1. School of Biomedical Sciences,Huaqiao University,Quanzhou 362021;2. Quanzhou Medical College,Quanzhou 362100)
Expression of eukaryotic genes is a complex process, achieved through the coordinated action of transcription regulatory elements. The screening and identification of transcriptional regulatory elements in genome scale have crucial significance for understanding the mechanism and biological function of gene expression regulation. However, the effectively screening and function verification is still the major challenges for researchers. Herein, based on the latest research findings, we reviewed the methods of prediction, screening, function analysis of transcription regulatory elements in genome.
Transcriptional regulatory element High throughput screening Biomark
2013-12-26
福建省教育廳科技項(xiàng)目(JA12434),泉州市科技計(jì)劃項(xiàng)目(2012Z71)
李文茂,男,碩士研究生,研究方向:肺癌靶向基因治療;E-mail:liwenmao@aliyun.com
許嶸,男,博士研究生,講師,研究方向:基因治療;E-mail:xurong197886@sina.com

