IQ基序突變對AtIQM1的鈣調素結合活性的影響
黃章科 張藝能 莫忠蓁 周玉萍 黃小玲 田長恩
(廣州大學生命科學學院 植物抗逆基因功能研究廣州市重點實驗室,廣州 510006)
IQ基序突變對AtIQM1的鈣調素結合活性的影響
黃章科 張藝能 莫忠蓁 周玉萍 黃小玲 田長恩
(廣州大學生命科學學院 植物抗逆基因功能研究廣州市重點實驗室,廣州 510006)
旨在確定IQ基序中關鍵氨基酸殘基突變對IQM1的CaM結合活性的影響,利用基因體外定點突變技術將LQ缺失或將L突變成S,并通過酵母雙雜交分析突變蛋白iqm1的CaM結合活性。結果顯示,用重組質粒pGADT7-IQM1CDS、pGADT7-IQM1Del113-114、pGADT7-IQM1L113S或pGBKT7-CaM5分別轉化酵母AH109,未出現自激活現象;用pGADT7-IQM1CDS和 pGBKT7-CaM5共轉化酵母能在選擇性培養基(SD/-Ade/-His/-Leu/-Trp/X-α-Gal)上形成藍色菌落;而用pGADT7-IQM1Del113-114和pGBKT7-CaM5或pGADT7-IQM1L113S和pGBKT7-CaM5共轉化酵母則不能在選擇性培養基(SD/-Ade/-His/-Leu/-Trp)上形成菌落。結果表明,IQ基序中LQ或者L是IQM1與CaM5結合的關鍵氨基酸殘基。
AtIQM1 IQ基序突變 酵母雙雜交 CaM結合
植物的生命過程受體內外各種信號的調控。Ca2+是細胞內1個非常重要的第二信使,它可以調控多種生理反應。鈣調素(calmodulin,CaM)作為細胞內Ca2+受體,絕大多數無酶活性,也無轉錄因子活性,更不是結構蛋白,只有通過其下游的鈣調素結合蛋白(calmodulin-binding protein,CaMBP)來調節細胞的生理功能[1-4]。CaMBP與CaM的結合可分為Ca2+依賴、不依賴和抑制3種類型[3],IQ基序是第一個被發現的Ca2+不依賴性CaM結合結構域(calmodulin-binding domain,CaMBD),其氨基酸序列為IQXXXRGXXXR,其中的I可變為L、V或M,第6位和11位的R可變為K或H,第7位的G同樣缺乏保守性,因此,IQ基序的完整序列可概括為[I,L,V]QXXXRXXXX[R,K][5]。但是,個別含IQ基序的蛋白質與CaM的結合也依賴Ca2+[6,7]。在植物中已經發現了5個含有IQ基序的蛋白質家
族[8]:myosin家族[9-11]、CAMTA家族[12,13]、CNGC家族[14,15]、IQD家族[6,16,17]和 IQM 家族[18-20],分別由17個、6個、20個、33個和6個成員組成。IQM家族成員的N-端與豌豆重金屬誘導蛋白6A(該蛋白的編碼基因受重金屬誘導而表達,但其功能未知)、C-端與天花粉素(一類使核糖體失活、具有RNA結合活性的小分子蛋白)具有較高的同源性[18]。IQM1(IQ motif containing 1)是IQM家族的1個成員,由At4g33050編碼,只含1個IQ基序,通過酵母雙雜交和雙分子熒光互補試驗證明IQM1在體內、外都能與CaM結合,并通過蛋白截短試驗證明IQ基序是其鈣調素結合所必需的結構域[20]。為了確證IQM1介導的CaM信號參與植物細胞生理或生長發育的調控,需要構建IQ基序中關鍵氨基酸殘基突變的突變互補載體,轉入IQM1基因的突變體中,觀察突變表型的恢復情況。若突變表型不能恢復,則說明通過IQ基序與CaM的結合,即IQM1介導的CaM信號參與了相關調控;否則相反。當然,在構建植物表達載體前,需要先證實IQ基序中關鍵氨基酸殘基點突變或缺失后iqm1的CaM結合活性。為此,本研究利用基因體外定點突變技術將IQM1的IQ基序中的關鍵氨基酸殘基LQ缺失或者將L突變成S,然后利用酵母雙雜交分析突變蛋白iqm1與CaM的結合活性,旨在確定IQ基序中關鍵氨基酸殘基突變對IQM1的CaM結合活性的影響。
1 材料與方法
1.1 材料
擬南芥(Arabidopsis thaliana)為Columbia(Col)生態型、大腸桿菌菌株為DH5α、重組質粒pUC19-P35S:eGFP:IQM1、pUC19-P35S:eGFP:CaM5為實驗室構建,酵母雙雜交系統(Matchmaker Gal4 Two-Hybrid System3)由中國科學院華南植物園張明永教授友好轉贈,Pfu DNA Polymerase購自Fermentas公司,Carrier DNA購自Sigma公司,pMD18-T Simple、膠回收試劑盒、限制性內切酶、Ex Taq DNA 聚合酶及PCR相關試劑均為TaKaRa公司產品。
1.2 方法
1.2.1 IQM1基因的CDS全長序列獲取 以pUC19-P35S:eGFP:IQM1為模板,利用IQM1基因引物(F1:5'-TACCCGGGTATGGGTCTTGAAGTTGGGTC-3',下劃線部分為Sma I酶切位點;R1:5'-CCATCGATTT AACACTTTACAGCCCTAGGGC-3',下劃線部分為Cla I酶切位點)進行PCR擴增,反應程序為:94℃預變性4 min;94℃變性30 s,64℃退火30 s,72℃延伸90 s,共35個循環;72℃再延伸5 min。反應結束后電泳,并用凝膠成像系統觀察、拍照。
1.2.2 pGADT7-IQM1CDS獵物載體的構建 切取目的片段所在的凝膠,用膠回收試劑盒回收IQM1 CDS。將回收產物與 pMD18-T連接,轉化E.coli DH5α,挑取氨芐青霉素抗性菌落,進行PCR 鑒定。取陽性克隆搖菌,提取質粒并用Sma I和Cla I進行雙酶切鑒定后送華大基因公司測序。然后,將經測序證實的pMD18-IQM1CDS和pGADT7用Sma I和Cla I雙酶切,電泳后割膠回收目的片段并連接,轉化E.coli DH5α,進行菌落PCR鑒定,并進一步提取質粒進行酶切鑒定,得到用于酵母雙雜交的獵物載體pGADT7-IQM1CDS。
1.2.3 IQ基序中LQ缺失和L突變成S的PCR擴增及其獵物載體的構建 用Pfu DNA Polymerase,以測序證實過的pMD18-IQM1CDS為模板,分別用LQ缺失 引物(F2:5'-GATGCAGCTGCAACTACGAAGGTGTACAAGAGTTAC-3',Leu113和Gln114缺失;R2:5'-GTAACTCTTGTACACCTTCGTAGTTGCAGCTGCATC-3',Leu113和Gln114缺失)和點突變引物(F3:5'-TTGATGCAGCTGCAACTACGTCACAAAAGGTGTACAAG-3',Leu113突變成Ser;R3:5'-CTTGTACACCTTTTGTGACGTAGTTGCAGCTGCATCAA-3',Leu113突變成Ser)進行PCR擴增,程序為:95℃ 2 min;95℃ 30 s,62℃/65℃ 30 s,72℃ 4 min,18個循環;72℃延伸5 min。擴增完成后用Dpn I消化有甲基化修飾的模板,體外擴增產物因無甲基化修飾而不被消化。模板消化后滅活Dpn I,轉化E.coli DH5α,經菌落PCR鑒定后搖菌提取質粒并經酶切鑒定后送華大基因公司測序。然后用Sma I和Cla I對測序驗證過的缺失突變質粒pMD18-IQM1Del113-114、點突變質粒pMD18-IQM1L113S及質粒pGADT7,進行雙酶切,割膠回收目的片段并連接轉化,經菌落PCR和質粒酶切鑒定,得到酵母雙雜交獵物載體pGADT7-
IQM1Del113-114和pGADT7-IQM1L113S。
1.2.4 CaM5的 PCR 擴增及其誘餌載體的構建 以pUC19-P35S:eGFP:CaM5為模板,用加有酶切位點的CaM5基因引物(5'-TTGAATTCATGGCAGATCAGCTCACCGATGATC-3',下劃線部分為EcoR I酶切位點;5'-TGGTCGACTCACTTTGCCATCATAACTTTGAC-3',下劃線部分為Sal I酶切位點)進行PCR擴增,程序為:94℃ 4 min;94℃ 30 s,61℃ 30 s,72℃ 30 s,共35個循環;72℃延伸 5 min。此后的載體構建方法同1.2.2。用卡那霉素進行篩選,EcoR I和Sal I雙酶切鑒定,得到酵母雙雜交誘餌載體pGBKT7-CaM5。
1.2.5 酵母感受態制備和轉化 參考 Clontech 公司Matchmaker Gal4 Two-Hybrid System3的操作方法制備AH109感受態細胞,取150 μL分裝于1.5 mL離心管,加入10 mg/mL 單鏈Carrier DNA 5 μL和各種質粒(圖3),混勻,再加入600 μL PEG/LiAc溶液(現配現用),渦旋混勻,于30℃、220 r/min培養30 min,加入70 μL DMSO,顛倒混勻,42℃水浴熱激15 min(每5 min輕混1次),冰上放置2 min后,室溫、12 000 r/min離心5 s,棄上清,用400 μL TE重懸細胞,涂布于SD/-Leu/-Trp培養基上,30℃培養3-5 d至菌落形成。
1.2.6 酵母雙雜交分析 從上述的SD/-Leu/-Trp培養基上挑取陽性克隆,涂于SD/-Ade/-His/-Leu/-Trp培養基上,30℃培養3-5 d,觀察菌落形成情況;再將其上長出的菌落轉涂于SD/-Ade/-His/-Leu/-Trp/X-α-Gal培養基上,30℃培養3-5 d,觀察菌落顏色變化情況。
2 結果
2.1 相關載體的構建與鑒定
IQM1基因由AT4G33050編碼,本試驗所用的CDS為AT4G33050.3,全長1 467 bp,其中IQ基序位于第二個外顯子末端(圖1)。盡管已通過Invitrogen的酵母雙雜交系統證實,IQM1能與CaM結合[20],但因本研究采用Clontech的酵母雙雜交系統,所以必須在IQM1的CDS的上下游分別加入合適的酶切位點。用分別添加Sma I和Cla I酶切位點的引物F1+R1,對pUC19-P35S:eGFP: IQM1進行PCR擴增,以克隆IQM1 CDS,并將其構建到pMD18-T,經過鑒定,IQM1 CDS未發生突變(圖2-A)。然后根據QuikChange?XL Site-Directed Mutagenesis Kit(Stratagene)的說明,將IQ基序中的關鍵氨基酸殘基進行突變,構建到pMD18-T中,測序結果顯示,點突變的IQM1的第338位堿基T突變成C,即第113位L(TTA)突變為S(TCA)(圖2-B);缺失突變的IQM1的第337-342位堿基(TTACAA)缺失,即第113、114位氨基酸(L、Q)缺失(圖2-C)。其它部分均不存在突變。最后把構建的pMD18-IQM1CDS、pMD18-IQM1Del113-114和pMD18-IQM1L113S通過Sma I和Cla I特異位點定向克隆到pGADT7,構建成pGADT7-IQM1CDS、pGADT7-IQM1Del113-114和 pGADT7-IQM1L113S,并經Sma I和Cla I雙酶切鑒定。同時,先將PCR擴增的CaM5 CDS克隆到pMD18-T,測序證實后再通過EcoR I和Sal I定向插入pGBKT7,得到pGBKT7-CaM5,并經EcoR I和Sal I雙酶切鑒定。
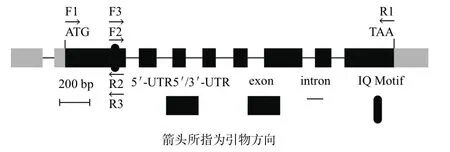
圖1 IQM1基因的結構及擴增引物示意圖

圖2 IQ基序中關鍵氨基酸殘基點突變或缺失
2.2 關鍵氨基酸突變對相互作用的影響分析
分別用圖3所示的1-9號質粒組合共轉化酵母AH109,涂布于培養基SD/-Trp/-Lue,30℃培養4 d,長出菌落(圖3-A),說明質粒共轉化成功。將所得轉化子分別轉涂于SD/-Trp/-Leu/-His/-Ade培養基培養,只有1號(陽性對照)和2號(IQM1×CaM5)組合的轉化子長出菌落,其他組合的轉化子均沒有生長(圖3-B);進而將1號、2號菌落轉移到SD/-Trp/-Leu/-His/-Ade/X-α-Gal培養基培養,二者均能生長且顯藍色(圖3-C)。以上結果說明,IQM1 和CaM5能在酵母細胞內結合,使轉錄因子GAL4的功能恢復,激活報告基因表達;而3號、4號組合的轉化子不能在SD/-Trp/-Leu/-His/-Ade培養基上生長,說明IQ基序中LQ缺失或L突變成S后的突變蛋白iqm1不能與CaM5結合,從而證實了IQ基序的LQ或L是IQM1與CaM5結合的關鍵氨基酸殘基,且L更為重要。

圖3 酵母雙雜交分析
3 討論
CaM作為細胞的Ca2+受體,在Ca2+/CaM信號轉導中需通過其靶標——CaMBP發揮作用。當前,研究蛋白質之間相互作用的技術方法主要有酵母雙雜交、免疫共沉淀、pull-down、雙分子熒光互補以及串聯親和純化等方法[21]。Fields 和Song[22]首先發明酵母雙雜交技術,由最初研究蛋白間的相互作用擴展到研究蛋白和DNA、RNA及其他小分子配體間的相互作用,現已衍生出許多試驗系統[23]。IQM1與CaM的結合活性已被本研究組先后利用Invitrogen[20]和Clontech的酵母雙雜交系統所證實。并通過蛋白截短試驗證明IQ基序是其CaM結合所必需的結構域[20];通過基因體外定點突變技術將其中的LQ缺失或L突變為S后所獲得的突變蛋白iqm1都不能與CaM結合。這說明IQ 基序是IQM1和CaM結合的關鍵結構域,其中的LQ尤其是L是決定結合活性的關鍵氨基酸殘基。本研究結果為構建IQ基序中關鍵氨基酸殘基點突變或缺失的突變互補載體,轉入iqm1突變體,觀察突變體表型恢復情況,以確證IQM1介導的CaM信號是否參與有關的生理或生長發育調控提供了支撐。相關思路可資其它CaMBP的功能研究者借鑒。
4 結論
利用酵母雙雜交系統分析了IQ基序中LQ缺失和L突變成S對于IQM1與CaM5結合活性的影響,試驗結果說明了無論是LQ缺失或L突變成S后,突變蛋白IQM1都不能與CaM5相互結合,從而確定了IQ基序的LQ或L是IQM1與CaM5結合的關鍵氨基酸殘基。
[1]McAinsh RM, Pittman JK. Shaping the calcium signature[J]. New Phytologist, 2009, 181(2):275-294.
[2]毛國紅, 宋林霞, 孫大業.植物鈣調素結合蛋白研究進展[J].植物生理與分子生物學報, 2004, 30(5):481-488.
[3]O’Day DH. CaMBOT:profiling and characterizing calmodulinbinding protein[J]. Cellular Signalling, 2003, 15(4):347-354.
[4]Sanders D, Pelloux J, Brownlee C, et al. Calcium at the crossroads of signaling[J]. Plant Cell, 2002, 14(Suppl.):S401-S417.
[5]B?hler M, Rhoads A. Calmodulin signaling via the IQ motif[J]. FEBS Letters, 2002, 513(1):107-113.
[6]Abel S, Savchenko T, Levy M. Genome-wide comparative analysis of the IQD gene families in Arabbidopsis thaliana and Oryza sativa[J]. BMC Evol Biol, 2005, 5:72-89.
[7]韋慧彥, 郭振清, 崔素娟.鈣不依賴性鈣調素結合蛋白的研究進展[J].生物化學與生物物理進展, 2007, 34(2):124-131.
[8]田長恩, 周玉萍.植物具IQ基序的鈣調素結合蛋白的研究進展[J].植物學報, 2013, 48(4):447-480.
[9]Reddy AS, Day IS. Analysis of the myosins encoded in the recently
completed Arabidopsis thaliana genome sequence[J]. Genome Biol, 2001, 2(7):research0024.1-research0024.17.
[10]Holweg C, Nick P. Arabidopsis myosin XI mutant is defective inorganelle movement and polar auxin transport[J]. Proc Natl Acad Sci USA, 2004, 101:10488-10493.
[11]Ojangu EL, J?rve K, Paves H, et al. Arabidopsis thaliana myosin XIK is involved in root hair as well as trichome morphogenesis on stems and leaves[J]. Protoplasma, 2007, 230(3-4):193-202.
[12]Bouché N, Scharlat A, Snedden W, et al. A novel family of calmodulin-binding transcription activators in multicellular organisms[J]. J Biol Chem, 2002, 277(24):21851-21861.
[13]Bouché N, Yellin A, Snedden WA, et al. Plant-specific calmodulinbinding proteins[J]. Annu Rev Plant Biol, 2005, 56:435-466.
[14]K?hler C, Merkle T, Neuhaus G. Characterisation of a novel gene family of putative cyclic nucleotide and calmodulin-regulated ion channels in Arabidopsis thaliana[J]. Plant J, 1999, 18(1):97-104.
[15]Talke IN, Blaudez D, Maathuis FJ, et al. CNGCs:prime targets of plant cyclic nucleotide signaling?[J]. Trends Plant Sci, 2003, 8(6):286-293.
[16]Levy M, Wang Q, Kaspi R, et al. Arabidopsis IQD1, a novel calmodulin-binding nuclear protein, stimulates glucosinolate accumulation and plant defense[J]. Plant J, 2005, 43(1):79-96.
[17]韋慧彥, 郭振清, 崔素娟.擬南芥鈣調素結合蛋白AtIQD26的分離鑒定[J].生物化學與生物物理進展, 2008, 35(6):703-711.
[18]Zhou Y, Fujibe T, Wang XL, et al. Initial characterization of Arabidopsis T-DNA insertion mutants of the iqm1 gene that encodes an IQ motif-containing protein[J]. Plant Cell Physiol, 2007, 48(Suppl.):s197.
[19]Zhou YP, Chen YZ, Duan J, et al. Sequence and expression analysis of the Arabidopsis IQM family[J]. Acta Physiol Plant, 2010, 32(1):191-198.
[20]Zhou YP, Duan J, Yamamoto KT, et al. AtIQM1, a novel calmodulin-binding protein, is involved in stomatal movement in Arabidopsis[J]. Plant Mol Biol, 2012, 79(4-5):333-346.
[21]陳謀通, 劉建軍.蛋白相互作用的研究方法[J].生物技術通報, 2009(1):50-68.
[22]Fields S, Song O. A novel genetic system to detect protein- protein interactions[J]. Nature, 1989, 340(6230):245-246.
[23]黃欣媛, 范紅波.酵母雙雜交及其衍生系統[J].生物技術通報, 2014(1):75-82.
(責任編輯 馬鑫)
Effects of Mutations in IQ Motif of AtIQM1 on Its Calmodulin Binding
Huang Zhangke Zhang Yi’neng Mo Zhongzhen Zhou Yuping Huang Xiaoling Tian Chang’en
(Guangzhou Key Laboratory for Functional Study on Stress-Resistant Genes in Plants,School of Life Sciences,Guangzhou University,Guangzhou 510006)
To confirm the effects of mutation of key amino acid residues in IQ motif of AtIQM1 on its calmodulin binding, the LQ was deleted or L was mutated to S through the mutagenesis technology in vitro. Then calmodulin binding of the mutant protein iqm1 was analyzed via yeast two-hybrid assay. The self-activation was not observed when the recombinant plasmid pGADT7-IQM1CDS, pGADT7-IQM1Del113-114, pGBKT7-CaM5 or pGADT7-IQM1L113Swas transformed into the yeast AH109 strain, respectively. Blue colonies were achieved on the selective agar plates(SD/-Ade/-His/-Leu/-Trp/X-α-Gal)when pGADT7-IQM1CDSand pGBKT7-CaM5 were co-transformed into AH109 yeast strain. However, the yeasts which were co-transformed with pGADT7-IQM1Del113-114and pGBKT7-CaM5 or pGADT7-IQM1L113Sand pGBKT7-CaM5 were not able to grow on the selective medium(SD/-Ade/-His/-Leu/-Trp). The results suggest that the LQ or L in IQ motif is required for AtIQM1 to combine with CaM5.
AtIQM1 Mutantion in IQ motif Yeast two hybrid Calmodulin binding
10.13560/j.cnki.biotech.bull.1985.2014.12.021
2014-05-26
國家自然科學基金項目(31170204),羊城學者科研項目(12A001G)
黃章科,男,碩士研究生,研究方向:植物分子遺傳學;E-mail:huang.zhangke@163.com
田長恩,男,博士,教授,研究方向:植物分子遺傳學;E-mail:changentian@aliyun.com

