中國(guó)葡萄酒產(chǎn)區(qū)酒酒球菌種質(zhì)資源遺傳多樣性分析
金 剛,王 華,2,3,張 昂,李 華,2,3,*
中國(guó)葡萄酒產(chǎn)區(qū)酒酒球菌種質(zhì)資源遺傳多樣性分析
金 剛1,王 華1,2,3,張 昂4,李 華1,2,3,*
(1.西北農(nóng)林科技大學(xué)葡萄酒學(xué)院,陜西 楊凌 712100;2.西北農(nóng)林科技大學(xué),合陽(yáng)葡萄試驗(yàn)示范站,陜西 合陽(yáng) 715300;3.陜西省葡萄與葡萄酒工程技術(shù)研究中心,陜西 楊凌 712100;4.秦皇島出入境檢驗(yàn)檢疫局,河北 秦皇島 066004)
為了解我國(guó)葡萄酒產(chǎn)區(qū)酒酒球菌種質(zhì)資源遺傳多樣性,對(duì)22株篩選自我國(guó)不同葡萄酒產(chǎn)區(qū)的乳酸細(xì)菌進(jìn)行了種特異性聚合酶鏈?zhǔn)椒磻?yīng)(species-specific polymerase chain reaction,PCR)分析、16S rRNA序列分析和擴(kuò)增片段長(zhǎng)度多態(tài)性分析(amplified fragment length polymorphism,AFLP)基因分型。種特異性PCR和16S rRNA序列分析表明22株分離株為酒酒球菌(Oenococcus oeni)。建立了O. oeni基于HindⅢ和MseⅠ為內(nèi)切酶的AFLP分析體系,對(duì)16對(duì)引物組合進(jìn)行了篩選,結(jié)果表明HT-MA、HT-MT、HT-MC、HG-MA、HG-MT、HC-MT為O. oeniAFLP分析的最佳引物組合,且實(shí)驗(yàn)重復(fù)性在98%以上。對(duì)O. oeni的AFLP分析結(jié)果顯示:22株O. oeni分為3個(gè)簇群,且簇群間遺傳相似性系數(shù)較小。所以,以HindⅢ和MseⅠ為內(nèi)切酶的AFLP技術(shù)是研究O. oeni基因分型的有效方法,我國(guó)葡萄酒產(chǎn)區(qū)的O. oeni具有豐富的遺傳多樣性,O. oeni菌株間的遺傳相似性系數(shù)不僅僅與其生態(tài)地理分布有關(guān),可能還與其所處的微生態(tài)和其他因素有關(guān)。
酒酒球菌;種特異性PCR;16S rRNA序列分析;擴(kuò)增片段長(zhǎng)度多態(tài)性分析技術(shù)
蘋(píng)果酸-乳酸發(fā)酵(malolactic fermentation,MLF)是葡萄酒釀造過(guò)程中基本的生物化學(xué)過(guò)程,它發(fā)生于酒精發(fā)酵之后,細(xì)菌把蘋(píng)果酸轉(zhuǎn)化成乳酸和二氧化碳[1]。酒酒球菌(Oenococcus oeni)被認(rèn)為是MLF的主要完成者,它能降低葡萄酒的酸度、改善葡萄酒的質(zhì)量和增強(qiáng)穩(wěn)定性[2]。
隨著我國(guó)葡萄酒產(chǎn)業(yè)的發(fā)展壯大,葡萄酒產(chǎn)業(yè)已開(kāi)始由單一的飲料葡萄酒到飲料葡萄酒和酒莊葡萄酒共同發(fā)展的局面。酒莊酒不僅對(duì)葡萄酒的質(zhì)量有較高要求,而且突出了對(duì)葡萄酒個(gè)性風(fēng)格的要求,葡萄酒的個(gè)性風(fēng)格是葡萄酒對(duì)不同產(chǎn)地的包括釀酒微生物的風(fēng)土條件的總體反映。所以,自然發(fā)酵和篩選當(dāng)?shù)匚⑸镞M(jìn)行接種發(fā)酵對(duì)于釀造高品質(zhì)的酒莊酒有重要意義。O. oeni在葡萄酒中的活性依賴于它對(duì)葡萄酒惡劣生境(高酸、高SO2含量、高酒精體積分?jǐn)?shù))的耐受性[1,3]。不同的菌株對(duì)于逆境的適應(yīng)性不同。我國(guó)葡萄酒產(chǎn)區(qū)分布比較廣泛,各產(chǎn)區(qū)生態(tài)地理?xiàng)l件差異很大,必然蘊(yùn)含著豐富和多樣性的O. oeni種質(zhì)資源。因此,分離篩選我國(guó)葡萄酒產(chǎn)區(qū)O. oeni,研究它們的遺傳多樣性及其與生態(tài)地理分布的關(guān)系對(duì)于我國(guó)葡萄酒的生產(chǎn)具有重要意義。
分子生物學(xué)的發(fā)展使得微生物的分類(lèi)鑒定變得更為高效和可靠。目前,各種分子生物學(xué)方法已廣泛應(yīng)用于微生物的分類(lèi)鑒定。不同種類(lèi)的乳酸菌可能存在于同一發(fā)酵過(guò)程中,所以對(duì)于乳酸菌種水平上的鑒定是必要的。近年來(lái),16S rRNA測(cè)序[4-5]、16S~23S rRNA間隔區(qū)序列分析[6-7]、種特異性聚合酶鏈?zhǔn)椒磻?yīng)(species-specific polymerase chain reaction,PCR)技術(shù)[4-5,8-9]被廣泛應(yīng)用于O. oeni的鑒定。
對(duì)于O. oeni菌株的基因分型,只有對(duì)于基因型微弱差異敏感的分子標(biāo)記方法才能完成。這些分子標(biāo)記方法包括隨機(jī)擴(kuò)增多態(tài)性DNA-PCR(random amplified polymorphic DNA-PCR,RAPD-PCR)[10-11],多位點(diǎn)序列分析(multiple locus sequence typing,MLST)[12-13],脈沖電場(chǎng)凝膠電泳(pulsed field gel electrophoresis,PFGE)[14],擴(kuò)增片段長(zhǎng)度多態(tài)性分析技術(shù)(amplified fragment length polymorphism,AFLP)[5,15-16]。它們已被越來(lái)越多地應(yīng)用于O. oeni及其他乳酸細(xì)菌的分類(lèi)鑒定研究中。
AFLP是基于對(duì)基因組限制性酶切片段選擇性PCR擴(kuò)增結(jié)果分析的基礎(chǔ)上來(lái)對(duì)不同基因組進(jìn)行分型的技術(shù)[17]。AFLP結(jié)合了限制性片段長(zhǎng)度多態(tài)性分析技術(shù)(restriction fragment length polymorphism,RFLP)和RAPD兩種技術(shù)的優(yōu)點(diǎn),被認(rèn)為是目前最有效最理想的一種DNA分子標(biāo)記技術(shù)。但它在葡萄酒微生物研究中仍然較少應(yīng)用。目前,在對(duì)O. oeni的基因分型研究時(shí)仍然應(yīng)用的是經(jīng)典的EcoRⅠ和MseⅠ內(nèi)切酶組合,本研究采用HindⅢ和MseⅠ兩種內(nèi)切酶組合,以期較好的將AFLP應(yīng)用于O. oeni基因研究中。
1 材料與方法
1.1菌株與培養(yǎng)基
供試菌株是分別從山東青島(SD-1a、SD-1b、SD-1c、SD-1d、SD-1e、SD-1f、SD-1g)、山東煙臺(tái)(SD-2a、SD-2b、SD-2d、SD-2ee、SD-2gf、SD-2gh、SD-2h、SD-2ji、SD-2kj)、河北沙城(HB-1a、HB-1b、HB-1c、HB-2b)、陜西楊凌(SX-1a、SX-1b)等地的葡萄酒中分離、初步鑒定得到的22株O. oeni。本實(shí)驗(yàn)收集了1株O. oeni的標(biāo)準(zhǔn)菌株作為陽(yáng)性對(duì)照,它是酒酒球菌31-DH,由中國(guó)食品發(fā)酵工業(yè)研究院提供。另外還收集了一株嗜酸乳桿菌(中國(guó)工業(yè)微生物菌種保藏管理中心,編號(hào)6005)作為陰性對(duì)照菌株,用于O. oeni的鑒定。
O. oeni的培養(yǎng)采用ATB培養(yǎng)基:蛋白胨10 g/L、酵母浸出物5 g/L、葡萄糖10 g/L、MgSO40.2 g/L、MnSO40.05 g/L、鹽酸半胱氨酸0.5 g/L,番茄汁250 mL/L,調(diào)節(jié)pH值至4.8,ATB固體培養(yǎng)基中加20 g/L的瓊脂,121℃滅菌15 min。
嗜酸乳桿菌的培養(yǎng)采用MRS培養(yǎng)基:蛋白胨10 g、酵母提取物5 g、牛肉膏10 g、葡萄糖20 g、乙酸鈉5 g、檸檬酸二銨2 g、吐溫-80 1 mL、MgSO40.58 g、MnSO40.05 g、K2HPO42 g,水1 000 mL。固體培養(yǎng)基加瓊脂粉17 g,121℃,1×105Pa滅菌20 min。
1.2試劑與儀器
DNA聚合酶 日本TaKaRa公司;內(nèi)切酶HindⅢ、MseⅠ,T4 DNA連接酶Fermentas公司;DNA聚合酶天根生化科技(北京)有限公司。
凝膠成像分析系統(tǒng) 英國(guó)Syngene公司;Veriti梯度基因擴(kuò)增儀 美國(guó)ABI公司;NanoDrop1000核酸蛋白測(cè)定儀 美國(guó)Thermo公司;JY 3000型電泳儀、JY-CX 2B型測(cè)序電泳槽 北京君意東方電泳設(shè)備有限公司。
1.3方法
1.3.1細(xì)菌的培養(yǎng)
1.3.1.1O. oeni的培養(yǎng)
固體培養(yǎng):在28℃厭氧箱培養(yǎng)箱中充CO2培養(yǎng),培養(yǎng)時(shí)間為5~7 d。液體培養(yǎng):28℃靜置培養(yǎng)72 h。
1.3.1.2嗜酸乳桿菌的培養(yǎng)
固體培養(yǎng):培養(yǎng)皿密封、倒置于37℃恒溫培養(yǎng)箱培養(yǎng)30 h。液體培養(yǎng):37℃靜置培養(yǎng)48 h[18]。
1.3.2細(xì)菌總DNA的提取
參照文獻(xiàn)中的十六烷基三甲基溴化銨(cetyl trimethyl ammonium bromide,CTAB)法和溶菌酶法[19]。
1.3.3O. oeni的種特異性PCR擴(kuò)增
參照Z(yǔ)apparoli等[8]設(shè)計(jì)的引物On1:5’-TAATGTGG TTCTTGAGGAGAAAAT-3’和On2:5’-ATCATCGTCAAA CAAGAGGCCTT-3’,引物由生工生物工程(上海)股份有限公司合成。反應(yīng)體系(2 0 ?L):10×PCR Buffer 2.0 ?L,dNTPs 1.6 ?L(2.5 mmol/L),Mg2+1.6 ?L(25 mmol/L),上下游引物各0.8 ?L(10 ?mol/L),模板DNA 1.0 ?L,DNA聚合酶0.1 ?L(5 U/?L),加ddH2O至終體積為20 ?L。反應(yīng)條件:94℃預(yù)變性2 min;94℃變性45 s,64℃退火2 min,72℃延伸2 min,30個(gè)循環(huán)。最后72℃延伸10 min。PCR產(chǎn)物上樣到1.0%的瓊脂糖凝膠上100 V電壓下電泳40 min,溴化乙錠染色,用凝膠成像分析系統(tǒng)照相。
1.3.4細(xì)菌16S rRNA序列分析
參照Klijn等[20]的引物P1(41~60:5’-GCGGCGTG C C T A A T A C A T G C-3’)和P 3(6 8 6~7 0 5:5’-ATCTACGCATTTCACCGCTA-3’),引物由生工生物工程(上海)股份有限公司合成。反應(yīng)體系(50 ?L):10×PCR Buffer 5.0 ?L、dNTPs 4 ?L(2.5 mmol/L)、Mg2+5 ?L(25 mmol/L)、上下游引物各5 ?L(10 ?mol/L)、模板DNA 2.0 ?L、DNA聚合酶0.3 ?L(5 U/?L),加ddH2O至終體積為50 ?L。反應(yīng)條件:94℃預(yù)變性5 min;94℃變性1 min,50℃退火1 min,72℃延伸1.5 min,35個(gè)循環(huán);最后72℃延伸10 min。PCR產(chǎn)物瓊脂糖凝膠電泳檢測(cè)后對(duì)合格樣品送南京金斯瑞生物科技有限公司進(jìn)行純化和測(cè)序。
1.3.5O. oeniAFLP分析方法的建立
1.3.5.1 DNA提取方法的建立
DNA模板的純度和質(zhì)量對(duì)AFLP的成功起著關(guān)鍵作用,實(shí)驗(yàn)中優(yōu)化和比較了CTAB法和溶菌酶法兩種常見(jiàn)的細(xì)菌DNA提取方法。
1.3.5.2雙酶切反應(yīng)體系的建立
通過(guò)實(shí)驗(yàn)優(yōu)化,獲得了O. oeni的HindⅢ和MseⅠ酶切和連接反應(yīng)體系:在20 ?L反應(yīng)體系中加入約200 ng基因組DNA、5 UMseⅠ和2 ?L 10×Buffer R,65℃酶切4 h,冷卻后加入10 UHindⅢ、MseⅠ接頭(5’-GACGATGAGTCCTGAG-3’;5’-TACT CAGGACTCAT-3’)各0.5 ?L(10 ?mol/L)、HindⅢ接頭(5’-CTCGTAGACTGCGTACC-3’;5’-AGCTGGTACGCAGTC-3’)各0.25 ?L(10 ?mol/L)、1 ?L ATP、10 U T4 DNA連接酶、1 ?L Buffer R、3 ?L T4 DNA連接酶Buffer,加ddH2O至30 ?L,37℃酶切鏈接4 h。產(chǎn)物稀釋5倍用于預(yù)擴(kuò)增反應(yīng)。
1.3.5.3 PCR擴(kuò)增反應(yīng)體系優(yōu)化
通過(guò)實(shí)驗(yàn)優(yōu)化反應(yīng)條件得到的預(yù)擴(kuò)增反應(yīng)體系(25 ?L):10×PCR Buffer 2.5 ?L、d N T P s 0.5 ?L(2.5 m m o l/L)、引物H 0(5’-G A C T G C G T A C C A G C T T-3’)1 ?L(10 ?mol/L)、M0(5’-GATGAGTCCTGAGTAA- 3’)1 ?L(10 ?mol/L)、模板4 ?L(酶切連接產(chǎn)物稀釋5倍)、D N A聚合酶0.6 ?L(2.5 U/?L)。反應(yīng)條件:94℃2 min;94℃30 s,56℃60 s,72℃1 min,30個(gè)循環(huán);最后72℃延伸7 min。預(yù)擴(kuò)增產(chǎn)物稀釋20倍用于選擇性擴(kuò)增。
通過(guò)實(shí)驗(yàn)優(yōu)化反應(yīng)條件得到的選擇性擴(kuò)增反應(yīng)體系(25 ?L):10×PCR Buffer 2.5 ?L、dNTPs 0.5 ?L(2.5 mmol/L)、引物Hx 1 ?L(10 ?mol/L)、My 1 ?L(10 ?mol/L)(引物組合見(jiàn)表2)、模板2.0 ?L(預(yù)擴(kuò)增產(chǎn)物稀釋20倍)、DNA聚合酶0.6 ?L(2.5 U/?L)。反應(yīng)條件:94℃2 min,66℃30 s(每個(gè)循環(huán)下降1℃),72℃2 min,10個(gè)循環(huán)。然后94℃20 s,56℃20 s,72℃2 min,20個(gè)循環(huán)。最后60℃延伸30 min。
1.3.5.4選擇性擴(kuò)增產(chǎn)物的聚丙烯酰胺凝膠電泳(polyacrylamide gel electrophoresis,PAGE)與銀染
選擇性擴(kuò)增產(chǎn)物加入1/3體積的變性上樣緩沖液(98%去離子甲酰胺、10 mmol/L pH 8.0乙二胺四乙酸、0.25%溴酚藍(lán)、0.25%二甲苯氰)混勻后95℃變性5 min,立即置于冰上。取5 ?L樣品于6%的PAGE上80 W恒功率進(jìn)行電泳檢測(cè),電泳結(jié)束后采用硝酸銀染色法顯色[21]。
1.3.5.5O. oeniAFLP引物組合的篩選
為篩選出O. oeniAFLP擴(kuò)增效率高、多態(tài)性好的引物組合,16對(duì)引物組合被用于O. oeni菌株的擴(kuò)增和分析。比較不同引物組合擴(kuò)增的條帶數(shù)以及多態(tài)性位點(diǎn)數(shù)。
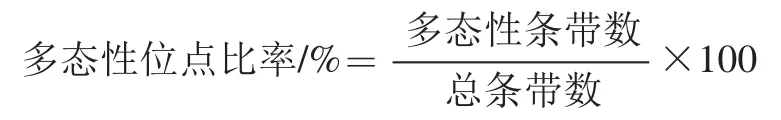
1.3.5.6中國(guó)葡萄酒產(chǎn)區(qū)篩選的O. oeni的AFLP分析
對(duì)從我國(guó)葡萄酒主產(chǎn)區(qū)篩選的22株O. oeni菌株進(jìn)行AFLP分析,了解我國(guó)葡萄酒產(chǎn)區(qū)酒酒球菌的遺傳多樣性。
1.4數(shù)據(jù)分析
對(duì)變性聚丙烯酰胺凝膠電泳圖譜中擴(kuò)增的條帶進(jìn)行人工統(tǒng)計(jì),按照等位基因有條帶的記為1,無(wú)條帶的記為0的方法,將AFLP指紋圖譜轉(zhuǎn)換成1和0構(gòu)成的數(shù)字矩陣,輸入到Microsoft Excel表格中。用NTSYS 2.10軟件進(jìn)行數(shù)據(jù)分析,用軟件中的SHAN程序和非加權(quán)配對(duì)算術(shù)平均法(unweighted pair-group method with arithmetic means,UPGMA)進(jìn)行聚類(lèi)分析,生成22株O. oeni遺傳相似性系數(shù)表,并通過(guò)系統(tǒng)發(fā)育樹(shù)生成聚類(lèi)圖。
2 結(jié)果與分析
2.1細(xì)菌的種特異性PCR鑒定
依據(jù)Zapparoli等[8]設(shè)計(jì)的引物,22株菌株都能擴(kuò)增得到一條長(zhǎng)1 025 bp的片段(圖1),陰性對(duì)照嗜酸乳桿菌(Lactobacillus acidophilus)不能擴(kuò)增出條帶。

圖1 222 株O..oeni的種特異性PPCCRR鑒定Fig.1 Identification of 22 O. oeni by species-specific PCR
2.2細(xì)菌的16S rRNA序列分析
使用擴(kuò)增細(xì)菌16S rRNA基因序列41~705 bp的一對(duì)引物,對(duì)22株細(xì)菌進(jìn)行16S rRNA測(cè)序,分析測(cè)序報(bào)告并完成序列處理,得到約600 bp的細(xì)菌16S rRNA基因序列。將這些序列與GenBank數(shù)據(jù)庫(kù)里的O. oeni菌株IOB 8413(登錄號(hào):AJ831548)的16S rRNA基因序列進(jìn)行比較,同源性為99.8%~100%。通過(guò)利用序列分析軟件ClustalX 1.83進(jìn)行比對(duì)分析22株分離菌及O. oeniIOB 8413的16S rRNA基因的73~650 bp序列,發(fā)現(xiàn)除SD-2d在76~77 bp處插入了堿基“C”外,其余堿基全部相同。這說(shuō)明O. oeni種內(nèi)菌株間16S rRNA基因差異小。
種特異性PCR和16S rRNA基因序列分析表明22 株細(xì)菌為O. oeni。
2.3O. oeniAFLP分析方法的建立
2.3.1適于O. oeniAFLP分析的DNA提取方法的建立

表1 兩種DNA提取方法所得DNA質(zhì)量濃度和純度的比較Table1 Concentrations and purities of DNA samples extracted by two different methods

圖2 O. oeni oeni兩種提取方法提取的DNA的PAGE結(jié)果AGEFig.2 Electrophoresis photograph of O. oeni DNA extracted by two different methods
本研究?jī)?yōu)化并比較了溶菌酶法和CTAB法兩種方法對(duì)O. oeniDNA的提取效果,結(jié)果表明溶菌酶法提取的O. oeniDNA濃度高、純度較差;CTAB法提取的DNA濃度低、純度很好(表1,圖2)。對(duì)于AFLP技術(shù),酶切連接的效果直接決定了AFLP是否成功,由于后續(xù)需要對(duì)基因組DNA進(jìn)行酶切鏈接,DNA提取物里的雜質(zhì)會(huì)嚴(yán)重影響酶切連接效果,所以高純凈度的基因組DNA對(duì)于AFLP的成功與否是起決定性作用的。而且,AFLP技術(shù)不需要很高的模板濃度,所以CTAB法提取的基因組DNA可適用于AFLP分析,后續(xù)的研究也表明CTAB法提取的基因組DNA酶切連接效果比溶菌酶法更穩(wěn)定。
2.3.2O. oeniAFLP分析引物組合的篩選
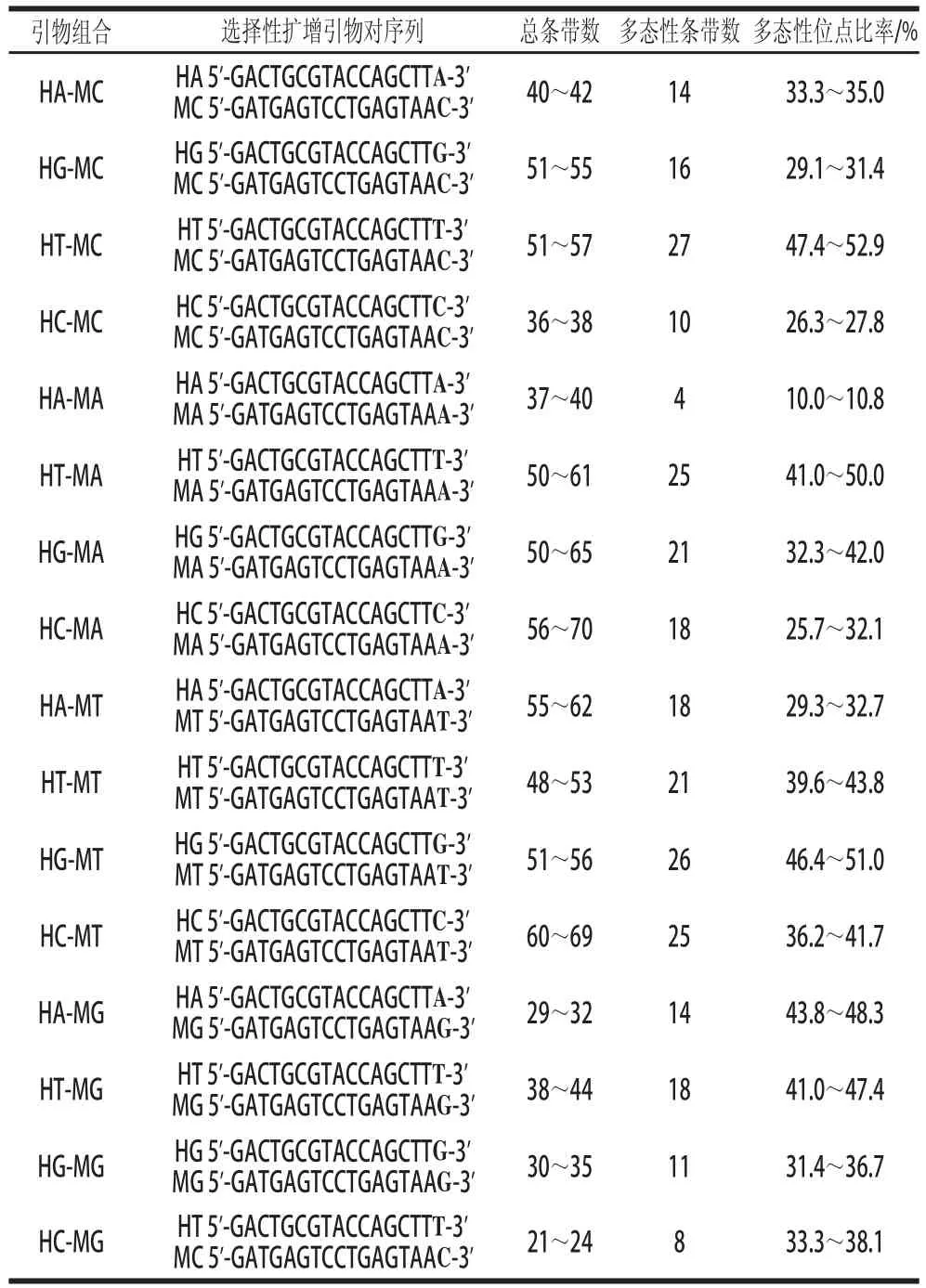
表2 16 對(duì)引物組合的序列及其相應(yīng)的O. oeni AFLP擴(kuò)增總條帶數(shù)和多態(tài)性比率Table2 Average number of fragments and polymorphism bands obtained from 16 pairs of selective primers and sequences to detect AFLPs among O. oeni strains isolated from Chinese wines
16對(duì)引物被用于O. oeni的AFLP分析,結(jié)果表明不同引物組合擴(kuò)增得到的總條帶數(shù)和多態(tài)性條帶數(shù)差別很大。引物組合中HT-MA、HG-MA、HC-MA、HA-MT、HC-MT能夠擴(kuò)增出最多的總條帶數(shù),其中HC-MA擴(kuò)增的條帶最多,達(dá)到最多70條,HC-MG擴(kuò)增的總條帶數(shù)最少,最少為21條。引物組合HT-MC、HT-MA、HT-MT、HG-MT、HC-MT、HA-MG、HG-MG具有較高的多態(tài)性位點(diǎn)比率,其中HT-MC多態(tài)性位點(diǎn)比率最高達(dá)到52.9%,HA-MA多態(tài)性位點(diǎn)比率最低,最低達(dá)到10%。綜合分析總條帶數(shù)和多態(tài)性位點(diǎn)比率,引物組合HT-MA、HT-MT、HT-MC、HG-MA、HG-MT、HC-MT為O. oeniAFLP分析的最佳引物組合(表2)。重復(fù)性實(shí)驗(yàn)表明,這些引物組合在O. oeniAFLP分析中的重復(fù)性為98%以上。
2.3.3中國(guó)葡萄酒產(chǎn)區(qū)篩選的O. oeni的AFLP分析
引物組合HC-MT被用于22株O. oeni的AFLP分析。由圖3可知,22株O. oeni的最小遺傳相似系數(shù)僅為0.45,表明不同產(chǎn)區(qū)O. oeni的種內(nèi)遺傳多樣性豐富。在相似性系數(shù)0.565處,22株O. oeni可分為3個(gè)簇群,第一簇群包括:HB-1a、SD-1g、SX-1a、HB-2b、SX-1b、SD-2ji、SD-1f、SD-1c、SD-2kj、SD-1b、HB-1c分離自河北、陜西、山東的11株O.oeni菌株。這些菌株間的遺傳相似性系數(shù)差異很大,遺傳多樣性豐富,其中HB-1a、SD-1g、SX-1a、HB-2b、SX-1b具有較高的相似性形成一個(gè)亞簇,且亞簇內(nèi)菌株間遺傳相似性系數(shù)差異也較大;第二簇群包括:SD-2gh、SD-2gf、SD-1a、SD-2d、SD-1e、HB-1b、SD-2b、SD-2a、SD-1d、SD-2ee分離自河北、山東的10株O. oeni,菌株間遺傳相似性系數(shù)差異小,遺傳多樣性單一;第三簇群僅有SD-2h一株,其與其他兩個(gè)簇群的相似系數(shù)僅有0.45。研究結(jié)果表明AFLP在O. oeni基因分型研究中具有很高的分辨率。
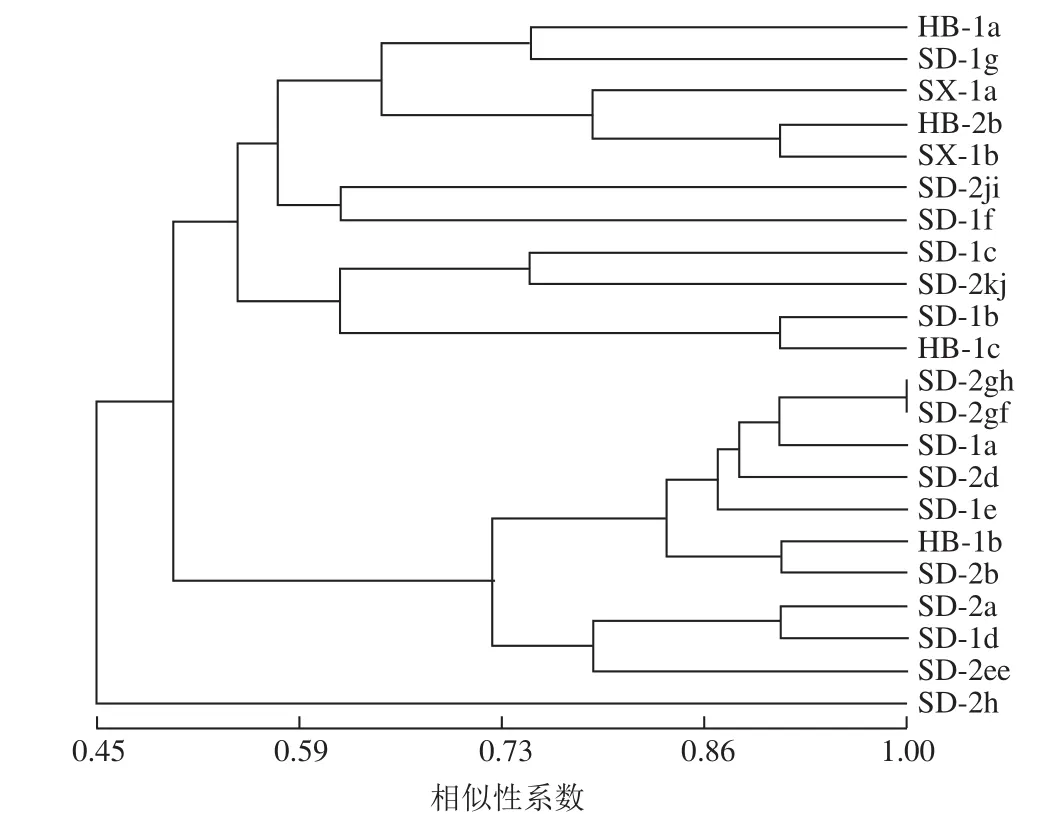
圖3 中國(guó)葡萄酒產(chǎn)區(qū)O. oeni的AFLP指紋圖譜聚類(lèi)分析Fig.3 UPGMA dendrogram derived from AFLP patterns of 22 O. oeni isolated from Chinese wines
3 討 論
本研究采用了目前最為高效和可靠的方法對(duì)從中國(guó)葡萄酒產(chǎn)區(qū)分離的22株O. oeni進(jìn)行了鑒定。同時(shí)首次發(fā)展了采用HindⅢ和MseⅠ兩種限制性內(nèi)切酶進(jìn)行酶切的高重現(xiàn)性的O. oeni的AFLP分析方法。
種特異性PCR和16S rRNA序列分析相結(jié)合是目前從種的水平上對(duì)O. oeni進(jìn)行鑒定最高效和可靠的方法,被廣泛應(yīng)用于O. oeni的鑒定。Lòpez等[22]從204株葡萄酒分離菌株中鑒定得到201株O. oeni;Cappello等[5]從葡萄酒中分離得到220株乳酸菌,鑒定出其中87株為O. oeni。本研究從107株葡萄酒分離菌株中鑒定得到22株O. oeni。
由于O. oeni菌株間差異較小,所以靈敏并且可靠的菌株區(qū)分方法對(duì)于O. oeni的分類(lèi)鑒定非常重要。一些經(jīng)典的分子生物學(xué)方法已經(jīng)被應(yīng)用于O. oeni的分類(lèi)鑒定研究中。在這些方法中,PFGE顯示出強(qiáng)大的菌株區(qū)分能力[23-24],但是PFGE對(duì)操作者的技術(shù)和設(shè)備有較高要求,而且其工作量很大,這些限制了其的廣泛應(yīng)用[25-26]。RAPD技術(shù)快速、實(shí)驗(yàn)成本低并且容易操作,然而PCR擴(kuò)增的固有缺陷使得RAPD的實(shí)驗(yàn)結(jié)果在不同實(shí)驗(yàn)室間重現(xiàn)性差[17,27]。AFLP是一項(xiàng)非常有用的技術(shù),它集合了PFGE和RAPD的優(yōu)點(diǎn),被證明是細(xì)菌包括乳酸菌種內(nèi)菌株間基因分型的最佳技術(shù)[26,28-29]。到目前為止,在有限的O. oeniAFLP研究中仍然使用傳統(tǒng)的EcoRⅠ和MseⅠ兩種內(nèi)切酶,但研究表明,EcoRⅠ和MseⅠ作為識(shí)別富含A+T序列的內(nèi)切酶,更適用于分析G+C含量低的基因組DNA。在常用的限制性內(nèi)切酶中,HindⅢ顯示出適用于消化G+C含量在40%~50%(物質(zhì)的量百分比)左右的基因組DNA,MseⅠ則被選用于消化G+C含量低于50%的基因組DNA,O. oeni基因組的G+C含量在40%左右,所以HindⅢ和MseⅠ會(huì)是分析O. oeni基因組的完美組合。研究結(jié)果也表明以HindⅢ和MseⅠ為內(nèi)切酶的O. oeniAFLP分析技術(shù)具有高擴(kuò)增效率、高分辨率、高重現(xiàn)性的特點(diǎn),而且以聚丙烯酰胺凝膠電泳和銀染體系作為檢測(cè)手段的AFLP技術(shù)對(duì)設(shè)備要求不高,適合于絕大多數(shù)實(shí)驗(yàn)室應(yīng)用。
目前,AFLP的檢測(cè)手段已發(fā)展為毛細(xì)管電泳檢測(cè),采用專(zhuān)門(mén)的軟件進(jìn)行數(shù)據(jù)分析,這大大提高了AFLP分析的效率,所以以HindⅢ和MseⅠ為內(nèi)切酶的AFLP分析技術(shù)是分析O. oeni遺傳多樣性的有效工具。
本研究中,22株O. oeni菌株可分為3個(gè)簇群,第一簇群包含的菌株最多,簇群內(nèi)菌株間遺傳多樣性豐富,可能跟菌株來(lái)源的多樣性有關(guān)(分別來(lái)源河北、陜西、山東)。第二簇群內(nèi)菌株間遺傳多樣性單一,可能跟菌株來(lái)源單一有關(guān)(90%來(lái)源于山東)。第三簇群只有一株來(lái)源于山東煙臺(tái)的SD-2h,其與其他21株菌株之間遺傳相似性系數(shù)較低,遺傳距離較遠(yuǎn),說(shuō)明來(lái)源于同一地區(qū)的菌株間也有可能遺傳差異很大,可能跟葡萄與葡萄酒微生態(tài)有相關(guān)性。
葡萄酒酒精發(fā)酵結(jié)束后,不同品種,甚至于不同發(fā)酵罐的葡萄酒酒精體積分?jǐn)?shù)、溫度、營(yíng)養(yǎng)物質(zhì)都不盡相同,這些微生態(tài)環(huán)境會(huì)影響葡萄酒中細(xì)菌的種類(lèi)、數(shù)量、細(xì)菌不同基因的表達(dá)等,進(jìn)而影響葡萄酒的品質(zhì)。所以需要更深入的研究來(lái)揭示生態(tài)地理和葡萄酒生態(tài)與釀酒微生物之間的關(guān)系。
參考文獻(xiàn):
[1] 李華. 現(xiàn)代葡萄酒工藝學(xué)[M]. 2版. 西安∶ 陜西人民出版社, 2000∶68-80.
[2] CLAIRE L M, ELISABETH B, ALINE L F. Tolerance to high osmolality of the lactic acid bacterium Oenococcus oeni and identification of potential osmoprotectants[J]. International Journal of Food Microbiology, 2007, 115(3)∶ 335-342.
[3] RENOUF V, DELAHERCHE A, CLAISSE O, et al. Correlation between indigenous Oenococcus oeni strain resistance and the presence of genetic markers[J]. Journal of Industrial Microbiology & Biotechnology, 2008, 35(1)∶ 27-33.
[4] du PLESSIS H W, DICKS L M T, PRETORIUS I S, et al. Identification of lactic acid bacteria isolated from South African brandy base wines[J]. International Journal of Food Microbiology, 2004, 91(1)∶ 19-29.
[5] CAPPELLO M S, STEFANI D, GRIECO F, et al. Genotyping by amplified fragment length polymorphism and malate metabolism performances of indigenous Oenococcus oeni strains isolated from Primitivo wine[J]. International Journal of Food Microbiology, 2008, 127(3)∶ 241-245.
[6] ZAVALETA A J, MARTINEZ-MURCIA A J, RODRI GUEZVALERA F. Intraspecific genetic diversity of Oenococcus oeni as derived from DNA fingerprinting and sequence analyses[J]. Applied and Environmental Microbiology, 1997, 63(4)∶ 1261-1267.
[7] HIRSCHH?USER S, FR?HLICH J, GNEIPEL A, et al. Fast protocols for the 5S rDNA and ITS-2 based identification of Oenococcus oeni[J]. FEMS Microbiology Letters, 2005, 244(1)∶ 165-171.
[8] ZAPPAROLI G, TORRIANI S, PESENTE P, et al. Design and evaluation of malolactic enzyme gene targeted primers for rapid identification and detection of Oenococcus oeni in wine[J]. Letters in Applied Microbiology, 1998, 27(5)∶ 243-246.
[9] MARIA A S, MARIA G B, GIOVANNI S. Isolation and characterization of Oenococcus oeni from Aglianico wines[J]. World Journal of Microbiology & Biotechnology, 2008, 24(9)∶ 1829-1835.
[10] ZAPPAROLI G, REGUANT C, BORDONS A, et al. Genomic DNA fingerprinting of Oenococcus oeni strains by pulsed-field gel electrophoresis and randomly amplified polymorphic DNA-PCR[J]. Current Microbiology, 2000, 40(6)∶ 351-355.
[11] LI Hua, ZHANG Chunhui, LIU Yanlin. Species attribution and distinguishing strains of Oenococcus oeni isolated from Chinese wines[J]. World Journal of Microbiology & Biotechnology, 2006, 22(5)∶ 515-518.
[12] de las RIVAS B, MARCOBAL A, MU?OZ R. Allelic diversity and population structure in Oenococcus oeni as determined from sequence analysis of housekeeping genes[J]. Applied and Environmental Microbiology, 2004, 70(12)∶ 7210-7219.
[13] DELAHERCHE A, BON E, DUP? A, et al. Intraspecific diversity of Oenococcus oeni strains determined by sequence analysis of target genes[J]. Applied Microbiology and Biotechnology, 2006, 73(2)∶ 394-403.
[14] MELANIE L, HARALD C, HELMUT K. Pulsed-field gel electrophoresis for the discrimination of Oenococcus oeni isolates from different wine-growing regions in Germany[J]. International Journal of Food Microbiology, 2008, 123(1/2)∶ 171-176.
[15] CAPPELLO M S, ZAPPAROLI G, STEFANI D, et al. Molecular and biochemical diversity of Oenococcus oeni strains isolated during spontaneous malolactic fermentation of Malvasia Nera wine[J]. Systematic and Applied Microbiology, 2010, 33(8)∶ 461-467.
[16] CAPPELLO M S, STEFANI D, LOGRIECO A, et al. Bio-molecular characterisation of indigenous Oenococcus oeni strains from Negroamaro wine[J]. Food Microbiology, 2014, 42∶ 142-148.
[17] VOS P, HOGERS R, BLEEKER M, et al. AFLP∶ a new technique for DNA fingerprinting[J]. Nucleic Acids Research, 1995, 23(21)∶4407-4414.
[18] 張帆, 王建華, 劉立恒, 等. 嗜酸乳桿菌的培養(yǎng)條件及其生物學(xué)特性[J].食品與發(fā)酵工業(yè), 2005, 31(3)∶ 43-46.
[19] 金剛. 中國(guó)葡萄酒主產(chǎn)區(qū)酒酒球菌的鑒定及SE-AFLP分析[D]. 楊凌∶ 西北農(nóng)林科技大學(xué), 2010∶ 17.
[20] KLIJN N, WEERKAMP A H, de VOS W G. Detection and characterization of lactose utilizing Lactococcus subsp. in natural ecosystems[J]. Applied and Environmental Microbiology, 1995, 61(2)∶788-792.
[21] 盧圣棟. 現(xiàn)代分子生物學(xué)實(shí)驗(yàn)技術(shù)[M]. 2版. 北京∶ 中國(guó)協(xié)和醫(yī)科大學(xué)出版社, 1999∶ 101-104.
[22] L?PEZ I, TENORIO C, ZARAZAGA M, et al. Evidence of mixed wild populations of Oenococcus oeni strains during wine spontaneous malolactic fermentation[J]. European Food Research and Technology, 2007, 226(1/2)∶ 215-223.
[23] LECHIANCOLE T, BLAIOTTA T, MESSINA D, et al. Evaluation of intra-specific diversities in Oenococcus oeni through analysis of genomic and expressed DNA[J]. Systematic and Applied Microbiology, 2006, 29(5)∶ 375-381.
[24] VIGENTINI I, PICOZZI C, TIRELLI A, et al. Survey on indigenous Oenococcus oeni strains isolated from red wines of Valtellina, a cold climate wine-growing Italian area[J]. International Journal of Food Microbiology, 2009, 136(1)∶ 123-128.
[25] GIAMMANCO G M, MAMMINA C, ROMANI C, et al. A Evaluation of a modified single-enzyme amplified fragment length polymorphism (SE-AFLP) technique for subtyping Salmonella enterica serotype Enteritidis[J]. Research in Microbiology, 2007, 158(1)∶ 10-17.
[26] DIMITROV Z P, MINKOVA S, MICHAYLOVA M. Comparative evaluation of three molecular typing methods in their applicability to differentiate Lactobacillus strains with human origin[J]. World Journal of Microbiology & Biotechnology, 2008, 24(8)∶ 1305-1312.
[27] SALMINEN S, ISOLAURI E, SALMINEN E. Clinical uses of probiotics for stabilizing the gut mucosal barrier∶ successful strains and future challenges[J]. Antonie Van Leeuwenhoek, 1996, 70(2/4)∶ 347-358.
[28] RICO A, ORTIZ-BARREDO A, RITTER E, et al. Genetic characterization of Erwinia amylovora strains by amplified fragment length polymorphism[J]. Journal of Applied Microbiology, 2004, 96(2)∶ 302-310.
[29] HONG Y, GARCA M, LEVISOHN S, et al. Differentiation of Mycoplasma gallisepticum strains using amplified fragment length polymorphism and other DNA-based typing methods[J]. Avian Diseases, 2005, 49(1)∶ 43-49.
Genetic Diversity Analysis of Oenococcus oeni Strains Isolated from Chinese Wines
JIN Gang1, WANG Hua1,2,3, ZHANG Ang4, LI Hua1,2,3,*
(1. College of Enology, Northwest A&F University, Yangling 712100, China; 2. Heyang Experimental Demonstration Station, Northwest A&F University, Heyang 715300, China; 3. Shaanxi Engineering Research Center for Viti-Viniculture, Yangling 712100, China; 4. Qinhuangdao Entry-Exit Inspection and Quarantine Bureau, Qinhuangdao 066004, China)
Oenococcus oeni(O. oeni)is considered as the key processer of malolactic fermentation (MLF), which will influence wine quality. Indigenous microorganisms are increasingly considered as one of the factors influencing the quality of individual wines. In order to understand the genetic diversity ofO. oeni, species-specific PCR and 16S rRNA sequence were used to identify 22 bacterial strains isolated from wines from different producing regions in China. Amplified fragment length polymorphism (AFLP) based onHindIIIandMseI was developed to analyze the genotypes of 22O. oenistrains.The results of species-specific PCR and 16S rRNA sequence analysis showed that the 22 isolates wereO. oeni. Double restriction enzyme digestion and ligation system, PCR reaction system, polyacrylamide gel electrophoresis were developed to analyzeO. oeni. The combinations of 16 AFLP primers were tested. The results showed that HT-MA, HT-MT, HT-MC, HG-MA, HG-MT, and HC-MT were the optimal primer combinations for analyzingO. oeni. The 22O. oenistrains were analyzed by AFLP, and cluster analysis indicated that the 22O. oenistrains fell into three groups and their genetic similarity was low. In conclusion, AFLP is a good method to genotypeO. oeniby usingHindIIIandMseI. TheO. oenistrains in China have very rich genetic diversity. The genetic similarity ofO.oenistrains is related to their ecological geographic distribution and other factors.
Oenococcus oeni; species-specific polymerase chain reaction; 16S rRNA sequence; amplified fragment length polymorphism (AFLP)
TS261.1
1002-6630(2015)17-0134-06
10.7506/spkx1002-6630-201517026
2015-01-13
國(guó)家自然科學(xué)基金面上項(xiàng)目(31471708)
金剛(1984—),男,博士研究生,研究方向?yàn)獒劸莆⑸铩-mail:gjinwine@hotmail.com
*通信作者:李華(1959—),男,教授,博士,研究方向?yàn)槠咸雅c葡萄酒學(xué)。E-mail:lihuawine@nwsuaf.edu.cn
- 食品科學(xué)的其它文章
- “互聯(lián)網(wǎng)+”潮中百舸爭(zhēng)流,調(diào)味品業(yè)內(nèi)究竟何為?
——2015第十一屆調(diào)味品博覽會(huì)營(yíng)銷(xiāo)峰會(huì)11月下旬將在上海召開(kāi) - 第二屆中國(guó)食品科技成果交流會(huì)暨第五屆中國(guó)食品產(chǎn)業(yè)產(chǎn)學(xué)研創(chuàng)新發(fā)展高峰論壇
- 我國(guó)水產(chǎn)品安全風(fēng)險(xiǎn)來(lái)源與風(fēng)險(xiǎn)評(píng)估研究進(jìn)展
- 食源性蛋白中免疫活性肽的研究進(jìn)展
- 轉(zhuǎn)基因組學(xué)分析技術(shù)研究進(jìn)展
- 多酚類(lèi)化合物對(duì)糖脂代謝影響的研究進(jìn)展

