下調受精卵中Clock基因對早期小鼠胚胎DNA甲基化的影響*
成姝婷 梁鑫 王正榮 李世平 劉延友 汪宇輝 江舟 肖靜 郭慧玲
(四川大學華西基礎醫學與法醫學院生物醫學工程學研究室,四川 成都 610041)
在哺乳動物中,至少有兩種不同的方式來反映局部染色質結構的信息,一種是通過對那些在DNA 復制過程中起作用的某些組蛋白特定殘基甲基化[1,2];另一種是通過對那些能夠復制DNA 的特定胞嘧啶殘基甲基化[3,4]。這兩種方式的印記都能夠很容易地復制給新合成的DNA 鏈,并且一代一代的傳遞下去,也就是所謂的表觀遺傳學修飾。表觀遺傳學修飾包括DNA 甲基化和組蛋白修飾,通過改變局部的染色質結構而調節基因表達,從而改變染色質和DNA 結合蛋白之間的相互作用,如轉錄激活子和抑制子與基因啟動子和增強子之間的結合[5]。不同于遺傳變異,表觀遺傳學修飾調節基因表達不會改變其核苷酸序列。值得注意的是,人們在對表觀遺傳學修飾的最初研究里并沒有把其與時序調節聯系在一起,因為通常都認為這些印記高度穩定,甚至傳代也不會改變。一直以來,研究人員對近日節律分子機制的研究通常也都是針對以轉錄/轉錄后反饋環路為基礎的這個復雜遺傳網絡[6,7]。然而,近期的研究證明,組蛋白和DNA 甲基化遠比從前所想的要活躍得多。例如:超過100個DNA 甲基化位點為了與細胞周期同步而發生近日震蕩[8],而細胞周期也常常受到近日生物鐘的控制[9,10]。這些研究讓研究者們開始認識到了表觀遺傳學調節與近日節律系統之間的聯系。DNA 甲基化一般是在胞嘧啶的CpG 位點上加上一個甲基團。這是哺乳動物胚胎發育接受表觀遺傳調節的一個重要環節。研究表明,DNA 甲基化對于動物胚胎發育階段的基因調節,特別是組織特異性基因,具有至關重要的作用[11]。值得注意的是,除了印記基因外,哺乳動物基因組的表觀遺傳學修飾在早期胚胎發育階段會被消除,并在之后的整個胚胎發育階段得到重建[12~14]。目前大多數對DNA 甲基化的研究,都是關于配子生成、發育、疾病和干細胞功能的,這些研究闡明了DNA 甲基化是如何調節基因表達和細胞分化的[15~18]。而關于節律基因在胚胎基因組DNA 甲基化中所起作用的研究則非常少。
本研究應用RNAi技術下調從二細胞期到囊胚期這段時間內小鼠受精卵中Clock 基因的表達,研究了7.5~11.5dpc之間的小鼠胚胎基因組DNA 甲基化水平的改變,以及在哺乳動物催化發育過程中去甲基化的CpG 位點重新甲基化的主要甲基轉移酶Dnmt3a和Dnmt3b。本研究結果顯示,Clock 基因干擾的小鼠胚胎基因組DNA 甲基化水平比陰性對照組顯著升高,而甲基轉移酶Dnmt3a和Dnmt3b的RNA 表達水平也顯著增加了。因此,這些結果為我們提供了一個新的信息,那就是節律基因Clock 能夠通過甲基轉移酶Dnmt3a和Dnmt3b調節早期小鼠胚胎基因組DNA 的甲基化水平。
1 材料與方法
1.1 材料
1.1.1 動物 SPF級ICR 雄性小鼠(6~8周)30只,雌鼠(4~6周)65只,由成都達碩實驗動物有限公司提供。所有雄鼠單只每籠,雌鼠5 只每籠,均單獨飼養于12L∶12D 光暗循環箱中,自由進食和飲水。
1.1.2 主要儀器 倒置生物顯微鏡TE 2000-U(日本,Nikon),CO2恒溫細胞孵箱(日本,SANYO),高效液相色譜儀(美國,VARIAN Pro Star),PCR 儀(德國,Eppendorf),電泳儀及水平電泳槽(美國,BIORAD),超凈細胞工作臺、凝膠成像儀、恒溫振蕩培養箱、微量注射器及眼科剪、眼科鑷等均為國產。
1.1.3 主要試劑 孕馬血清促性腺激素PMSG、注射用絨促性腺激素HCG(寧波市激素制品有限公司),Clock干擾質粒和HK 陰性對照質粒(武漢市晶賽生物工程技術有限公司),DMEM 高糖培養液、Opti-MEMI培養液和胎牛血清(美國,HyClone),LipofectamineTM2000細胞轉染試劑、Trizol(美國,Invitrogen),基因組DNA 提取試劑盒(美國,OMEGA),核酸酶P1、堿性磷酸酶、2′-脫氧胞苷dC 和5-甲基-2′-脫氧胞苷mdC(美國,Sigma),第一鏈cDNA 合成試劑盒、Taq酶(美國,Fermentas),其它試劑為國產分析純。
1.2 方法
1.2.1 獲得胚胎樣本 本實驗為了對小鼠受精卵中Clock基因的表達進行干擾,特采用人工授精(IVF)的方法先在體外得到受精卵,轉染干擾質粒后,再將其移植回假孕雌鼠子宮中以得到所需胚齡(dpc)的小鼠胚胎。
1.2.1.1 人工授精 在進行IVF前64小時,對每只實驗用雌鼠腹腔注射10IU 的PMSG,48小時后再對這些雌鼠腹腔注射10IU 的HCG。頸椎脫臼法處死40只雌鼠,取出輸卵管,放進裝有0.5ml T6 培養液(含20mg/ml BSA)的培養皿內,撕開膨大的壺腹部,釋放出卵丘卵母細胞復合體,將其轉移到mCZB 受精滴中,放入37℃的CO2孵箱中孵育得到成熟卵子。頸椎脫臼法處死20只雄鼠,摘取附睪,用眼科剪刀將每段附睪尾剪成4 段,輕輕擠壓出精液,然后將其放置在37℃的CO2孵箱中孵育30 分鐘,讓精子充分游出,然后將表面皿中的精子懸液移至1.5ml的EP 管中,繼續放置在37℃的CO2孵箱中孵育60分鐘以獲能。調整精子濃度到1×106/ml,每滴含有成熟卵子的受精滴加入10μl獲能的小鼠精子懸液,繼續放回37℃的CO2孵箱中孵育6小時,觀察并分離出受精滴中的二細胞受精卵,以備后續實驗。
1.2.1.2 質粒干擾 將分離出的受精卵隨機分為Clock干擾組(Clock組)和陰性對照組(HK 組)兩組,以LipofectamineTM2000細胞轉染試劑分別將Clock干擾質粒和HK 陰性對照質粒轉染到二細胞受精卵中。將兩組轉染后的受精卵放置在37℃的CO2孵箱中孵育到第5天得到囊胚期胚胎。
1.2.1.3 囊胚移植 在囊胚移植前20天,將10只雄鼠行輸精管結扎術,14天后與25只促排卵雌鼠交配獲得假孕雌鼠。將假孕5 天的受體雌鼠用水合氯醛麻醉后,剔除其后腹部的毛并用75%酒精消毒,作一個長約1cm 的切口,輕輕牽拉出兩側子宮角,用4號針頭在子宮角壁上刺一小孔,分別把12 個胚胎注入兩側子宮角,將子宮角放回腹腔后縫合肌肉、皮膚。
1.2.1.4 獲取胚胎 從7.5dpc開始,每天頸椎脫臼法處死Clock干擾組和HK 對照組的受孕雌鼠各2只,剝離其子宮內的小鼠胚胎,冷凍于-80℃低溫冰箱,到11.5dpc結束。
1.2.2 胚胎基因組DNA 甲基化水平 本實驗參照《分子克隆實驗指南》中檢測DNA 甲基化的標準方法,先將DNA 樣品水解成堿基,水解產物通過液相色譜柱,所得結果與標準品相比較,并用紫外光測定其吸收峰值及出峰面積,計算mdC/(mdC+dC)的積分面積,從而得到基因組整體的甲基化水平。
1.2.2.1 提取DNA 使用OMEGA 基因組DNA 提取試劑盒(D3396-02E.Z.N.A.TM Tissue DNA Kit)提取7.5~11.5dpc胚胎的DNA。
1.2.2.2 水解DNA 將DNA 樣本濃度調節到0.5μg/μl,每個樣本吸取3μl開水浴2min 后快速冷卻,加入0.75μl ZnSO4(10mM)和1.25μl核酸酶P1,混勻后在37℃孵 育16 小 時;再加入1.25μl Tris(0.5M,pH 8.3)和0.75μl堿性磷酸酶[50U/ml溶于2.5M 的(NH4)2SO4],37℃孵育2小時,離心后取上清。
1.2.2.3 高效液相色譜法檢測 本實驗采用25cm的C18反相色譜柱,柱溫設定為35℃,泵A 緩沖液為甲醇,泵B緩沖液為0.05M KH2PO4(pH 4.0);流動相為8%甲醇+92%0.05M KH2PO4(pH 4.0),流速為1ml/min,UV 波長為280nm,運行時間為30min。標準品采用mdC 和dC,注入樣品為水解DNA 所得單核苷。樣本中甲基化的定量以總胞苷中mdC 的百分比來計算。
1.2.3 甲基轉移酶水平 用酚:氯仿法提取7.5~11.5dpc胚胎的RNA,使用Fermentas的第一鏈cDNA 合成試劑盒將其逆轉錄為cDNA。引物序列如下:Dnmt3a:5′-TCACAAGGAAGTTTACACCGAC-3′和5′-GAGGCTCCCACATGAGATACA-3′;Dnmt3b:5′-CGCTCAAGGAGTTGGGTATT-3′和5′-TCTTTGCGGGCAGGATT-3′;Gapdh:5′-TCACTGCCACCCAGAAGA-3′和5′-AAG TCG CAG GAG ACA ACC-3′。電泳PCR 的擴增產物,用PCR 儀自帶的分析軟件讀取條帶灰度值,并以Gapdh為參照,比較這段時間內兩種甲基轉移酶的表達變化。
2 結果
2.1 小鼠胚胎獲取結果 人工授精實驗中觀察并分離出的二細胞受精卵共528枚。分別轉染Clock干擾質粒和HK 陰性對照質粒后,有37 枚沒有發育到囊胚;另隨機抽取10枚檢測轉染效果,剩余481枚受精卵移植到假孕雌鼠子宮內。25只造模的雌鼠僅得到20只假孕受體。Clock 組和HK 組所得到的7.5~11.5dpc胚齡的小鼠胚胎數量見表1。
2.2 受精卵中Clock 基因下調對早期胚胎基因組DNA 甲基化水平的影響 我們應用高效液相色譜法得到了Clock 組和HK 組胚胎基因組DNA 中mdC和dC堿基的出峰面積,并通過公式:計算得到基因組整體的甲基化水平。結果發現,除了7.5dpc之外,從8.5~11.5dpc 的DNA 甲基 化水平均是Clock組顯著高于HK 組(P<0.05),見圖1。
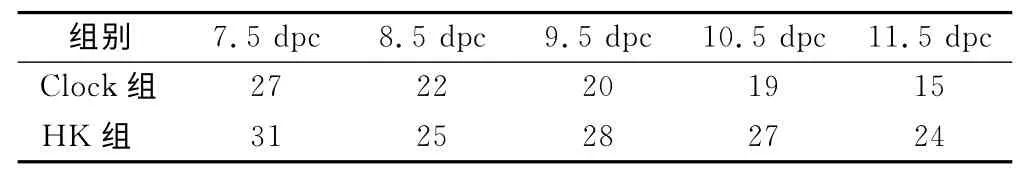
表1 兩組小鼠取得各胚齡(dpc)胚胎數量(個)Table 1 Embryonic numbers in each dpc of the two groups
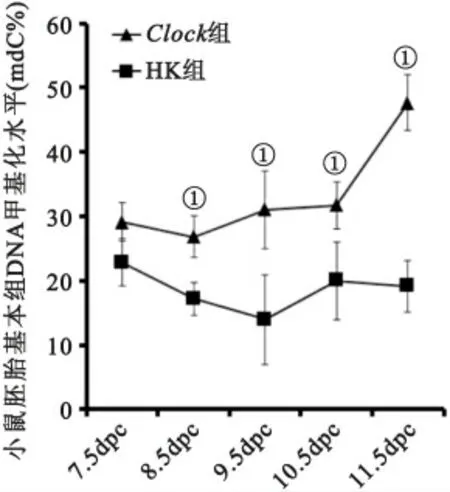
圖1 小鼠胚胎基因組DNA甲基化水平變化Figure 1 Methylation level changes of genomic DNA in mice embryos
2.3 干擾受精卵Clock基因表達對早期胚胎中DNA甲基轉移酶的影響 Clock組小鼠胚胎中甲基轉移酶Dnmt3a的表達從8.5dpc開始到11.5dpc均顯著高于HK 組(P<0.05),而其在7.5dpc時則無顯著差異(P>0.05),見圖2A。而甲基轉移酶Dnmt3b則從7.5~11.5dpc均是Clock組顯著高于HK 組(P<0.05),見圖2B。
3 討論
目前已有研究證明,近日節律系統與哺乳動物的生殖存在千絲萬縷的聯系。本實驗室在前期研究中已經證實干擾雄性小鼠圓形精子中Clock基因的表達后,可以引起雄鼠的生殖能力降低,這種影響不僅表現在與雄鼠交配的雌鼠胎仔數的減少、體外受精實驗中囊胚形成率的降低,還表現在其子代胚胎發育過程中早期吸收胎的增多[19]。早期胚胎發育的過程包含了許多重要細胞命運的決定,還有染色質狀態的改變。在這段期間,多種表觀遺傳學機制的運行改變了染色質的結構,使得某些基因的表達激活,而另一些基因的表達沉默[20]。通常情況下,發育中的胚胎DNA 甲基化模式在個體的整個發育階段以及之后的成年時期都會保持穩定[11]。然而個別位點DNA 甲基化的改變是存在的,并且這種改變能夠影響某些在發育和病理過程中具有重要生物功能的基因表達[15,21],如全基因組范圍或是特定基因位點5-甲基胞嘧啶水平的變化會增加疾病的易感性,DNA 甲基化的失調與心血管疾病、Ⅱ型糖尿病以及腫瘤有密切的關聯[21~23]。
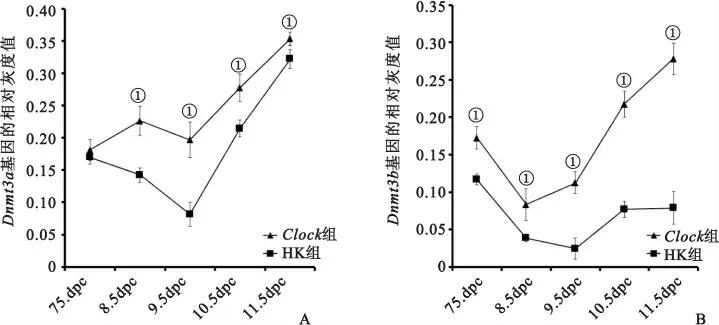
圖2 甲基轉移酶Dnmt3a和Dnmt3b在RNA水平的表達變化Figure 2 Expression changes of methyltransferase Dnmt3aand Dnmt3bat RNA level
本實驗通過人工授精技術在動物體外得到了二細胞受精卵,以RNAi干擾技術下調其Clock 基因的表達,再將其培育到囊胚并移植到假孕受體雌鼠體內。我們對收獲的7.5~11.5dpc的小鼠胚胎整體進行基因組DNA 甲基化水平的檢測,結果發現Clock干擾組胚胎的甲基化水平顯著高于對照組(P <0.05)。這就說明二細胞受精卵中的Clock 基因對于小鼠胚胎的早期甲基化是具有影響的。DNA 甲基化就是一個將甲基團共價結合到哺乳動物基因組CpG位點上的胞嘧啶殘基上的過程。這個過程需要一組被稱為DNA 甲基轉移酶(Dnmts)的酶類參與[24]。由于哺乳動物催化發育過程中去甲基化的CpG 位點重新甲基化的主要甲基轉移酶是Dnmt3a 和Dnmt3b[20],所以我們又用PCR 檢測了這些胚胎中Dnmt3a和Dnmt3b的RNA 表達水平。結果發現,這兩種酶的變化與其DNA 甲基化水平的變化具有相似性,且也是Clock 組顯著高于對照組(P<0.05)。因此,我們認為二細胞受精卵中Clock 基因可以通過調節甲基轉移酶Dnmt3a和Dnmt3b對小鼠早期胚胎基因組DNA 甲基化水平造成影響,它們之間具體的作用機制仍需要進一步研究。
4 結論
在本實驗中,我們利用RNAi技術干擾了小鼠二細胞受精卵中Clock 基因的表達,并成功將這些處理后的受精卵移植到假孕雌鼠子宮內,從而得到了由這些受精卵發育而來的各胚齡(7.5~11.5dpc)小鼠胚胎。對這些胚胎基因組DNA 甲基化水平和甲基轉移酶的檢測結果顯示,Clock干擾組無論是基因組DNA甲基化水平還是甲基轉移酶Dnmt3a、Dnmt3b的表達量均較對照組高。由此我們認為,受精卵中Clock 基因表達的下調可以使得甲基轉移酶Dnmt3a和Dnmt3b的表達量增加,進而導致其早期胚胎(7.5~11.5dpc)基因組DNA 甲基化水平上升。
[1]Fischle W,Wang Y,Allis C D.Histone and chromatin crosstalk[J].Current opinion in cell biology,2003,15(2):172-83.
[2]Kikuchi H,Barman H K,Nakayama M,et al.Participation of histones,histone modifying enzymes and histone chaperones in vertebrate cell functions[J].Sub-cellular biochemistry,2006,40:225-243.
[3]Klose R J,Bird AP.Genomic DNA methylation:the mark and its mediators[J].Trends in biochemical sciences,2006,31(2):89-97.
[4]Bird AP,Wolffe AP.Methylation-induced repression--belts,braces,and chromatin[J].Cell,1999,99(5):451-454.
[5]Goldberg AD,Allis CD,Bernstein E.Epigenetics:a landscape takes shape[J].Cell,2007,128(4):635-638.
[6]Ko CH,Takahashi JS.Molecular components of the mammalian circadian clock[J].Hum Mol Genet,2006,15(2):271-277.
[7]Young MW,Kay SA.Time zones:a comparative genetics of circadian clocks[J].Nature reviews Genetics,2001,2(9):702-715.
[8]Brown SE,Fraga MF,Weaver IC,et al.Variations in DNA methylation patterns during the cell cycle of HeLa cells[J].Epigenetics :official journal of the DNA Methylation Society,2007,2(1):54-65.
[9]Merrow M,Roenneberg T.Cellular clocks:coupled circadian and cell division cycles[J].Current biology:CB,2004,14(1):25-26.
[10]Nagoshi E,Saini C,Bauer C,et al.Circadian gene expression in individual fibroblasts:cell-autonomous and self-sustained oscillators pass time to daughter cells[J].Cell,2004,119(5):693-705.
[11]Reik W.Stability and flexibility of epigenetic gene regulation in mammalian development[J].Nature,2007,447(7143):425-432.
[12]Howlett SK,Reik W.Methylation levels of maternal and paternal genomes during preimplantation development[J].Development,1991,113(1):119-127.
[13]Olek A,Walter J.The pre-implantation ontogeny of the H19 methylation imprint[J].Nature genetics,1997,17(3):275-276.
[14]Okano M,Bell DW,Haber DA,et al.DNA methyltransferases Dnmt3aand Dnmt3bare essential for de novo methylation and mammalian development[J].Cell,1999,99(3):247-257.
[15]Movassagh M,Choy MK,Goddard M,et al.Differential DNA methylation correlates with differential expression of angiogenic factors in human heart failure [J].PloS one,2010,5(1):e8564.
[16]Takahashi K,Tanabe K,Ohnuki M,et al.Induction of pluri-potent stem cells from adult human fibroblasts by defined factors[J].Cell,2007,131(5):861-872.
[17]Takahashi K,Yamanaka S.Induction of pluripotent stem cells from mouse embryonic and adult fibroblast cultures by defined factors[J].Cell,2006,126(4):663-676.
[18]Ueda Y,Okano M,Williams C,et al.Roles for Dnmt3bin mammalian development:a mouse model for the ICF syndrome[J].Development,2006,133(6):1183-1192.
[19]Liang X,Cheng S,JIANG X,et al.The noncircadian function of the circadian Clock gene in the regulation of male fertility[J].J Biol Rhythms,2013,28(3):208-217.
[20]Geiman TM,Muegge K.DNA methylation in early development[J].Mol Reprod Dev,2010,77(2):105-113.
[21]Shahrzad S,Bertrand K,Minhas K,et al.Induction of DNA hypomethylation by tumor hypoxia [J].Epigenetics:official journal of the DNA Methylation Society,2007,2(2):119-125.
[22]Watson JA,Watson CJ,Mccrohan AM,et al.Generation of an epigenetic signature by chronic hypoxia in prostate cells[J].Human molecular genetics,2009,18(19):3594-3604.
[23]Waterland RA,Michels KB.Epigenetic epidemiology of the developmental origins hypothesis[J].Annual review of nutrition,2007,27:363-388.
[24]Gopalakrishnan S,Van Emburgh BO,Robertson KD.DNA methylation in development and human disease[J].Mutation research,2008,647(1-2):30-38.

