基于網絡藥理學的姜黃素抗炎作用機制研究
趙萱, 傅超美,甘彥雄,何瑤
基于網絡藥理學的姜黃素抗炎作用機制研究
趙萱, 傅超美,甘彥雄,何瑤
目的:闡明姜黃素的抗炎機制。方法:通過基因本體論(GO)以及復合分子檢測法(MCODE),建立的姜黃素蛋白質相互作用網絡具有482個節點與1688個作用點。結果:其中兩個模塊與炎癥密切相關,姜黃素的抗炎機制可能與SMAD、ERG和調節TLR系統有關,特別是TLR9作用最為顯著。結論:TLR9可能為姜黃素起抗炎作用的蛋白質作用靶點。
姜黃素;功能模塊;蛋白質相互作用;抗炎機制;生物信息學
姜黃(Curcuma longa Linn.)與莪術(Curcuma phaeocaulis Val.)作為傳統常用中藥,已有上千年的臨床應用歷史,許多古籍中有詳細記載其具有活血化瘀、通絡止痛的功效。姜黃素是姜黃、莪術等姜黃屬植物的主要活性成分之一。現代研究表明,姜黃素具有抗炎[1]、抗癌[2]、抗氧化[3]以及神經保護等作用。其中炎癥與心血管疾病關系密切[4],表現為腫脹與疼痛。同時,姜黃素的抗炎機制研究有助于明確中藥在治療炎癥有效性的機理。因此,本文將借助網絡模塊及基因本體論(GO)的分析方法,系統的闡釋姜黃素的抗炎機制。
蛋白質在生物機體內執行著各式各樣的功能,且相互間合作密切。信號蛋白通常形成活性蛋白質對,實現多元功能、構成細胞間的信號通路以及細胞形態發育[5]。蛋白質相互作用(PPIs)是許多生物過程的關鍵,而基因本體論(GO)是目前被認為對于蛋白質相互作用的詮釋的最有力的指標[6]。
在本研究中,我們將利用網絡藥理學的研究方法來分析姜黃素的抗炎機制。使用PPIs來構建生物網絡,并分析其無標度、小范圍的網絡模塊特性。通過模塊化網絡分析,闡釋姜黃素的抗炎機制。
1 方法
1.1 網絡構建
靶點信息來源于ChEMBL (https://www.ebi.ac.uk/ chembl/#)和STITCH4.0 (http://stitch.embl.de/)數據庫。ChEMBL是一個有關藥物及具藥物類小分子屬性的生物活性分子的化學數據庫[7],其數據主要從原始文獻中提取,然后進一步的優化數據,使得網站數據的品質得到最優保障。STITCH[8]是一個研究化學物質與蛋白質相互作用的數據庫,它集成了眾多實驗資源,挖掘文獻信息及預測相互作用來構建數據庫。
蛋白質相互作用信息從實時更新的網絡數據庫String 9.1(http://string-db.org)獲取,該網站用于檢索對于靶點的預期作用[9]。在String 9.1中的所有可獲取的關聯數據將有信度評分。靶點的置信度大于0.7時將被篩選應用于PPI網絡的建立。
1.2 網絡分析
拓撲信息在獲取可視化合成組織與結構的復雜網絡時被廣泛應用[10]。因此,我們將用Cytoscape軟件中的network analyzer[11]分析聚類系數、連接組件、度分布、平均最短路徑等參數。基于拓撲參數,并與隨機網絡相比,分析網絡無標度、小范圍和模塊化等特性。
復合分子檢測法(MCODE)將被用來進一步對PPI進行模塊的劃分,(網絡中蛋白質)節點連接度的臨界值大于3。此算法優于其他具有微調功能但卻不能兼顧網絡其他部分的圖聚類制導模式方法,并且允許檢查蛋白質集群的相互關聯性[12]。基于識別模塊,運用Cytoscape軟件中的BingoBingo插件[13]進行GO富集分析,當超幾何檢驗閾值P小于0.05時,基因本體論的注釋功能與全面分析功能將被很好的展示。
2 結果與討論
2.1 網絡的構建
從STITCH 4.0提取了10個靶點,從ChEMBL提取了68個靶點。去掉重復靶點,共得到67個姜黃素靶點。姜黃素對ALPI與TLR9的有效劑量分別為100μM、8.36μM。因為姜黃素會抑制或激活其他蛋白,剩余靶點的IC50無法獲取[13]。表1中列出了靶點的信息。PPI的靶點信息導入Cytoscape[14]。然后進行union計算和去重復,選擇相關性最大的子圖作為姜黃素的蛋白質網絡,網絡包含482個節點與1688個邊,見圖1。節點代表蛋白質,邊則表示蛋白質間的相互作用關系。灰色節點表示種子節點,藍色則為與種子節點相互作用的節點。由于目前研究的局限性,一些人體蛋白質相互作用仍不清楚。所以網絡構建的研究并不全面,因此選擇最大連通子圖進行研究。
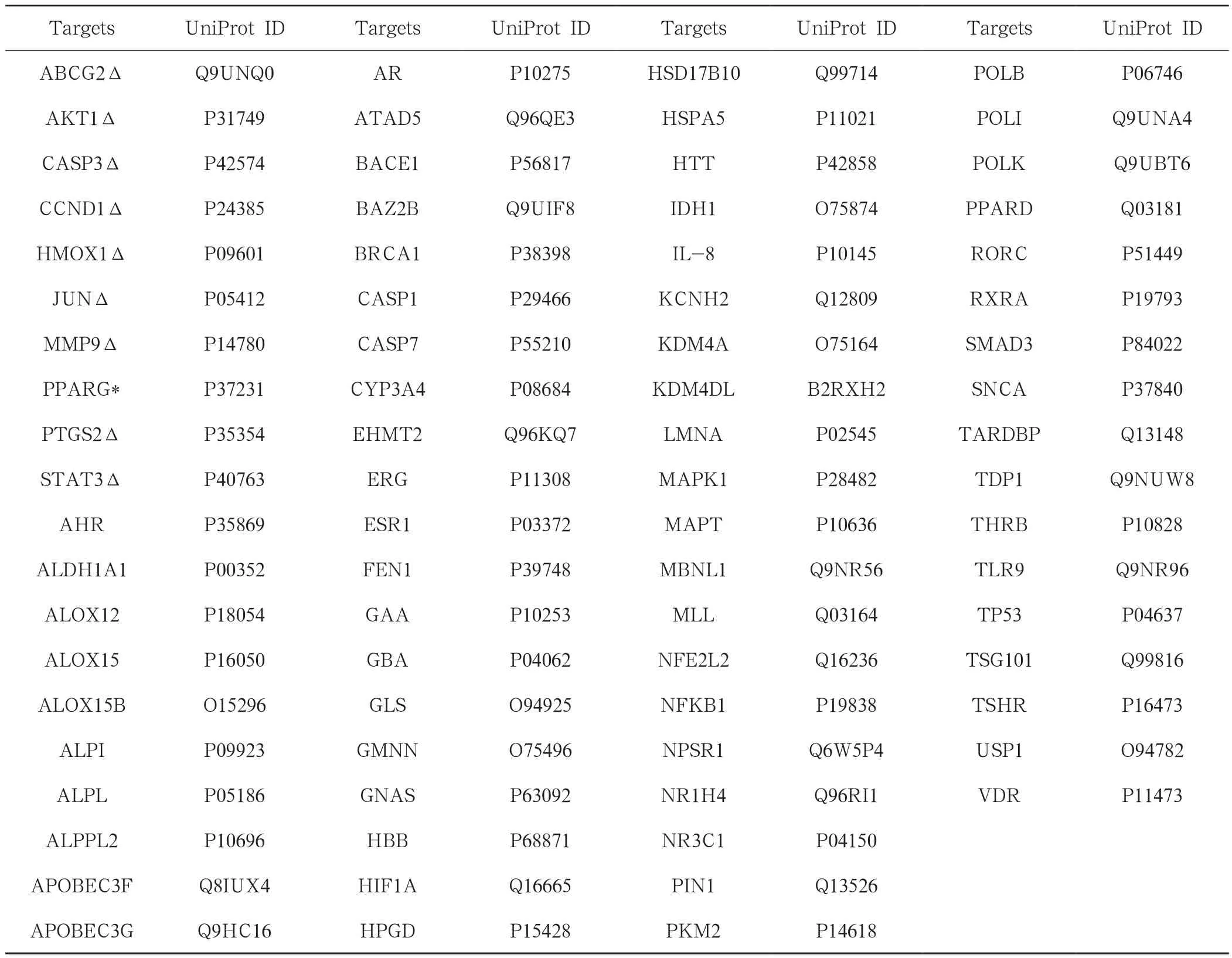
表1 姜黃素的靶點信息表
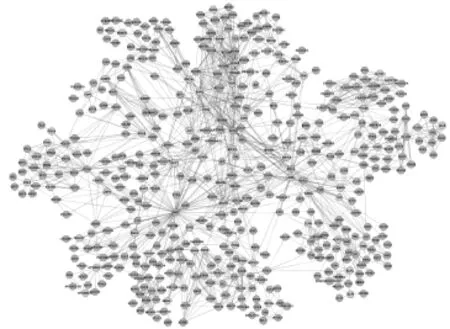
圖1 姜黃素的蛋白相互作用網絡
2.2 網絡分析
2.2.1 拓撲分析
計算得到的拓撲參數信息見表2。

表2 關于姜黃素蛋白質相互作用網絡與隨機網絡的參數
通過對網絡中各種蛋白質間的關聯數統計,用計算機求得度分布[15]。姜黃素的PIN度分布遵循冪律分布,直線方程為y=218.67x-1.359,如圖2A。表明姜黃素的PIN為無標度網絡。
平均最短路徑指的是所有成對節點之間最短路徑的平均密度[16]。如圖2B,網絡路徑長度主要是集中在3-5步。任意兩個蛋白質之間的最短路徑長度為4.394。大多數蛋白質的聯系非常密切,表明姜黃素的PIN具有小世界性質。
聚類系數是指節點的平均密度的區域[17]。聚類系數越高,網絡的模塊化程度越高。對比姜黃素PIN節點數目與邊數目相同的隨機網絡,姜黃素的PIN聚類系數更高,表明姜黃素具有模塊化的性質。綜上所述,姜黃素PIN網絡具有無標度、小范圍和模塊化的體系結構。
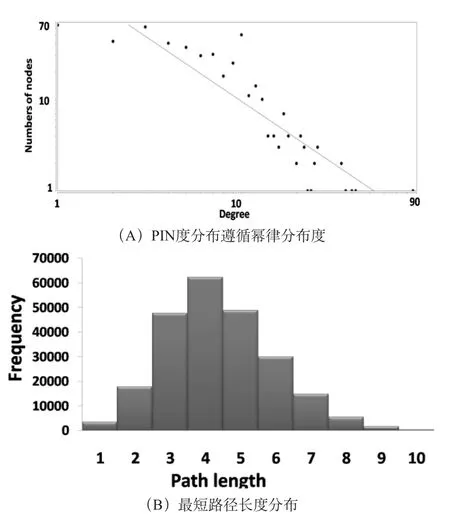
圖2 姜黃素蛋白質相互作用網絡與隨機網絡的參數
2.2.2 集群和基因本體論的富集分析
如圖3所示,通過MCODE算法,獲得19個模塊。灰色節點表示源節點,其他為連接節點。
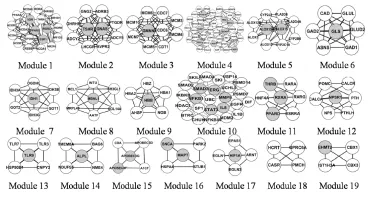
圖3 GO 生物過程的模塊顯示部分
使用Bingo功能富集分析的結果如表3所示。結果表明,姜黃素在生物過程發揮了藥效學作用,如轉錄調節、細胞周期、凝血酶的反向調節、過氧化氫代謝、抗炎作用等過程。其中模塊10與模塊13與抗炎作用有關。

表3 GO生物過程的模塊顯示部分
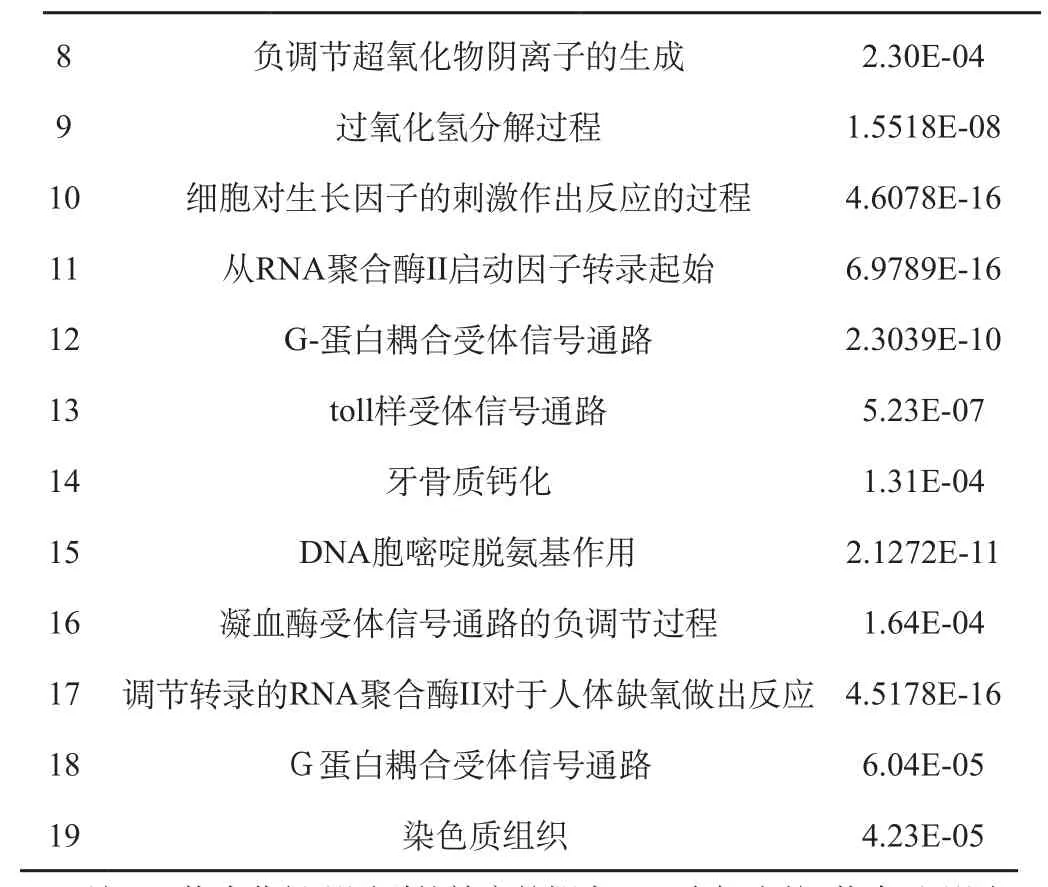
注:P 值為獲得觀測到的效應的概率,一個極小的P值表示觀測到的效應極有可能純屬偶然,因此對無效的假設提供了證據。
模塊10含有的蛋白質如IL-8、NF-κB、STAT3、SMAD3、ERG等。IL-8是引發局部炎癥的關鍵參數[18]。核因子kappa B(NF-κB) 是炎癥反應中的一個關鍵信號分子。STAT3被激活后可應對包括IL - 6、IL -10等各種細胞因子和生長因子。已有報道稱,姜黃素通過減少IL-8水平[19]、作為NF-κB抑制劑[20]、抑制STAT3[21]從而起到抗炎作用。因此,在一定程度上證明網絡模塊的分析方法結果的可靠性。SMAD3的表達與激活蛋白激酶(MAPK)有關[22]。姜黃素可以通過抑制MAPK而表現出抗炎的活性[23]。所以姜黃素的抗炎活性可能與SMAD3的表達有關。ERG是紅細胞完成特定轉換(ETS)中的轉錄因子之一,可用于調節炎癥[23]。C-jun可通過雙磷酸化被氨基末端激酶(JNK)通道激活,將會導致炎癥,ERG也被證明與C-jun具有相互作用[24]。同時,姜黃素可通過抑制JNK的活性發揮抗炎作用[25]。因此,姜黃素的抗炎作用可能與ERG有關。模塊10的分析表明,姜黃素的抗炎作用可能與SMAD3與ERG相關。
模塊13與toll樣受體家族密切相關,包括TLR3,TLR7,TLR9,TLR3[26]可誘導激活IRF3,最終誘導的I型干擾素(IFNS)的產生。IFNS可以激活信號傳感器與轉錄復合物(STATs),啟動了Janus kinase-STAT(JAK-STAT)信號通路)。有資料顯示JAK-STAT通路參與了抗炎反應[27]。TLR7和TLR9識別也使激活的細胞開始促炎反應,導致I型干擾素等細胞因子的產生[28]。此外,TLR9為種子節點,姜黃素對其有效劑量(IC50)為8.36 μM。因此,通過調節TRL蛋白群,姜黃素可能發揮抗炎作用,對于姜黃素治療炎癥TLR9可能是一個潛在的靶點。
3 結論
中藥的療效來源于歷史悠久的臨床實踐,為保障中華民族的健康作出了重大貢獻。隨著現代研究的不斷深入,越來越多的作用機制會被闡明。尤其對于像ChEMBL和STITCH這樣的數據庫而言,可為我們提供大量的可供快速篩選的蛋白質信息。
本研究基于網絡拓撲結構的無標度、小世界性質、模塊化性質,研究了姜黃素蛋白質相互作用。進而提出了一種模塊化網絡分析方法來研究姜黃素的抗炎機制。最終得到結論,姜黃素的抗炎機制可能與SMAD蛋白家族、ERG蛋白家族以及TLR調節有關。TLR9可能是姜黃素發揮抗炎作用的潛在靶點。此外,本研究還是運用模塊化蛋白網絡分析的例證。顯然,對于預測目標化合物,這是一個行之有效的研究方法。
[1] Jurenka JS. Anti-infammatory properties of curcumin, a major constituent of curcuma longa: A review of preclinical and clinical research [J]. Alternative medicine review : a journal of clinical therapeutic, 2009,14(2):141.
[2] Wilken R, Veena MS, Wang MB, et al. Curcumin: A review of anti-cancer properties and therapeutic activity in head and neck squamous cell carcinoma [J]. Molecular cancer, 2011,10.
[3] Sun YM, Zhang HY, Chen DZ, et al. Theoretical elucidation on the antioxidant mechanism of curcumin: A dft study [J]. Organic letters, 2002,4(17):2909.
[4] Libby P. Inflammation and cardiovascular disease mechanisms [J]. The American journal of clinical nutrition, 2006,83(2):456s.
[5] Wang Z, Nakayama T. Inflammation, a link between obesity and cardiovascular disease [J]. Mediators of inflammation, 2010,535.
[6] Yeh JT, Binari R, Gocha T, et al. Papti: A peptide aptamer interference toolkit for perturbation of protein-protein interaction networks [J]. Scientifc reports, 2013,3:1156.
[7] Wang T, Ming Z, Xiaochun W, et al. Rab7: Role of its protein interaction cascades in endo-lysosomal traffic [J]. Cellular signalling, 2011,23(3):516.
[8] Maetschke SR, Simonsen M, Davis MJ, et al. Gene ontologydriven inference of protein-protein interactions using inducers [J]. Bioinformatics, 2012,28(1):69.
[9] Ashburner M, Ball CA, Blake JA, et al. Gene ontology: Tool for the unifcation of biology. The gene ontology consortium [J]. Nature genetics, 2000,25(1):25.
[10] Zhang QC, Petrey D, Deng L, et al. Structure-based prediction of protein-protein interactions on a genome-widescale [J]. Nature, 2012,490(7421):556.
[11] Ren Z, Wang X, Wang S, et al. Mechanism of action of salvianolic acid b by module-based network analysis [J]. Biomedical materials and engineering, 2014,24(1):1333.
[12] Gaulton A, Bellis LJ, Bento AP, et al. Chembl: A large-scale bioactivity database for drug discovery [J]. Nucleic acids research, 2012,40(Database issue):D1100.
[13] Kuhn M, Szklarczyk D, Pletscher-Frankild S, et al. Stitch 4: Integration of protein-chemical interactions with user data [J]. Nucleic acids research, 2014,42(Database issue):D401.
[14] Franceschini A, Szklarczyk D, Frankild S, et al. String v9.1: Protein-protein interaction networks, with increased coverage and integration [J]. Nucleic acids research, 2013,41(Database issue):D808.
[15] Albert R. Scale-free networks in cell biology [J]. Journal of cell science, 2005,118(Pt 21):4947.
[16] Almaas E. Biological impacts and context of network theory [J]. The Journal of experimental biology, 2007,210(Pt 9):1548.
[17] Barabasi AL, Oltvai ZN. Network biology: Understanding the cell's functional organization [J]. Nature reviews Genetics, 2004,5(2):101.
[18] Bader GD, Hogue CW. An automated method for finding molecular complexes in large protein interaction networks [J]. BMC bioinformatics, 2003,4.
[19] Shannon P, Markiel A, Ozier O, et al. Cytoscape: A software environment for integrated models of biomolecular interaction networks [J]. Genome research, 2003,13(11):2498.
[20] Jin W, Wang J, Zhu T, et al. Anti-inflammatory effects of curcumin in experimental spinal cord injury in rats [J]. Inflammation research : official journal of the European Histamine Research Society [et al], 2014,63(5):381.
[21] Kloesch B, Becker T, Dietersdorfer E, et al. Antiinflammatory and apoptotic effects of the polyphenol curcumin on human fibroblast-like synoviocytes [J]. International immunopharmacology, 2013,15(2):400.
[22] Assenov Y, Ramirez F, Schelhorn SE, et al. Computing topological parameters of biological networks [J]. Bioinformatics, 2008,24(2):282.
[23] Manke T, Demetrius L, Vingron M. Lethality and entropy of protein interaction networks [J]. Genome informatics International Conference on Genome Informatics, 2005,16(1):159.
[24] Przulj N. Biological network comparison using graphlet degree distribution [J]. Bioinformatics, 2007,23(2):e177.
[25] Estrada E. Virtual identifcation of essential proteins within the protein interaction network of yeast [J]. Proteomics, 2006,6(1):35.
[26] Vlahopoulos S, Boldogh I, Casola A, et al. Nuclear factor-kappab-dependent induction of interleukin-8 gene expression by tumor necrosis factor alpha: Evidence for an antioxidant sensitive activating pathway distinct from nuclear translocation [J]. Blood, 1999,94(6):1878.
[27] Klawitter M, Quero L, Klasen J, et al. Curcuma dmso extracts and curcumin exhibit an anti-infammatory and anticatabolic effect on human intervertebral disc cells, possibly by infuencing tlr2 expression and jnk activity [J]. Journal of infammation, 2012,9(1):29.
[28] Shehzad A, Ha T, Subhan F, et al. New mechanisms and the anti-inflammatory role of curcumin in obesity and obesityrelated metabolic diseases [J]. European journal of nutrition, 2011,50(3):151.
[29] Ross KR, Corey DA, Dunn JM, et al. Smad3 expression is regulated by mitogen-activated protein kinase kinase-1 in epithelial and smooth muscle cells [J]. Cellular signalling, 2007,19(5):923.
[30] Patwardhan RS, Checker R, Sharma D, et al. Dimethoxycurcumin, a metabolically stable analogue of curcumin, exhibits anti-inflammatory activities in murine and human lymphocytes [J]. Biochemical pharmacology, 2011,82(6):642.
[31] Murakami K, Mavrothalassitis G, Bhat NK, et al. Human erg-2 protein is a phosphorylated DNA-binding protein--a distinct member of the ets family [J]. Oncogene, 1993,8(6):1559.
[32] Verger A, Buisine E, Carrere S, et al. Identifcation of amino acid residues in the ets transcription factor erg that mediate erg-jun/fos-DNA ternary complex formation [J]. The Journal of biological chemistry, 2001,276(20):17181.
[33] Alexopoulou L, Holt AC, Medzhitov R, et al. Recognition of double-stranded rna and activation of nf-kappab by toll-like receptor 3 [J]. Nature, 2001,413(6857):732.
[34] Platanias LC. Mechanisms of type-i- and type-ii-interferonmediated signalling [J]. Nature reviews Immunology, 2005,5(5):375.
[35] Busch-Dienstfertig M, Gonzalez-Rodriguez S. Il-4, jak-stat signaling, and pain [J]. Jak-stat, 2013,2(4):e27638.
[36] Akira S, Uematsu S, Takeuchi O. Pathogen recognition and innate immunity [J]. Cell, 2006,124(4):783-801.
(責任編輯:何瑤)
Research on the anti-infammatory mechanisms of Curcumin based on network pharmacology
ZHAO Xuan, FU Chaomei, GAN Yan-xiong, HE Yao//(School of Pharmacy, Chengdu University of Traditional Chinese Medicine; Key Laboratory of Standardization for Chinese Herbal Medicine, Ministry of Education; National Key Laboratory Breeding Base of Systematic Research, Development and Utilization of Chinese Medicine Resources, Chengdu 611137, Sichuan)
Objective:To elucidate the anti-infammatory mechanism of curcumin based on the analysis of protein interaction network (PIN).Method:Through the Gene ontology (GO) enrichment based on Molecular complex detection (MCODE), a PIN of curcumin with 482 nodes and 1688 interactions was created and analyzed.Result:Two modules were found to be intimately associated with infammation. In addition, the anti-infammatory effect of curcumin may be related to SMAD, ERG and mediate TLR family.Result:TLR9 might be a potential target of curcumin to treat infammation.
curcumin; module-based; PPI; anti-infammatory; bioinformatics
R28
A
1674-926X(2015)04-019-05
成都中醫藥大學藥學院中藥材標準化教育部重點實驗室 中藥資源系統研究與開發利用省部共建國家重點實驗室培育基地,四川 成都 611137
趙萱(1986-),女,四川德陽,碩士研究生,講師,研究方向:中藥新制劑和新劑型的研究Email:626098860@qq.com
傅超美(1961-),男,四川簡陽,教授,博導,研究方向:中藥新制劑和新劑型的研究Email:chaomeifu@126.com
2014-12-06

