長江草魚不同群體EST-SSR多態性標記的篩選及其遺傳結構分析
鄭國棟 陳 杰 蔣霞云 鄒曙明
(上海海洋大學農業部淡水水產種質資源重點實驗室, 上海 201306)
長江草魚不同群體EST-SSR多態性標記的篩選及其遺傳結構分析
鄭國棟 陳 杰 蔣霞云 鄒曙明
(上海海洋大學農業部淡水水產種質資源重點實驗室, 上海 201306)
為對我國不同地域草魚群體進行鑒定和遺傳多樣性分析, 在2400條草魚EST序列中, 對其二堿基至五堿基重復序列進行篩選, 共篩選出微衛星位點181個, 選取其中的46個進行引物的設計與合成, 獲得呈多態性的微衛星引物9對。利用這9對引物對8個長江水系草魚群體和1個紅色草魚自然突變群體進行了遺傳結構分析。結果顯示, 長沙草魚、安慶草魚、嘉興草魚、靖江草魚、石首草魚、松江草魚、瑞昌草魚、邗江草魚和紅色突變草魚9個群體的平均等位基因數(Na)分別為4.67、5.22、5.33、5.00、4.89、4.78、4.89、4.67和2.56, 平均期望雜合度(He)分別為0.6397、0.6543、0.6831、06356、0.6737、0.6483、0.6664、0.7129和0.4696, 平均多態信息含量(PIC)分別為0.5787、0.6126、0.6283、0.5894、0.6217、0.5956、0.6136、0.6582和0.3949, 表明邗江草魚的遺傳多樣性最高, 而紅草魚的遺傳多樣性最低。聚類分析表明, 8個長江水系草魚群體首先聚類, 最后與紅色草魚聚類; 其中, 安慶草魚與松江草魚首先聚為一支, 遺傳距離較近, 為 0.0725; 紅色草魚與長沙草魚的遺傳距離最遠, 為0.5217。研究結果對我國草魚種質資源保存、種群鑒定和良種選育具有重要意義。
草魚; EST-SSR; 遺傳結構; 長江水系
草魚(Ctenopharyngodon idella), 鯉形目鯉科雅羅魚亞科草魚屬的唯一一種魚類。草魚作為我國的四大家魚之一, 因具有生長迅速, 個體大, 肉質肥嫩, 肌間刺少等特點而備受廣大養殖戶和消費者喜愛。草魚廣泛分布于我國江河、湖泊中, 在我國淡水養殖業中占有重要的經濟地位。草魚的種質資源在我國曾非常豐富, 近幾十年來, 由于過度捕撈、魚類病害、盲目近親交配以及草魚生境的破壞, 使我國的草魚天然種質資源發生了嚴重的衰退[1—3]。因此, 保留中國優良草魚品種, 以及增加其生物多樣性是解決草魚種質資源退化的根本措施。目前, 針對長江水系草魚群體, 王解香等[4]利用EST-SSR標記, 范玉頂等[5]、廖小林等[6]、張志偉等[7]及傅建軍等[8]利用SSR標記分別對長江水系草魚進行過遺傳多樣性研究。另在同工酶[9]、(RAPD)[11]、(mtDNARFLP)[10]、細胞色素B序列多態性標記[12]方面, 也做過相關的研究。但得出的結論不盡相同, 因此運用新的分子標記對其進行補充研究仍有重要意義。
目前, 關于EST-SSR標記用于草魚的研究還比較少。相對于傳統的基因組微衛星標記, EST-SSR具有在基因組中分布均勻、呈共顯性遺傳、數量豐富、實驗重復性好等特點[13], 同時由于部分EST來源于基因的編碼區, 所以能更直接、準確地標記功能基因, 更真實地反映該物種的遺傳多樣性[14—17], EST-SSR已成功應用于群體遺傳結構分析、QTL定位和分子標記輔助選擇育種等研究領域[18—20]。本研究擬通過從草魚的眾多EST序列中篩選出優良的EST-SSR標記, 以8個長江水系草魚群體和1個紅色草魚突變群體為研究材料, 進行遺傳結構的分析, 從而為我國草魚種質資源保存、種群鑒定和良種選育奠定基礎。
1 材料與方法
1.1 實驗材料
9個草魚群體包括 8個長江水系群體(長沙草魚、安慶草魚、嘉興草魚、靖江草魚、石首草魚、松江草魚、瑞昌草魚和邗江草魚)和1個草魚紅色自然突變群體(圖1), 均保存于上海海洋大學水產動物遺傳育種中心。其中, 長沙草魚、嘉興草魚、靖江草魚、石首草魚、瑞昌草魚和邗江草魚系原種, 安慶群體和松江群體系自繁群體, 紅色草魚突變體系紅色自發突變體自繁后代, 具體的采樣數量信息見表1。每個群體隨機剪取30尾魚鰭, 共270尾, 放入95%的酒精中, 保存于–20℃備用。
1.2 微衛星引物的篩選
根據 NCBI系統中的草魚 EST數據庫, 應用SSR軟件在線查找EST-SSR, 查找中使用的標準是:二堿基重復單元重復次數為5次以上(包括5次), 三堿基以上重復單元重復次數均為 4 次以上(包括 4次)[4], 包括復合型微衛星。使用Primer5.0進行引物設計, 引物長度控制在18—24 bp 之間, GC含量在40%—60%, Tm值控制在 50℃—65℃, 產物長度控制在100—350 bp, 最后合成EST-SSR引物46對。引物由上海生工生物技術有限公司合成。
1.3 基因組DNA的制備
參照北京天根生物科技有限公司生產的海洋動物組織基因組DNA提取試劑盒(離心柱型)說明書介紹的方法提取樣品基因組DNA。基因組DNA提取完成后, 用0.8%的瓊脂糖凝膠電泳和分光光度計檢測DNA質量和濃度, 保存于–20℃備用。
1.4 PCR反應體系與擴增程序
反應體系 10 μL, 包含 5 μL含染料的 2×Taq PCR MasterMix (Taq DNA Polymerase: 0.1 U/μL;MgCl2: 4 mmol/L; dNTPs each: 0.4 mmol/L), 上下游引物各0.5 μL (10 μmol/L), 0.5 μL 模板DNA(30—50 ng), 3.5 μL ddH2O。PCR反應在Eppendorf Mastercycler ep gradients型 PCR儀上進行, 反應程序為: 94℃預變性 5min, 94℃ 30s, 52—62℃(可據表 1進行調整)30s, 72℃ 30s, 30個循環, 最后72℃延伸10min。
1.5 PCR產物凝膠電泳檢測
PCR 產物在 8%的非變性聚丙烯酰胺凝膠上進行電泳, 膠片大小為 195 mm (長)×120 mm (寬)× 1 mm (厚)。產物上樣量均為 1 μL, DNA Marker (pBR322 DNA/Msp Ι)上樣量為 0.5 μL。電泳條件:電泳緩沖液為1×TBE, 電壓200 V, 電泳1.5—2h (具體時間根據 PCR產物分子量大小而定)。電泳完成后, 進行硝酸銀染色, 染色方法參照張倩倩等[21]的方法進行。最后將膠片平鋪于觀片燈箱上, 拍照并保存。

圖1 普通草魚(上)和紅色草魚(下)Fig. 1 Common grass carp (upper) and red grass carp (lower)

表1 九個草魚群體采集地點及基本信息Tab. 1 Sampling sites and information of the grass carp samples
1.6 數據統計與分析
用Quantity One凝膠圖像分析軟件分析微衛星條帶大小, 根據每個個體產生的條帶位置確定基因型。用Popgene(Version 1.32)軟件進行分析, 計算每個微衛星座位分別在 6個群體中的等位基因數(Number of alleles, Na)和期望雜合度(Expected heterozygosity, He), 并計算群體間的Nei’s遺傳相似性(Genetic identity)和遺傳距離(Genetic distance), 并基于該遺傳距離利用MEGA 5.1軟件對9個草魚群體進行基于非加權組平均法(Unweighted pair-group method with arithmetic means, UPGMA) 的樹狀聚類圖的繪制。最后用Cervus 3.0軟件計算多態信息含量(PIC)。
2 結果
2. 1 EST-SSR引物的篩選
根據 NCBI系統中的草魚 EST數據庫, 應用SSR軟件在線查找EST-SSR, 在2400條EST序列中共發現微衛星位點181個, 占EST序列的7.54%,其中包括二堿基重復序列 116條, 三堿基重復序列45條, 四堿基重復序列19條, 五堿基重復序列1條; 二堿基重復序列最為豐富, 共占64.09%, 其中(AC/CA/TG/GT)n形式在二堿基重復中最為常見(表2)。

表2 草魚EST-SSR的類型及分布特點Tab. 2 The type and number of EST-SSRs
2.2 EST-SSR引物的設計及多態性檢測
在 181條含有微衛星的 EST序列中, 選取 46條微衛星序列進行了引物的設計、合成, 對30尾草魚樣本進行PCR擴增, 最終獲得呈多態性的引物9對(表 3), 另有 37對引物只能擴增出單一條帶或無條帶, 多態性引物占所有設計引物的19.57%。多態性引物中雙堿基重復占88.89%, 雙堿基重復序列又以(AC/GT)n為主。這9對引物在所研究的9個草魚群體中均具有群體間相容性高、多態性良好等特征,部分引物在草魚群體中的擴增圖譜(圖2)。
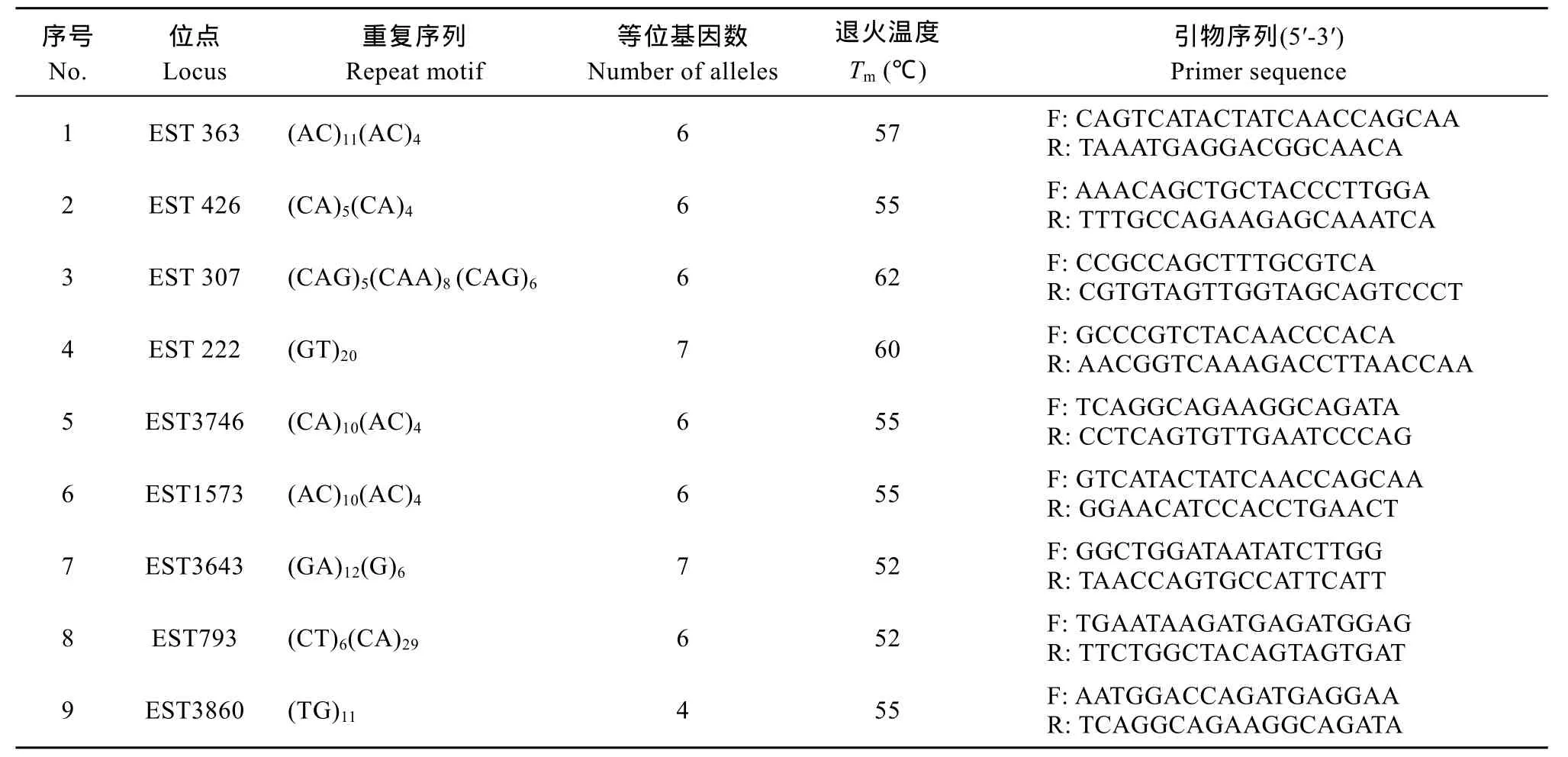
表3 微衛星引物特征Tab. 3 Characteristics of microsatellite primers
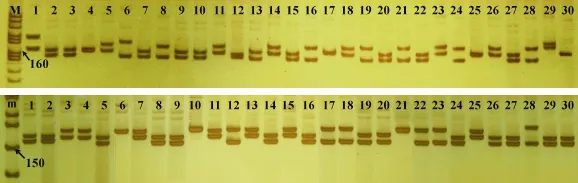
圖2 EST307(上圖)在長沙草魚和EST3746 (下圖)在紅草魚群體中的PAGE圖譜Fig. 2 PAGE analysis by primers EST307 (upper) in Changsha group and EST3746 (lower) in red group
2.3 九個草魚群體的SSR擴增結果
本研究共計開發出了9對呈多態性的EST-SSR引物, 利用這9對微衛星引物對8個長江水系草魚群體和1個紅色草魚自然突變群體共270尾樣品進行擴增分析, 每個位點檢測到的等位基因數4—7個不等, 共檢測出54個等位基因。在9個草魚群體中,長沙草魚、安慶草魚、嘉興草魚、靖江草魚、石首草魚、松江草魚、瑞昌草魚、邗江草魚、紅色草魚9個群體的平均等位基因數(Na)分別為4.67、5.22、5.33、5.00、4.89、4.78、4.89、4.67和2.56, 平均期望雜合度(He)分別為0.6397、0.6543、0.6831、06356、0.6737、0.6483、0.6664、0.7129和0.4696, 平均多態信息含量(PIC)分別為 0.5787、0.6126、0.6283、0.5894、0.6217、0.5956、0.6136、0.6582和0.3949,除了紅色草魚突變群體外基本都為較高的多態性(PIC>0.5), 能夠在分子水平上準確反映各地理群體間和群體內的遺傳關系(表4、表5)。從這些遺傳參數中可得出, 這 9個群體的遺傳多樣性由高到低依次為邗江草魚>嘉興草魚>石首草魚>瑞昌草魚>安慶草魚>松江草魚>靖江草魚>長沙草魚>紅色草魚,即邗江草魚的遺傳多樣性最高, 紅色草魚突變群體的遺傳多樣性最低。
2.4 不同草魚群體間的遺傳距離和聚類分析
用Popgene(Version 1.32)軟件計算草魚9個群體間的Nei’s相似性和遺傳距離(表6)。9個草魚群體間的Nei’s相似系數為0.5935—0.9300, 遺傳距離為0.0725—0.5217。其中安慶群體和松江群體的相似性系數最高, 為 0.9300, 長沙群體和紅色草魚突變群體的遺傳相似性最低, 為 0.5935。基于不同群體間Nei’s遺傳距離, 利用MEGA5.1軟件構建了9個草魚群體間的UPGMA聚類關系(圖3)。結果顯示, 8個長江水系草魚群體首先聚類, 最后才與紅色草魚突變群體聚類; 其中, 安慶群體和松江群體首先聚為一支, 瑞昌群體和邗江群體聚為一支, 然后這兩支聚為一支后又與嘉興群體聚為一支; 靖江群體和石首群體聚為一支, 這支再與前一大支聚類; 最后與長沙群體聚類。其中紅色草魚突變群體與長沙群體的遺傳距離最遠, 為0.5217。
3 討論
3.1 草魚EST-SSR標記的篩選
本研究從草魚2400個EST 序列中查找獲得181個微衛星位點, 占整個ESTs 數據庫的7.54%, 這與其他學者在EST中所篩選到的微衛星比率基本一致,其中王解香等[4]在草魚EST中獲得的微衛星比例為2.12%, 王艷紅等[22]在凡納濱對蝦(Litopenaeus vannamei)EST中獲得的微衛星的比例是7.80%; 孫國華等[23]在刺參EST 中獲得的微衛星比例為6.48%;Wang等[25]在太平洋牡蠣(Crassostrea gigas)EST中獲得的微衛星的比例為4.50%; 石耀華等[24]在馬氏珠母貝(Pinctada martensii Dunker)EST中獲得含有微衛星的EST的比例為3.48%; 徐鵬等[26]對中國對蝦(Fenneropenaeus chinensis)的整個EST數據庫進行分析, 含微衛星序列的比例為2.19%。總的看來, 微衛星在EST序列中的比例大都在10%以下, 但不同物種之間存在差異, 這可能是由于不同物種基因組的結構組成、容量大小不同造成的。同一物種之間也存在一定差異, 原因可能是每個研究人員在研究中所使用的EST序列來源不盡相同造成的。
本研究所獲得的EST-SSR中雙堿基重復占有較大的比例, 占64.09%, 其中以(AC/CA/TG/GT)n形式的二堿基重復序列最為常見。這個結論與其他學者所篩選到的微衛星堿基組成類型及比例基本一致,如王艷紅等[22]在凡納濱對蝦EST中發現雙堿基重復占總EST-SSR序列的80.20%; Wang等[27]、魯翠云等[28]發現在鯉( Cyprinus carpio)中, (AC/GT)n占所有雙堿基重復EST-SSR的67.57%; 一些學者在鯉微衛星中發現雙堿重復的微衛星所占比例達到了96.75%, (AC)n所占的比例是(CT)n的7倍[29—31]。這些結果表明,以(AC/CA/TG/GT)n形式存在的雙堿基重復序列在魚類的EST-SSR中占主要部分, 應該成為篩選魚類EST微衛星的側重點。

表4 草魚微衛星位點的等位基因數、期望雜合度Tab. 4 Number of alleles (Na) and expected heterozygosity (He) of microsatellite loci

表5 草魚微衛星位點的多態信息含量Tab. 5 The polymorphism information content (PIC) of microsatellite loci

圖3 不同草魚群體的UPGMA 聚類圖Fig. 3 Dendrogram of relationships among grass carp using UPGMA method of clustering
3.2 九個草魚群體的遺傳結構分析
從遺傳角度來講, 等位基因的數目(Na)、遺傳雜合度(He)和多態性信息含量(PIC)等遺傳參數可從多個角度反映群體的遺傳多樣性和遺傳潛力[32]。并且,通常認為PIC>0.5為高度多態, 0.25<PIC<0.5為中度多態, PIC<0.25為低度多態[33]。本研究中長沙草魚、安慶草魚、嘉興草魚、靖江草魚、石首草魚、松江草魚、瑞昌草魚、邗江草魚和紅草魚9個群體的平均等位基因數(Na)為 2.56—5.33, 平均期望雜合度(He)為 0.4696—0.7129, 平均多態信息含量(PIC)為 0.3949—0.6582, 除紅色草魚突變群體外, 9個長江水系的原種或自繁群體均表現為較高的多態性; 紅色草魚突變群體的遺傳多樣性最低(平均Na、He和PIC依次為2.56、0.4696和0.3949), 表現為較低的遺傳多樣性, 這可能與紅色草魚原始突變親本較少和存在一定的近親交配有關。
利用UPGMA 法對這9個草魚群體進行聚類分析, 其中, 安慶群體和松江群體首先聚為一支, 瑞昌群體和邗江群體聚為一支, 然后這兩支聚為一支后又與嘉興群體聚為一支; 靖江群體和石首群體聚為一支, 這支再與前一大支聚類; 最后與長沙群體聚類。其中紅色草魚群體單獨聚為一支, 與其他8個群體遺傳距離較大, 與長沙群體的遺傳距離最遠。由于紅色草魚群體遠離長江水系, 而其他8個群體均屬于長江水系, 地理隔離導致了紅色草魚群體產生了較大偏離。而在同屬于長江水系的8個草魚群體中, 長沙群體與其他7個群體遺傳距離較遠, 可能是因為長沙群體的主要生活水域為洞庭湖, 洞庭湖水域相對穩定, 流動性較小, 久而久之就與長江干流群體產生了分化, 而長江干流群體因流動性大, 基因交流程度大, 所以其遺傳相似性大。總的看來, 所有長江水系群體的遺傳距離與其地理距離也不盡相符, 這可能與取樣的隨機性有關, 不同江段的樣本自身也存在差異, 可能還和所用的微衛星引物及研究所用樣本的數量有關, 因為標記數和樣本數量對遺傳參數的影響很大[34]; 另外, 不同地域之間草魚的人工遷移可能也對研究結果有影響。
3.3 紅色草魚突變體的產生原因及意義
體色是魚類的一個重要表型性狀, 體色變異在遺傳學、育種學以及功能基因發掘方面有著重要的研究價值。魚類的不同體色與其體內色素細胞(黑色素細胞、虹彩細胞等)直接相關[35]。而硬骨魚類色素細胞合成色素時受神經和內分泌的調控。Fujii等[36]研究發現, 釋放兒茶酚胺的神經原控制腦垂體分泌促黑色素細胞生成激素, 進而影響體色。腎上腺素、去甲腎上腺素和多巴胺等兒茶酚胺是應激時調控魚類體色變化的主要激素, 而酪氨酸酶則是黑色素生成的關鍵酶, 酪氨酸酶的含量和活性較高的部位體色較深。本研究中的草魚紅色突變體, 體表鱗片和鰭均呈淡紅色, 且經過一年的養殖之后體色并沒有變化, 應屬于不可逆性突變。所以, 黑色素主控基因酪氨酸酶基因的突變或者酪氨酸酶在體內受到抑制可能是草魚紅色突變體產生的根本原因。
目前, 魚類等生物優良品種選育的一個主要途徑就是發現并利用自然突變體, 然后通過繼續選育來強化和穩定優良的突變基因, 也可用雜交的方法將突變體的優良基因整合到我們所需要的目標魚體當中。從本質而言, 這均是利用自然界已存在的攜帶優良性狀基因的突變體, 在此基礎上加以選擇、固定, 從而建立優良品系; 因此, 攜帶優良性狀突變體的獲得對魚類基因庫的擴展及縮短育種年限具有重要意義[37]。
[1] Li M L, Huang S L. Analysis of the effects and countermeasures of large hydraulic structures on fishery resources in Yangtze River [J]. Journal of Shanghai Ocean University, 2009, 18(6): 759—764 [李美玲, 黃碩琳. 大型水工建筑對長江漁業資源影響及對策淺析. 上海海洋大學學報, 2009, 18(6): 759—764]
[2] Chen D Q, Duan X B, Liu S P, et al. On the dynamics of fishery resources the Yangtze River and its management [J]. Acta Hydrobiologica Sinica, 2002, 26(6): 685—690 [陳大慶,段辛斌, 劉紹平, 等. 長江漁業資源變動和管理對策. 水生生物學報, 2002, 26(6): 685—690]
[3] Li S H, Chen D Q, Duan X B, et al. Genetic effects of released mature Ctenopharyngodon idellus on natural populations based on the mitochondrial DNA markers in the middle reaches of the Yangtze River [J]. Freshwater Fisheries, 2014, 44(3): 45—50 [李樹華, 陳大慶, 段辛斌,等. 基于線粒體DNA 標記的長江中游草魚親本增殖放流的遺傳效果評估. 淡水漁業, 2014, 44(3): 45—50]
[4] Wang J X, Yu L Y, Bai J J, et al. Development of EST-SSR markers and analysis of genetic diversity in five populations of grass carp (Ctenopharyngodon idella) [J]. Chinese Journal of Zoology, 2011, 46(5): 24—32 [王解香, 于凌云,白俊杰, 等. 草魚EST-SSR標記及5個不同地域群體的遺傳結構分析. 動物學雜志, 2011, 46(5): 24—32]
[5] Fan Y D, Zhang Y, Wang D Q, et al. Microsatellite markers isolation and genetic analysis of wild grass carp (Ctenopharyngodon idellus) in Yangtze River [J]. Freshwater Fisheries, 2010, 40(6): 3—8 [范玉頂, 張燕, 汪登強, 等. 長江草魚多態微衛星位點的分離及后備親魚的遺傳多樣性分析. 淡水漁業, 2010, 40(6): 3—8]
[6] Liao X L, Yu X M, Tan D Q, et al. Microsatellite and analysis of analysis of genetic diversity of grass carp in Yangtze River system [J]. Acta Hydrobiologica Sinica, 2005, 29(2): 113—119 [廖小林, 俞小牧, 譚德清, 等. 長江水系草魚遺傳多樣性的微衛星 DNA分析. 水生生物學報, 2005, 29(2): 113—119]
[7] Zhang Z W, Cao Z M, Yang H, et al. Microsatellites analysis on genetic variation between wild and cultured populations of Ctenopharyngodon idella [J]. Zoological Research, 2006, 27(2): 189—196 [張志偉, 曹哲明, 楊弘, 等. 草魚野生和養殖群體間遺傳變異的微衛星分析. 動物學研究, 2006, 27(2): 189—196]
[8] Fu J J, Li J L, Shen Y B, et al. Genetic variation analysis of wild populations of grass carp (Ctenopharyngodon idella) using microsatellite markers [J]. Heredity, 2013, 35(2): 192—201 [傅建軍, 李家樂, 沈玉幫, 等. 草魚野生群體遺傳變異的微衛星分析. 遺傳, 2013, 35(2): 192—201]
[9] Zhao J L, Li S F. Isoenzyme analysis of population divergence of silver carp, bighead carp, grass carp and black carp in the middle and lower stream of Changjiang River [J]. Journal of Fisheries of China, 1996, 20(2): 104—110 [趙金良, 李思發. 長江中下游鰱, 鳙, 草魚, 青魚種群分化的同工酶分析. 水產學報, 1996, 20(2): 104—110]
[10] Li S F, Lü G Q, Louis B. Diversitiy of mitochondrial DNA in the populations of silver carp, bighead carp, grass carp and black carp in the middle and lower reaches of the Yangtze River [J]. Acta Zoologica Sinica, 1998, 44(1): 82—93 [李思發, 呂國慶. 長江中下游鰱鳙草青四大家魚線粒體 DNA多樣性分析. 動物學報, 1998, 44(1): 82—93]
[11] Zhang S M, Deng H, Wang D Q, et al. Population structure and genetic diversity of silver carp and grass carp from populations of Yangtze River system revealed by RAPD [J]. Acta Hydrobiologica Sinica, 2001, 25(4): 324—330 [張四明, 鄧懷, 汪登強, 等. 長江水系鰱和草魚遺傳結構及變異性的 RAPD 研究. 水生生物學報, 2001, 25(4): 324—330]
[12] Ye X, Bai J J, Lao H H, et al. Clone and characterization of mitochondrial cytochrome b gene from grass carpCtenopharyngodon idella [J]. Journal of Fishery Science of China, 2002, 9(3): 193—197 [葉星, 白俊杰, 勞海華. 草魚線粒體細胞色素b基因的克隆與序列分析. 中國水產科學, 2002, 9(3): 193—197]
[13] Powel W, Machray G C, Provan J. Polymorphism revealed by simple sequence repeats [J]. Trends in Plant Science, 1996, 1(7): 215—222
[14] Hu J, Wang X, Hu X, et al. Existence of microsatellites in expressed sequence tags of common carp (Cyprinus carpio L.) available in GenBank dbEST Database [J]. Journal of Ocean University of China, 2006, 5(1): 12—20
[15] Wang M Q, Hu J J, Zhuang Y Y, et al. In silico screening for microsatellite markers from expressed sequence tags of Porphyrayezoensis (Bangiales, Rhodophyta) [J]. Journal of Ocean University of China, 2007, 6(2): 161—166
[16] Ji X S, Chen S L, Ma H Y, et al. Isolation and characterization of 19 EST-linked ploymorphic microsatellite loci for olive flounder (Paralichthy solivaceus) [J]. Aquaculture Research, 2009, 40(8): 980—983
[17] Yu H, Li Q. Exploiting EST databases for the development and characterization of EST-SSRs in the Pacific oyster (Crassostre agigas) [J]. Journal of Heredity, 2008, 99(2): 208—214
[18] Varshney R K, Sigmund R, Brner A, et al. Interspecific transferability and comparative mapping of barley EST-SSR markers in wheat, rye and rice [J]. Plant Science, 2005, 168(1): 195—202
[19] Perez F, Ortiz J, Zhinaula M, et al. Development of ESTSSR markers by data mining in three species of shrimp: Litopenaeus vannamei, Litopenaeus stylirostris and Trachypenaeus birdy [J]. Marine Biotechnology, 2005, 7(5): 554—569
[20] Wang H X, Li F H, Xiang J H. Polymorphic EST-SSR markers and their mode of inheritance in Fenneropenaeus chinensis [J]. Aquaculture, 2005, 249(1—4): 107—114
[21] Zhang Q Q, Chen J, Zou S M, et al. Establishment of DNA fingerprinting and analysis on genetic structure of different Parabramis and Megalobrama populations with microsatellite [J]. Journal of Fisheries of China, 2014, 38(1): 15—22 [張倩倩, 蔣霞云, 鄒曙明, 等. 不同鳊魴魚類群體微衛星DNA指紋圖譜的構建和遺傳結構分析. 水產學報, 2014, 38(1): 15—22]
[22] Wang Y H, Hu C Q, Zhang L P, et al. A preliminary study on microsatellite markers screening from EST sequences of Litopenaeus vannamei [J]. Journal of Fisheries of China, 2011, 35(7): 969—976 [王艷紅, 胡超群, 張呂平, 等. 凡納濱對EST微衛星標記初步篩選. 水產學報, 2011, 35(7): 969—976]
[23] Sun G H, Yang J M, Song Z L, et al. Isolation of microsatellite markers from Apostichopus japonicus ESTs [J]. Oceanologia et Limnologia Sinica, 2010, 41(1): 133—139 [孫國華, 楊建敏, 宋志樂, 等. 刺參(Apostichopus japonicus) EST序列中微衛星分布分析及其標的篩選. 海洋與湖沼, 2010, 41(1): 133—139]
[24] Shi Y H, Hong K, Guo X M, et al. Microsatellite markers screening from EST sequences of Pinctada martensii Dunker [J]. Journal of Fisheries of China, 2008, 32(2): 174—181 [石耀華, 洪葵, 郭希明, 等. 馬氏珠母貝EST微衛星的篩選. 水產學報, 2008, 32(2): 174—181]
[25] Wang Y H, Ren R, Yu Z N. Bioinformatic mining of EST-SSR loci in the Pacific oyster, Crassostrea gigas [J]. Animal Genetics, 2008, 39: 287—289
[26] Xu P, Zhou L H, Tian L P, et al. Isolation of microsatellite markers from Fenneropenaeus chinensis ESTs [J]. Journal of Fisheries of China, 2003, 27(3): 213—218 [徐鵬, 周令華,田麗萍, 等. 從中國對蝦ESTs中篩選微衛星標記的研究.水產學報, 2003, 27(3): 213—218]
[27] Wang D, Liao X L, Cheng L, et al. Development of novel EST-SSR markers in common carp by data mining from public EST sequences [J]. Aquaculture, 2007, 271(1—4) : 558—574
[28] Lu C Y, Quan Y C, LI D Y, Population structure analysis of common carp of Yangtze River and Heilongjiang River using EST-SSRs markers [J]. Journal of Agricultural Biotechnology, 2007, 15(6): 47—52 [魯翠云, 全迎春, 李大宇, 等. 用鯉魚 EST-SSRs分子標記分析長江黑龍江鯉種群結構. 農業生物技術學報, 2007, 15(6): 47—52]
[29] Edwards Y J K, Elgar G, Clark M S, et al. The identification and characterization of microsatellites in the compact genome of the Japanese pufferfish, Fugurubripes: perspectives in functional and comparative genomic analyses [J]. Journal of Molecular Biology, 1998, 278(4): 843—854
[30] David L, Rajasekaran P, Fang J, et al. Polymorphism in ornamental and common carp strains (Cyprinus carpio L.) as revealed by AFLP analysis and a new set of microsatellite markers [J]. Molecular Genetics and Genomics, 2001, 266(3): 353—362
[31] Serapion J, Kucuktas H, Feng J N, et al. Bioinformatic mining of type I microsatellites from expressed sequence tags of channel catfish (Ictalurus punctatus) [J]. Marine Biotechnology, 2004, 6(4): 364—377
[32] Yang H, Li D Y, Cao X, et al. Genetic potential analysis of six Tilapia populations by microsatellite DNA markers [J]. Heredity, 2011, 33(7): 768—775 [楊弘, 李大宇, 曹祥, 等.微衛星標記分析羅非魚群體的遺傳潛力. 遺傳, 2011,33(7): 768—775]
[33] Botstein D, White R L, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. American Journal of Human Genetics, 1980, 32(3): 314—331
[34] Li O, Zhao Y Y, Sun X W, et al. Effects of sample size and loci number on genetic diversity in wild population of grass carp revealed by SSR [J]. Zoological Research, 2009, 30(02): 121—130 [李鷗, 趙瑩瑩, 孫效文, 等. 草魚種群SSR分析中樣本量及標記數量對遺傳多度的影響. 動物學研究, 2009, 30(02): 121—130]
[35] Fujii R. Cytophysiology of fish chromatophores [J]. International Review of Cytology-A Survey of Cell Biology, 1993, 143: 191—255
[36] Fujii R, Hiroaki K, Noriko O. Control of chromatophore movements in dermal chromatic units of blue damselfish-II. The motile iridophore [J]. Comparative Biochemistry and Physiology Part C: Comparative Pharmacology, 1986, 83: 1—7
[37] Fujii K, Hasegawa O, Honda K, et al. Marker-assisted breeding of a lymphocystis disease-resistant Japanese flounder (Paralichthys olivaceus) [J]. Aquaculture, 2007, 272(1—4): 291—295
DETECTION OF EST-SSRS MARKERS AND GENETIC STRUCTURE OF DIFFERENT POPULATIONS OF GRASS CARP IN YANGTZE RIVER SYSTEM
ZHENG Guo-Dong, CHEN Jie, JIANG Xia-Yun and ZOU Shu-Ming
(Key Laboratory of Freshwater Aquatic Genetic Resources, Shanghai Ocean University, Shanghai 201306, China)
In this study, we selected 181 microsatellite loci from 2400 EST sequences to perform germplasm identification and genetic diversity analysis in different regions of grass carp, Ctenopharyngodon idella. Nine pairs of primers, which could give stable and polymorphic amplification profiles, were screened out from 46 microsatellite loci and used to analyze the genetic structure of C. idella. The 9 pairs of primers were used to analyze the genetic structure of 8 populations of the Yangtze River (CSC, AQC, JXC, JJC, SSC, SJC, RCC and HJC) and 1 red population (HC). The mean value of number on alleles (Na) of CSC, AQC, JXC, JJC, SSC, SJC, RCC, HJC and HC was 4.67, 5.22, 5.33, 5.00, 4.89, 4.78, 4.89, 4.67 and 2.56, respectively. The mean value of expected heterozygosity (He) was 0.6397, 0.6543, 0.6831, 0.6356, 0.6737, 0.6483, 0.6664, 0.7129 and 0.4696, respectively. And the average value of polymorphism information content (PIC) was 0.5787, 0.6126, 0.6283, 0.5894, 0.6217, 0.5956, 0.6136, 0.6582 and 0.3949, respectively. It demonstrated that the genetic diversity of HJC was the highest and HC was the lowest among the nine populations. Using unweighted pair-group method with arithmetic means method (UPGMA) based on their genetic distances, the cluster analysis in nine populations showed that 8 populations of the Yangtze River first grouped together, then they clustered with the red population. In addition, the genetic distance between AQC and SJC was 0.0725, which was the nearest, the genetic distance between HC and CSC was 0.5217, which was the farthest. Our results will have important value in germplasm resources preservation, germplasm identification and breeding of C. idella.
Grass carp; EST-SSR; Genetic structure; Yangtze River system
Q346+.5
A
1000-3207(2015)05-1003-09
10.7541/2015.131
2014-07-14;
2015-04-12
“十二五”國家863計劃主題項目(2011AA100403); 公益性行業(農業)科研專項(200903045); 水產動物遺傳育種中心上海市協同創新中心(2F1206)資助
鄭國棟(1987—), 男, 山東濰坊人; 碩士; 主要從事動物遺傳育種與繁殖研究。E-mail: zgdong.521@163.com
鄒曙明, Tel: 021-61900345; E-mail: smzou@shou.edu.cn

