基于轉錄組數據的馬氏珠母貝EST-SSR位點的信息分析及其多態性檢測*
王忠良 丁 燏 許尤厚 簡紀常 魯義善王 蓓 陳 剛 吳灶和,
(1. 廣東海洋大學水產學院 湛江 524088; 2. 廣東海洋大學南海水產經濟動物增養殖重點實驗室 湛江 524088; 3. 廣西北部灣海洋生物多樣性養護重點實驗室(欽州學院) 欽州 535000; 4. 廣東省水產經濟動物病原生物學及流行病學重點實驗室湛江 524088; 5. 仲愷農業工程學院 廣州 510225)
馬氏珠母貝(Pinctada martensii)又稱合浦珠母貝(Pinctada fucata), 是人工培育海水珍珠的主要母貝。自 1965年我國成功開展馬氏珠母貝人工育苗以來,海水珍珠養殖業發展迅速, 尤其是進入20世紀90年代后, 海水珍珠養殖已成為廣東、廣西和海南沿海地區的支柱產業(史兼華等, 2006; Wang et al, 2009)。然而, 由于長期的人工養殖和近親繁殖, 馬氏珠母貝種質已呈現明顯的退化現象, 如種苗活力下降、死亡率增加、育珠貝變小、珍珠質量差等(Miyazaki et al, 1999;Tomaru et al, 2001; He et al, 2008)。為改善這一狀況,馬氏珠母貝的遺傳改良和優良品種培育是當前海水珍珠養殖業的首要任務, 而篩選穩定可靠的分子標記評價其遺傳多樣性對馬氏珠母貝良種選育和種質資源保護都具重要的指導意義。
簡單重復序列(Simple Sequence Repeats, SSR),又稱微衛星DNA, 是指由1-6個核苷酸組成的簡單串聯重復DNA序列(Powell et al, 1996; Liu et al, 2013)。與諸多分子標記相比, 具有數量豐富、多態性高、多等位性、共顯性和可重復性高等優點, 已廣泛應用于動植物遺傳圖譜構建、種質鑒定、基因定位、遺傳多樣性、分類與進化及分子標記輔助育種等研究領域(趙瑩瑩等, 2006; 徐美佳等, 2009; 李云峰等, 2010;孫國華等, 2010; 劉博等, 2012; 王東等, 2014)。根據來源, SSR標記又可分為基因組 SSR(Genomic SSR,gSSR)和表達序列標簽 SSR(Expressed sequence tag SSR, EST-SSR)兩種。與gSSR標記相比, EST-SSR標記無需構建基因組文庫, 節省了大量人力物力, 開發成本較低, 且來源于基因的轉錄區, 其多態性與基因功能可能直接相關, 同時在相近物種間具良好的通用性(Varshney et al, 2005; 李琪, 2006; 張瓊等,2010)。
近年來, 隨著EST和cDNA大規模測序技術的快速發展, 多種海產經濟貝類 SSR標記的開發利用研究已相繼展開(李紅蕾等, 2003; Sato et al, 2005; 李琪,2006; 李云峰等, 2010), 但現有的SSR標記還非常有限, 仍不能滿足進一步研究的需要。為此, 本文基于前期Illumina高通量測序技術獲得的馬氏珠母貝轉錄組數據(未發表), 利用計算機軟件輔助進行大規模EST-SSR標記的發掘, 并對其組成、分布及特征等信息進行了分析, 同時對 SSR標記的多態性進行了評價, 以期為馬氏珠母貝遺傳多樣性、遺傳圖譜構建及分子輔助育種研究提供有效工具, 促進馬氏珠母貝種質資源保護、優良品種培育和珍珠養殖業的健康發展。
1 材料與方法
1.1 馬氏珠母貝的暫養及血細胞采集
馬氏珠母貝(平均殼長 70mm)購自廣東省湛江市徐聞邁陳珍珠貝養殖場, 暫養于室內玻璃鋼水槽中(200L水體), 每槽飼養20只; 水溫25°C, 鹽度28, 飼養期間連續充氣; 暫養過程中以螺旋藻粉為餌料投喂,每天換水(100%)一次; 室內暫養一周后采集血淋巴。
利用 1mL無菌注射器于馬氏珠母貝閉殼肌處采集血淋巴, 每只 0.5mL; 采集的血淋巴分裝至 1.5mL離心管中, 每管1mL; 4°C, 800g 離心10min 收集血細胞, 并立即進行DNA提取。
1.2 馬氏珠母貝轉錄組數據來源
馬氏珠母貝轉錄組數據來源于課題組前期利用Illumin/Hiseq-2000高通量測序平臺對馬氏珠母貝血細胞進行的全轉錄組測序。轉錄組測序共獲得70407878條原始讀數(Raw reads), 經去除含有接頭、重復及測序質量較低的原始讀數后, 獲得 56345139條純凈讀數(Clean reads)。使用轉錄組de novo組裝軟件Trinity(Grabherr et al, 2011)對Clean reads進行組裝,并進行去冗余處理和進一步拼接, 共得到74007條非冗余獨立基因(Unigene)。(以上轉錄組數據尚未發表,轉錄組Raw reads已提交至NCBI的SRA數據庫, 登錄號為SRP041567。)
1.3 馬氏珠母貝轉錄組EST-SSR位點篩選
為檢測馬氏珠母貝轉錄組中 EST-SSR位點, 使用軟件MIcroSAtellite (MISA) (Lu et al, 2013)對組裝得到的Unigenes序列進行SSR 搜索和定位。所檢測SSR 位點共6類, 分別為單核苷酸重復、二核苷酸重復、三核苷酸重復、四核苷酸重復、五核苷酸重復和六核苷酸重復, 其篩選標準為: 單核苷酸重復≥10次; 二核苷酸重復≥6次; 三核苷酸重復、四核苷酸重復、五核苷酸重復和六核苷酸重復≥5次。
1.4 馬氏珠母貝轉錄組EST-SSR引物設計
使用Primer3軟件進行SSR引物設計, 每條SSR產生3對引物; 引物設計的主要參數設置如下: 引物長 18—23bp, 最適長度 23 bp; PCR擴增產物長度100—300bp; GC含量40%—65%, 最適含量50%; 退火溫度55—65°C, 最佳退火溫度55°C。
1.5 馬氏珠母貝轉錄組EST-SSR多態性檢測
1.5.1 模板DNA的制備 采用Universal Genomic DNA Mini-Isolation Kit試劑盒(上海生工生物工程有限公司), 并按照說明書方法進行血細胞 DNA的提取。經紫外分光光度計定量后, –20°C保存備用。
1.5.2 PCR擴增及產物檢測 為了篩選和驗證所設計的SSR引物, 隨機挑選80對引物(引物由上海生工生物工程有限公司合成), 以上述血細胞DNA為模板進行PCR擴增。PCR反應體系為20 μL, 包括2×ES Taq MIX 10 μL, 上下游引物(10 μmol/L)各 1 μL, 模板DNA 1 μL, ddH2O補足至 20 μL。PCR擴增程序為:94°C預變性5 min, 30個循環(94°C變性30s, 最佳Tm退火 30s, 72°C延伸 30s), 最后 72°C 延伸 5 min。PCR擴增產物用8%非變性聚丙烯酰胺凝膠電泳結合銀染法顯色進行檢測。
2 結果與分析
2.1 馬氏珠母貝轉錄組EST-SSR的分布及頻率
利用MISA軟件對馬氏珠母貝轉錄組中的74007條Unigenes序列進行搜索, 共檢測到9872個SSR位點, 分布于8135條Unigenes序列中。SSR 發生頻率(含SSR的Unigene數/Unigene總數)為10.99%, SSR出現頻率(檢出SSR位點數/Unigene總數)為13.34%;平均每5102bp含有1個SSR位點(檢測序列總堿基數50368155 bp)。其中, 1350條Unigene含有1個以上SSR位點, 復合型SSR數目為518個。
馬氏珠母貝 EST-SSR重復類型豐富, 除六核苷酸重復外, 1—5核苷酸重復均有發現。其中, 單核苷酸重復有4種類型, 二核苷酸重復有11種類型, 三核苷酸重復有 52 種類型, 四核苷酸重復有 58種類型,五核苷酸重復有7種類型(表1)。從各類型SSR位點數量看, 出現最多的為 1—3核苷酸重復, 占總 SSR位點數的98.32%。其中, 單核苷酸重復比例最高, 占81.46%; 其次為二核苷酸重復和三核苷酸重復, 分別占10.48%和6.38%; 4—5核苷酸重復SSR數量較少,共占 1.68%(表 1)。
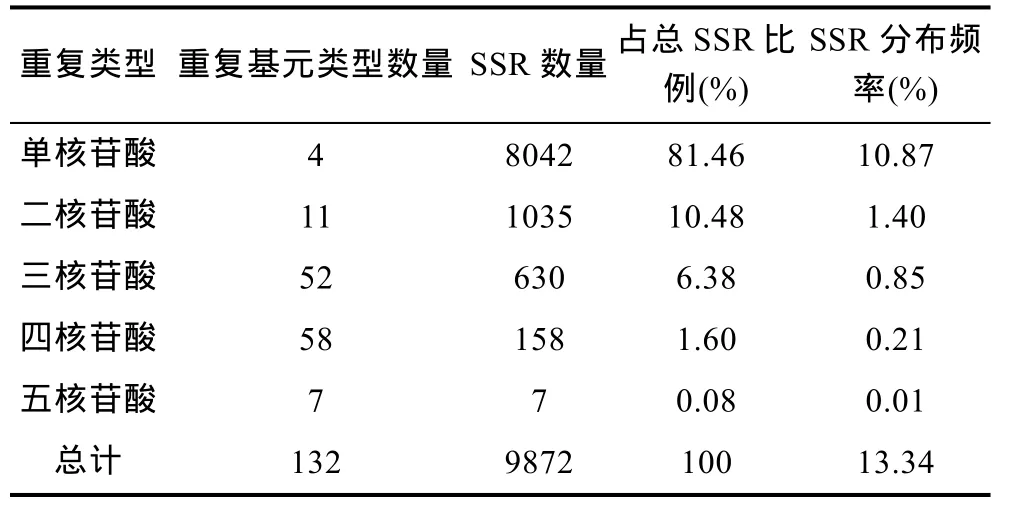
表1 馬氏珠母貝EST-SSRs不同重復基元分布情況Tab.1 Distribution of different repeat motifs of EST-SSRs in P.martensii transcriptome
馬氏珠母貝轉錄組中 EST-SSR的長度在 12—20bp的有3677條, 其中單核苷酸重復SSR共計2226條; 長度超過20bp的SSR有336條, 并以單核苷酸重復SSR最多, 達129條(表2)。SSR位點重復次數方面, 以重復 10次(3883)最多, 占總 SSR位點的39.33%; 其次為重復11次和12次, 分別占17.12%和8.88%; 重復5—9次、13—17次及18—24次的SSR位點個數分別為1671、1279和472(圖1)。
2.2 馬氏珠母貝轉錄組EST-SSR的特征
在馬氏珠母貝轉錄組9872個SSR位點中, 共檢測到 132種重復基元(表 1)。其中, 以單核苷酸重復基元 A/T和 C/G最多, 分別占總 SSR的 71.27%和10.19%, 發生頻率分別為 9.507%和 1.359%; 其次為二核苷酸重復基元AT/AT和AG/CT, 分別占4.66%和4.24%; 三核苷酸重復單元中, 以 ATC/ATG、AAG/CTT和AAC/GTT出現次數最多, 分別占總SSR的2.07%、1.29%和1.16%; 四核苷酸和五核苷酸重復基元種類較多, 但數量較少, 頻率較低, 共占SSR總數的 1.67%(表 3)。
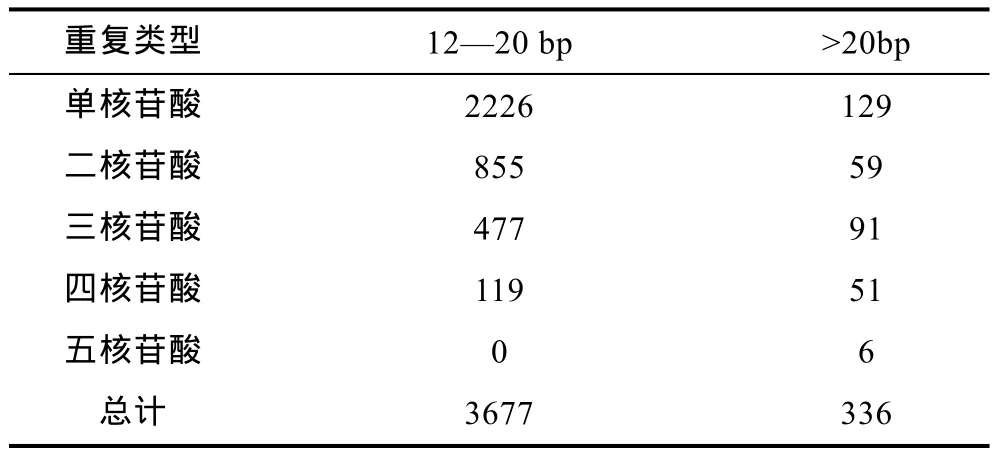
表2 馬氏珠母貝EST- SSRs長度分布情況Tab.2 Lengthdistribution of EST-SSRs in P. martensii transcriptome
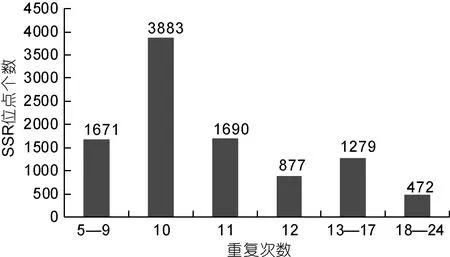
圖1 馬氏珠母貝EST-SSRs重復次數分布圖Fig.1 Distribution of EST-SSR repeats frequency in P.martensii transcriptome
2.3 馬氏珠母貝轉錄組EST-SSR引物設計及多態性檢測
為更好地利用馬氏珠母貝 SSR位點, 本文應用Primer3軟件對上下游序列均不小于 150 bp的EST-SSR設計引物, 每條序列產生 3對引物。經SSRFinder校驗及除去不符合條件的引物后, 共為5922條EST-SSR序列成功設計出17766對引物, 占馬氏珠母貝SSR總數的59.99%。
從上述設計引物中隨機挑選 80對引物, 并以隨機挑選的20只健康馬氏珠母貝血細胞基因組DNA為模板進行PCR擴增以驗證設計的EST-SSR引物。結果表明, 共有 62對引物成功擴增出穩定條帶, 占引物總數的 77.5%, 但其中有 23對引物擴增產物片段與預期產物片段大小不符; 在62個SSR位點中, 以二堿基、三堿基重復類型的 SSR位點擴增成功率較高; 此外, 17對 EST-SSR引物(占擴增引物總數的21.25%)表現出個體多態性(表 4), 多態性比率為27.42%; 17對多態性引物所在SSR位點中, 有7個為二堿基重復, 6個為三堿基重復, 2個為單堿基重復,四堿基重復和五堿基重復各1個。
3 討論
目前, 有關貝類微衛星標記的研究主要是通過構建cDNA文庫來獲得含有微衛星的序列, 并且設計引物對微衛星標記進行篩選(Sato et al, 2005; 李春艷等, 2009)。如石耀華等(2008)從馬氏珠母貝9個組織的SMART cDNA文庫中共獲得6979條EST序列, 并篩選到268個SSR位點, SSR出現頻率為3.48%; 同樣, 李云峰等(2010)從蝦夷扇貝(Patinopecten yessoensis) cDNA文庫中發現了74個SSR重復序列(2007條EST序列), SSR出現頻率為3.69%。與上述傳統 EST-SSR標記開發技術相比, 基于高通量轉錄組測序的 SSR標記開發技術不僅可以借助更全面的數據信息, 提高遺傳多樣性研究的準確性, 而且開發的SSR標記同樣具有EST-SSR標記的諸多優點。王學穎(2012)從馬氏珠母貝珍珠囊轉錄組的99127條無冗余EST序列中共發現了6595個SSR位點, SSR出現頻率為6.65%; 同樣, 本文利用 MISA軟件從馬氏珠母貝轉錄組74007條Unigenes中共檢索出9872個SSR位點, 分布于8135條Unigenes中, SSR出現頻率高達 13.34%。因此, 結合其它動植物 SSR開發相關文獻和本研究結果表明, 基于轉錄組數據篩選 SSR標記是發掘非模式生物 EST-SSR的一種快速、高效的方法, 應用前景廣闊。
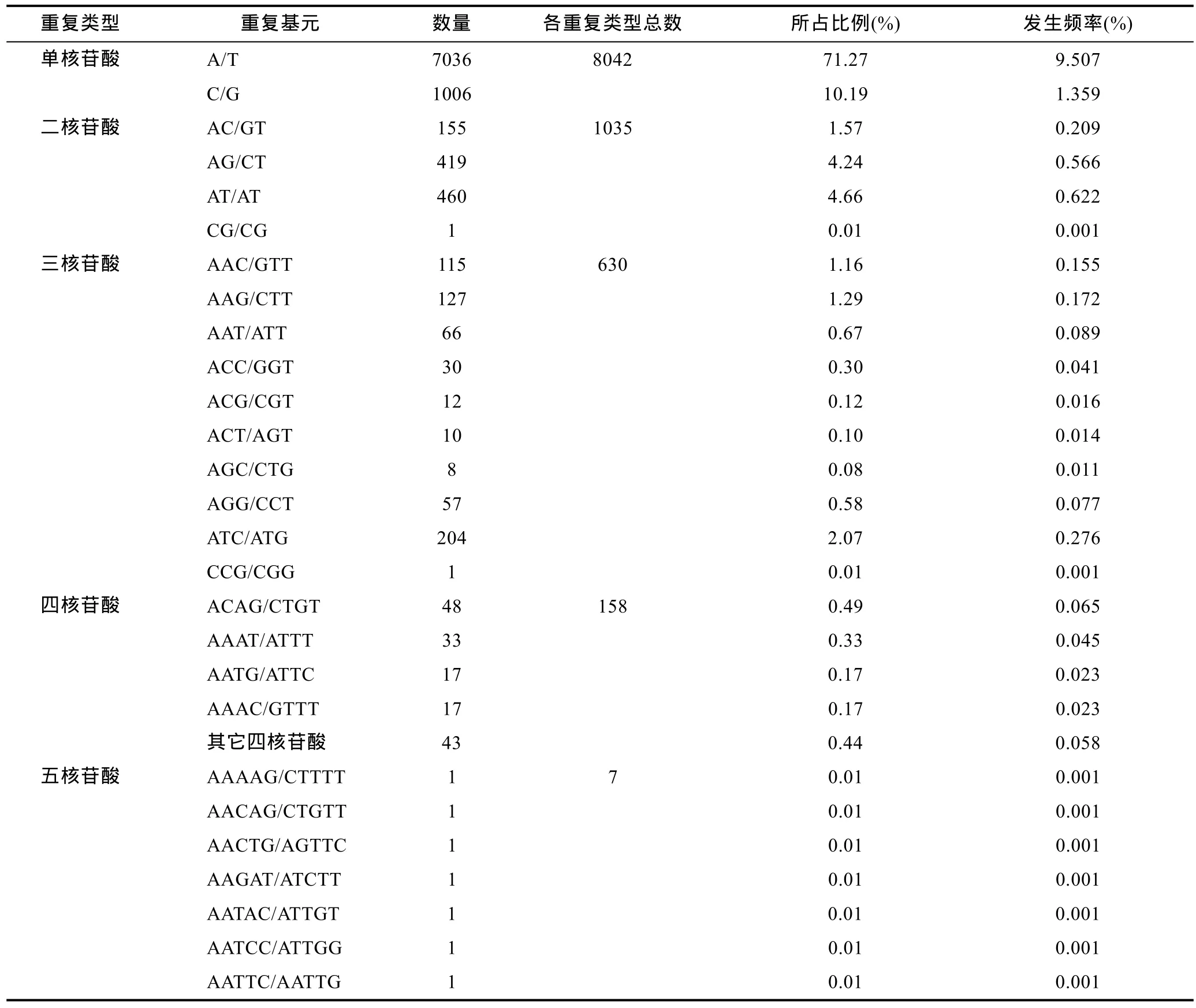
表3 馬氏珠母貝EST-SSRs重復基元的類型及頻率Tab.3 EST-SSR repeats motifs and the frequency in P. martensii transcriptome
根據現有報道, 蝦夷扇貝和馬氏珠母貝的EST-SSR均以二核苷酸重復基元為主, 其所占比例分別為 40.54%和 48.5% (石耀華等, 2008; 李云峰等,2010); 縊蟶(Sinonovacula constricta)SSR以三核苷酸重復SSR比列最高, 為37.13%(劉博等, 2012); 此外,基于馬氏珠母貝珍珠囊轉錄組的 SSR分析中, 四堿基重復基元占總EST-SSR的40%(石耀華等, 2008)。而本研究中以單核苷酸重復基元為主, 比列達到81.46%。造成以上差異的原因, 除EST序列獲取方法不同外(王學穎, 2012), 在一定程度上與搜索 SSR時設置的參數有很大的關系(Wei et al, 2011)。Aggarwal等(2007)發現, 如果改變 SSR搜索標準(提高二核苷酸重復基元的重復次數, 或降低三核苷酸重復基元的重復次數), 原本二核苷酸重復 SSR占優勢的研究中, 便會出現三核苷酸重復SSR占優勢的情況; 同樣,若本研究中提高單核苷酸 SSR的重復次數, 則 SSR將以二核苷酸重復基元為主。因此, 亟需建立一個統一的 SSR檢測參數標準以便有效比較不同研究中同一或相近物種SSR分布的類型與特點。
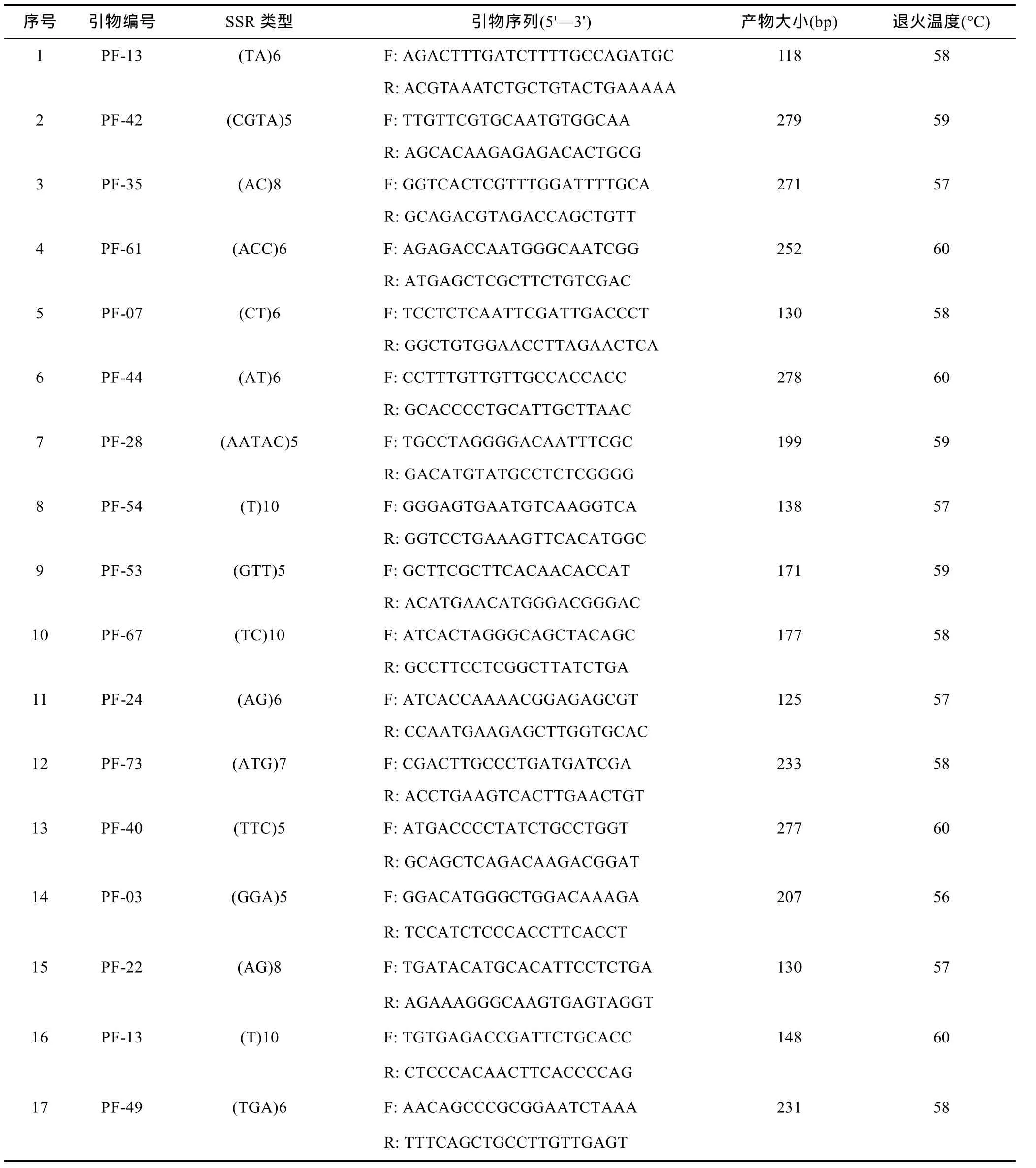
表4 17對多態性EST-SSRs引物序列及擴增參數Tab.4 Sequences and amplification parameters of the 17 EST-SSRs primers
研究表明, SSR標記可用性的重要依據——多態性的高低, 主要取決于的其長度大小(李珊等, 2010)。當SSR長度≥20bp時其多態性較高, 長度在12—20bp之間的SSR則呈現中等多態性, 而長度<12bp時多態性較低(楊華等, 2011)。本研究中, 長度在 12—20bp之間的SSR有3677條(占SSR總數的37.25%), 這些SSR具中等多態性; 而長度≥20bp的SSR有336條(占SSR總數的 3.4%), 此類SSR呈較高的多態性。此外, 本研究中20bp以上的低級重復基元(一、二、三核苷酸重復基元)較多, 共計 279條(占 20bp以上SSR的83.04%)。由此可預見馬氏珠母貝轉錄組來源的 SSR具有較高的多態性潛能, 在分子標記研究方面將具有較高的利用價值(Dreisigacker et al, 2004)。
為檢測轉錄組中EST-SSR的多態性, 隨機挑選了80對引物進行PCR擴增, 其中62對引物成功擴增出穩定條帶, 但仍有18條引物未能擴增出產物。這可能是由于所設計的引物序列位于兩個外顯子上, 或者基因組對應序列含有內含子而不具備SSR序列特征等原因造成(Varshney et al, 2005)。此外,本研究出現了 23對引物擴增產物片段與預期產物片段大小不符的現象, 而該現象同樣出現在櫛孔扇貝和縊蟶的 EST-SSR擴增產物中(李紅蕾等, 2003;劉博等, 2012)。究其原因, 有學者認為其與引物的非特異性結合(錯配)或基因組 DNA序列含有內含子等相關(李紅蕾等, 2003; Saha et al, 2004)。17對多態性引物所在SSR位點中, 多為二、三核苷酸重復, 這從某種程度上也驗證了低級重復基元多態性高于高級重復基元多態性的推斷(Dreisigacker et al,2004)。
4 結論
本研究利用馬氏珠母貝的轉錄組數據, 篩選獲得了9872個EST-SSR位點, 位點出現頻率達13.34%;同時, 共為5922條EST-SSR序列成功設計出17766對引物; 隨機選擇的 80對擴增引物中, 共有 17對EST-SSR引物表現出個體多態性, 多態性比率為27.42%。以上研究結果對于豐富馬氏珠母貝分子標記類型、加速功能基因資源的利用、探究種群遺傳結構、分析遺傳多樣性、保護種質資源和培育優良品種均具有重要意義。
王 東, 曹玲亞, 高建平, 2014. 黨參轉錄組中SSR位點信息分析. 中草藥, 45(16): 2390—2394
王學穎, 2012. 馬氏珠母貝微衛星引物的批量開發及選育系和珍珠囊的標記分析. 湛江: 廣東海洋大學碩士學位論文
石耀華, 洪 葵, 郭希明等, 2008. 馬氏珠母貝EST微衛星的篩選. 水產學報, 32(2): 174—181
史兼華, 何毛賢, 黃良民等, 2006. 馬氏珠母貝選育系遺傳變異的AFLP分析. 高技術通訊, 16(5): 534—538
劉 博, 邵艷卿, 滕爽爽等, 2012. 縊蟶(Sinonovacula constricta)EST-SSR分布特征及引物開發利用. 海洋與湖沼, 43(1): 132—137
孫國華, 楊建敏, 宋志樂等, 2010. 刺參(Apostichopus japonicus)EST 序列中微衛星分布分析及其標記的篩選.海洋與湖沼, 41(1): 133—139
李 珊, 周天華, 趙桂仿等, 2010. 馬蹄香表達序列標簽資源的SSR信息分析. 中草藥, 41(3): 464—468
李 琪, 2006. 海洋貝類微衛星 DNA標記的開發及其在遺傳學研究中的應用. 中國水產科學, 13(3): 502—509
李云峰, 劉衛東, 高祥剛等, 2010. 蝦夷扇貝外套膜和腎臟組織cDNA文庫構建以及EST 的初步分析. 中國水產科學,17(3): 578—585
李紅蕾, 宋林生, 王玲玲等, 2003. 櫛孔扇貝EST中微衛星標記的篩選. 高技術通訊, 13(12): 72—75
李春艷, 丁 君, 常亞青等, 2009. 蝦夷扇貝微衛星標記的分離及其養殖群體的遺傳結構分析. 中國水產科學, 16(1):39—46
楊 華, 陳 琪, 韋朝領等, 2011. 茶樹轉錄組中SSR位點的信息分析. 安徽農業大學學報, 38(6): 882—886
張 瓊, 劉小林, 李喜蓮等, 2010. EST-SSR分子標記在水生動物遺傳研究中的應用. 水產科學, 29(5): 302—306
趙瑩瑩, 朱曉琛, 孫效文等, 2006. 蝦夷扇貝的多態性微衛星座位. 動物學報, 52(1): 229—233
徐美佳, 張 妍, 魯翠云等, 2009. 利用 EST-SSR座位對鯉魚 4種生長性狀的單標記回歸分析. 水產學雜志, 22(1): 15—18
Aggarwal R K, Hendre P S, Varshney R K et al, 2007.Identification, characterization and utilization of EST-derived genic microsatellite markers for genome analyses of coffee and related species. Theor Appl Genet,114(2): 359—372
Dreisigacker S, Zhang P, Warburton M L et al, 2004. SSR and pedigree analyses of genetic diversity among CIMMYT wheat lines targeted to different megaenvironments. Crop Sci, 44(2): 381—388
Grabherr M G, Haas B J, Yassour M et al, 2011. Full-length transcriptome assembly from RNA-Seq data without a reference genome. Nat Biotechnol, 29(7): 644—652
He M X, Guan Y Y, Yuan T et al, 2008. Realized heritability and response to selection for shell height in the pearl oyster Pinctada fucata (Gould). Aquac Res, 39(8): 801—805
Liu T M, Zhu S Y, Fu L L et al, 2013. Development and characterization of 1, 827 expressed Sequence tag-derived simple sequence repeat markers for ramie (Boehmeria nivea L. Gaud). PLoS One, 8(4): e60346
Lu X, Wang H X, Liu B Z et al, 2013. Three EST-SSR markers associated with QTL for the growth of the clam Meretrix meretrix revealed by selective genotyping. Mar Biotechnol,15(1): 16—25
Miyazaki T, Goto K, Kobayashi T et al, 1999. Mass mortalities associated with a virus disease in Japanese pearl oysters Pinctada fucata martensii. Dis Aquat Org, 37(1): 1—12
Powell W, Machray G C, Provan J, 1996. Polymorphism revealed by simple sequence repeats. Trends Plant Sci, 1(7):215—222
Saha M C, Rouf Mian M A, Eujayl I et al, 2004. Tall fescue EST-SSR markers with transferability across several grass species. Theor Appl Genet, 109(4): 783—791
Sato M, Kawamata K, Zaslavskaya N et al, 2005. Development of microsatellite markers for Japanese scallop (Mizuhopecten yessoensis) and their application to a population genetic study. Mar Biotechnol, 7(6): 713—728
Tomaru Y, Kawabata Z, Nakano S, 2001. Mass mortality of the Japanese pearl oyster Pinctada fucata martensii in relation to water temperature, chlorophyll a and phytoplankton composition. Dis Aquat Org, 44(1): 61—68
Varshney R K, Graner A, Sorrells M E et al, 2005. Genic microsatellite markers in plants: features and applications.Trends Biotechnol, 23(1): 48—55
Wang Z L, Wu Z H, Jian J C et al, 2009. Cloning and expression of heat shock protein 70 gene in the haemocytes of pearl oyster (Pinctada fucata, Gould 1850) responding to bacterial challenge. Fish Shellfish Immunol, 26(4): 639—645
Wei W L, Qi X Q, Wang L H et al, 2011. Characterization of the sesame (Sesamum indicum L.) global transcriptome using Illumina paired-end sequencing and development of EST-SSR markers. BMC Genomics, 12: 451

