甜蕎重組自交系(RIL)親本基因組RAPD標(biāo)記的多樣性分析
郭菊卉+田娟+石桃雄+陳慶富

摘要:以貴州師范大學(xué)蕎麥產(chǎn)業(yè)技術(shù)研究中心建立的兩個(gè)甜蕎(Fagopyrum esculentum)重組自交系群體(Homo×甜自100和Lorena-3×甜自21-1)的親本為研究對(duì)象,選取了5個(gè)多態(tài)性好、帶型清楚的隨機(jī)引物進(jìn)行RAPD分析,計(jì)算了每對(duì)親本之間的相似度系數(shù),并構(gòu)建了各親本的RAPD指紋圖譜。結(jié)果表明,在Homo×甜自100和Lorena-3×甜自21-1的親本中,平均相似度系數(shù)分別為39.13%和42.69%;在構(gòu)建的RAPD指紋圖譜中,有29條譜帶為Homo(P1)所獨(dú)有,27條譜帶為甜自100(P2)所特有;有26條譜帶為L(zhǎng)orena-3(P1)所獨(dú)有,25條譜帶為甜自21-1(P2)所特有。蕎麥特異譜帶可以作為品種鑒定的分子標(biāo)記。
關(guān)鍵詞:甜蕎(Fagopyrum esculentum);RAPD;多樣性;DNA指紋圖譜
中圖分類號(hào):S517 ? ? ? ? ? ?文獻(xiàn)標(biāo)識(shí)碼:A ? ? ? ?文章編號(hào):0439-8114(2015)02-0284-05
DOI:10.14088/j.cnki.issn0439-8114.2015.02.008
The Diversity Analysis of Parents of Common Buckwheat RIL Populations
with RAPD Markers
GUO Ju-hui,TIAN Juan,SHI Tao-xiong,CHEN Qing-fu
(Research Center of Buckwheat Industry Technology/Institute of Plant Genetics and Breeding, School of Life Science,
Guizhou Normal University, Guiyang 550001, China)
Abstract: The genomic polymorphisms of four parents of two common buckwheat RIL populations were analyzed with RAPD markers of 5 random primers. The results showed that there were the average index of similarity of 39.13% and 42.69% in the parents of the crosses Homo×Tianzi 100 and Lorena-3×Tianzi 21-1, respectively. There were 29 unique bands for Homo(P1) and 27 for Tianzi 100 in the first cross,and 26 for Lorena-3 and 25 for Tianzi 21-1 in the second cross.
Key words:common buckwheat; RAPD; diversity; DNA fingerprint
甜蕎(Fagopyrum esculentum)為蕎麥大粒組類群7個(gè)生物學(xué)物種之一[1]。Chen[2,3]的試驗(yàn)研究表明,普通蕎麥中存在著大量的形態(tài)變異,而對(duì)這些變異進(jìn)行深入研究將在很大程度上有利于遺傳育種。基因控制著生物體的性狀表達(dá)和變異,研究DNA分子的多態(tài)性也會(huì)促進(jìn)對(duì)形態(tài)變異的進(jìn)一步探索,并在遺傳育種中為選擇強(qiáng)優(yōu)勢(shì)組合的親本進(jìn)行雜交和蕎麥資源的鑒定提供科學(xué)依據(jù)[4]。
RAPD技術(shù)是以PCR為基礎(chǔ)的一項(xiàng)檢測(cè)物種DNA多態(tài)性的分子技術(shù)[5]。該技術(shù)的優(yōu)點(diǎn)是DNA樣品需求量小;無(wú)種屬和基因組結(jié)構(gòu)特異性,一套引物可用于不同生物基因組分析;試驗(yàn)成本低;試驗(yàn)技術(shù)簡(jiǎn)單,檢測(cè)速度較快。RAPD在蕎麥中應(yīng)用主要集中在蕎麥的遺傳多樣性和種間系統(tǒng)關(guān)系研究方面[6]。通過(guò)建立RAPD指紋圖譜,尋找每個(gè)種類的恒定譜帶和獨(dú)特譜帶,可以迅速準(zhǔn)確地對(duì)蕎麥種類進(jìn)行鑒別。近年來(lái),RAPD技術(shù)已應(yīng)用于多種植物雜交組合后代鑒定[7,8]和種子純度的檢測(cè)等。Ohinshi等[9]、Tsuji等[10]利用AFLP和RAPD技術(shù)研究苦蕎栽培品系和天然種群間的系統(tǒng)發(fā)育關(guān)系。Sharma[11]應(yīng)用RAPD技術(shù)研究了蕎麥屬14個(gè)種之間的遺傳多樣性。Kump等[12,13]采用RAPD技術(shù)進(jìn)行了苦蕎33個(gè)栽培種之間遺傳關(guān)系的分析。許瑾等[4]利用RAPD技術(shù)對(duì)蕎麥屬的9個(gè)品種進(jìn)行基因組指紋圖譜分析。譚萍等[14]采用RAPD方法對(duì)國(guó)內(nèi)10個(gè)蕎麥栽培種進(jìn)行基因型分析,并建立了指紋圖譜。Pan等[15]以F. esculentum Moench, F. esculentum var. homotropicum 及其雜交所建立的 225 株 F2代分離群體為材料,進(jìn)行了 RAPD 與 STS 標(biāo)記分析和重要形態(tài)性狀遺傳分析,并由此構(gòu)建遺傳標(biāo)記連鎖圖譜,共涉及87個(gè)RAPD標(biāo)記,12個(gè)STS標(biāo)記。覆蓋基因組長(zhǎng)度655.2 cM。總的說(shuō)來(lái),由于普通蕎麥自交不親和,相關(guān)研究主要以蕎麥品種或品系為材料,主要運(yùn)用RAPD等標(biāo)記。
在重組自交系中,利用親本DNA樣品進(jìn)行RAPD 標(biāo)記引物篩選,并將重復(fù)性好的引物進(jìn)行其可育F2代分離群體樣本的PCR擴(kuò)增,用以分析甜蕎RAPD標(biāo)記多態(tài)性和構(gòu)建起分子遺傳連鎖圖譜。本研究對(duì)普通蕎麥重組自交系群體親本組合(Homo×甜自100,Lorena-3×甜自21-1)的親本用5種隨機(jī)引物的RAPD譜帶進(jìn)行多態(tài)性分析和指紋圖譜建立,以期為其F2代自交系群體的進(jìn)一步研究打下基礎(chǔ)。endprint
1 ?材料與方法
1.1 ?試驗(yàn)材料
本研究選用了甜蕎重組自交系的4個(gè)親本甜蕎種,分別為L(zhǎng)orena-3、甜自21-1、甜自100和Homo。重組自交系的親本組合為:Homo×甜自100,Lorena-3×甜自21-1。甜蕎品種由貴州師范大學(xué)蕎麥產(chǎn)業(yè)技術(shù)研究中心提供。
1.2 ?DNA提取
取2~3周苗齡的新鮮健康葉片約300 mg置于研缽中,倒入液氮,研成粉末。將該粉末直接轉(zhuǎn)入裝有預(yù)熱的含有15 μL β-巰基乙醇的1 000 μL 2% CTAB提取緩沖液(質(zhì)量濃度2% CTAB,100 mmol/L pH 8.0 Tris-HCl,50 mmol/L EDTA-Na2,1.4 mol/L NaCl,質(zhì)量濃度2% PVP)的5 mL離心管中,輕輕搖動(dòng)使其充分混勻,置于65 ℃水浴中保溫60 min(每隔20 min輕輕顛倒混勻1次),加入酚/氯仿/異戊醇混合液(25∶24∶1,V/V/V),充分混勻,常溫下靜置10 min,10 000 r/min 離心10 min。將上清液加入氯仿/異戊醇混合液(24∶1,V/V),輕輕混勻,室溫下10 000 r/min離心10 min。將上清液轉(zhuǎn)移到干凈的離心管中,加入2倍體積-20 ℃冰凍的無(wú)水乙醇,輕輕顛倒離心管直至出現(xiàn)白色絮狀沉淀,將離心管放在 ? ?-20 ℃中沉淀10 min。離心沉淀DNA并將沉淀用 -20 ℃冰凍的體積分?jǐn)?shù)為70%的乙醇洗滌兩次,在室溫下使DNA沉淀干燥。加入600 μL TE緩沖液,完全溶解后,置于-20 ℃冰箱保存?zhèn)溆谩?/p>
1.3 ?引物
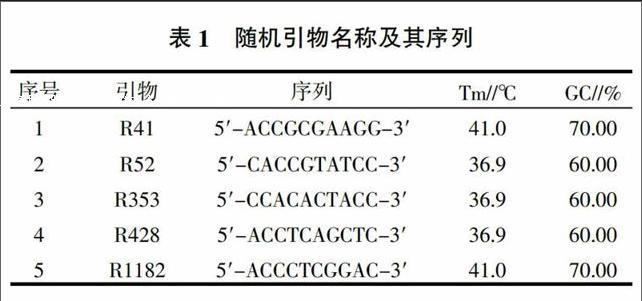
共使用了5個(gè)隨機(jī)引物,各引物序列見表1。該引物從貴州師范大學(xué)蕎麥產(chǎn)業(yè)技術(shù)研究中心設(shè)計(jì)的25個(gè)引物中選取,以親本間多態(tài)性好、電泳帶型清晰、重復(fù)性好為主要篩選原則。
1.4 ?PCR反應(yīng)體系
試驗(yàn)中,RAPD檢測(cè)使用的PCR反應(yīng)體系見表2。PCR反應(yīng)程序?yàn)椋?4 ℃預(yù)變性10 min;94 ℃變性1 min,Tm(引物不同溫度不同)退火1 min,72 ℃延伸2 min,38個(gè)循環(huán);72 ℃延伸10 min。
1.5 ?統(tǒng)計(jì)分析方法
擴(kuò)增產(chǎn)物于0.8%的瓊脂糖凝膠中電泳,在BioSenSC805型全自動(dòng)凝膠成像系統(tǒng)的Analysis工具欄中,點(diǎn)擊消除背景圖標(biāo)以消除圖像本底的干擾;點(diǎn)擊自動(dòng)檢測(cè)所有泳道的條帶圖標(biāo)以進(jìn)行各泳道內(nèi)條帶檢測(cè);點(diǎn)擊相對(duì)分子質(zhì)量計(jì)算圖標(biāo),以Marker泳道內(nèi)的Marker條帶的相對(duì)分子質(zhì)量為標(biāo)準(zhǔn)設(shè)定相對(duì)分子量曲線圖,關(guān)閉Marker窗口,則會(huì)顯示各個(gè)條帶的相對(duì)分子質(zhì)量,統(tǒng)計(jì)各泳道內(nèi)的條帶數(shù)。
參考文獻(xiàn)[16],采用條帶相似度系數(shù)分析條帶的多樣性。相似度系數(shù)SD=(2nAB)/(nA+nB),nA為A泳道內(nèi)的DNA條帶數(shù),nB為B泳道內(nèi)的DNA條帶數(shù),nAB是A、B泳道內(nèi)共有的條帶數(shù)。
2 ?結(jié)果與分析
2.1 ?親本甜蕎的RAPD電泳圖譜
不同引物擴(kuò)增的親本甜蕎的RAPD電泳圖譜如圖1和圖2所示。
2.2 ?親本甜蕎RAPD標(biāo)記的多樣性分析
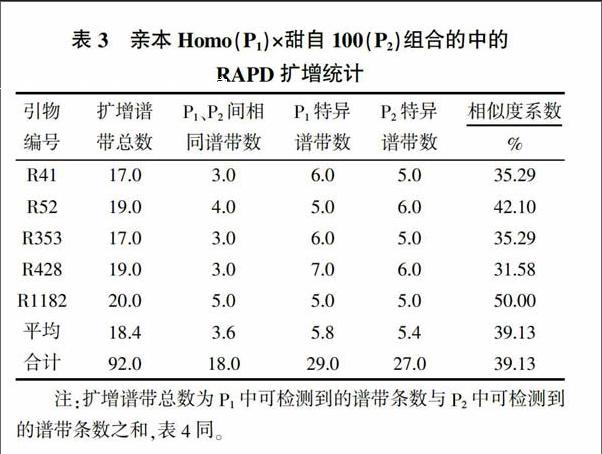
根據(jù)篩選出的5個(gè)隨機(jī)引物的RAPD擴(kuò)增結(jié)果,可以對(duì)每對(duì)雜交組合中的兩個(gè)親本進(jìn)行擴(kuò)增條帶的多樣性分析,兩對(duì)雜交組合親本中各引物的擴(kuò)增情況與相似度系數(shù)見表3和表4。
由表3可知,對(duì)親本Homo(P1)×甜自100(P2)的組合進(jìn)行RAPD擴(kuò)增,5個(gè)引物共擴(kuò)增出92條譜帶,平均每個(gè)隨機(jī)引物產(chǎn)生了18.4條譜帶。這些引物的擴(kuò)增產(chǎn)物為17~20條譜帶,其中擴(kuò)增條帶數(shù)目最多的是引物R1182,共有20條譜帶;其次為引物R52和R428,均為19條譜帶;最少的為引物R41和R353,擴(kuò)增出17條譜帶。
對(duì)親本Homo(P1)×甜自100(P2)組合的所有隨機(jī)引物的RAPD標(biāo)記多樣性進(jìn)行檢測(cè),結(jié)果(表3)表明,在5個(gè)隨機(jī)引物獲得的92條譜帶中,有18條譜帶在兩親本間表現(xiàn)為相同譜帶,RAPD標(biāo)記的相似度系數(shù)為39.13%(36/92)。對(duì)于每一條引物的相似度系數(shù)來(lái)說(shuō),其變化范圍為31.58%(6/19)~50.00%(10/20)。其中RAPD標(biāo)記的相似度系數(shù)最高的引物是R1182,達(dá)到了50.00%,引物R428的標(biāo)記相似度系數(shù)最低,為31.58%(6/19)。因此,本研究所采用的雜交組合親本Homo(P1)×甜自100(P2)在基因組DNA水平上存在較低的RAPD標(biāo)記相似度系數(shù),即DNA水平上條帶的多樣性較為豐富。有利于其子代群體獲得更為廣泛的性狀變異。
由表4可知,對(duì)親本Lorena-3(P1)×甜自21-1(P2)的組合進(jìn)行RAPD擴(kuò)增,5個(gè)引物共擴(kuò)增出89條譜帶,平均每個(gè)隨機(jī)引物產(chǎn)生了17.8條譜帶。這些引物的擴(kuò)增產(chǎn)物為15~20條譜帶,其中擴(kuò)增條帶數(shù)目最多的是引物R1182,共有20條譜帶;而后依次為R52、R353、R41、R428,擴(kuò)增出的譜帶數(shù)分別為19、18、17、15。
對(duì)Lorena-3(P1)×甜自21-1(P2)的親本組合所有隨機(jī)引物的RAPD標(biāo)記多樣性進(jìn)行檢測(cè),結(jié)果(表4)表明,在5個(gè)隨機(jī)引物獲得的89條譜帶中,有51條譜帶在兩親本間的表現(xiàn)存在差異性,RAPD標(biāo)記的相似度系數(shù)為42.69%(38/89)。對(duì)每條引物的標(biāo)記相似度系數(shù)來(lái)說(shuō),其變化范圍為40.0%(6/15)~47.06%(8/17)。其中RAPD標(biāo)記的相似度系數(shù)最低的引物為R428和R1182,相似度系數(shù)為40.00%,R41的標(biāo)記相似度系數(shù)最高,達(dá)47.06%(8/17)。因此,本研究所采用的雜交組合親本Lorena-3(P1)×甜自21-1(P2)基因組DNA水平上同樣存在較低的RAPD標(biāo)記相似度,也存在較高的多樣性,將有利于其子代群體獲得更為廣泛的性狀變異。endprint
2.3 ?親本甜蕎的RAPD指紋圖譜
根據(jù)全部隨機(jī)引物擴(kuò)增產(chǎn)物的數(shù)據(jù)統(tǒng)計(jì)分析結(jié)果,將能在親本組合中穩(wěn)定擴(kuò)增出現(xiàn)的、且具有差異的特異性譜帶用來(lái)構(gòu)建親本組內(nèi)P1和P2的RAPD標(biāo)記指紋圖譜。在Homo(P1)×甜自100(P2)的親本中,共擴(kuò)增出56條差異性譜帶(表5);在Lorena-3(P1)×甜自21-1(P2)的親本中,共擴(kuò)增出51條差異性譜帶(表6)。
由表5和表6可知,Homo(P1)×甜自100(P2)和Lorena-3(P1)×甜自21-1(P2)組合親本RAPD指紋圖譜存在較明顯的差異性。前者中有28條譜帶為Homo(P1)所獨(dú)有,27條譜帶為甜自100(P2)所特有;后者中有24條譜帶為L(zhǎng)orena-3(P1)所獨(dú)有,24條譜帶為甜自21-1(P2)所特有。因此,在每對(duì)雜交組合中,存在差異的譜帶可看作是兩親本的特征性譜帶,而由RAPD指紋圖譜所反應(yīng)的這些特征譜帶可以作為鑒別親本的依據(jù),也可為進(jìn)一步對(duì)雜交組合中親本的DNA多態(tài)性及其他分子遺傳研究打下基礎(chǔ)。
3 ?討論
Deng等[17]利用7個(gè)隨機(jī)引物對(duì)19個(gè)蕎麥(包括甜蕎和苦蕎)品種進(jìn)行RAPD分析,結(jié)果表明其平均多態(tài)性達(dá)94.89%,而本研究結(jié)果表明,甜蕎重組自交系的兩對(duì)親本組合的平均相似度系數(shù)分別為39.13%(Homo×甜自100)和42.69%(Lorena-3×甜自21-1),再次在DNA水平上說(shuō)明蕎麥種間的多態(tài)性遠(yuǎn)高于種內(nèi)多態(tài)性。同時(shí),對(duì)同屬甜蕎品種的兩個(gè)親本組合而言,其低于50%的相似度系數(shù)也從DNA的角度解釋了甜蕎異花授粉、自交不親和等生殖學(xué)特征所帶來(lái)的物種基因重組的多態(tài)性現(xiàn)象。在對(duì)蕎麥進(jìn)行種類劃分的研究中,部分種類難以從形態(tài)學(xué)上進(jìn)行識(shí)別,分類特征也常存在較大變異,難以把握。本研究在DNA水平上進(jìn)行的RAPD遺傳標(biāo)記分析可對(duì)蕎麥品種鑒別和遺傳歧化研究提供依據(jù)。
此外,本研究以重組自交系親本為研究對(duì)象進(jìn)行RAPD分析,發(fā)現(xiàn)自交系群體親本之間存在較多的特異RAPD譜帶,也可為以重組自交系為研究對(duì)象進(jìn)行的相關(guān)農(nóng)藝學(xué)特征遺傳及分子標(biāo)記研究奠定基礎(chǔ)。
參考文獻(xiàn):
[1] 陳慶富.蕎麥屬植物科學(xué)[M].北京:科學(xué)出版社,2012.
[2] CHEN Q F. Hybridization between Fagopyrum(Polygonaceae) species native to China[J]. Botanical Journal of the Linnean Society,1999,131:177-185.
[3] CHEN Q F.A study of resource of Fagopyrum(Polygonaceae) native to China[J]. Botanical Journal of the Linncan Socicty,l999,130:53-64.
[4] 許 ?瑾,周小梅,范玲娟,等.蕎麥RAPD指紋圖譜的建立及在品種鑒定中的應(yīng)用[J].山西大學(xué)學(xué)報(bào)(自然科學(xué)版),2006,29(2):194-197.
[5] WILLIAMS J G K,KUBELIK A R,LIVAK K J,et al. DNA polymorphisms amplified by arbitrary primers are useful as genetic markers[J]. Nucleic Acids Res,1990,18:6531-6535.
[6] SHARMA T R,JANA S.Random amplified polymorphic DNA(RAPD)variation in Fagopyrum tataricum Gaerm.accessions from China and the Himalayan region[J]. Euphytica,2002,127:327-333.
[7] 李文彬,梁紅健,孫勇如,等.RAPD鑒定栽培稻和野生稻體細(xì)胞雜種[J]. 生物工程學(xué)報(bào),1996,12(4):390-393.
[8] 趙同金,向鳳寧,聶 ?卉,等.5SrDNA間隔序列分析和RAPD技術(shù)用于鑒定體細(xì)胞雜種[J].植物研究,2004,24(1):120-124.
[9] OHINSHI O,MATSUOKA Y.Search for the wild ancestor of buchwheat ll.Taxonomy of Fagopyrum(Polygonaeeae) species based on morphology, isozymes and cpDNA variability[J].Genes Genet.Syst,1996,71:383-390.
[10] TSUJI K,OHNISHI O. Origin of cultivated Tartary buckwheat(Fagopyrum tarieum Gaertn.) revealed by RAPD analyses[J]. Genetics Resources and Crop Evolution, 2000,47:431-438.
[11] SHARMA T R. Species relationships in Fagopyrum revealed by PCR-based DNA fingerprinting[J]. Theor APPl Genet,2002,105:306-312.
[12] KUMP B,JAVORNIK B.Evaluation of genetic vaiability among common buckwheat(Fagopyrum eseulentum Moeneh), population by RAPD makers[J]. Plant seience,1996,114:149-158.
[13] KUMP B, JAVORNIK B. Genetic diversity and relationships among cultivated and wild aceessions of tartary buckwheat (Fagopyrum tataricum Gaertn.) as revealed by RAPD markers[J].Genetic Resources and Crop Evolution,2002,49:565-572.
[14] 譚 ?萍,王玉株.十種栽培苦蕎麥的隨機(jī)擴(kuò)增多態(tài)性DNA(RAPD)研究[J],種子,2006,25(7):46-49.
[15] PAN S J, CHEN Q F. Genetic mapping of common buckwheat using DNA,protein and morphological markers[J]. Hereditas,2010,147:27-33.
[16] EICHNER C A,ERB R W,TIMMIS K N.Thermal gradient gel electrophoresis analysis of bioprotection from pollutant shocks in the activated sludge microbial community[J]. Appl Environ Microbiol,1999,65:102-109.
[17] DENG L Q,ZHANG K,HUANG K F,et al. RAPD analysis for genetic diversity of nineteen common and Tartary Buckwheat[J]. Agricultural Science&Technology,2011,12(1):65-69.endprint

