利用染色體片段置換系群體檢測水稻產量相關性狀QTL
周麗慧, 張亞東, 朱 鎮, 陳 濤, 趙慶勇, 姚 姝, 趙 凌, 趙春芳, 于 新,王才林
(江蘇省農業科學院糧食作物研究所/江蘇省優質水稻工程技術研究中心/國家水稻改良中心南京分中心,江蘇南京 210014)
利用染色體片段置換系群體檢測水稻產量相關性狀QTL
周麗慧, 張亞東, 朱 鎮, 陳 濤, 趙慶勇, 姚 姝, 趙 凌, 趙春芳, 于 新,王才林
(江蘇省農業科學院糧食作物研究所/江蘇省優質水稻工程技術研究中心/國家水稻改良中心南京分中心,江蘇南京 210014)
為了發掘單株產量及其構成因素相關性狀的數量性狀基因座(QTL),本研究以9311/日本晴染色體片段置換系群體為材料,調查了單株產量及單株產量構成因素(單株實粒數、單株總穎花數、結實率、每穗實粒數、有效穗數、每穗穎花數、千粒質量等8個性狀)。利用IciMapping v3.1軟件,將分子標記檢測結果與田間性狀調查值相結合,定位了與單株產量、單株實粒數、千粒質量、每穗穎花數、結實率、單株總穎花數6個性狀有關的QTL,未定位到與有效穗數(EPN)和每穗實粒數(GPP)有關的QTL。共定位到的10個相關QTLs,分別分布于第1條、第2條、第5條、第7條、第8條染色體的7個區間,貢獻率為7.52%~44.59%,其中4個QTLs的貢獻率大于10.00%。單株產量qGY1,單株實粒數qGN1,結實率qSSR1.2、qSSR2和qSSR8,加性效應值為負值,表明9311的等位基因表現為增效作用;單株總穎花數qSN2,結實率qSSR1.1,千粒質量qTGW5,每穗穎花數qSPP5和qSPP7,加性效應值為正值,表明日本晴的等位基因表現為增效作用。10個QTLs位點中,除qSN2、qTGW5、qSPP5與已克隆的LP、qSW5、OsNADH-GOGAT2可能位于同一區域外,其余7個位點均未被克隆或精細定位。
水稻;染色體片段置換系;數量性狀基因座;產量
追溯近六十年水稻品種的改良史,從上世紀五十年代評選出的地方良種到六十年代矮化育種、七十年代的雜交水稻直到目前正在研究的超高產水稻品種,都是以產量性狀的提高作為前提和目標[1],高產一直是水稻育種的第一目標。近年來由于靠進一步擴大水稻種植面積的潛力已經有限,今后進一步增加產量的途徑只能依賴于單位面積產量的提高。對單株個體而言,單株產量是由單株的有效穗數、每一單穗的穎花數、結實率和千粒質量4個因素決定。
育種研究是以有利基因的發現和利用為前提而開展的。目前關于單株產量及構成因素相關性狀基因或QTL的克隆或精細定位有較多相關的報道,如每穗實粒數基因Gnla[2]和qGN4-1[3],密直立穗和每穗實粒數基因DEP[4]、DEP3[5],每穗穎花數基因Ghd7[6]或qSSP7[7]、SPP1[8],大穗基因LP[9],每穗粒數NADH-谷氨酸合酶基因OsNADH-GOGAT2[10]。控制分蘗或穗數的OsTB1或FC1[11-12]、HTD2、D88或D14[13-15]和DST[16]。理想株型基因IPA1[17],蔗糖轉運蛋白質基因OsSUT2[18]在分蘗數、千粒質量上表現一因多效,OGR1基因影響分蘗、降低育性[19]。粒寬和粒質量基因GW2[20]、qSW[21]、GW5[22],粒長和粒質量基因PGL1[23]、qGL3[24]、GS3[25],粒質量基因gw3.1[26]、TGW6[27]、HGW[28],粒寬、粒質量和結實率基因GS5[29]。影響結實率基因OsSIZ1[30],與磷轉運有關同時影響結實率基因OsPT8[31]等。
盡管大量基因或QTL的發掘為水稻產量性狀的改良提供了先決條件,但是相關基因資源在產量上的育種利用仍然效率不高,可能與產量性狀為數量性狀的特點(如位點的數量較多及位點間的互作等)、背景不同、基因的多效性、產量相關性狀的此長彼消、產量以外其他性狀的極端表型等有關。因此選擇不同的材料配置合適的群體挖掘更多與之有關的位點,對今后研究產量性狀相關基因及其遺傳特點,有著極其重要的意義。
本研究利用豐產性好的秈稻品種揚稻6號(9311)與普通粳稻品種日本晴所構建的CSSLs群體,研究QTL定位,以期為單株產量相關性狀的改良及超高產育種提供新的基因資源。
1 材料與方法
1.1 供試材料
受體親本秈稻品種9311及以其為背景、粳稻品種日本晴為供體的染色體片段置換系(CSSLs), CSSLs的構建過程為:以秈稻品種9311(Oryza sativa L.ssp.indica cv.9311)為受體親本,粳稻品種日本晴(Oryza sativa L.ssp.japonica cv.Nipponbare)為供體親本,得到雜交后代BC4F1,用覆蓋水稻全基因組的親本間具多態性的SSR(Simple sequence repeat)標記檢測BC4F1單株的基因型,選擇基因組內含有少量雜合片段單株的種子種植得到BC4F2群體,通過對BC4F2群體中單株的基因型檢測,獲得一套純合的高代回交置換系[32]。CSSLs共119個純系,置換片段總長度(去除重疊片段)為1 202 cM,占染色體總長度的比例,約為78.6%[33]。
1.2 田間種植與性狀調查
于2010年夏季在江蘇省農業科學院種植CSSLs群體及其親本,所有材料5月至11月種植于院內試驗田中,隨機區組設計,2個重復,每小區種植3行,每行10株,單本栽插,株、行距分別為26.6 cm、33.3 cm,正常田間管理。
成熟后每個小區取中間5株,自然晾干后調查水稻產量相關性狀,包括單株產量、單株實粒數、千粒質量、有效穗數、每穗實粒數、每穗穎花數、單株總穎花數、結實率。相關產量性狀的測定與計算參考Jiang等[34]的方法,有效穗數為單株內實粒數在5粒以上的穗的數目;單株總穎花數為單株全部穎花的數目;單株實粒數為單株除去空癟粒以外的所有粒的數目;單株產量調查方法為單株脫粒后所有實粒的質量;千粒質量=單株產量/單株實粒數×1 000 (g);結實率=單株實粒數/單株總穎花數×100%;每穗實粒數=單株實粒數/有效穗數;每穗穎花數=單株總穎花數/有效穗數。
1.3 數據分析與QTL定位
采用SPSS17.0軟件進行性狀相關分析、差異性比較等;采用Wang等[35]的RSTEP-LRT方法,利用其開發軟件QTL IciMapping v3.1(LOD值為2.0),將分子標記檢測的結果和田間性狀的調查值相結合,對CSSLs群體各株系進行全基因組范圍內單株產量及其構成因素QTL分析研究,QTL的命名遵循McCouch等[36]的原則。
2 結果與分析
2.1 CSSLs群體表型變異
t測驗顯示,絕大部分性狀在雙親之間呈現極顯著差異,僅有效穗數在雙親間差異不顯著,與粳稻品種日本晴相比,9311是豐產性較好的常規秈稻品種,除有效穗數接近一致以外,產量各性狀均較大程度優于日本晴(表1)。單株產量在CSSLs群體中變幅較大,單株總穎花數、單株實粒數、每穗穎花數、每穗實粒數、千粒質量、結實率等都出現類似情況,各株系出現明顯的超親分離現象,表現出較大幅度的變異(圖1)。
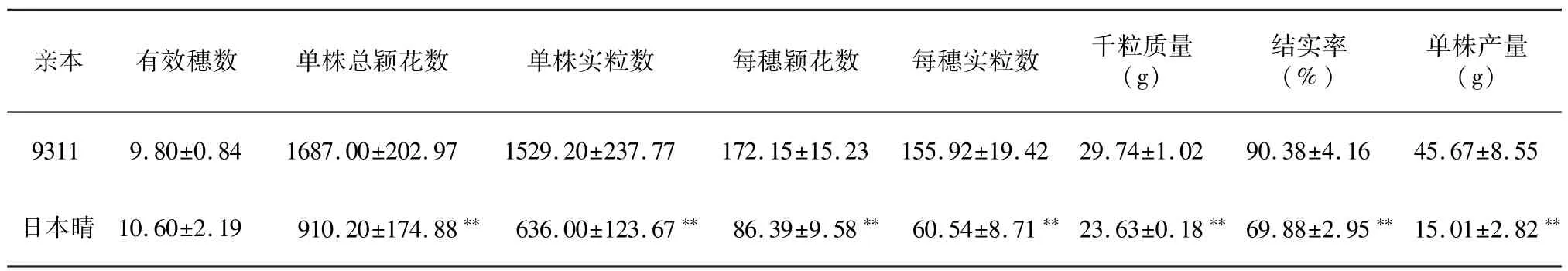
表1 雙親產量性狀Table 1 Yield-related traits of rice 9311 and Nipponbare
2.2 產量相關性狀間相關分析
對各系產量相關性狀的相關分析結果見表2。單株產量除與千粒質量、結實率相關關系不顯著外,與其構成因素各性狀間均呈極顯著正相關;單株實粒數與其構成因素(有效穗數和每穗實粒數)均呈極顯著正相關;同樣地單株總穎花數及其構成因素(有效穗數和每穗穎花數)間情況一致。千粒質量與單株產量相關關系不顯著,與結實率呈極顯著正相關外,而與產量其余性狀均呈極顯著負相關。結實率與有效穗數、單株總穎花數、每穗穎花數均呈極顯著負相關,與實粒數沒有顯著相關性。有效穗數與每穗穎花數和每穗實粒數均呈極顯著負相關。
各系產量相關性狀與其構成因素之間的關系從圖2可以看出,圖2A反映了單株實粒數與千粒質量之間的此長彼消,散點圖中某一點與兩坐標軸圍成方形面積的大小可反映單株產量的大小;圖2B中的任一點與兩坐標軸(單株總穎花數與結實率)圍成方形面積的大小可表明單株實粒數的大小;圖2C中的任一點與兩坐標軸(每穗實粒數與有效穗數)圍成方形面積的大小可表明單株實粒數的大小;圖2D中的任一點與兩坐標軸(每穗穎花數與結實率)圍成方形面積的大小可表明每穗實粒數的大小。圖2中,點所在的橫坐標值和縱坐標值雖存在負向效應,但仍可通過兩者協調找到最佳的組合方式,實現面積(產量)的最大化。
2.3 產量相關性狀的QTLs定位
利用IciMapping v3.1軟件,將分子標記檢測結果與田間性狀調查值相結合,共檢測到10個產量相關性狀QTLs(表3),分布于第1、2、5、7、8染色體共7個區間(圖3),貢獻率在7.52%~44.59%,其中貢獻率大于10.00%的QTL有4個,大部分貢獻率接近10.00%,沒有檢測到有效穗數和每穗實粒數有關的QTLs。
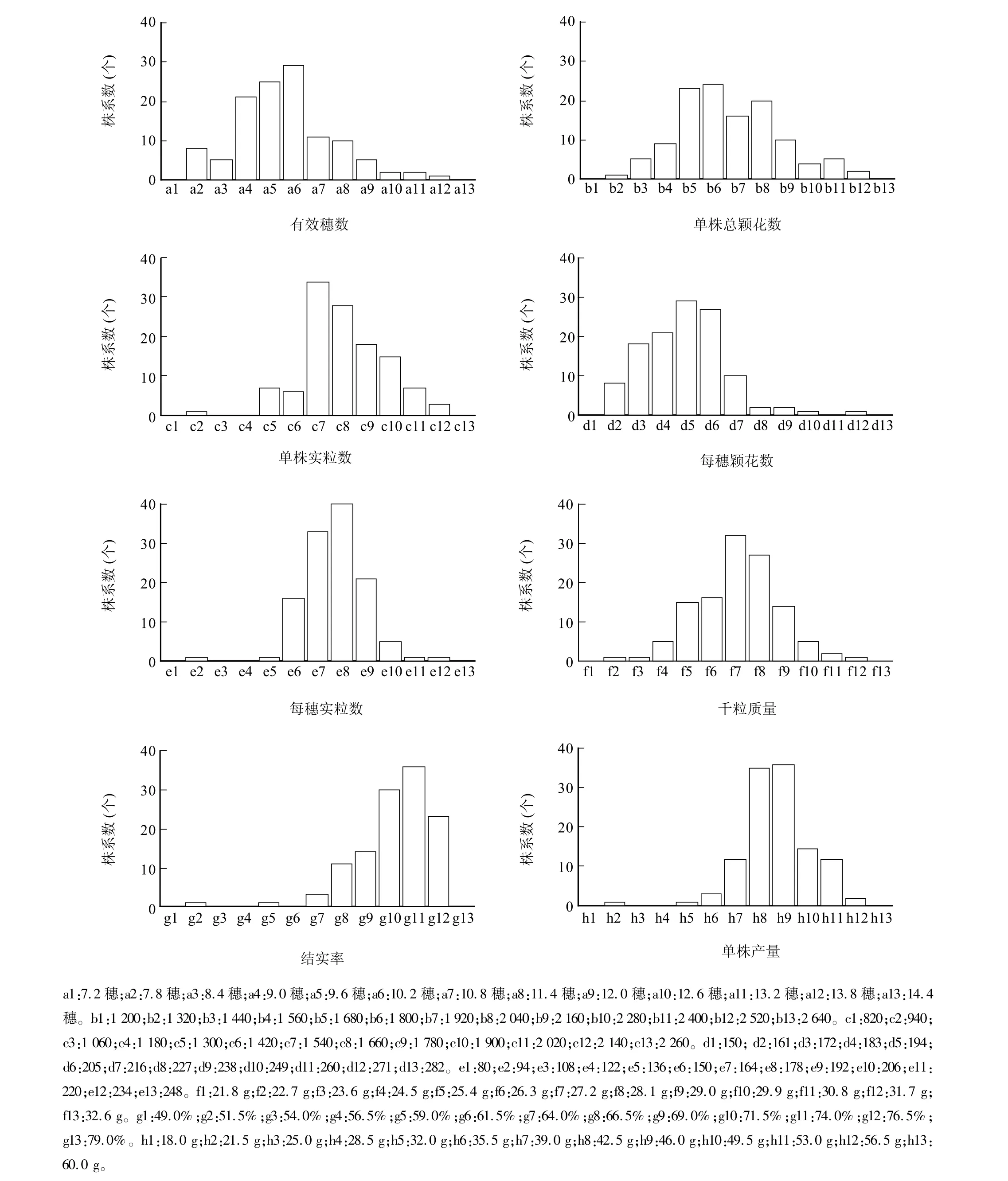
圖1 各系產量性狀頻率分布圖Fig.1 The number of CSSLs corresponding to different yield-related traits

表2 產量性狀間的相關系數Table 2 Correlation coefficients between yield-related traits
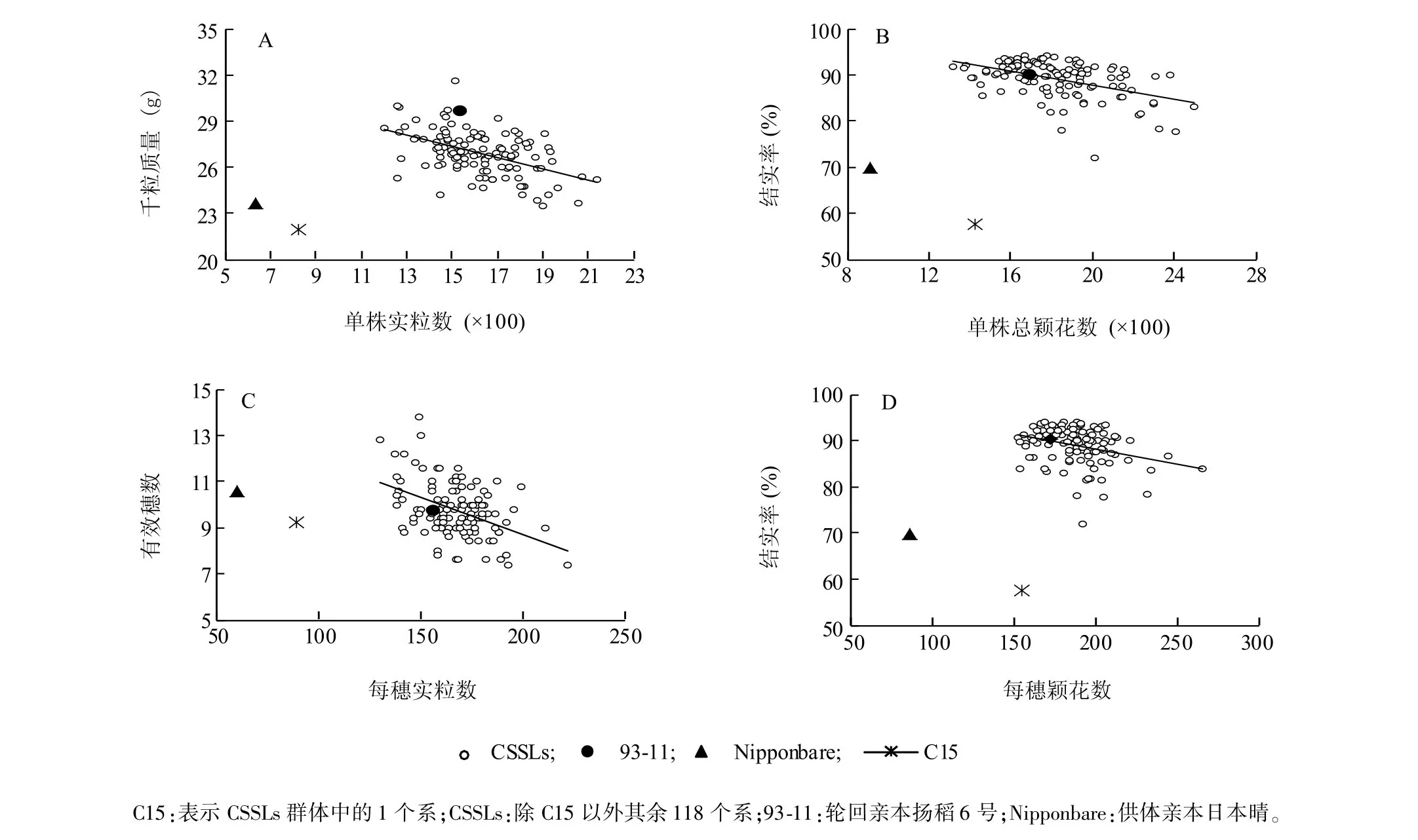
圖2 群體2個產量性狀之間的散點圖Fig.2 Scatter diagram between two yield-related traits in CSSLs
單株產量qGY1被定位在第1染色體RM1387附近,加性效應為-5.58,可解釋表型變異為12.39%,來自供體親本日本晴的替換片段對單株產量的作用表現為減產,純合系的單株產量比輪回親本單株減產11.16 g。單株實粒數qGN1與qGY1定位在同一區域,加性效應為-208.13,可解釋表型變異為10.42%,來自日本晴的替換片段對單株實粒數的作用表現為減少,從加性效應值可看出純合系的單株實粒數比輪回親本減少416粒。這一區域還定位到結實率qSSR1.2,加性效應為-10.34,可解釋表型變異為44.59%,來自供體親本日本晴的替換片段對結實率的作用表現為降低,純合系的單株產量比輪回親本單株降低20.68%。RM1387附近同一區域內的位點,可能由于一因多效作用,結實率降低,從而引起單株實粒數的下降,最終導致單株產量的下降。
結實率其余的3個QTLs中有2個QTLs分別位于第2染色體RM7286附近、第8染色體RM6863附近的qSSR2與qSSR8,加性效應為負,貢獻率小于qSSR1.2,來自供體親本日本晴的替換片段表現為結實率降低。另1個QTL qSSR1.1與qSSR1.2相鄰,其加性效應為6.13,可解釋表型變異為15.68%,該位點的日本晴替換片段表現為結實率提高。相鄰2個QTLs qSSR1.1與qSSR1.2效應值相反且數值接近,也部分解釋了C15只含有來自日本晴qSSR1.2,結實率表現為嚴重下降(圖1、圖2B、圖2C),而C13、C16 2個系均含有來自日本晴qSSR1.1和qSSR1.2,結實率表現正常。
單株總穎花數qSN2被定位在第2染色體RM7286附近,位置與結實率加性效應負向位點qSSR2位于同一替換片段上,加性效應為正,數值為190.44,可解釋表型變異為7.97%,來自供體親本日本晴的替換片段對單株總穎花數的作用表現為增加,純系增加的幅度理論上可以達到381個;千粒質量qTGW5被定位在第5染色體RM2422附近,加性效應為2.38,可解釋表型變異為8.20%,來自供體親本日本晴的替換片段對千粒質量的作用表現為增大,理論上純合系的千粒質量比輪回親本增大4.76 g。檢測到2個每穗穎花數QTLs qSPP5和qSPP7分別位于第5染色體RM26附近、第7染色體RM427附近,加性效應分別為21.77、28.60,可解釋表型變異分別為8.64%、7.52%,相應日本晴替換片段對該性狀的作用為增效。
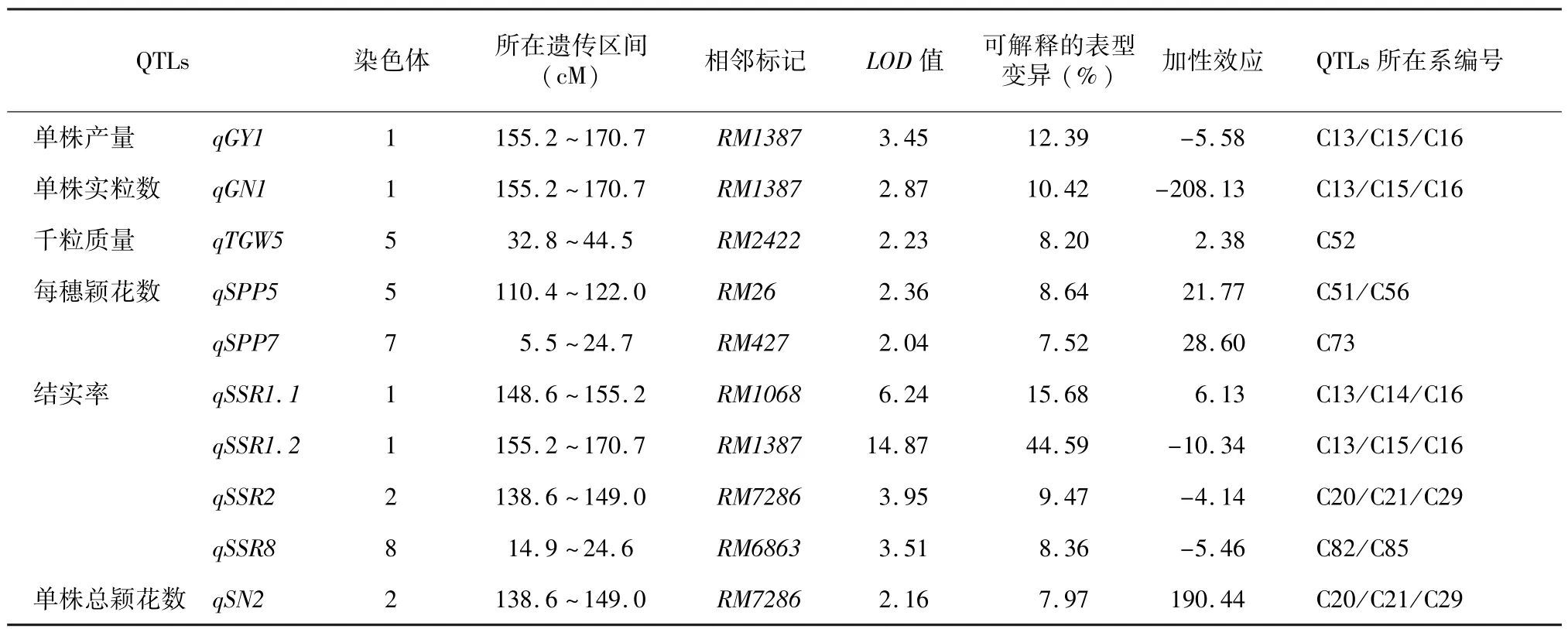
表3 產量相關性狀QTLs定位、遺傳參數估算及QTLs所在系編號Table 3 QTLs of yield-related traits,estimation of genetic coefficients and the lines
3 討論
高產歷來是作物科學研究者最重要的目標之一,水稻產量相關基因的研究也一直是水稻育種和水稻分子生物學的重點研究內容。單位面積稻谷產量是由這一面積范圍內稻株分化的穎花量、結實率和千粒質量3個因素決定的,穎花量又可分解為有效穗數及每穗穎花數。本研究以9311與日本晴構建的119個CSSLs群體為研究對象,初步進行了單株產量及其構成因素QTL分析。與前人的研究結果相比,本研究中單株總穎花數qSN2與從中花11輻射誘變突變體第2染色體RM475標記附近克隆到一個大穗基因LP[9]位于同一個區域范圍內。千粒質量qTGW5與水稻品種Asominori中克隆的gw5[22]位于同一個區域范圍內,與日本晴中克隆的粒寬和粒質量QTL qSW5[21]也位于同一區域,對應9311測序圖譜的位置gw5與qSW5基本是同一位置,但由于gw5在日本晴中是缺失的,且本研究中qTGW5與Shomura等[21]發現的qSW5均來自日本晴,大體可確定qTGW5是qSW5的1個很好的候選基因。每穗穎花數qSPP5與水稻品種日本晴中NADH-谷氨酸合酶基因OsNADH-GOGAT2位于同一區域,OsNADH-GOGAT2基因參與編碼鐵氧化還原蛋白質,日本晴中該基因敲除后引起葉片中氮素含量的下降,同時導致穗粒數的下降[10]。第7染色體每穗穎花數qSPP7與明恢63中克隆的控制抽穗期和穗粒數基因Ghd7或qSPP7[6-7]相比,位置位于其上方,不在同1個位點。第1染色體上有研究者克隆了與穗粒數、穎花數等有關的QTLs,如Gn1a[2]、SPP1[8]等,但本研究中第1染色體中2個結實率QTLs、1個單株產量QTL和1個單株實粒數QTL均未被精細定位或克隆,另外第2染色體和第8染色體中結實率qSSR2、qSSR8也是未被精細定位或克隆的新位點。以上研究結果為新基因的進一步定位和利用奠定了基礎。為了進一步確定這些QTLs的準確性,我們將利用攜帶此QTL的CSSL與9311雜交,通過分析F2的性狀分離比例以及性狀與QTL的連鎖關系進行驗證。

圖3 產量相關性狀QTLs在染色體上的定位Fig.3 Location of QTLs of yield-related traits on chromosomes
通過對CSSLs各系產量及產量構成因素間的分析,不難看出產量相關性狀間有2個相互關系:性狀與構成因素間的正向關系、部分互為某一性狀構成因素的性狀間的負向關系。這與栽培、生理等研究得出的相關結論較為一致:(1)水稻產量與產量構成因素之間的關系有較多的研究,一般認為水稻的超高產應該是在一定穗數的基礎上主要依靠成穗質量(高成穗、高結實和高粒質量),挖掘大穗的潛力來進一步提高產量[37-40]。(2)穎花數過大易導致結實率下降,千粒質量的提高與穎花及實粒數的增加互為矛盾,增穗不一定能增加穎花數、實粒數,也可能影響千粒質量。超高產遺傳研究和超高產栽培技術研究,需要遺傳和育種研究發現和利用有利基因(或基因組合)培育高產品種,栽培和生理研究通過后天的措施實現產量構成因素之間的協調發展,最終實現進一步的高產。
本研究中發現低值親本日本晴的產量相關性狀的表型遠不及另一親本9311,但其后代中出現了一些系,其產量性狀有所改善,少數系甚至優于9311。這可能對育種利用有所啟發:性狀較優的材料與該性狀表現較差的材料雜交的后代中可能挑選到更好的材料;育種中應該重視不同資源的利用,特別是差異較大的材料,比如秈稻與粳稻之間的基因交流等。在綜合考慮其他性狀如米質、育性恢復性、綜合農藝性狀等基礎上,可以對一些產量及產量構成因素等性狀優于9311的系進行直接育種利用,比如作為常規秈稻和作為雜交秈稻恢復系配置組合等。CSSLs群體中有部分系的產量相關性狀較輪回親本9311有所改善,但定位的增效位點卻很少,可能與原背景檢測分子標記密度有限,未能發現系中其他更小片段的存在有關,期望通過相關系與輪回親本構建F2分離群體,對產量相關性狀QTLs的進一步定位,同時將定位中加密標記后獲得的雜交純合后代補充到CSSLs群體中,使CSSLs進一步完善。
[1] 顧銘洪.水稻高產育種中一些問題的討論[J].作物學報, 2010,36(9):431-439.
[2] ASHIKARI M,SAKAKIBARA H,LIN S Y,et al.Cytokinin oxidase regulates rice grain production[J].Science,2005,309 (741):741-745.
[3] DESHMUKH R,SINGH A,JAIN N,et al.Identification of candidate genes for grain number in rice(Oryza sativaL.)[J]. Functional&Integrative Genomics,2010,10(3):339-347.
[4] HUANG X Z,QIAN Q,LIU Z B,et al.Natural variation at theDEP1locus enhances grain yield in rice[J].Nature Genetics, 2009,41(4):494-497.
[5] QIAO Y L,PIAO R H,SHI J X,et al.Fine mapping and candidate gene analysis ofdense and erect panicle 3,DEP3,which confers high grain yield in rice(Oryza sativaL.)[J].Theor Appl Genet,2011,122(7):1439-1449.
[6] XUE W Y,XING Y Z,WENG X Y,et al.Natural variation inGhd7is an important regulator of heading date and yield potential in rice[J].Nature Genetics,2008,40(6):761-767.
[7] XING Y Z,TANG W J,XUE W Y,et al.Fine mapping of a major quantitative trait loci,qSSP7,controlling the number of spikelets per panicle as a single Mendelian factor in rice[J].Theor Appl Genet,2008,116(6):789-796.
[8] LIU T M,MAO D H,ZHANG S P,et al.Fine mappingSPP1,a QTL controlling the number of spikelets per panicle,to a BAC clone in rice(Oryza sativa)[J].Theor Appl Genet,2009,118 (8):1509-1517.
[9] LI M,TANG D,WANG K J,et al.Mutations in the F-box gene larger panicle improve the panicle architecture and enhance the grain yield in rice[J].Plant Biotechnology Journal,2011,9(9): 1002-1013.
[10]TAMURA W,KOJIMA S,TOYOKAWA A,et al.Disruption of a novel NADH-glutamate synthase 2 gene caused marked reduction in spikelet number of rice[J].Frontiers in Plant Nutrition,2011, 2:57.
[11]GUO S Y,XU Y Y,LIU H H,et al.The interaction between Os-MADS57 and OsTB1 modulates rice tillering via DWARF14[J]. Nature Communications,2013,4(3):1566.
[12]MINAKUCHI K,KAMEOKA H,YASUNO N,et al.FINE CULM1(FC1)works downstream of strigolactones to inhibit the outgrowth of axillary buds in rice[J].Plant and Cell Physiology, 2010,51(7):1127-1135.
[13]LIU W Z,WU C,FU Y P,et al.Identification and characterization ofHTD2:a novel gene negatively regulating tiller bud outgrowth in rice[J].Planta,2009,230(4):649-658.
[14]GAO Z Y,QIAN Q,LIU X H,et al.Dwarf 88,a novel putative esterase gene affecting architecture of rice plant[J].Plant Molecular Biology,2009,71(3):265-276.
[15]ARITE T,UMEHARA M,ISHIKAWA S,et al.d14,a Strigolactone-Insensitive mutant of rice,shows an accelerated outgrowth of tillers[J].Plant and Cell Physiology,2009,50(8):1416-1424. [16]LI S Y,ZHAO B R,YUAN D Y,et al.Rice zinc finger protein DST enhances grain production through controllingGn1a/OsCKX2expression[J].Proceedings of the National Academy of Sciences, 2013,110(8):3167-3172.
[17]JIAO Y Q,WANG Y H,XUE D W,et al.Regulation ofOsSPL14by OsmiR156 defines ideal plant architecture in rice[J]. Nature Genetics,2010,42(6):541-544.
[18]EOM J S,CHO J I,REINDERS A,et al.Impaired function of the tonoplast-localized sucrose transporter in rice,OsSUT2,limits the transport of vacuolar reserve sucrose and affects plant growth [J].Plant Physiology,2011,157(1):109-119.
[19]KIM S R,YANG J I,MOON S,et al.RiceOGR1encodes a pentatricopeptide repeat-DYW protein and is essential for RNA editing in mitochondria[J].The Plant Journal,2009,59(5):738-749. [20]SONG X J,HUANG W,SHI M,et al.A QTL for rice grain width and weight encodes a previously unknown RING-type E3 ubiquitin ligase[J].Nature Genetics,2007,39:623-630.
[21]SHOMURA A,IZAWA T,EBANA K,et al.Deletion in a gene associated with grain size increased yields during rice domestication [J].Nature Genetics,2008,40(8):1023-1028.
[22]WENG J F,GU S H,WAN X Y,et al.Isolation and initial characterization ofGW5,a major QTL associated with rice grain width and weight[J].Cell Research,2008,18(12):1199-1209.
[23]HEANG D,SASSA H.Antagonistic actions of HLH/bHLH proteins are involved in grain length and weight in rice[J].PLoS ONE,2012,7(2):e31325.
[24]ZHANG X J,WANG J F,HUANG J,et al.Rare allele ofOsPPKL1associated with grain length causes extra-large grain and a sig-nificant yield increase in rice[J].Proceedings of the National A-cademy of Sciences,2012,109(52):21534-21539.
[25]MAO H L,SUN S Y,YAO J L,et al.Linking differential domain functions of the GS3 protein to natural variation of grain size in rice [J].Proceedings of the National Academy of Sciences,2010, 107(45):19579-19584.
[26]LI J M,THOMSON M,MCCOUCH S R.Fine mapping of a grainweight quantitative trait locus in the pericentromeric region of rice chromosome 3[J].Genetics,2004,168(4):2187-2195.
[27]ISHIMARU K,HIROTSU N,MADOKA Y,et al.Loss of function of the IAA-glucose hydrolase geneTGW6enhances rice grain weight and increases yield[J].Nature Genetics,2013,45(6): 707-711.
[28]LI J,CHU H W,ZHANG Y H,et al.The riceHGWgene encodes a ubiquitin-associated(UBA)domain protein that regulates heading date and grain weight[J].PLoS ONE,2012,7(3): e34231.
[29]LI Y B,FAN C C,XING Y Z,et al.Natural variation inGS5plays an important role in regulating grain size and yield in rice [J].Nature Genetics,2011,43(12):1266-1269.
[30]THANGASAMY S,GUO C L,CHUANG M H,et al.RiceSIZ1, a SUMO E3 ligase,controls spikelet fertility through regulation of anther dehiscence[J].New Phytologist,2011,189(3):869-882.
[31]JIA H,REN H,GU M,et al.The phosphate transporter geneOs-Pht1;8is involved in phosphate homeostasis in rice[J].Plant Physiology,2011,156(3):1164-1175
[32]ZHU W Y,LIN J,YANG D W,et al.Development of chromosome segment substitution lines derived from backcross between two sequenced rice cultivars,Indica recipient 93-11 and japonica donar Nipponbare[J].Plant Mol Bio Rep,2009,27(2):126-131.
[33]周麗慧,趙春芳,趙 凌,等.利用染色體片段置換系群體檢測水稻葉片形態QTL[J].中國水稻科學,2013,27(1):26-34.
[34]JIANG G H,XU C G,LI X H,et al.Characterization of genetic basis for the yield and its component traits of rice using doubled haploid population[J].Acta Genet Sinica,2004,31(1):63-72. [35]WANG J K,WAN X,LI H,et al.Application of identified QTL-marker associations in rice quality improvement through a designbreeding approach[J].Theor Appl Genet,2007,115:87-100.
[36]MCCOUCH S R,CHO Y G,YANO M,et al.Report on QTL nomenclature[J].Rice Genet Newsl,1997,14:11-13.
[37]凌啟鴻,張洪程,蔡建中,等.水稻高產群體質量及其優化控制探討[J].中國農業科學,1993,26(6):1-11.
[38]凌啟鴻.水稻群體質量理論與實踐[M].北京:中國農業出版社,1995:108-220.
[39]蔣彭炎,馮來定,洪曉富.水稻三高一穩栽培法論叢[M].北京:中國農業科技出版社,1993:1-3.
[40]黃仲青,李奕松,蔣之塤.關于水稻“四少四高”栽培模式的探討[C]//高佩文,談 松.水稻高產理論與實踐.北京:中國農業出版社,1994:127-130.
(責任編輯:袁 偉)
Quantitative trait locus(QTL)detection for rice yield-related traits using chromosome segment substitution lines
ZHOU Li-hui, ZHANG Ya-dong, ZHU Zhen, CHEN Tao, ZHAO Qing-yong, YAO Shu, ZHAO Ling, ZHAO Chun-fang, YU Xin, WANG Cai-lin
(Institute of Food Crops,Jiangsu Academy of Agricultural Sciences/Jiangsu High Quality Rice Research and Development Center/Nanjing Branch of Chinese National Center for Rice Improvement,Nanjing 210014,China)
To identify the quantitative trait locus (QTL)referringtoyieldanditscomponentsfor improvement of yield,a population of chromosome segment substitution lines(CSSLs)derived from backcross betweenindicarecipient 9311 andjaponicadonor Nipponbare were employed,and the yield-related traits,such as grain yield per plant(GY),grain number per plant(GN),spikeletnumber per plant(SN),seed setting rate(SSR),grain number per panicle(GPP),effective panicles number(EPN), spikelet number per panicle(SPP),and 1 000-grain weight(TGW)were measured.A total of ten QTLs for the yield-related traits except for EPN and GPP were located at seven regions on chromosomes 1,2,5,7 and 8,with explained phenotypic variations(EPV)ranging from 7.52%to 44.59%,four of which with EPV above 10.00%.qGY1,qGN1,qSSR1.2,qSSR2andqSSR8derived from 9311 allele showed negative effect,while the others,includingqSN2,qSSR1.1,qTGW5,qSPP5andqSPP7derived from Nipponbare allele,exhibited positive effect.Three QTLs,qSN2,qTGW5,qSPP5were located on the same regions as the reported cloned lociLP,qSW5,OsNADH-GOGAT2,respectively,and the rest seven QTLs were not cloned or fine mapped.
rice;chromosome segment substituted line(CSSL);quantitative trait locus(QTL);yield
S511.03
A
1000-4440(2015)01-0001-09
周麗慧,張亞東,朱 鎮,等.利用染色體片段置換系群體檢測水稻產量相關性狀QTL[J].江蘇農業學報,2015,31(1):1-9.
10.3969/j.issn.1000-4440.2015.01.001
2014-07-28
國家水稻產業技術體系項目(CARS-01-47);江蘇省農業科技自主創新基金項目[CX(11)1022]
周麗慧(1981-),女,湖南沅江人,碩士,助理研究員,主要從事水稻遺傳育種研究。(Tel) 13645176538;(E-mail) zhoulihui@jaas.ac.cn
王才林,(E-mail)clwang@jaas.ac.cn

