植物iPGAM的研究進展
謝鑫 劉娜 魏鳳菊
(河北農業大學生命科學學院 河北省植物生理與分子病理學重點實驗室,保定 071001)
植物iPGAM的研究進展
謝鑫 劉娜 魏鳳菊
(河北農業大學生命科學學院河北省植物生理與分子病理學重點實驗室,保定071001)
輔因子非依賴型磷酸甘油酸變位酶(Cofactor-independent phosphoglycerate mutase,iPGAM)是在真菌和高等植物中發現的金屬酶,是糖酵解和糖異生過程中的一個關鍵酶。隨著原核生物、無脊椎動物等物種中iPGAM基因被克隆、蛋白晶體結構的解析及其生物學功能的陸續報道,近些年,植物iPGAM的功能開始受到關注。闡述了植物iPGAM蛋白的結構特點及作用機制,著重對已報道的擬南芥、水稻和玉米中iPGAMs的系統進化關系以及生物學功能進行了概述。
植物;輔因子非依賴型;磷酸甘油酸變位酶
糖類是生物能量的主要來源,糖代謝是生物新陳代謝物質代謝中的核心問題之一,它的合成與分解代謝關系到植物的形態建成、逆境脅迫應答等生命過程。磷酸甘油酸變位酶(Phosphoglycerate mutase,PGAM)是糖酵解和糖異生過程中的關鍵酶之一,它能夠催化3-磷酸甘油酸(3-phosphoglycerate,3-PGA) 和2-磷 酸 甘 油 酸(2-phosphoglycerate,2-PGA)之間的相互轉化。1982年Fothergill等[1]首先在酵母中發現了PGAM,隨著其氨基酸序列和晶體結構被揭示,人們在多種生物體中均發現了該蛋白,并根據其在催化反應中與輔因子2,3-二磷酸甘油酸(2,3-bisphosphoglycerate,2,3-BPG)的依賴關系,分為兩種類型:輔因子依賴型PGAM(Cofactor-dependent PGAM,dPGAM)和輔因子非依賴型PGAM(Cofactor-independent PGAM,iPGAM)。前一種酶存在于所有的脊椎動物、大部分無脊椎動物、某些真菌和細菌,尤其是革蘭氏陰性菌中,其在酶催化反應時需要2,3-BPG作為輔因子;后一種酶存在于所有植物、一些無脊椎動物、某些真菌和細菌,尤其是革蘭氏陽性菌中[2],其在酶催化反應時不需要2,3-BPG作為輔因子[3]。根據其氨基酸的序列分析顯示,dPGAM屬于酸性磷酸酶家族,而iPGAMs屬于堿性磷酸酶超家族。盡管iPGAMs屬于堿性磷酸酶超家族,但它和堿性磷酸酶(Alkaline phosphatase,AP)的活性有著實質性的差異。堿性磷酸酶在蛋白保守區AP上包含有典型的磷酸轉移殘基,具有磷酸轉移活性;iPGAM在蛋白保守區具有磷酸酶的活性殘基,具有磷酸酶活性[4-6]。
在過去的幾十年中,人們對于動物、真菌、細菌中的dPGAMs研究較多,尤其對哺乳動物和人類dPGAMs的蛋白結構、生化機理及其功能特性等已有比較深入的了解[7];與dPGAM所不同的是,植物中廣泛分布的iPGAMs,而在脊椎動物中沒有發現。自從第一次從玉米中分離純化到PGAM-i后[8],人們開始關注植物中的iPGAMs。由于植物來源的iPGAMs不穩定性導致其分離純化困難,對其研究報道亦相對較少,目前對其功能研究尚處于初級階段。結合近年對植物來源的iPGAMs的相關報道,對其結構特點、作用機制和生物學功能(重點介紹了擬南芥、玉米及水稻)進行闡述。
1 iPGAM蛋白的生化特性
1.1 結構特征
蛋白晶體結構的分析結果顯示iPGAM具有典型的保守域。首個報道的iPGAM結構是嗜熱脂肪芽孢桿菌(Bacillus stearothermophilus)中的[3],它含有兩個顯著分離的結構域,分別具有磷酸酶和磷酸轉移酶的活性。隨后,在細菌、真菌及動物中都陸續有iPGAM晶體結構解析,并證實了結構域的典型特征,但在植物來源的iPGAM中,其成員的晶體結構未見研究報道。植物iPGAMs相對于dPGAM,分子稍大,一般由約500個氨基酸殘基組成,以60 kD的單體形式存在,其一級結構較為保守。在iPGAMs的1-76位和311-511位的氨基酸殘基形成磷酸酶的活性中心,該結構與堿性磷酸酶和硫酯酶結構相近,包含多個活性位點和兩個金屬離子結合位點(Pfam domain PF01676)[9],其中的金屬離子結合位點是區別于dPGAM的特有的結構域[10],且和堿性磷酸酶的金屬離子結合位點序列一致性很低。102-332位是屬于2,3-BPG不依賴區域(Pfam domain,PF0-6415),包含一個保守的磷酸化位點絲氨酸殘基[9](圖1)。77-310位氨基酸殘基區域形成磷酸轉移酶區,但與細菌iPGAMs蛋白一級結構中氨基酸序列存在差異,這也體現了生物進化過程中蛋白分子的進化過程。
1.2 活性調節
磷酸甘油酸變位酶iPGAMs的活性受金屬離子的影響。較早的研究結果顯示,細菌中的iPGAMs是金屬離子依賴的酶,可以結合Mg2+[11]、Mn2+[12],Ni2+或Co2+等二價陽離子[13],且每個酶分子可以結合兩個金屬離子[14]。而植物中的iPGAMs是否需要金屬離子來保持活性,這在當時一直存在爭議。起初報道的小麥胚芽中的iPGAM受EGTA抑制[15,16],其活性的恢復需要Mn2+和Co2+[17]。與這一結果相反,蓖麻種子中的iPGAM活性不受金屬離子影響[18]。迄今為止,還未有報道在反應體系中把金屬離子完全去除來檢測酶活性。鑒于植物和細菌中iPGAM序列的高度相似性,推測植物中的iPGAMs也屬于金屬離子依賴的酶[11]。現今的研究報道中,直接稱植物中的iPGAMs為金屬酶。
1.3 催化機理
iPGAM催化反應不需要2,3-BPG作為輔因子,其酶的活性位點是絲氨酸。底物直接與絲氨酸結合并使該位點磷酸化形成酶-底物中間體,然后通過這個磷酸絲氨酸中間體來催化磷酸基團在磷酸甘油酸分子內的轉移[19]。其中2-磷酸甘油酸第2個碳所結合的磷酸根來自變位酶iPGAM,而后第3個碳的磷酸根再結合到iPGAM上。可見,磷酸基團并非從3號碳原子直接轉移到2號碳原子上(圖2)。
2 iPGAM s家族
2.1 家族成員
在高等植物的研究報道中,對擬南芥的iPGAMs研究較多。早在2003年,TAIR(http://www.arabid opsis.org/)網站將At1g09780、At1g22170和At3g08-590定名為PGAMs,KEGG(http://www.genome.jp/ kegg/)網站公布了4個PGAMs:At1g09780、At3g-50520、At3g08590和At5g04120。同年,Mazarei等[20]研究發現擬南芥中存在5個PGAMs:At1g58280、At2g17280、At3g50520、At5g04120和At5g64460[21],較早報道的這些成員中既包含iPGAM也包含dPGAM。后續的研究中,擬南芥PGAM家族成員逐漸增多,同時也將發現的蛋白進行了細致的鑒別[10,22]。截至目前,文獻信息報道及網站信息顯示擬南芥中的iPGAMs成員共計21個,其中兩個被正式命名為iPGAM1(At1g09780)和iPGAM2(At3g08590),并證實二者具有磷酸甘油酸變位酶活性;另外,人們推測還有兩個成員At4g09520和At3g30841具有磷酸甘油酸變位酶活性[23]。其余一些成員是否具有磷酸甘油酸變位酶的活性,尚待進一步檢測驗證。玉米和水稻的iPGAMs家族成員報道相對較少。玉米中的一些家族成員已經被克隆,第一個報道的玉米iPGAM成員是PGAM-i,通過篩選cDNA表達文庫獲得了該成員的cDNA序列,經序列比對發現PGAM-i與小麥胚芽、蘑菇、蜘蛛中的iPGAMs成員序列相似性很低[24]。兩年之后,又成功克隆了玉米PGAM-i的基因組序列[25]。目前通過NCBI(http://www.ncbi.nlm.nih.gov/)信息查詢,注釋為玉米iPGAM的蛋白共9個。而Gramene(http://ensembl.gramene.org/genome_browser/index. htm l)信息顯示,編碼玉米iPGAM的基因存在多個轉錄本,同時發現多個基因可能轉錄生成一種蛋白產物。由于真核生物中基因發生可變剪切的比例較高,至于玉米的iPGAMs基因在何種情況下轉錄翻譯成有功能的iPGAMs蛋白,我們不得而知。除去NCBI中注釋的9個蛋白成員,與AtiPGAM1蛋白序列相似性較高的成員(>70%)有7個,關于這些成員的深入研究還未見報道。水稻數據庫中標注為iPGAM的蛋白共3個,這些蛋白的功能還未知,活性也未曾被驗證。與AtiPGAM1、AtiPGAM2蛋白序列相似性較高(>70%)的成員共6個,經結構域分析,這些蛋白都具有BPG-非依賴的結構域,推測這些蛋白成員可能屬于水稻的iPGAM家族。

圖1 iPGAM s中的2,3-BPGA不依賴的區域(Pfam domain PF06415)

圖2 iPGAM催化反應機理
其他植物如巴旦杏[9]、煙草、蓖麻[26]及橡膠樹[27]中都克隆了iPGAMs的cDNA序列,在草菇[28]、歐洲油菜[29]、大青楊[30]等植物中也發現了iPGAMs。
2.2 iPGAMs家族的進化
蛋白質演變是生物進化的體現之一,其中iPGAMs家族在真核生物中衍化出很多成員,或許各個成員之間功能已有了精細差異。Stechmann等[31]對真核生物、真菌和古細菌的iPGAMs蛋白序列中的394個氨基酸位點進行比對獲得了系統進化樹,結果表明iPGAMs與很多生物的側向基團轉移有關。為了分析不同植物物種中iPGAMs家族成員的進化關系,將已知的水稻、玉米、擬南芥的iPGAMs成員與最早報道的嗜熱脂肪芽孢桿菌中的iPGAMs一起進行系統發育樹構建,結果印證了上述觀點,同種來源的iPGAMs未必包括在同一個亞群中,如擬南芥的At3g30841(NP_850651)和At4g09520(NP_ 192690)與細菌的蛋白序列相似性較高,而已報道的在保衛細胞中發揮功能的At3g08590(NP_187471)和At1g09780(NP_563852)與細菌的序列相似性較低,它們分別位于不同的亞群。玉米和水稻的iPGAMs蛋白表現出與擬南芥已知功能iPGAMs:At3g08590(NP_187471)和At1g09780(NP_563852)的序列相似性較高,位于一個亞群(圖3),推測這些成員的功能可能較為相近。
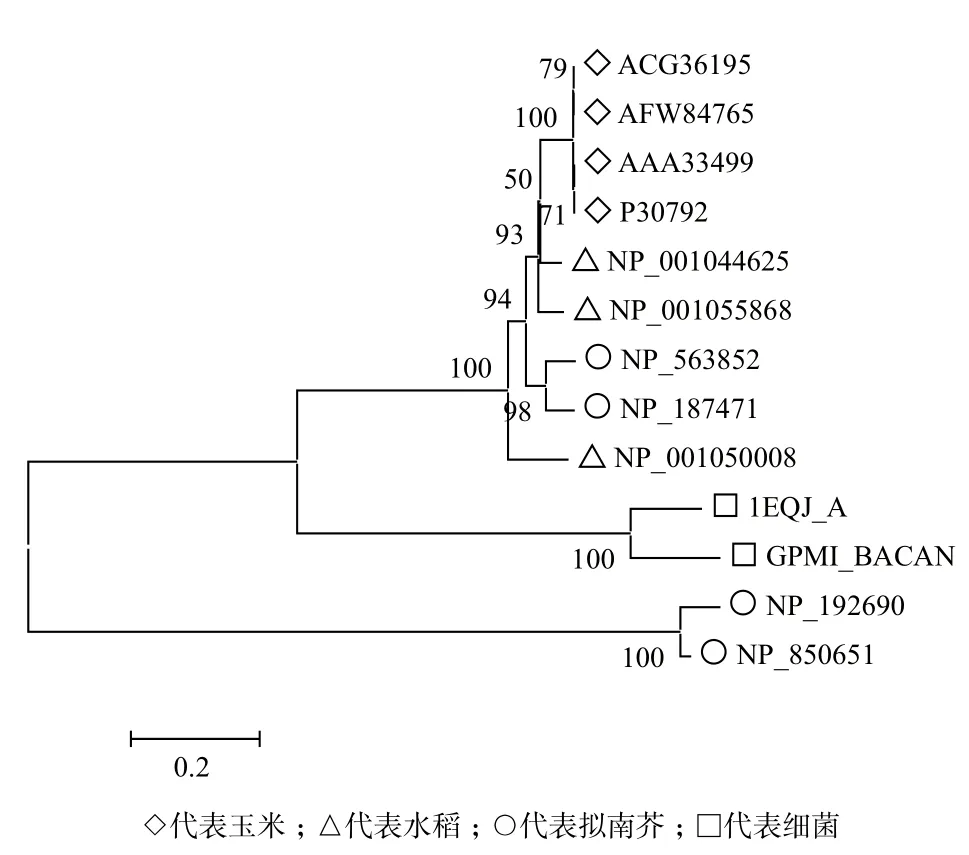
圖3 不同物種iPGAM s系統進化樹
3 iPGAM s的定位
iPGAMs分布于多種器官組織。在正常情況下,擬南芥AtPGAM特異性分布于莖尖和根尖分生組織;囊包線蟲侵染后,在入侵部位的合胞體中磷酸甘油酸變位酶活性顯著增強,并且根部組織也有明顯AtPGAM表達活性。另外,分布在分生組織的AtPGM受多種刺激調控,表明AtPGM與分生組織功能及細胞周期活動有關[20]。免疫金標結果顯示麝香百合中的iPGAM(與玉米中的iPGAM同源性較高)在根尖和花柱傳遞細胞中表達量很高,推測這可能與這部分細胞的代謝活動旺盛相關[32]。
研究發現iPGAMs定位于多種細胞器。糖酵解的發生部位是細胞質,因此通常認為參與糖酵解途徑的酶定位于細胞質,而檢測結果與這一觀點存在差異。通過對擬南芥線粒體蛋白質組學分析發現,糖酵解途徑中的10個酶中有7個也在線粒體中存在,AtiPGAM1(At1g09780)便是其中之一,該酶定位于線粒體外膜和膜間隙[33]。糖酵解代謝的產物——丙酮酸的重要去路就是進入線粒體基質,參與三羧酸循環為機體產生能量。糖酵解過程中的酶定位在線粒體,推測這將有助于丙酮酸用于線粒體中的進一步代謝。除線粒體之外,也有定位于其他細胞器的iPGAMs,Wang等[32]通過免疫金標技術觀察到麝香百合iPGAMs在根的頂端分生組織中定位于細胞質、質體及細胞核中。iPGAM定位于多個細胞器預示著iPGAMs可能在細胞中發揮多種功能。
4 iPGAM s的生物學功能
植物iPGAMs參與多種生長發育過程,如氣孔運動、花粉形成及營養生長等。在擬南芥氣孔中表達的兩個基因iPGAM1(At1g09780)和iPGAM2(At3g08590)[34],具有90%的蛋白序列相似性,其雙突變體在黑暗條件下低濃度CO2誘導的氣孔開放和藍光促使的氣孔開放過程中表現出極為敏感的表型,這一實驗結果首次提供了遺傳學方面的實驗證據,表明糖酵解過程中的酶(iPGAMs)在氣孔響應環境信號過程中發揮重要作用。另外,兩個單突變體在酶活性上沒有差異,表型與野生型也沒有差別。而雙突變體沒有檢測到iPGAM的酶活性,其營養生長和生殖生長都受到嚴重影響,表明糖酵解代謝過程中的兩個酶iPGAM1和iPGAM2是能量代謝或者代謝物提供途徑的重要組分[23],且兩個成員之間存在著功能冗余。擬南芥非磷酸化的3-磷酸甘油醛脫氫酶(NP-GAPDH)在植物生長和發育過程中發揮重要作用,其缺失后表現為第三、四個花蕾不能自花授粉,經檢測編號為At3g30841的iPGAM基因表達量明顯下降,預示著糖酵解過程受到了抑制[35],使開花受精所需的能量供應不足,表明At3g30841基因在花粉授粉過程中的能量代謝方面發揮著重要功能。
在非生物脅迫響應過程中iPGAMs發揮著重要功能。如擬南芥中的AtiPGAM1(At1g09780),不僅在生長發育過程中起作用,而且參與了響應非生物脅迫的過程。在擬南芥受低溫脅迫時,AtiPGAM1表達量明顯增高[36],預示AtiPGAM1在植物響應冷脅迫過程中起正調節因子的作用。但也有一些iPGAM起負調節作用,如耐冷性較強的水稻赤霉素不敏感突變體gid1(Gibberellin-insensitive dwarf1),其懸浮細胞中iPGAM(Accession No. BAB64833)的表達量降低[37];在水稻花藥三核期響應冷脅迫時iPGAM呈現部分降解[37,38],暗示水稻的iPGAM(Accession No. BAB64833)是水稻響應冷脅迫的負調節因子。
生物脅迫過程中也有iPGAM成員的參與。在卷心菜曲葉病毒(Cabbage leaf curl virus,CaLCuV)侵染擬南芥12 d時,上位葉片表現出小、狹窄和卷曲等癥狀;芯片結果顯示iPGAM成員At5g64460表達量下降[21],推測這一基因可能在病毒侵染過程參與了葉片的形態建成。
5 展望
從最初對iPGAMs的酶活性檢測,到現階段對家族成員的挖掘以及功能的分析驗證,經過幾十年的不斷研究,人們對于植物中的iPGAMs有了一定的認識,但仍有很多問題需要去探究。植物中各物種iPGAM家族成員鑒定還不完全;與已知iPGAM成員氨基酸序列相似性較高的成員是否屬于iPGAM家族;對于已報道的多數擬南芥的家族成員,還不清楚它們是否具有磷酸甘油酸變位酶活性;如果沒有變位酶的活性,是否作為另外一種酶發揮其功能;至今對于植物中磷酸甘油酸變位酶的功能知之甚少,作為催化基礎代謝過程中不可或缺的一類酶,iPGAMs參與了植物生命中的哪些生理過程,以及iPGAMs參與生理過程的分子機制是什么。隨著各種生物技術的日臻成熟與完善,后續研究中更多植物的iPGAMs將會被關注,相信以上科學問題會被科研工作者逐一揭示。
[1] Fothergill LA, Harkins RN. The amino acid sequence of yeast phosphoglycerate mutase[J]. Proc R Soc Lond B Biol Sci, 1982,215(1198):19-44.
[2] Jedrzejas MJ. Structure, function, and evolution of phosphoglycerate mutases:comparison with fructose-2, 6-bisphosphatase, acid phosphatase, and alkaline phosphatase[J]. Prog Biophys Mol Biol, 2000, 73(2-4):263-287.
[3] Fothergill-Gilmore LA, Watson HC. The phosphoglycerate mutases[J]. Adv Enzymol Relat Areas Mol Biol, 1989, 62:227-313.
[4] Breathnach R, Knowles JR. Phosphoglycerate mutase from wheat germ:studies with18O-labeled substrate, investigations of thephosphatase and phosphoryl transfer activities, and evidence for aphosphoryl-enzyme intermediate[J]. Biochemistry, 1977, 16(14):3054-3060.
[5] Bond CS, Clements PR, Ashby SJ, et al. Structure of a human lysosomal sulfatase[J]. Structure, 1997, 5(2):277-289.
[6] Lukatela G, Krauss N, Theis K, et al. Crystal structure of human arylsulfatase A:the aldehyde function and the metal ion at the active site suggest a novel mechanism for su lfate ester hydrolysis[J]. Biochemistry, 1998, 37(11):3654-3664.
[7] Wang Y, Wei Z, Bian Q, et al. Crystal structure of human bisphosphoglycerate mutase[J]. J Biol Chem, 2004, 279(37):39132-39138.
[8] Gra?a X, Ure?a J, Ludevid D, et al. Purification, characterization and immunological properties of 2, 3-bisphosphoglycerate-independent phosphoglycerate mutase from maize(Zea mays)seeds[J]. Eur J Biochem, 1989, 186(1-2):149-153.
[9] Gra?a X, Pérez de la Ossa P, Broce?o C, et al. 2, 3-Bisphosphoglycerate-independent phosphoglycerate mutase is conserved among different phylogenic kingdoms[J]. Comp Biochem Physiol B Biochem Mol Biol, 1995, 112(2):287-293.
[10] Andriotis VM, Kruger NJ, Pike MJ, et al. Plastidial glycolysis in developing Arabidopsis embryos[J]. New Phytol, 2010, 185(3):649-662.
[11] Galperin MY, Bairoch A, Koonin EV. A superfam ily of metalloenzymes unifies phosphopentomutase and cofactorindependent phosphoglycerate mutase with alkaline phosphatases and sulfatases[J]. Protein Sci, 1998, 7(8):1829-1835.
[12] Singh RP, Setlow P. Purification and properties of phosphoglycerate phosphomutase from spores and cells of Bacillus megaterium[J]. J Bacteriol, 1979, 137(2):1024-1027.
[13] Hammer-Jespersen K, Munch-Petersen A. Phosphodeoxyribomutase from Escherichia coli. purification and some properties[J]. Eur J Biochem, 1970, 17(3):397-407.
[14] Kuhn NJ, Setlow B, Setlow P, et al. Cooperative manganese(II)activation of 3-phosphoglycerate mutase of Bacillus megaterium:a biological pH-sensing mechanism in bacterial spore formation and germination[J]. Arch Biochem Biophys, 1995, 320(1):35-42.
[15] Leadlay PF, Breathnach R, Gatehouse JA, et al. Phosphoglycerate mutase from wheat germ:studies with isotopically labeled 3-phospho-D-glycerates showing that the catalyzed reaction is intramolecular. Appendix:phosphoglycerate mutase from wheat germ:isolation, crystallization, and properties[J]. Biochemistry,1977, 16(14):3045-3053.
[16] Smith GC, Hass LF. Wheat germ phosphoglycerate mutase:purification, polymorphism, and inhibition[J]. Biochem Biophys Res Commun, 1985, 131(2):743-749.
[17] Smith GC, McWilliams AD, Hass LF. Wheat germ phosphoglycerate mutase:evidence for a metalloenzyme[J]. Biochem Biophys Res Commun, 1986, 136(1):336-340.
[18] Botha FC, Dennis DT. Isozymes of phosphoglyceromutase from the developing endosperm of Ricinus communis:isolation and kinetic properties[J]. Arch Biochem Biophys, 1986, 245(1):96-103.
[19] Jedrzejas MJ, Chander M, Setlow P, et al. Mechanism of catalysis of the cofactor-independent phosphoglycerate mutase from Bacillus stearothermophilus. Crystal structure of the complex with 2-phosphoglycerate[J]. J Biol Chem, 2000, 275(30):23146-23153.
[20] Mazarei M, Lennon KA, Puthoff DP, et al. Expression of an Arabidopsis phosphoglycerate mutase homologue is localized to apical meristems, regulated by hormones, and induced by sedentary plant-parasitic nematodes[J]. Plant Mol Biol, 2003, 53(4):513-530.
[21] Ascencio-Ibá?ez JT, Sozzani R, Lee TJ, et al. Global analysis of Arabidopsis gene expression uncovers a complex array of changes impacting pathogen response and cell cycle during geminivirus infection[J]. Plant Physiol, 2008, 148(1):436-454.
[22] Stein M, Gabdoulline RR, Wade RC. Cross-species analysis of the glycolytic pathway by comparison of molecular interaction fields[J]. Mol Biosyst, 2010, 6(1):152-164.
[23] Zhao Z, Assmann SM. The glycolytic enzyme, phosphoglycerate mutase, has critical roles in stomatal movement, vegetative growth,and pollen production in Arabidopsis thaliana[J]. J Exp Bot,2011, 62(14):5179-5189.
[24] Gra?a X, de Lecea L, el-Maghrabi MR, et al. Cloning and sequencing of a cDNA encoding 2, 3-bisphosphoglycerateindependent phosphoglycerate mutase from maize. Possible relationship to the alkaline phosphatase family[J]. J Biol Chem,1992, 267(18):12797-12803.
[25] Pérez de la Ossa P, Grana X, Ruiz-Lozano P, et al. Isolation and characterization of cofactor-independent phosphoglycerate mutasegene from maize[J]. Biochem Biophys Res Commun, 1994, 203(2):1204-1209.
[26] Huang Y, Blakeley SD, McAleese SM, et al. Higher-plant cofactorindependent phosphoglyceromutase:purification, molecular characterization and expression[J]. Plant Mol Biol, 1993, 23(5):1039-1053.
[27] 龍翔宇, 董緒濃, 方永軍, 等. 橡膠樹PGAM基因的克隆及表達特性分析[J]. 熱帶作物學報, 2013, 34(10):1895-1901.
[28] 劉朋虎, 鄧優錦, 江玉姬, 等. 草菇PGAM基因克隆、結構及其在同核、異核菌株中的表達量分析[J]. 福建農業學報,2012, 27(3):252-256.
[29] Hajduch M, Casteel JE, Hurrelmeyer KE, et al. Proteomic analysis of seed filling in Brassica napus. Developmental characterization of metabolic isozymes using high-resolution two-dimensional gel electrophoresis[J]. Plant Physiol, 2006, 141(1):32-46.
[30] 李春明, 白卉, 于文喜. 低溫馴化過程中大青楊葉片差異蛋白質分析[J]. 東北林業大學學報, 2011, 39(10):45-49.
[31] Stechmann A, Baumgartner M, Silberman JD, et al. The glycolytic pathway of trimastix pyriformis is an evolutionary mosaic[J]. BMC Evol Biol, 2006, 6(1):101.
[32] Wang JL, Walling LL, Jauh GY, et al. Lily cofactor-independent phosphoglycerate mutase:purification, partial sequencing, and immunolocalization[J]. Planta, 1996, 200(3):343-352.
[33] Giegé P, Heazlewood JL, Roessner-Tunali U, et al. Enzymes of glycolysis are functionally associated with the mitochondrion in Arabidopsis cells[J]. Plant Cell, 2003, 15(9):2140-2151.
[34] Zhao Z, Zhang W, Stanley BA, et al. Functional proteomics of Arabidopsis thaliana guard cells uncovers new stomatal signaling pathways[J]. Plant Cell, 2008, 20(12):3210-3226.
[35] Rius SP, Casati P, Iglesias AA, et al. Characterization of an Arabidopsis thaliana mutant lacking a cytosolic non-phosphorylating glyceraldehyde-3-phosphate dehydrogenase[J]. Plant Mol Biol,2006, 61(6):945-957.
[36] Amme S, Matros A, Schlesier B, et al. Proteome analysis of cold stress response in Arabidopsis tha liana using DIGE-technology[J]. J Exp Bot, 2006, 57(7):1537-1546.
[37] Tanaka N, Matsuoka M, Kitano H, et al. gid1, a gibberellininsensitive dwarf mutant, shows altered regulation of probenazoleinducible protein(PBZ1)in response to cold stress and pathogen attack[J]. Plant Cell Environ, 2006, 29(4):619-631.
[38] Imin N, Kerim T, Rolfe BG, et al. Effect of early cold stress on the maturation of rice anthers[J]. Proteomics, 2004, 4(7):1873-1882.
(責任編輯 狄艷紅)
Research Progress on Cofactor-independent Phosphoglycerate M utase in Plants
Xie Xin Liu Na Wei Fengju
(Hebei Province Key Laboratory of Plant Physiology and Moleculars Pathology College of Life Sciences,Agriculture University of Hebei,Baoding071001)
Cofactor-independent phosphoglycerate mutase(iPGAM)is a metalloenzyme discovered in fungi and higher plants, i.e., a key enzyme in glycolysis and gluconeogenesis. With the progress on cloning of iPGAM gene, analysis of its protein crystal structure and biological functions in prokaryotes and invertebrates, the function of plant’s iPGAM has captured the attentions. This article reviewed the structural characteristics, action mechanism of plant iPGAM proteins while emphatically introducing the phylogenetic relationships and biological functions of iPGAMs in Arabidopsis, rice and corn.
plant;cofactor-independent;phosphoglycerate mutase
10.13560/j.cnki.biotech.bull.1985.2015.09.001
2015-03-02
國家自然科學基金項目(31040052,31101022),中國農業大學開放課題(SKLPPBKF1504),河北農業大學青年科學基金(QJ201234)
謝鑫,女,碩士研究生,研究方向:植物抗旱分子機制;E-mail:xiexincg@163.com
魏鳳菊,女,博士,講師,研究方向:植物抗逆分子機制;E-mail:weifj98@126.com

