嗜熱鏈球菌的特性與應用研究進展
田輝 梁宏彰 霍貴成 Evivie Sm ith Etareri
(東北農業大學 乳品科學教育部重點實驗室,哈爾濱 150030)
嗜熱鏈球菌的特性與應用研究進展
田輝 梁宏彰 霍貴成 Evivie Sm ith Etareri
(東北農業大學乳品科學教育部重點實驗室,哈爾濱150030)
嗜熱鏈球菌是一種重要的工業用乳酸菌,廣泛應用于發酵乳制品生產。近年來,對嗜熱鏈球菌的研究已不僅僅停留于應用方面,而有關該菌基因組學等深層次特性機理的研究越來越多,對嗜熱鏈球菌的研究和應用開發具有較好的參考價值。綜述了嗜熱鏈球菌的認定和應用、基因組學特征、發酵特性、噬菌體防御和共生機制等方面的研究進展,并展望了現代生物技術在嗜熱鏈球菌特性研究中的應用前景。
嗜熱鏈球菌;基因組;發酵;噬菌體;共生
嗜熱鏈球菌是一種重要的工業用乳酸菌,可用于生產酸奶和奶酪等多種發酵乳制品。長期以來,人們對嗜熱鏈球菌的研究和應用僅限于傳統的菌株篩選和低效的反復試驗之中,難以滿足現代食品工業的需求。而隨著人類基因組計劃的實施和完成,基因組學和各種現代生物技術以嶄新的面貌呈現,并以前所未有的速度解析多種生物規律和機理。截至2014年底,京都基因與基因組百科全書(Kyoto encyclopedia of genes and genomes,KEGG)共公布了3 511條全基因組序列,其中細菌3 036條,古細菌176條,真核生物299條[1]。
現代生物技術能夠使人們將某一物種的基因組序列等靜態數據,與在不同環境中的基因轉錄、翻譯及蛋白質修飾等動態數據系統地結合,從整體上對物種的生長代謝性能進行分析和預測。近幾年,人們應用基因組學、代謝組學、蛋白質組學和轉錄組學等方法深入研究了嗜熱鏈球菌的特性,為嗜熱鏈球菌在現代食品工業中的應用提供了豐富的依據。本文從嗜熱鏈球菌的種屬認定和應用、嗜熱鏈球菌的基因組學特征和發酵特性、嗜熱鏈球菌的防御體系、以及與保加利亞乳桿菌的共生機制等方面,依據現代生物技術的研究,對嗜熱鏈球菌的特性和應用展開論述,為乳品微生物研究提供數據支持。
1 嗜熱鏈球菌的認定與應用
乳酸菌是一類公認安全的食品級微生物,普遍存在于水果、蔬菜、谷物、酒、牛乳和人類胃腸道及泌尿生殖道等諸多環境中。乳酸菌能夠代謝乳糖和葡萄糖等產生乳酸,被廣泛應用于發酵乳制品、肉制品和青貯飼料等生產中。乳酸菌對人類健康具有重要作用,如人體腸道內的乳酸菌通過產生細菌素拮抗有害微生物。另外,乳酸菌發酵產生的短鏈脂肪酸和胞外多糖等代謝產物都是有益物質。乳酸菌是一類細菌的統稱,所包含的種屬涉及到乳球菌屬(Lactococcus)、腸球菌屬(Enterococcus)、酒球菌屬(Oenococcus)、片球菌屬(Pediococcus)、鏈球菌屬(Streptococcus)、明串珠菌屬(Leuconostoc)、和乳桿菌屬(Lactobacillus)等[2]。
嗜熱鏈球菌作為一種重要的工業用乳酸菌,廣泛應用于發酵乳制品的生產之中,其重要性僅次于第一位的乳酸乳球菌,其每年的市場份額約為400億美元[3]。鑒于嗜熱鏈球菌在食品發酵中具有悠久歷史,歐洲食品安全局(European food safety authority,EFSA)認定其具有安全資格(Qualified presumption of safety,QPS)[3,4]。作為最經典的酸奶發酵劑菌種,嗜熱鏈球菌常與保加利亞乳桿菌(Lactobacillus bulgaricus)一起用于酸奶生產,通過共生作用,二者相互促進,大大提高生長速度并改善酸奶品質[5]。嗜熱鏈球菌是一種耐高溫型乳酸菌,在酸奶和硬質奶酪生產中可耐受較高的發酵和加工溫度[6],與同為耐高溫型乳酸菌的保加利亞乳桿菌或瑞士乳桿菌(Lactobacillus helveticus)一起用于生產,共同作用,快速產酸,賦予產品良好的質構特性和風味[7]。
人們對嗜熱鏈球菌的分離與研究工作開始于上個世紀初。1991年,首次報道了嗜熱鏈球菌的部分特性:革蘭氏陽性、不運動、無芽孢、過氧化氫酶陰性、兼性厭氧、同型乳酸發酵、可生存于牛乳房黏膜和生牛乳中,而通過分析對比16S rRNA和超氧化物歧化酶sodA基因得知,與嗜熱鏈球菌親緣相近的是唾液鏈球菌和前庭鏈球菌,因此,嗜熱鏈球菌曾一度被劃為唾液鏈球菌的一個亞種,直到1991年通過DNA-DNA重組實驗才將嗜熱鏈球菌恢復到種的水平[6,8]。隨后,嗜熱鏈球菌被美國和歐盟確定為93個鏈球菌屬中唯一的公認安全菌種[9]。現在,人們每年攝食的活體嗜熱鏈球菌約1021個,在注重飲食營養與安全的現代生活中,由嗜熱鏈球菌發酵生產的酸乳已成為一種遍布世界各地的產業化健康美食[6]。
發酵乳制品的產業化使得人們對發酵劑菌種(嗜熱鏈球菌、保加利亞乳桿菌和乳酸乳球菌)提出更嚴格的要求,如何開發利用嗜熱鏈球菌,研制出滿足要求的工業發酵劑,是嗜熱鏈球菌研究的新內容。現代乳制品產業多采用的固定菌株,以及酸奶發酵劑的單一化,導致產品種類不足、生產過程易受噬菌體侵染等諸多缺點,因此分離和篩選新菌種作為發酵劑成為亟待解決的問題[10];而另一方面,對候選菌種特性的了解程度,會直接影響到其產品質量的安全性與穩定性。所以,如何全面了解候選菌種的多方面特性是新菌株篩選所面臨的一大難題。
2 嗜熱鏈球菌的基因組學研究
2.1 嗜熱鏈球菌的基因組特征
從2001年第一株乳酸菌(乳酸乳球菌IL1403)的全基因組測序完成,以后每年公布的乳酸菌全基因組序列的數量不斷增加。2008年,Mayo等[11]報道乳酸菌的全基因組序列有屬于15個種類的20多株被公布,其中包括嗜熱鏈球菌CNRZ1066、LMG13811和LMD9。2011-2013年公布測序全基因組完成的嗜熱鏈球菌有JIM8232、MN-ZLW-002、ND03和MTCC5461[12,13]。截至2014年底,在National Center for Biotechnology Information(NCBI) 網站能夠查詢到詳細信息的嗜熱鏈球菌有7株,分別是CNRZ 1066(基因組編號CP000024)、LMG 13811(基因組編號CP000023)、LMD-9(基因組編號CP000419)、MN-ZLW-002(基因組編號CP003499)、ND 03(基因組編號CP002340)、JIM 8232(基因組編號FR875178)及ASCC 1275(基因組編號CP006819),見表1[14]。通過統計分析表明:其基因組大小在1.85 Mb左右,含單一環狀染色體,不含或含少量質粒,G+C含量低,約含有2 000個基因,不同嗜熱鏈球菌的基因組在堿基水平上的相似度約95%[15]。嗜熱鏈球菌與真核生物相比具有基因組小、不含內含子、富含多種營養物質運載體基因、代謝底物合成和分解基因以串聯形式組成的操縱子等特點,為基因的高效轉錄、表達產物快速發揮作用奠定了基礎,也為其組學研究創造了有利條件。

表1 七株嗜熱鏈球菌的全基因組信息
2.2 嗜熱鏈球菌的基因組進化
嗜熱鏈球菌CNRZ 1066和LMG 13811分離自法國和英國傳統酸奶,是首次完成全基因組測序的嗜熱鏈球菌,其序列特征見表2。CNRZ 1066和LMG 13811菌株都含有一個約1.80 Mb的染色體,含約1 900個編碼序列,其中近80%與鏈球菌屬中其他菌種的序列同源,證實了嗜熱鏈球菌在細菌分類中的重要地位。而兩株菌(CNRZ 1066和LMG 13811)間也存在90%以上的同源序列,表明兩者可能由共同的祖先菌株進化而來,根據兩者基因組特征和自然突變速率估測,如果按照每天細胞裂殖1-10代計算,兩株菌的共同祖先可追溯至107代之前,即3 000-30 000年前,與人們開始制作發酵乳制品的時間相吻合,說明此時人們已經開始使用嗜熱鏈球菌了。Bolotin等[12]于2004年對這兩株菌(CNRZ 1066和LMG 13811)的全基因組序列進行了比較發現,嗜熱鏈球菌與鏈球菌屬的其他菌種相比,基因組中明顯發生了基因退化,其中10%的假基因多為糖代謝相關基因。因此,推測嗜熱鏈球菌發生基因退化之前,其糖代謝基因可正常發揮作用,使菌株具有廣泛的環境適應能力,其部分糖代謝基因失活是由于長期被人們應用于乳制品的發酵中,對糖類貧乏的乳環境(只含乳糖)的產生了適應;另一方面,嗜熱鏈球菌基因組缺少存在于致命性鏈球菌中的諸多致病相關基因,如肺炎鏈球菌(Streptococcus pneumoniae)、生膿鏈球菌(Streptococcus pyogenes)、無乳鏈球菌(Streptococcus agalactiae)和引起齲齒的變形鏈球菌(Streptococcus mutans)[12,20]。因此,現代生物技術的組學在嗜熱鏈球菌研究中的應用,明確了嗜熱鏈球菌的進化進程,也證實了嗜熱鏈球菌的安全性。

表2 嗜熱鏈球菌CNRZ 1066和LMG 13811的基因組特征
2.3 基因的水平轉移
不同菌株的基因組之間存在差異,可能是由基因丟失、基因重疊和基因的水平轉移等引起的[2]。2011年,Delorme等[18]對一株分離自乳中的嗜熱鏈球菌JIM 8232進行了基因組測定分析。由于能夠產生黃色色素,因此該菌株具有較高的特異性。通過比較基因組學分析發現,JIM 8232基因組中存在一段長度為129 kb的特異性序列,這段序列可分為2.6-55 kb長度不等的9個區域,其中胞外多糖基因簇、限制/修飾系統和規律成簇的間隔短回文重復(Clustered regularly interspaced short palindromic repeats,CRISPR)序列為該段的高變異區域,另外3個區域與抗氧化代謝相關,這6個區域的兩側都有基因座等移動元件;而最后3個區域為整合區域,包括剪接酶合成基因以及兩個主要的基因島(色素合成操縱子和prtS蛋白酶基因)。這些區域的特性說明它們是通過基因水平轉移的方式整合到基因組上的,說明基因水平轉移在嗜熱鏈球菌基因組進化中起到了重要作用。
嗜熱鏈球菌CNRZ 1066和LMG 13811是兩株親緣較近的菌株,然而它們在胞外多糖合成、限制/修飾系統、原噬菌體和CRISPR基因座等基因序列上存在差異[12]。2011年,在荷蘭召開的第10屆乳酸菌專題討論會上,Goh等[9]介紹了將一株性能優良的嗜熱鏈球菌LMD-9與兩株菌CNRZ 1066和LMG 13811進行基因組比較分析,發現菌株LMD-9也擁有一段長度為114 kb的特異性序列,其中包括組氨酸合成、細胞表面蛋白酶、宿主防御機制和原噬菌體、假定粘液粘附蛋白、肽類運載體以及胞外多糖合成的相關基因,根據同源性分析表明這些特異性基因是菌株在特定乳環境中通過水平基因轉移獲得。事實上,嗜熱鏈球菌的一些重要特性與基因水平轉移是密不可分的,如胞外多糖的合成,細菌素的產生,限制性修飾系統及耐氧力等[2]。
3 嗜熱鏈球菌發酵特性
牛乳富含多種營養物質,適合多種微生物生長和繁殖,而嗜熱鏈球菌發酵牛乳時,具有快速生長產酸的特點,從而創造適合另一部分乳酸菌生長的酸性環境。嗜熱鏈球菌能夠在眾多微生物中占據初期生長優勢,主要在于其發酵過程中獨特的糖代謝和蛋白質代謝能力,轉錄組學和蛋白質組學為人們對其發酵特性的研究提供了重要技術手段。
3.1 嗜熱鏈球菌的基因表達特征
Derzelle等[21]在2005年首次測定了嗜熱鏈球菌LMG 18311在牛乳和M17中生長的蛋白質組,分析了菌株在兩種基質中的蛋白質表達差異,重點研究了與氨基酸攝入相關的酶,如肽酶PepX和一些氨基酸合成酶。研究發現菌株在兩種基質中生長1.5 h后,主要蛋白表達差異是丙酮酸鹽甲酸鹽裂合酶(Pfl)、參與氨基酸與嘌呤合成的蛋白質,推測可能是由于乳中缺少必要的遺傳物質,嗜熱鏈球菌在乳中生長時Pfl高表達,因為它參與嘌呤的合成過程,而嘌呤是重要的遺傳代謝物質。通過Northern blot對pfl基因的mRNA進一步研究發現,在乳中生長時,pfl基因表達上調,導致Pfl酶的含量增加,而向乳中添加甲酸和嘌呤堿基后,pfl基因的表達水平降低,Pfl酶的合成減弱,證明了Pfl酶在以甲酸鹽為底物的合成反應中的重要作用,探索了Pfl酶的表達調控特性[21,22]。隨后Salzano等[23]測定了嗜熱鏈球菌ATCC 19258的細胞質蛋白和細胞膜蛋白,鑒定了對應于458個基因的蛋白質,包括參與碳源、脂肪酸、氨基酸和核酸代謝的酶類,基因復制、轉錄、翻譯、細胞壁合成的蛋白酶類,以及與乳制品加工性能有關的重要蛋白質(如乳糖的攝入和胞外多糖的合成),實驗結果有利于進一步分析和對比不同菌體的蛋白質組,為工業生產中優良菌株的篩選提供參照。Herve-Jimenez等[22]測定了關于LMG 18311在乳中生長的細胞質蛋白質組圖譜,鑒定了203個不同的蛋白質,占理論蛋白質組的32%。通過轉錄組學和蛋白質組學方法分析了菌株在乳中不同生長階段(2.5 h和5.5 h)的生理狀況,確定了菌株生長后期上調表達的蛋白質分別為:(1)肽類與氨基酸的運載體,及特定氨基酸尤其是含硫氨基酸的合成;(2)糖類代謝中的相關蛋白質。新技術的應用使人們對嗜熱鏈球菌在牛乳環境中生長的基因表達特征有了全局認識,為嗜熱鏈球菌代謝特性的研究提供了更為深入的數據。
3.2 糖類代謝的研究
嗜熱鏈球菌能夠快速代謝牛乳中乳糖生成乳酸的能力,是其廣泛應用于酸奶和奶酪生產的重要特性之一[21]。嗜熱鏈球菌長期生長在牛乳環境中使其糖類代謝力退化,可利用的糖類較少,主要有乳糖、蔗糖、葡萄糖、半乳糖和果糖等,其中能夠利用半乳糖和果糖的只占少數[6]。嗜熱鏈球菌糖類代謝類型是同型乳酸發酵,代謝產物除L-乳酸外,尚有微量的甲酸鹽、乙偶姻(3-羥基-2-丁酮)、丁二酮、乙醛和醋酸鹽等[6]及與發酵乳風味形成的相關物質。
3.2.1 乳糖代謝系統 牛乳中的糖種類單一,但其中的乳糖含量相對比較豐富,長期的進化使嗜熱鏈球菌對乳糖具有較強的偏好性[24]。當分別以3種糖(乳糖、蔗糖和葡萄糖)為單一碳源時,嗜熱鏈球菌A147在葡萄糖基質中生長時會出現遲滯期延長、生長速率為緩慢的現象。以蔗糖和果糖為代謝底物時,其糖轉運系統中可以檢測到磷酸烯醇式丙酮酸依賴型磷酸轉移酶系統(PTS)代謝活力。而以葡萄糖、乳糖和半乳糖為底物時則沒有出現該現象,進一步研究表明,乳糖和半乳糖的轉運是通過乳糖滲透酶(LacS)二級轉運蛋白進行的,該菌的糖轉運特征如表3所示[24]。對于大部分乳酸菌,葡萄糖是典型的PTS碳源,能夠被迅速攝取并利用,而對于嗜熱鏈球菌,葡萄糖卻是一種非PTS碳源,并且其利用率顯著低于另一種非PTS糖類——乳糖[25]。Bolotin等[12]在菌株CNRZ 1066和LMG 13811的基因組中發現,有7個基因座編碼PTS系統(ptsG、fruA、bglP、treP、manLMN、celB和scrA)、2個基因座編碼糖類ABC轉運體(malEF,stu/str0808-811)、2個編碼糖類轉運體lacS和stu/str0855,但是這些基因中含有大量的假基因以及無編碼功能基因,說明嗜熱鏈球菌產生了基因丟失性進化[6],因此,結合嗜熱鏈球菌A147糖轉運系統的研究可知,引起上述差異的主要原因在于嗜熱鏈球菌在進化過程中丟失了葡萄糖PST轉運系統相關基因,大大降低了葡萄糖攝取能力,而乳糖的代謝基因卻得到保留,包括乳糖滲透酶(LacS)和β-半乳糖苷酶(LacZ),其中LacS是一種轉運乳糖的非磷酸系統轉移酶[21,25],而β-半乳糖苷酶是乳糖/半乳糖的反向運載體[6]。乳糖進入菌體后被β-半乳糖苷酶分解為半乳糖和葡萄糖,其中的葡萄糖可被嗜熱鏈球菌株利用,而半乳糖一般情況下不被利用,經LacZ排出體外。
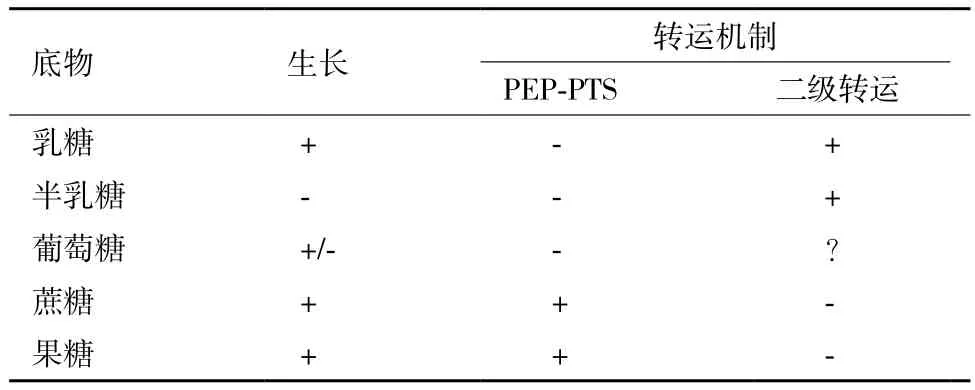
表3 嗜熱鏈球菌A147的糖類轉運機制
嗜熱鏈球菌不能代謝半乳糖的特性也從基因水平上得到了解釋。嗜熱鏈球菌中多數是不能利用半乳糖的,然而van den Bogaard等[26]發現在半乳糖代謝陰性菌株中也存在編碼半乳糖代謝(Leloir途徑)的基因(galK-TEM),與陽性突變菌株的基因進行比較發現,限制大多數嗜熱鏈球菌半乳糖代謝的主要原因并非是缺少相關的代謝基因,而是其表達的半乳糖激酶活力較低所致[27,28](圖1)。該發現揭示了嗜熱鏈球菌糖代謝的重要機理,改變了以往人們的觀點,為以后的代謝調控提供了數據參考,指明了正確的研究途徑。
3.2.2 糖代謝調控因子 嗜熱鏈球菌的乳糖代謝基因簇是高度保守的,包括半乳糖代謝(Leloir途徑)基因和乳糖代謝(轉運和分解)基因,這些基因連接在一起組成一個基因簇,即galKTEMlacSZ。在galK基因上游有一個轉錄調控子編碼基因galR,它能對兩個操縱子(galKTEM和lacSZ)的轉錄進行正調控[27]。半乳糖代謝陽性菌株CNRZ 302能夠較高水平地表達galKTE,經過分析表明,其galK啟動子區域出現了特定變異[27]。兩個操縱子除了受galR的正調控外,其中lac S啟動子還受分解代謝控制蛋白CcpA的調控。CcpA通過lac S啟動子區的cre(CcpA感應因子)而產生作用,抑制lacSZ表達,減少乳糖的攝入,同時又能提高糖酵解過程中3種關鍵酶(乳酸脫氫酶、磷酸果糖激酶和丙酮酸激酶)的表達,加速胞內糖的酵解。CcpA突變菌株的乳糖攝入和分解能力大大增加,但卻將大量半乳糖和葡萄糖排出胞外,從而導致乳酸生成速率和菌株生長速率大大降低,這說明CcpA對lacS啟動子的調控能力可以使乳糖攝入和分解過程與整個糖酵解過程達到更好的平衡[25]。通過DNA微陣列實驗對比嗜熱鏈球菌野生菌株和CcpA突變菌株發現,CcpA的突變影響了大量的轉錄調控子,其中包括金屬離子轉運系統的一些調控子,說明CcpA不僅能調控糖代謝,還參與其他代謝途徑[6]。
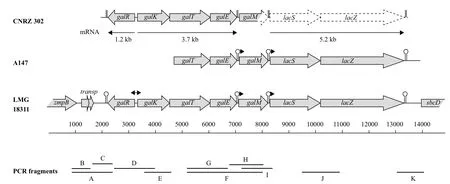
圖1 嗜熱鏈球菌gal-lac基因簇[26]
3.2.3 胞外多糖合成 胞外多糖能夠改善發酵乳制品組織和流變特性,防止脫水收縮,產生絲滑柔順的組織特性,賦予產品良好的口感和味覺[6]。多種細菌具有產生胞外多糖的能力,如果胞外多糖結合于細胞表面稱為莢膜多糖(Capsular polysaccharides,CPS),如果脫離細胞而釋放到培養基中則稱為胞外多糖(Extracellular polysaccharide,EPS)。胞外多糖又可根據其組成單元是一種單糖還是不同單糖而分為同型多聚糖和雜型多聚糖。首次被測定結構的EPS產自菌株CNCMI 733、734和735,它們產生的胞外多糖中D-半乳糖∶D-葡萄糖∶2-乙酰-氨基-2-脫氧-D-半乳糖的比例為2∶1∶1[29],大多數嗜熱鏈球菌產生雜多聚糖的胞外多糖,也有一些菌株產生的是莢膜多糖[6,30]。嗜熱鏈球菌的EPS結構多樣,組成單元主要由半乳糖、葡萄糖和鼠李糖組成,但也有N-乙酰基-半乳糖胺、果糖和乙酰基半乳糖[30]。嗜熱鏈球菌產生胞外多糖的特性具有菌株特異性,與生長速率相關,而且其合成與分子結構明顯受生長條件,如溫度、pH、碳源種類、濃度及C/N比等影響[31]。
目前有至少12個不同的eps基因簇序列被測定和公布,但通過限制性片段長度多態性(Restriction fragment length polymorphism,RFLP)分析,卻發現有60多個不同的eps基因簇存在[32]。Eps基因簇主要包括調控基因(epsA、epsB)、糖鏈長度控制基因(epsC、epsD)、重復性片段單元的合成基因(epsE、epsF、epsG、epsH和epsI)、單元聚合及多糖釋放控制基因(epsK、epsL和epsM)[30,31]。嗜熱鏈球菌胞外多糖合成途徑相對保守,并與同屬中其他鏈球菌相似[31],由葡萄糖-6-磷酸開始,生成葡萄糖-1-磷酸,經UDP-葡萄糖焦磷酸化酶形成UDP-葡萄糖參與合成EPS[6]。Rasmussen等[7]基于已公布的2 200個基因設計了DNA微陣列,分析了47株鏈球菌的基因組,發現幾乎所有菌株都含有deoD-epsABCD基因,7株菌沒有epsE(編碼半乳糖-1磷酸或葡萄糖-1-磷酸轉移酶),2個菌株不含epsA(編碼轉錄調控子),但在其他胞外多糖合成基因和整個基因簇組織上存在多種形式,證明eps基因簇結構的高度可變性。比較CNRZ 1066、LMG 18311和LMD-9三株嗜熱鏈球菌的全基因組發現,嗜熱鏈球菌LMD-9有兩個eps基因簇,分別為eps基因簇和rgp基因簇,3株嗜熱鏈球菌和一株唾液鏈球菌(SK126)的eps基因序列相似度在90%以上,尤其是在5'和3'兩端的區域,而且兩端的保守區域分別與deoD(編碼嘌呤核苷磷酸酶)和bglH(編碼β-糖苷酶)毗鄰[9]。嗜熱鏈球菌eps基因簇中一些移動元件也發現于其他鏈球菌和乳酸乳球菌,說明了eps基因簇的可移動性。嗜熱鏈球菌CNRZ 1066和Sfi6基因組中的eps基因簇的同源[32],也說明了這個問題。eps基因簇的高變異性可能由于其在水平基因轉移或基因重組過程中插入片段或丟失片段等原因[30]。大量關于嗜熱鏈球菌全基因組的分析都證明eps基因簇與基因水平轉移相關,如JIM 8232[18],以及在ND03基因組中發現eps基因簇存在多拷貝的IS元件,而這些元件也導致菌株eps基因具有較高特異性[17]。
許多乳酸菌產EPS的性狀是不穩定的,甚至可能會永久性丟失。在乳酸乳球菌中,由于多糖控制基因存在于質粒上,因此可以用質粒的不穩定來解釋EPS的性狀丟失,但嗜熱鏈球菌的eps合成基因全部存于染色體上,其性狀不穩定可能是一些插入元件插入在eps基因簇的內部或者與其相連所致,也可能是由于整體基因組不穩定性導致EPS的產生具有可變性[30]。而從另外一個角度來看,其可變性又為陰性嗜熱鏈球菌獲得產EPS的特性提供了機會。
3.3 蛋白質代謝的研究
3.3.1 關鍵代謝基因prtS 乳中氨基酸和短肽含量雖然相對較少,但卻富含大量蛋白質,主要包括酪蛋白和乳清蛋白,其中酪蛋白通過鈣離子交聯以膠束形式存在,因此不能直接被乳酸菌攝入,所以嗜熱鏈球菌通過多種蛋白酶及氨基酸肽類轉運系統將乳中酪蛋白水解為二肽、三肽及多肽,攝入至胞內參與氨基酸和蛋白質的合成、降解等代謝[22]。與乳酸乳球菌蛋白質水解系統相比,嗜熱鏈球菌對外源氨基酸的攝入能力較弱[21]。乳酸乳球菌主要依靠細胞壁蛋白酶(PrtP)分解乳中酪蛋白為自身提供所需求的氨基酸,而嗜熱鏈球菌中僅有少數菌株具有細胞外蛋白酶(PrtS)。PrtS是枯草桿菌蛋白酶家族的一種絲氨酸蛋白酶,能分解乳蛋白,提供肽類和氨基酸,使嗜熱鏈球菌在乳中快速生長,當菌株含有該酶時,菌體密度能夠迅速達到109CFU/mL,而不含該酶的菌株最多只能達到108CFU/mL[22,33]。在發酵乳制品生產和優良發酵劑的評估中,嗜熱鏈球菌的生長速率可由產酸速率來體現,產酸速率越高代表發酵劑的生長性能越好。因此,PrtS蛋白酶的存在與否將決定嗜熱鏈球菌是快產酸型菌株或是慢產酸型菌株,尤其當嗜熱鏈球菌以單菌株作為發酵劑時,PrtS更能起到關鍵作用。嗜熱鏈球菌作為生長起始菌株,能快速生長產酸,使pH值迅速降低,因此常選用產酸性能較強的嗜熱鏈球菌作為工業生產用菌株[10]。而在INRA所收集的135株菌中,僅有21株為細胞壁蛋白酶陽性菌株,表明了prtS基因在嗜熱鏈球菌中的稀有性。嗜熱鏈球菌中prtS是一個長為15 kb的基因,存在于菌株LMD 9,而不存在于菌株LMG 18311和CNRZ 1066[6,33],可能通過水平基因轉移獲得。Dandoy等[33]將PrtS陽性菌株的prtS基因轉化到3株產酸較慢的PrtS陰性菌株(LMG 18311、DGCC 7666和DGCC 7891)中,使重組菌株產酸速率提高2.4倍,遲滯期縮短,產酸至pH 5.2的時間縮短2-3倍。該實驗的成功為乳品發酵劑定向改造提供了新思路,即可以將不同的乳品工業相關特征(快速產酸、組織結構的影響、細菌素的產生、抗噬菌體等)組合到單株菌中,使人們在生產中利用單菌株即可達到產品要求,既簡化了生產步驟,又節約了成本投入。
3.3.2 氨基酸合成特性 由上可知,大部分嗜熱鏈球菌不能水解酪蛋白,而依靠自身氨基酸合成和細胞內肽酶的作用來滿足自身氨基酸需求主要。氨基酸的合成對嗜熱鏈球菌的生長具有關鍵作用,尤其是支鏈氨基酸的合成,是其在天然牛乳環境中生長的關鍵途徑之一[22]。雖然一些菌株能夠滿足自身的氨基酸需要,但是也有一部分菌株不能合成特定的氨基酸。對菌株LMD 9與LMG 18311和CNRZ 1066的基因組比較分析發現,其基因組缺少谷氨酸合成基因,這一結果解釋了該菌株不能在谷氨酸和谷氨酰胺都缺少的環境中生長的現象[6]。盡管氨基酸合成對嗜熱鏈球菌具有重要作用,在基因組序列中氨基酸合成的基因序列也是高度保守的,但其中也含有一定數量的假基因,如在菌株LMG 18311中,假基因包括參與合成丙氨酸、賴氨酸、支鏈氨基酸和含硫氨基酸的alaD、dapE、ilvD和yhcE基因,另外菌株CNRZ 1066中的天冬氨酸氨基轉移酶aspC3也是假基因[6]。生物信息學預測嗜熱鏈球菌LMG 18311和CNRZ 1066具有合成除了組氨酸外的其他氨基酸的所有基因,但是經過實驗驗證的只有參與支鏈氨基酸和脯氨酸合成的基因。由于大部分嗜熱鏈球菌都能在混合有谷氨酰胺、甲硫氨酸、亮氨酸、異亮氨酸、纈氨酸和組氨酸的底物中生長,因此推測其他氨基酸可以由菌株自身合成[6]。更多關于CNRZ 1066和LMG 13811的基因組序列的分析研究表明,嗜熱鏈球菌有20多種潛在的胞內肽酶[12]。其中一些肽酶的基因序列已被克隆并研究,包括兩個氨肽酶基因(pepN和pepC)、一個內肽酶基因(pepO)、一個X-脯氨酰-二肽酰基-氨基肽酶基因(pepX)和一個氨肽酶(pepS)[34]。一株營養缺陷嗜熱鏈球菌中,由于肽的轉運、支鏈氨基酸和嘌呤的合成基因失活,使得該菌株不能在乳中生長,因此,嗜熱鏈球菌的正常生長需要具有自身轉運肽類并轉化為氨基酸或合成氨基酸的能力[21]。
3.3.3 氮代謝調控因子 菌株LMG 18311和CNRZ 1066的基因組中發現了乳酸乳球菌中調控基因的同系物,如CodY(Stu1635)、ArgR(Stu0048)、GlnR(Stu0177)、AhrC(Stu1214)以及FhuR(Stu0520、Stu0452和Stu0872)。CodY蛋白是存在于革蘭氏陽性細菌中的一種上游轉錄調控因子,它能調控蛋白水解系統中相關基因的表達,當細胞內支鏈氨基酸(異亮氨酸、亮氨酸和纈氨酸)的濃度增大時,CodY 能夠結合到蛋白水解系統相應基因操縱子的上游序列,從而阻止這些基因的表達。在營養物質缺乏時支鏈氨基酸濃度降低,菌株生長進入穩定期,此時蛋白水解系統的基因被誘導表達,幫助菌體適應營養僵乏的環境。本實驗室根據已公布的嗜熱鏈球菌CNRZ1066和LMD-9的基因組信息,推斷嗜熱鏈球菌中也應含有一個類似的調控蛋白CodY,并根據嗜熱鏈球菌基因組序列設計了引物Co1和Co2,分別對5株嗜熱鏈球菌的CodY基因進行PCR擴增,證實了codY基因的存在,嗜熱鏈球菌的codY基因序列具有非常保守的特點。實驗進一步證實了CodY蛋白在嗜熱鏈球菌蛋白代謝中的調控作用,為高效酸奶發酵劑的開發提供了理論依據[35]。
4 嗜熱鏈球的防御體系
生產環境較為安全、雜菌污染較少的乳制品生產發酵過程中,嗜熱鏈球菌遭受噬菌體污染也成為了一個難以避免的難題。
嗜熱鏈球菌對于噬菌體存在一定的防御機制。其中成簇的短間隔回文重復序列以及其相關核酸內切酶系統(CRISPR-Cas),是人們研究最多的噬菌體防御系統。該系統的Cas(CRISPR-associated)核酸內切酶通過CRISPR序列相關RNA(crRNA)的定向誘導對外源DNA片段進行特異性切割,達到使噬菌體侵染流產的目的。另外,切割后產生的外源DNA碎片會整合到CRISPR序列中,加強crRNA的定向誘導作用。因此,該系統是一個天然的分子免疫系統。
通過比較分析發現,嗜熱鏈球菌中存在多種類型的CRISPR-Cas系統,而且幾乎都含有1-4個不同的CRISPR序列。本實驗室鄧凱波等[36]研究實驗室菌種庫中8株嗜熱鏈球菌的CRISPR序列,分別檢測了菌株中的CRISPR序列的存在,并分析了序列同源性,結果表明除1株菌含有1個CRISPR結構外,其余7株菌均含有3個結構,序列長度最長為2 853 bp,最短僅為101 bp。通過同源性比對發現,CRISPR序列中的重復序列與遠緣菌種具有較高同源性,表明CRISPR序列可能通過水平基因轉移獲得。該研究對我國野生嗜熱鏈球菌抗噬菌體性能的研究提供參考,有利于高品質自主發酵劑研究的進展。
5 與保加利亞乳桿菌的共生作用機制
嗜熱鏈球菌和保加利亞乳桿菌在發酵牛乳的過程中,能夠相互提供營養物質和生長因子,使生長和產酸速度得到很大提升,被稱為共生作用。這種共生作用體現在多方面,其中最為明顯的是保加利亞乳桿菌利用細胞壁蛋白酶水解乳蛋白,產生肽類和游離氨基酸,為蛋白水解力相對較差的嗜熱鏈球菌提供氮源需求[21];同時嗜熱鏈球菌為保加利亞乳桿菌提供葉酸、甲酸和CO2,作為合成嘌呤的前體或輔助因子,促進保加利亞乳桿菌快速生長[37,38]。
Herve-Jimenez等[22]在嗜熱鏈球菌LMG 18311與保加利亞乳桿菌共同培養,研究對嗜熱鏈球菌表達特性的影響,結果表明嗜熱鏈球菌生長后期的糖代謝基因、肽類和氨基酸運載體基因上調表達未受影響,說明保加利亞乳桿菌了對嗜熱鏈球菌生長的無抑制作用。劉夢晉等(Liu)[39]通過計算機模擬技術分析嗜熱鏈球菌和保加利亞乳桿菌的全基因組序列,預測了兩者之間的水平基因轉移,結果表明,在酸奶生產歷史中,嗜熱鏈球菌和保加利亞乳桿菌由于長期在同一環境中共存,導致兩者之間更容易發生水平基因轉移,使菌株在基因水平上產生優化,有利于更好地相互作用和生長,其中EPS合成基因從嗜熱鏈球菌轉移到保加利亞乳桿菌,而含硫氨基酸代謝的cbs-cblB(cglB)-cysE基因簇(或稱為cysM2-metB2-cysE2)則從保加利亞乳桿菌轉移到嗜熱鏈球菌。
Herve-Jimenez等[40]及Sieuwerts等[38]通過轉錄組學和蛋白質組學研究發現,嗜熱鏈球菌和保加利亞乳桿菌的相互作用涉及到嘌呤、含硫氨基酸和短鏈脂肪酸的代謝,通過計算機模擬技術、轉錄組學和功能基因組學方法分析嗜熱鏈球菌LMD-9雙組份系統,測定出6個具有HK和RR的完整TCS(TCS 2、4、5、6、7和9),以及兩個不完整的HK系統(RR01和08)。在對數生長期時(pH 5.5)的嗜熱鏈球菌添加保加利亞乳桿菌,并以單菌株嗜熱鏈球菌作對照發現,由于保加利亞乳桿菌的加入,嗜熱鏈球菌的4個RR表達上調,導致嗜熱鏈球菌的調控產生變化[41],表明在酸奶生產中,嗜熱鏈球菌與保加利亞乳桿菌的共生作用,就是通過雙組份系統感應并調控自身代謝。
Sieuwerts等[38]通過轉錄組學對兩株菌(CNRZ 1066、ATCC BAA-365)進行深層次研究發現,共生過程中保加利亞乳桿菌只存在一個對數生長期,而嗜熱鏈球菌則出現了不同的生長曲線,分別為延遲期、第一個對數期、過渡期、第二個對數期和穩定期4個生長步驟;實驗還發現,在共同培養過程中,支鏈氨基酸和含硫氨基酸合成基因發生上調,說明兩種菌株難以得到足夠的此類氨基酸,兩個菌種雖然相互提供一定的營養物質,但是由于酪蛋白中僅能釋放出較低含量的支鏈氨基酸(6%-7%)、精氨酸(4%)和半胱氨酸殘基(0.35),所以只依靠桿菌的蛋白質水解作用不能為兩個菌種提供足夠的含硫氨基酸和支鏈氨基酸。共生過程中上調表達的還有eps基因,EPS主要在嗜熱鏈球菌的發酵后期產生,可能是培養基低pH值誘導產生EPS,基因epsA-M在第二個對數期及穩定期時才產生高表達,且EPS產量也相應增加。
6 展望
現代生物技術的應用使嗜熱鏈球菌的遺傳和生長代謝特性研究得到了豐碩成果,為嗜熱鏈球菌發酵開發過程中的關鍵基因和代謝途徑的發掘提供了大量信息。在不斷發展更新的生物技術的促進下,以基因組學為核心,與轉錄組學、蛋白質組學與代謝組學等共同組成了能夠全面理解生物本質和特性的系統生物學。而系統生物學將依靠互聯網技術,逐漸地整合生物學理論、數學模型和應用計算機等,形成生物信息學和系統生物學的整體分析模式,為嗜熱鏈球菌的研究引入新的階段。不久的將來,圍繞嗜熱鏈球菌如何提高產胞外多糖、風味、共生機制、抗噬菌體等性能,利用最新的生物技術和多學科交叉,會出現越來越多的高新成果,為發酵乳制品的發展注入新活力。
[1] KEGG Organisms:Complete Genomes[EB/OL]. http://www. genome. jp/kegg/catalog/org_list. htm l.
[2] Makarova K, Slesarev A, Wolf Y, et al. Comparative genomics of the lactic acid bacteria[J]. Proc Natl Acad Sci USA, 2006, 103(42):15611-15616.
[3] El-Sharoud WM, Delorme C, Darwish MS, et al. Genotyping of Streptococcus thermophilus strains isolated from traditional Egyptian dairy products by sequence analysis of the phosphoserine phosphatase(serB)gene with phenotypic characterizations of the strains[J]. J Appl Microbiol, 2011, 112(2):329-337.
[4] Delorme C. Safety assessmen t of dairy m icroorganisms:Streptococcus thermophilus[J]. Int J Food Microbiol, 2008, 126(3):274-277.
[5] Tamine AY, Robinson RK. Yoghurt:science and technology[G]. Cambridge:Woodhead Publishing Limited, 2000.
[6] Hols P, Hancy F, Fontaine L, et al. New insights in the molecular biology and physiology of Streptococcus thermophilus revealed by comparative genomics[J]. FEMS Microbiol Rev, 2005, 29(3):435-463.
[7] Rasmussen TB, Danielsen M, Valina O, et al. Streptococcus thermophilus core genome:comparative genome hybridization study of 47 strains[J]. Appl Environ Microbiol, 2008, 74(15):4703-4710.
[8] Schleifer KH, Ehrmann M, Krusch U, et al. Revival of the Species Streptococcus thermophilus(ex Orla-Jensen, 1919)nom. rev. [J]. Systematic and Applied Microbiology, 1991, 14(4):386-388.
[9] Goh YJ, Goin C, O’Flaherty S, et al. Specialized adaptation of a lactic acid bacterium to the milk environment:the comparative genomics of Streptococcus thermophilus LMD-9[J]. Microb Cell Fact, 2011, 10(Suppl 1):S22.
[10] Erkus O, Okuklu B, Yenidunya AF, et al. High genetic and phenotypic variability of Streptococcus thermophilus strains isolated from artisanal Yuruk yoghurts[J]. LWT-Food Science and Technology, 2014, 58(2):348-354.
[11]Mayo B, van Sinderen D, Ventura M. Genome analysis of food grade lactic Acid-producing bacteria:from basics to applications[J]. Curr Genomics, 2008, 9(3):169-183.
[12] Bolotin A, Quinquis B, Renault P, et al. Complete sequence and comparative genome analysis of the dairy bacterium Streptococcus thermophilus[J]. Nat Biotechnol, 2004, 22(12):1554-1558.
[13] Prajapati JB, Nathani NM, Patel AK, et al. Genomic analysis of dairy starter culture Streptococcus thermophilus MTCC 5461[J]. J Microbiol Biotechnol, 2013, 23(4):459-466.
[14] http://www. ncbi. nlm. nih. gov/genome/genomes/420[EB/OL].
[15] Callanan MJ, Ross RP. Starter Cu ltures:Genetics[M]. Academic Press, 2004.
[16] Kang X, Ling N, Sun G, et al. Complete genome sequence of Streptococcus thermophilus strain MN-ZLW-002[J]. J Bacteriol,2012, 194(16):4428-4429.
[17] Sun Z, Chen X, Wang J, et al. Complete genome sequence of Streptococcus thermophilus strain ND03[J]. J Bacteriol, 2011,193(3):793-794.
[18] Delorme C, Bartholini C, Luraschi M, et al. Complete genome sequence of the pigmented Streptococcus thermophilus strain JIM8232[J]. J Bacteriol, 2011, 193(19):5581-5582.
[19] Wu Q, Tun HM, Leung FC, et al. Genomic insights into high exopolysaccharide-producing dairy starter bacterium Streptococcus thermophilus ASCC 1275[J]. Sci Rep, 2014, 4:4974.
[20] Hols P, Hancy F, Fontaine L, et al. New insights in the molecular biology and physiology of Streptococcus thermophilus revealed by comparative genomics[J]. FEMS Microbiology Reviews, 2005,29(3):435-463.
[21] Derzelle S, Bolotin A, Mistou MY, et al. Proteome analysis of Streptococcus thermophilus grown in milk reveals pyruvate formatelyase as the major upregulated protein[J]. Appl Environ Microbiol, 2005, 71(12):8597-8605.
[22] Herve-Jimenez L, Guillouard I, Guedon E, et al. Physiology of Streptococcus thermophilus during the late stage of milk fermentation with special regard to sulfur am ino-acid metabolism[J]. Proteomics, 2008, 8(20):4273-4286.
[23] Salzano AM, Arena S, Renzone G, et al. A widespread picture of the Streptococcus thermophilus proteome by cell lysate fractionation and gel-based/gel-free approaches[J]. Proteomics, 2007, 7(9):1420-1433.
[24] Poolman B. Energy transduction in lactic acid bacteria[J]. FEMS Microbiol Rev, 1993, 12(1-3):125-147.
[25] van den Bogaard PT, Kleerebezem M, Kuipers O P, et al. Control of lactose transport, beta-galactosidase activity, and glycolysis by CcpA in Streptococcus thermophilus:evidence for carbon catabolite repression by a non-phosphoenolpyruvate-dependent phosphotransferase system sugar[J]. J Bacteriol, 2000, 182(21):5982-5989.
[26] van den Bogaard PT, Hols P, Kuipers OP, et al. Sugar utilisation and conservation of the gal-lac gene cluster in Streptococcus thermophilus[J]. Syst Appl Microbiol, 2004, 27(1):10-17.
[27] Vaughan EE, van den Bogaard PT, Catzeddu P, et al. Activation of silent gal genes in the lac-gal regulon of Streptococcus thermophilus[J]. J Bacteriol, 2001, 183(4):1184-1194.
[28] Vaillancourt K, LeMay JD, Lamoureux M, et al. Characterization of a galactokinase-positive recombinant strain of Streptococcus thermophilus[J]. Appl Environ Microbiol, 2004, 70(8):4596-4603.
[29] Navarini L, Abatangelo A, Bertocchi C, et al. Isolation and characterization of the exopolysaccharide produced by Streptococcus thermophilus SFi20[J]. International Journal of Biological Macromolecules, 2001, 28(3):219-226.
[30] Broadbent JR, McMahon DJ, Welker DL, et al. Biochemistry,genetics, and applications of exopolysaccharide production in Streptococcus thermophilus:a review[J]. J Dairy Sci, 2003, 86(2):407-423.
[31] Iyer R, Tomar SK, Uma Maheswari T, et al. Streptococcus thermophilus strains:Multifunctional lactic acid bacteria[J]. International Dairy Journal, 2010, 20(3):133-141.
[32] Stingele F, Neeser JR, Mollet B. Identification and characterization of the eps(Exopolysaccharide)gene cluster from Streptococcus thermophilus Sfi6[J]. J Bacteriol, 1996, 178(6):1680-1690.
[33] Dandoy D, Fremaux C, de Frahan MH, et al. The fast m ilk acidifying phenotype of Streptococcus thermophilus can be acquired by natural transformation of the genomic island encoding the cellenvelope proteinase PrtS[J]. Microb Cell Fact, 2011, 10(Suppl 1):S21.
[34] Anastasiou R, Papadelli M, Georgalaki MD, et al. Cloning and sequencing of the gene encoding X-prolyl-dipeptidyl aminopeptidase(PepX)from Streptococcus thermophilus strain ACA-DC 4[J]. J Appl Microbiol, 2002, 93(1):52-59.
[35] 劉芳. CodY蛋白在嗜熱鏈球菌蛋白水解系統調控中的作用[D]. 哈爾濱:東北農業大學, 2009.
[36] 鄧凱波, 霍貴成. 嗜熱鏈球菌中CRISPR序列的檢測與同源性分析[J]. 食品科學, 2013(3):153-157.
[37] van de Guchte M, Penaud S, Grimaldi C, et al. The complete genome sequence of Lactobacillus bulgaricus reveals extensive and ongoing reductive evolution[J]. Proc Natl Acad Sci USA, 2006,103(24):9274-9279.
[38] Sieuwerts S, Molenaar D, van Hijum SA, et al. Mixed-culture transcriptome analysis reveals the molecular basis of mixedculture growth in Streptococcus thermophilus and Lactobacillus bulgaricus[J]. Appl Environ Microbiol, 2010, 76(23):7775-7784.
[39] Liu M, Siezen RJ, Nauta A. In silico prediction of horizontal gene transfer events in Lactobacillus bulgaricus and Streptococcus thermophilus reveals protocooperation in yogurt manufacturing[J]. Appl Environ Microbiol, 2009, 75(12):4120-4129.
[40] Herve-Jimenez L, Guillouard I, Guedon E, et al. Postgenomic analysis of Streptococcus thermophilus cocultivated in milk with Lactobacillus delbrueckii subsp. bulgaricus:involvement of nitrogen, purine, and iron metabolism[J]. Appl Environ Microbiol, 2009, 75(7):2062-2073.
[41] Thevenard B, Rasoava N, Fourcassie P, et al. Characterization of Streptococcus thermophilus two-component systems:In silico analysis, functional analysis and expression of response regulator genes in pure or mixed culture with its yogurt partner, Lactobacillus delbrueckii subsp. bulgaricus[J]. Int J Food Microbiol, 2011,151(2):171-181.
(責任編輯 狄艷紅)
Research Progress on the Property and App lication of Streptococcus thermophilus
Tian Hui Liang Hongzhang Huo Guicheng Evivie Smich Ecareri
(Key Lab of Dairy Science of Ministry of Education,Northeast Agricultural University,Harbin150030)
Streptococcus thermophilus is one of the most important species of lactic acid bacteria in industry, and it is widely used in the process of producing fermented milk. This review discusses researches on the qualification and application of S. thermophilus, the properties of genome and fermentation, phage defense system, and symbiotic mechanism. In the end, the potential application of modern biological technologies in the study of S. thermophilus properties is prospected.
Streptococcus thermophilus;property;genome;fermentaion;phage;protocooperation
10.13560/j.cnki.biotech.bull.1985.2015.09.006
2014-11-14
國家“863”計劃項目(2011AA100902,2012AA022108)
田輝,男,博士研究生,研究方向:乳品科學與工程;E-mail:qingdaoyun08@163.com
霍貴成,男,教授,博士生導師,研究方向:食品科學;E-mail:gchuo58@126.com

