葡萄酒中葡萄的DNA提取和分子鑒定研究進展
高蘇娟,李志勇,高東微,劉 津,*
(1.仲愷農業工程學院輕工食品學院,廣東 廣州 510225;2.廣東檢驗檢疫技術中心,廣東 廣州 510623)
葡萄酒中葡萄的DNA提取和分子鑒定研究進展
高蘇娟1,李志勇2,高東微2,劉 津2,*
(1.仲愷農業工程學院輕工食品學院,廣東 廣州 510225;2.廣東檢驗檢疫技術中心,廣東 廣州 510623)
分子生物學技術能夠對葡萄酒真偽和品質鑒別提供直接的判別依據。解決葡萄酒中葡萄DNA提取的技術難題,并以此為基礎利用分子標記技術進行葡萄酒中葡萄品種的鑒定,具有重大的研究和應用價值。因此,本文綜述了近10余年來分子生物學技術在葡萄酒分子鑒定方面的研究進展,從葡萄酒中葡萄DNA提取、DNA質量評價和品種鑒定三方面進行了闡述,尤其是將文獻中的DNA提取和下游檢測技術方案進行了系統比較,就DNA提取部分按照葡萄DNA的富集、DNA粗提和多糖多酚的去除以及DNA純化回收3 個步驟進行了深入的分析,并詳細介紹了以微衛星標記為代表的品種鑒定技術現狀。在此基礎上,本文還探討了基因檢測技術在葡萄酒真偽和品質鑒定方面的應用前景。
葡萄酒;DNA提取;微衛星標記;品種鑒定
葡萄酒已然成為百姓餐桌上不可或缺的飲品,近兩年,我國進口葡萄酒總銷量達325 億元,增長超過70%[1],然而“以假充真”、“以次充好”等頻頻見諸報端的葡萄酒品質安全問題,不僅侵害到消費者的利益,也嚴重制約了葡萄酒行業的健康發展。
葡萄酒的價值與其品質密切相關,高品質的葡萄酒如莊園酒和特定產區的葡萄酒價值從一瓶幾百元到上萬元不等,而普通的餐酒許多只需幾十元,即使享譽世界的法國拉菲莊園出產的波爾多葡萄酒也有不同級別檔次,其定價也從兩三百元到萬余元不等,葡萄酒品質鑒定則能為有效規范葡萄酒銷售市場提供科學依據和技術支撐。實際上,葡萄酒的品質與葡萄品種、產地微氣候、釀造技術及其風味有密切關系,其中最基本的、也是最容易量化考核的即是釀酒所用的葡萄品種及比例。目前,基于化學分析的葡萄酒品質檢測技術已相對成熟,迄今蛋白質或氨基酸[2-5]、酚類物質[6]、礦物質[7]和芳香類物質[8]已用來分析和鑒定葡萄的品種或地理來源。多元醇、有機酸、多酚、單寧等多種葡萄酒中主要風味化學成分的分析技術已經建立并廣泛應用于葡萄酒行業[9-12],但葡萄酒中上述化學物質的存在與多寡易受土壤成分、溫度條件以及發酵和陳釀過程的影響,只能從側面、間接地反映葡萄酒的真偽和品質[13]。由于釀酒葡萄品種對于葡萄酒品質至關重要[14],基于基因水平的、能夠提供相對直觀鑒別依據的葡萄酒真偽和品質分析技術備受關注。隨著葡萄品種基因鑒別方法的逐步發展和漸成體系,其在葡萄酒真偽和品質鑒別方面的應用性研究已有突破性的進展。隨著葡萄酒中葡萄DNA提取技術的建立,以分子標記為代表的葡萄酒鑒別檢測技術也逐漸發展起來。本文對近10余年來葡萄酒中葡萄DNA的提取和分子鑒定的研究進展進行綜合評述,旨在為葡萄酒基因鑒偽技術的研究提供參考。
1 概 述
葡萄酒的分子鑒定包括葡萄酒中葡萄DNA的提取和下游分子生物學分析兩個部分,其中從葡萄酒中獲得高質量的葡萄DNA是進行下游分子生物學分析的基礎。植物基因組DNA提取方法已較為成熟,基于十六烷基三甲基溴化銨法(cetyltrimethyl ammonium bromide,CTAB)[15]和十二烷基硫酸鈉(sodium dodecyl sulfate,SDS)法[16]等傳統方法,適于不同類型植物材料的DNA提取方法[17-18]已經廣泛應用,甚至開發成為商品化的植物DNA提取試劑盒[18]。然而,這些DNA提取方法均不適用于葡萄酒中的葡萄DNA提取。葡萄酒為高度深加工產品,其生產過程歷經發酵、轉瓶、陳釀、澄清和過濾等諸多環節,成品葡萄酒中DNA含量極微,且由葡萄漿果引入葡萄酒中的大量的多酚類(單寧)、多糖、有機酸等復雜的化學物質,嚴重影響DNA提取質量及下游的分子生物學分析,目前尚未見報道一種能夠達到應用水平的葡萄酒DNA提取方法,也沒有成熟的商品化試劑盒。2000年至今,關于果汁、新酒或陳釀的葡萄酒中DNA提取及分析的研究報道[19-22]層出不窮。已有研究[13,23-27]表明:即使經過發酵,葡萄酒中依然存在微量的葡萄DNA,采用分子生物學手段進行用果汁或葡萄酒中的葡萄DNA分析是可行的;從葡萄酒中提取出足量且優質的葡萄DNA和篩選出葡萄特異性的聚合酶鏈式反應(polymerase chain reaction,PCR)引物是應用分子生物學手段鑒別葡萄酒中葡萄品種的前提,也是目前葡萄酒基因鑒偽技術研究的關鍵點和難點;在成功地提取出適宜進行下游分子生物學實驗的葡萄DNA后,可以進行葡萄特異性基因的檢測,以判斷是否用葡萄原汁釀造;可以進行微衛星位點擴增,通過與相關葡萄微衛星位點數據庫比對,從而鑒別出葡萄酒中的葡萄品種。
2 葡萄酒中葡萄DNA的提取
2.1釀造環節對葡萄酒中葡萄DNA的影響
研究表明,葡萄酒生產過程的發酵、轉瓶、陳釀、澄清和過濾等步驟都會減少葡萄酒中葡萄DNA的含量并破壞其質量。在葡萄酒發酵過程的不同階段取樣提取DNA的效果差別很大:在發酵進行的初期,雖然細胞被破壞、發酵的葡萄組織逐漸消融,但是提取的葡萄DNA卻有所上升;但是在發酵的中后期,DNA的質和量都急劇下降[19]。大多數研究者認為在進行澄清和過濾等步驟后,葡萄酒中的DNA含量會驟然減少[22],但是Drábek等[28]認為澄清和過濾的步驟對葡萄酒中DNA的含量影響不大,因為大多數葡萄DNA已經在澄清步驟之前損失,能夠存留的DNA則完全溶解于液相,直徑只有2 nm,能夠通過孔徑0.4 ?m的濾膜[12,28]。市售葡萄酒裝瓶的時間越久,從中提取出葡萄DNA的難度越大[13,19,25]。
Siret等[19-20]從發酵階段的葡萄酒中每天取樣并提取其DNA,發現在整個發酵過程中,利用從發酵罐里的固相中提取獲得的DNA能夠有效地進行微衛星序列擴增,而從液相中提取的DNA在進行微衛星序列擴增時重復性較差。對不同加工工藝的市售葡萄酒進行DNA提取和微衛星序列擴增,提取和擴增效果類似于發酵階段液相中的情況。Savazzini等[25]從發酵后陳釀一年的葡萄酒中提取出了葡萄DNA并進行了微衛星位點分析。Pereira等[13]從處于發酵階段的葡萄酒以及裝瓶后2~6 a的市售葡萄酒中均成功提取了葡萄DNA,并能成功地進行微衛星序列的擴增。
可見,從初始發酵的葡萄果汁至發酵各個階段乃至市售的陳年葡萄酒中均可能提取得到適于微衛星序列擴增的葡萄DNA,這是對葡萄酒進行葡萄基因鑒別檢測的物質基礎。只有解決了葡萄酒中葡萄DNA提取的難題,開展葡萄酒基因鑒偽技術研究才有探討的可能。
2.2葡萄酒DNA提取及純化
葡萄酒中葡萄DNA提取是葡萄酒基因鑒偽技術研究首要解決的問題,主要的難點有兩方面:即如何將微量DNA從相對大量的酒樣中富集,以及如何將DNA與葡萄酒中大量的多酚類、多糖等雜質分離。本文將目前已報道文獻中的葡萄酒中DNA提取方法按照DNA富集、DNA粗提、DNA純化回收3 個階段進行了分類匯總列于表1。目前已報道的葡萄酒或葡萄汁中DNA提取方法可分為三類,即以CTAB法為基礎進行改良的方法[13,19-22,25,28,31-32]、以SDS法為基礎改良的方法[26-27]和硅石(silica)吸附法[23],其中以CTAB法改良的報道居多。除了采用硅石吸附法[23]提得的DNA未進行下游的分子生物學實驗外,其余方法均用提取得到的DNA成功進行微衛星序列分析,少部分文獻中[25,27-28,31]還成功地進行了葡萄特異性基因的PCR或實時熒光定量PCR分析,表明基因水平進行葡萄酒品質分析鑒別盡管難度較大但并非不可行。表1中各種葡萄酒中DNA提取方法中采用的技術策略分析如下。
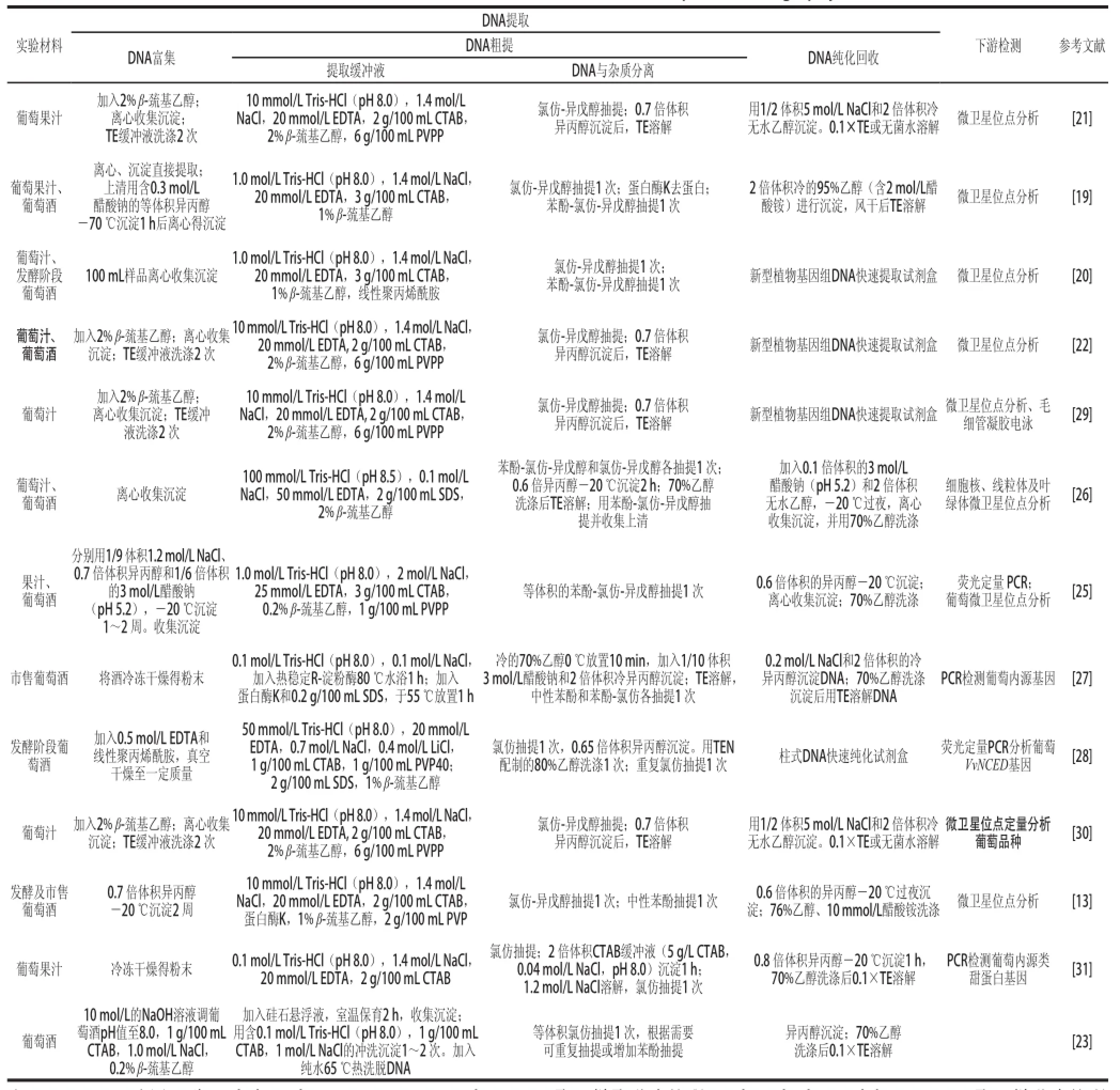
表1 葡萄果汁和葡萄酒中葡萄DNA提取和下游檢測技術方案Table 1 Summaries of DNA extraction and downstream detection protocols for grape juice and wines
2.2.1葡萄酒中DNA富集
葡萄酒中微量DNA片段的富集是DNA提取的第一個關鍵環節。由表1可知,4 ℃低溫離心取沉淀、真空冷凍干燥以及硅石吸附是目前富集葡萄酒中DNA的3 種有效方式。為了更充分富集溶于液相中分子質量較小的DNA片段,提高DNA的得率,Savazzini等[25]在原酒中分別加入1/9 體積1.2 mol/L NaCl、0.7 倍體積異丙醇或1/6 體積醋酸鈉并于-20 ℃放置1~2 周,然后進行DNA提取,結果表明3 種試劑均能顯著提高DNA得率,且異丙醇沉淀后提取的DNA得率稍遜于NaCl和醋酸鈉沉淀,但純度較高;Pereira等[13]比較了添加0.7 倍體積異丙醇-20 ℃放置24 h、48 h、1 周或2 周對DNA得率以及下游PCR效果的影響,認為-20 ℃放置2 周為最佳,對于紅葡萄酒尤其如此。Drábek等[28]在真空冷凍干燥市售葡萄酒時,加入0.5 mol/L的EDTA和線性聚丙烯酰胺,也獲得了可用于熒光定量PCR分析的DNA。
2.2.2DNA粗提以及多糖和多酚的去除
葡萄酒中富含源于葡萄漿果的多糖和多酚等物質[33],不僅干擾DNA的提取,也抑制下游的分子生物學檢測[27]。CTAB是一種去污劑,它既可以裂解細胞,又可以有效沉淀多糖,因此,在Tris-HCl-EDTA(pH 8.0)的弱堿性緩沖液中,1~3 g/100 mL的CTAB配合1.4 mol/L NaCl是普遍采用的葡萄酒中DNA提取緩沖液(表1),Zeng Jie等[34]認為對于多糖與次生物質含量較高的材料,DNA提取液中提高CTAB質量濃度至3 g/100 mL則效果更好。SDS法是經典的DNA提取方法,由于SDS不具有沉淀多糖的能力,較少用于多糖多酚含量較多的材料中DNA的提取[17],但經改良(加入2%(體積分數,下同)β-巰基乙醇)可以從葡萄酒和葡萄汁中提取獲得可用于微衛星位點分析的DNA[26],另有Nakamura等[27]將SDS法與耐熱R-淀粉酶和蛋白酶K合用,從市售葡萄酒中提取獲得了DNA,并成功進行葡萄特異性基因的PCR分析。
由表1可知,數種改良的CTAB法和SDS方法中,普遍添加了1%~2% β-巰基乙醇、PVP40或PVPP 1~6 g/100 mL。因為β-巰基乙醇為抗氧化劑,可避免酚類氧化;PVP或PVPP則能絡合多酚和萜類物質,可阻止多酚物質氧化為醌,兩者配合使用能有效防止多酚的污染[13,17]。經DNA提取緩沖液裂解得粗提DNA后,通常用苯酚-氯仿-異戊醇(25∶24∶1,V/V)抽提或者中性水飽和苯酚與氯仿-異戊醇(24∶1)分次抽提,以去除蛋白質和多糖等雜質。Pereira等[13]通過實驗表明,盡管酚抽提會增加DNA中酚污染的機率,但該環節依然是葡萄酒中DNA提取過程中不可或缺的,另外加入蛋白酶K以去除蛋白也是必要的。
2.2.3DNA純化回收
通常采用添加0.6~0.8 倍體積異丙醇或2 倍體積無水乙醇至DNA粗提液,并于低溫-20 ℃過夜沉淀的方式獲得相對較純的DNA,NaCl、醋酸銨以及醋酸鈉等鹽類物質能夠幫助達到更好的沉淀效果(表1);另外,商業化的DNA提取或純化試劑盒被用來進一步純化所提得的DNA,以滿足下游微衛星位點分析或熒光定量PCR實驗對DNA純度的需求[20,22,28-29]。
3 DNA質量評價
在優化葡萄酒中DNA提取條件的過程中,合適的DNA質量評價指標尤為重要。分子生物學實驗中通常采用分光光度法測定提取產物260 nm與280 nm波長處的吸光度,據此計算DNA的濃度并初步判定其純度。然而,從葡萄酒中提取的DNA,除了含有源于葡萄漿果的葡萄DNA,還有源于發酵過程中酵母等微生物的DNA,因此,分光光度法并不能判定提取的DNA是否含有葡萄內源DNA。采用微衛星序列分析、普通PCR或熒光定量PCR進行葡萄特異性基因分析則能夠直接判定所提得的DNA中是否含有葡萄特異性基因。表2列出了目前文獻中報道的可用于葡萄酒中葡萄特異性基因分析的PCR引物與探針信息,以供參考。

表2 葡萄內源基因PCR引物和探針Table 2 Primers and probes for PCR of endogenous genes in grape
4 葡萄品種的鑒定
葡萄酒的品質與葡萄品種、水土和釀造技術等密切相關,特別是高品質葡萄酒對釀酒采用的葡萄品種及其配比有著嚴格的要求。為了保障高級葡萄酒的品質,許多國家如法國、意大利和葡萄牙等都實行了原產地保護規定,只允許使用特定產區有限的少數幾個葡萄品種進行釀酒[14]。近年來,分子標記技術如限制性片段長度多態性(restriction fragment length polymorphism,RFLP)、隨機擴增多態性DNA(random amplified polymorphic DNA,RAPD)、擴增片段長度多態性(amplified fragment length polymorphism,AFLP)和微衛星標記(simple sequence repeats,SSR)等已經成功的應用于葡萄品種鑒定[35]。SSR標記技術利用SSR兩端的保守序列設計出一對特異性引物進行PCR擴增,將擴增產物通過凝膠電泳或毛細管電泳等手段檢測出擴增條帶的多態性,從而鑒別出不同的葡萄品種。SSR分子標記技術具有對DNA的濃度和純度要求較低,即便部分降解的DNA也能進行分析鑒定等特點,因此廣泛應用于葡萄酒中葡萄品種的鑒定。
Siret等[20]利用從發酵階段的葡萄酒中提取的DNA,用VVMD5、VVMD7、VVMD21、VVMD24和VVMD32這5 個微衛星位點進行檢測,能夠有效的鑒別單一品種發酵葡萄酒中的Chardonnay B、Clairette B、Syrah N和Grenache,對兩個葡萄品種混合發酵的葡萄酒能夠成功的鑒別出含量為30%的異源葡萄品種。Siret等[20]還提出對于葡萄酒這種深加工的產品,應該將檢測目標針對線粒體DNA(mitochondrial DNA,mtDNA)或葉綠體DNA(chloroplast DNA,cpDNA),因其含量相對于細胞核DNA更豐富,且線粒體和葉綠體DNA為環形,對熱處理等具有較高的抗性。Savazzini等[25]能夠成功地從發酵后超過1 a的葡萄酒中擴增出微衛星序列,但用Genotyper軟件分析發現擴增得到的微衛星序列與不同葡萄品種葉片的微衛星序列仍存在差異,認為可能與低DNA濃度和高多酚、多糖和其他PCR抑制劑有關;Baleiras-Couto等[26]應用6 種細胞核SSR和2 種葉綠體SSR微衛星標記對5 種單一品種發酵和2 種雙品種混合發酵葡萄酒進行了分析,發現在雙品種發酵的葡萄酒中某一葡萄品種比例越高則擴增信號越強,且葉綠體SSR比細胞核SSR更適用于葡萄酒的檢測。García-Beneytez等[22]在一個PCR反應體系中同時擴增VVS5、VVMD7、VVS2、VVMD5、ZAG47、ZAG62、ZAG79和VVS29共7 個微衛星序列,并成功地鑒別了單一品種發酵的葡萄酒中葡萄品種,該方法依據上述微衛星位點擴增產物片段大小差異或PCR時標記的熒光不同,可以在同一反應體系中同時進行不同微衛星位點的檢測,從而有效地節省DNA、時間和金錢,在微衛星標記鑒別未知樣品中存在的多種葡萄品種方面也具有較好的應用潛力。
獲取SSR分子標記的相關信息可以通過查閱文獻,也可以通過訪問SSR分子標記數據庫,主要的公開的葡萄微衛星標記數據庫見表3。瑞士葡萄SSR分子標記數據庫(Swiss Vitis Microsatellite Database)截至2014年6月收錄了171 份本國葡萄品種的SSR信息,該數據庫中利用6 個SSR標記(VVMD5、VVMD7、VVMD27、VVS2、VrZAG62、VrZAG79),區分了171 份葡萄品種。意大利葡萄SSR分子標記數據庫(Grape Microsatellite Collection,GMC)能通過葡萄品種名稱、微衛星位點和名稱首字母進行檢索,檢索的結果包括微衛星位點、等位基因1和2的長度、品種名稱、作者、引用文獻和片段分析方法這7 項內容。

表3 葡萄微衛星標記數據庫Table 3 Databases of grapevine microsatellite markers
3 結 語
隨著人們對葡萄酒文化的逐漸熟悉和鐘愛,我國進口葡萄酒消費市場急速膨脹。令人不齒的是,市場上出現了“以假亂真”、“以次充好”、甚至“葡萄酒里不含葡萄原汁”等種種亂象,深刻影響了葡萄酒文化的宣傳推廣,極大打擊了葡萄酒消費市場的健康發展,嚴重侵害了消費者的正當權益。面對目前葡萄酒魚龍混雜的局面,研究并建立葡萄酒真偽和品質鑒別檢驗技術能夠為葡萄酒市場的科學監管提供有效的技術武器。
高品質葡萄酒對葡萄品種及比例均有具體的要求,通過基因檢測技術可以有效的鑒別出葡萄酒中所含有的葡萄品種及其比例,從而能夠為鑒別葡萄酒的真偽和品質提供一種直接判定的依據。經過諸多研究者的共同努力,葡萄酒中葡萄的DNA提取和分子鑒定已取得喜人進展,主要表現為:1)果汁和發酵階段葡萄酒中葡萄DNA提取以及SSR技術鑒定部分葡萄品種的方法已基本確立(表1);2)能夠從市售葡萄酒中提取獲得葡萄的DNA,并在品種鑒定方面進行了有益嘗試。
同時,從已有的研究報道中也凸顯出實現從基因水平簡便、快速、直接地對葡萄酒品質進行鑒別仍有不少技術難題亟待解決:1)目前尚無成熟穩定的葡萄酒DNA提取方法體系建立,尤其是市售和紅葡萄酒中高質量DNA提取仍是發展葡萄酒基因鑒別技術的關鍵及限速環節。在該環節,可以從以下方面進行有益的嘗試,諸如通過溫度梯度控制或加入糖原等助沉劑以縮短DNA富集(沉淀)時間;選用磁珠和特異性吸附膜等方法進行DNA的純化,以避免常規純化過程中大量使用苯酚、氯仿等有機試劑造成的污染;針對不同發酵階段、白葡萄酒或紅葡萄酒DNA提取的共性難點開發相應的DNA提取試劑盒等方面以優化DNA的提取。2)葡萄酒中葡萄DNA品種鑒別依舊困難重重,其主要限制性體現在難以獲得葡萄品種特異性的基因序列,且各品種微衛星位點擴增產物差異較小,某些品種間產物大小差異僅為幾個堿基對,甚至僅是個別堿基的不同,即使借助于分辨率相對較高的毛細管電泳或芯片凝膠電泳也難以鑒別,而將擴增產物測序,通過序列分析確定品種則不失為更可行的方法。因此,建立并共享釀酒葡萄微衛星序列數據庫勢在必行。
綜上所述,基因檢測技術將在葡萄酒的真偽和品質鑒別檢測方面發揮越來越重要的作用,隨著研究方法和技術的日新月異,從基因水平簡便、快速、直接地判別葡萄酒是否由葡萄原汁釀制,以及由何種釀制, 建立葡萄酒“指紋”體系指日可待。
[1] 張亮. 進口葡萄酒: 繁榮之下難掩“泡沫”[J]. 中國檢驗檢疫, 2014(2):46-47.
[2] GONZALEZ-LARA R, CORREA I, POLO M C, et al. Classification of variety musts by statistical analysis of their electrophoretic protein pattern[J]. Food and Chemical Toxicology, 1989, 34: 103-110.
[3] PUEYO E, POLO M C, DIZY M. Varietal differentiation of musts and wines by means of protein fraction[J]. American Journal of Enology and Viticulture, 1993, 44: 255-260.
[4] MORENO-ARRIBAS M V, CABELLO F, POLO M C, et al. Assessment of the native electrophoretic analysis of total grape must proteins for the characterization of Vitis vinifera L. cultivars[J]. Journal of Agricultural and Food Chemistry, 1999, 47(1): 114-120.
[5] VASCONCELLOS A M P, CHAVES das NEVES H J. Characterization of elementary wines of Vitis vinifera varieties by pattern recognition of three amino acids profiles[J]. Journal of Agricultural and Food Chemistry, 1989, 37: 931-937.
[6] CABELLO F, REVILLA E, GARCIA-BENEYTEZ E, et al. Value of high-performance liquid chromatographic analysis of anthocyanins in the differentiation of red grape cultivars and red wines made from them[J]. Journal of Chromatography A, 2001, 915(1/2): 53-60.
[7] DAY M P, ZHANG B L, MARTIN G J. The use of trace element data to c omplement stable-isotope methods in the characterization of grape musts[J]. American Journal of Enology and Viticulture, 1994, 45: 79-85.
[8] GONZAL EZ-BARREIRO C, RIAL-OTER O R, CANCHO-GRANDE B,e t al. Wine aroma compounds in grapes: a critical review[J]. Critical Reviews in Food Science and Nutrition, 2015, 55(2): 202-218.
[9] COETZEE P P, VANHAECKE F. Classifying wine according to geographical origin via quadrupole-based ICP-mass spectrometry measurements of boron isotope ratios[J]. Analytical and Bioanalytical Chemistry, 2005, 383(6): 977-984.
[10] MONACI F, BARGAGLI R, FOCARDI S. Element concentrations in Chianti Classico appellation wines[J]. Journa l of Trace Elements in Medicine and Biology, 2003, 17(Suppl 1): 45-50.
[11] GARCIA-BENEYTEZ E, CABELLO F, REVILLA E. Analysis of grape a nd wine anthocyanins by HPLC-MS[J]. Journal of Agricultural and Food Chemistry, 2003, 51(19): 5622-5629.
[12] ALMEIDA C M, VASC ONCELOS M T. Multielement composition of wines and their precursors including provenance soil and their potentialities as fingerprints of wine origin[J]. Journal of Agricultural and Food Chemistry, 2003, 51(16): 4788-4798.
[13] PEREIRA L, GUEDES-PINTO H, MARTINS-LOPES P. An enhanced method for Vitis vinifera L. DNA extraction from wines[J]. American Journal of Enology and Viticulture, 2011, 62(4): 547-552.
[14] 崔熏, 李海英, 吳少惠. 與葡萄酒的相遇[M]. 濟南: 山東人民出版社, 2009: 92-98.
[15] DOYLE J J, DOYLE J L. A rapid DNA isolation procedure for small quantities of fresh leaf tissue[J]. Phytochemical Bulletin, 1987, 19: 11-15.
[16] 蔡朝輝, 李萍, 董婷霞 , 等. 貝母的分子生物學鑒定方法的研究[J].藥學學報, 2000, 35(4): 309-312.
[17] 孫璐宏, 魯周民, 張麗. 植物基因組DNA提取與純化研究進展[J].西北林學院學報, 2010, 25(6): 102-106.
[18] 李金璐, 王碩, 于婧, 等. 一種改良的植物DNA提取方法[J]. 植物學報, 2013, 48(1): 7 2-78.
[19] SIRET R, BOURSIQUOT J M, MERLE M H, et al. Toward the authentication of varietal wines by the analysis of grape (Vitis vinifera L.) residual DNA in must and wi ne using microsatellite markers[J]. Journal of Agricultural and Food Chemistry, 2000, 48(10): 5035-5040.
[20] SIRET R, GIGAUD O, ROSEC J P, et al. Analysis of grape Vitis vinifera L. DNA in must mixtures and experimental mixed wines using micros atellite markers[J]. Journal of Agricultural and Food Chemistry,2002, 50(13): 3822-3827.
[21] FARIA M A, MAGALHAES R, FERREIRA M A, et al. Vitis vinifera must varietal authentication using microsatellite DNA analysis (SSR)[J]. Journal of Agricultural and Food Che mistry, 2000, 48(4): 1096-1100.
[22] GARC?A-BENEYTEZ E, MORENO-ARRIBAS M V, BORREGO J,et al. Application of a DNA analysis method for the cultivar identification of grape musts and experimental and commercial wines of Vitis vinifera L. using microsatellite markers[J]. Journal of Agricultural and Food Chemistry, 2002, 50(21): 6090-6096.
[23] 葉永忠, 李春奇, 王偉. 葡萄酒中DNA的提取方法: 中國,200610128466.5[P]. 2008-07-02.
[24] STUMMER B E, ZANKER T, HARVEY P R, et al. Detection and quantification of Erysiphe necator DNA in wine grapes and resultant must and juice[J]. Mycological Research, 2006, 110(Pt 10): 1184-1192.
[25] SAVAZZINI F, MARTINELLI L. DNA analysis in wines:development of methods for enhanced extr action and real-time polymerase chain reaction quantification[J]. Analytica Chimica Acta,2006, 563(1/2): 274-282.
[26] BALEIRAS-COUTO M M, EIRAS-DIAS J E. Detection and identification of grape varieties in must and wine using nuc lear and chloroplast microsatellite markers[J]. Analytica Chimica Acta, 2006,563: 583-591.
[27] NAKAMURA S, HARAGUCHI K, MITANI N, et al. Novel preparation method of template DNAs from wine for PCR to diff erentiate grape (Vitis vinifera L.) cultivar[J]. Journal of Agricultural and Food Chemistry, 2007, 55(25): 10388-10395.
[28] DR?BEK J, ST?VEK J, JAL?VKOVá M, et al. Quantification of DNA during winemaking by florimetry and Vitis vinifera L.-specific quantitative PCR[J]. European Food Research and Technology, 2008,226: 491-497.
[29] RODRíGUEZ-PLAZA P, GONZáLEZ R, MORENO-ARRIBAS M V, et al. Co mbining microsatellite markers and capillary gel electrophoresis with laser-induced fluorescence to identify the grape(Vitis vinifera) variety of musts[J]. European Food Research and Technology, 2006, 223: 625-631.
[30] FARIA M A, NUNES E, OLIVEIRA M B P P. Relative quantification of Vitis vinifera L. varieties in musts by microsatellite DNA analysis[J]. European Food Research and Technology, 2008, 2 27: 845-850.
[31] HAN Jianxun, WU Yajun, HUANG Wensheng, et al. PCR and DHPLC methods used to detect juice ingredient from 7 fruits[J]. Food Control, 2012, 25: 696-703.
[32] ENGLEN M D, KELLEY L C. A rapid DNA is olation procedure for the identification of Campylobacter jejuni by the polymerase chain reaction[J]. Letters in Applied Microbiology, 2000, 31(6): 421-426.
[33] CABANIS J C, CABANIS M T, CHEYNIER V, et al. Tables de composition[M]//F LANZY C. Oenologie: fondements scientifiques et technologiques. Paris: Techniques et Documentation, 1999: 315-336.
[34] ZENG Jie, ZOU Yuping, BAI Jiayu, et al. Preparation of total DNA from recalcitrant plant taxa[J]. Acta Botanica Sinica, 2002, 44(6): 694-697.
[35] ROUBELAKIS-ANGELAKIS K A. Grapevine molecular physiology and biotechnology[M]. 2nd ed. Netherlands: Kluwer Academic Publication, 2009: 565-586.
A Review on Grape (Vitis vinifera L.) DNA Extraction and Molecular Identification for Wines
GAO Sujuan1, LI Zhiyong2, GAO Dongwei2, LIU Jin2,*
(1. College of Food Science and Technology, Zhongkai University of Agriculture and Engineering, Guangzhou 510225, China;2. Guangdong Inspection and Quarantine Technology Center, Guangzhou 510623, China)
Molecular biology techniques can provide direct authenticity and quality identification of wines. The resolution of technical problems on grape DNA extraction from wines and the subsequent application of molecular marker techniques in grapevine identification are of great interest among researchers and of high practical value. A literature review on molecular biology techniques used for grapevine identification for wines in recent decades is given in this paper, including grape DNA extraction from wines, DNA quality evaluation and grapevine identification. A systematic comparison of DNA extraction and downstream detection protocols reported in the literature is provided as well as a detailed analysis of the three steps involved in DNA extraction namely DNA enrichment, crude DNA extraction and subsequent removal of polyphenol and polysaccharide, and DNA purification. The technical advances in grapevine identification represented by microsatellite marker technology are displayed. Meanwhile, the application prospects of gene detection technologies in authenticity and quality identification of wines are discussed in this paper.
wine; DNA extraction; microsatellite marker; grapevine identification
TS255.7
A
1002-6630(2015)15-0282-06
10.7506/spkx1002-6630-201515052
2014-08-08
國家質檢總局科技計劃項目(2014IK102);廣東出入境檢驗檢疫局科技計劃項目(2013GDK24);仲愷農業工程學院大學生創新基金項目(2014A17)
高蘇娟(1981—),女,講師,博士,研究方向為生物工程。E-mail:gaoshj@126.com
劉津(1983—),女,工程師,碩士,研究方向為食品檢測。E-mail:ivfhonline@126.com

