去除紫花苜蓿葉片高豐度蛋白的方法及其應用
陳晶,韓貴清,*,尚晨,張海玲,李佶愷,劉慧瑩,張月學
(1.哈爾濱師范大學生命科學與技術學院,黑龍江 哈爾濱150025;2.黑龍江省農業科學院草業研究所,黑龍江 哈爾濱150080)
去除紫花苜蓿葉片高豐度蛋白的方法及其應用
陳晶1,韓貴清1,2*,尚晨2,張海玲2,李佶愷2,劉慧瑩2,張月學2
(1.哈爾濱師范大學生命科學與技術學院,黑龍江 哈爾濱150025;2.黑龍江省農業科學院草業研究所,黑龍江 哈爾濱150080)
摘要:紫花苜蓿葉片中大量的高豐度蛋白(核酮糖-l,5-二磷酸羧化/加氧酶,Rubisco)干擾了蛋白質的動態分辨率,嚴重影響蛋白質組學研究中功能蛋白的檢測與鑒定。為了探究去除高豐度蛋白的適宜方法,本研究利用Mg/NP-40與聚乙二醇(PEG)預分離紫花苜蓿葉片蛋白,通過雙向凝膠電泳法比較了不同濃度PEG對葉片高豐度蛋白的分離情況。電泳圖譜顯示0,15%,17.5%,20%PEG處理的蛋白質中分別可以檢測到(335±17),(417±3),(445±7),(459±11)個蛋白質點,0,15%,17.5%處理組間差異顯著(P<0.05),17.5%和20%PEG 處理組間沒有差異(P<0.05)。然而,17.5%PEG能夠檢測到更多的差異蛋白質點,證明其更能有效沉淀高豐度蛋白,便于檢測被Rubisco遮蓋的蛋白質點。將該方法應用于紫花苜蓿葉片響應低溫脅迫的蛋白質組學研究中檢驗其應用效果,與三氯乙酸/丙酮法提取的全蛋白相比,去除高豐度蛋白后鑒定出8個新的蛋白質差異點,證明該方法適用于實際的蛋白質組學研究。可見,Mg/NP-40與17.5%PEG法是最適宜去除紫花苜蓿葉片高豐度蛋白的方法。
關鍵詞:高豐度蛋白;核酮糖-l,5-二磷酸羧化/加氧酶;聚乙二醇;紫花苜蓿;蛋白質組學
DOI:10.11686/cyxb2015044
Chen J, Han G Q, Shan C, Zhang H L, Li J K, Liu H Y, Zhang Y X. Proteomic methods for removing high-abundance proteins in alfalfa leaf. Acta Prataculturae Sinica, 2015, 24(7): 131-138.
陳晶,韓貴清,尚晨,張海玲,李佶愷,劉慧瑩,張月學. 去除紫花苜蓿葉片高豐度蛋白的方法及其應用. 草業學報, 2015, 24(7): 131-138.
http://cyxb.lzu.edu.cn
收稿日期:2015-01-21;改回日期:2015-04-09
基金項目:黑龍江省博士后特殊基金項目(LBH-TZ1209)和“十二五”支撐項目(2011BAD17B04-2)資助。
作者簡介:陳晶(1984-),女,黑龍江哈爾濱人,在讀博士。E-mail:ccyj15@163.com
通訊作者*Corresponding author. E-mail:ccyj200@163.com
Abstract:The higher content of high-abundance proteins (Ribulose-l, 5-bisphosphate carboxylase/oxygenase; RuBisCo) interferes with the dynamic resolution of proteins in two-dimensional electrophoresis (2-DE), affecting the detection and identification of functional proteins in proteomics. To explore suitable methods for removing high-abundance protein, Mg/NP-40 and different concentrations of polyethylene glycol (PEG) were used for protein pre-fractionation and the treatments’ influence on the separation of proteins in alfalfa leaf was compared. The results indicated that (335±17), (417±3), (445±7) and (459±11) spots were detected in treatments of 0, 15%, 17.5% and 20% PEG respectively. There were significant differences between the 0-17.5% treatments but no significant differences were found between the 17.5% and 20% treatments. More differential protein spots were detected in the 17.5% treatment, which thus proved the most effective way of removing RuBisCo proteins. In order to inspect the applicability of this result, a proteomic study of alfalfa in response to low temperatures was under taken. Compared with total proteins extracted by TCA/acetone, eight new protein spots were identified after PEG treatment to remove high-abundance proteins. The research thus indicates that pre-fractionation with Mg/NP-40 and17.5% PEG is suitable for proteomic studies of alfalfa.
Proteomic methods for removing high-abundance proteins in alfalfa leaf
CHEN Jing1, HAN Gui-Qing1,2*, SHAN Chen2, ZHAN Hai-Ling2, LI Ji-Kai2, LIU Hui-Ying2, ZHANG Yue-Xue2
1.CollegeofLifeSciencesandTechnology,HarbinNormalUniversity,Harbin150025,China; 2.InstituteofGrassResearch,HeilongjiangAcademyofAgricultureSciences,Harbin150080,China
Key words: high-abundance protein; Rubisco; polyethylene glycol; alfalfa; proteomics
蛋白質組學是21世紀的前沿熱點領域之一。近十年來,其從高度專業化轉變為常規的技術手段應用于植物學研究[1]。大量蛋白質數據的挖掘在植物基因功能分析和特殊蛋白質鑒定上發揮了重要作用,也在一定程度上為基因組和代謝組間構建了“溝通”橋梁[2]。紫花苜蓿(Medicagosativa)是多年生豆科牧草,其豐富的蛋白質含量及優良的適應性為草業和畜牧業的可持續發展提供了有利條件[3]。對其非生物性(低溫、鹽堿、干旱、藥害等)逆境因子應答反應和抗、耐性機理的蛋白質組學研究具有重要意義。但紫花苜蓿葉片中大量高豐度蛋白[如,核酮糖-l,5-二磷酸羧化/加氧酶(Rubisco)]的存在降低了蛋白質的動態分辨率,極大地影響了逆境應答和代謝過程中功能蛋白的分析與鑒定。
Rubisco作為一種“超大量”蛋白,普遍存在于綠色植物中。在對擬南芥(Arabidopsisthaliana)和水稻(Oryzasativa)葉片的蛋白質組學研究中發現,其高豐度蛋白Rubisco及其變體分別占蛋白總量的12%和35.3%,極易遮蓋鄰近蛋白,使一些低豐度蛋白無法被識別[4-5]。近年來,國內外科學家一直致力于尋求提高低豐度蛋白分辨率的方法。Cellar 等[6]利用Rubisco免疫耗竭柱去除了葉片組織中90%~98%的Rubisco,增強了低豐度蛋白的檢測和鑒定;但該方法復雜、耗時且費用較高。Cho 等[7]發現高濃度二硫蘇糖醇(dithiothretiol, DTT)可以有效去除水稻葉片中的Rubisco酶,提高雙向凝膠電泳的分辨率;但DTT 沉淀法原理及其在其他作物中的有效性還需要進一步研究和檢驗[8]。另外,也有研究發現肌醇六磷酸鈉結合鈣離子法可以去除植物葉片中的大部分Rubisco酶,其在不同植物中有效性及適宜的反應溫度也需要檢驗和篩選[8-9]。PEG是一種直鏈大分子聚合物,能夠破壞蛋白質分子表面的水化層而使蛋白發生沉淀作用,最早應用于一些細菌和病毒蛋白的提純[10-11]。Kim等[12]在2001年采用聚乙二醇(PEG)沉淀法預分離了水稻葉片蛋白,去除了大部分Rubisco。此后該方法又被成功應用于擬南芥[13]、水稻[14-15]和甜瓜(Cucumis)[16]的蛋白質組學研究中,有效提高了低豐度蛋白的分辨率,也是目前應用最多的去除植物高豐度蛋白的方法。與其他方法相比,PEG沉淀法更加快捷、簡單、費用低廉且具有較廣的適用性。到目前為止,尚沒有探究紫花苜蓿高豐度蛋白去除方法的相關報道。因此,本試驗在前人的基礎上,利用不同濃度的PEG預分離紫花苜蓿葉片蛋白,旨在篩選去除高豐度蛋白的適宜方法;并將該方法應用于紫花苜蓿響應低溫的蛋白質組學研究中,檢測其可應用性。
1材料與方法
1.1 材料
紫花苜蓿WL525HQ種子由黑龍江省農業科學院草業研究所提供。2014年,紫花苜蓿種子播種于混有草炭土∶蛭石=2∶1的塑料花盆中,置于晝/夜溫度 25℃/20℃,光周期 16 h/8 h,濕度約為80%,光照強度 12000 lx的人工氣候箱中。培養至60 d苗齡,移至溫度 4℃,其他條件相同的恒溫箱中處理12 h。于低溫處理0和12 h時分別剪取紫花苜蓿葉片若干,液氮速凍,-80℃保存備用。
1.2 高豐度蛋白的去除
1.2.1蛋白質提取取凍存葉片(低溫4℃處理0 h)2 g,于液氮中研磨成粉末后加入8 mL 預冷的Mg/NP-40蛋白提取液[0.5 mol/L pH 8.3 Tris-HCl,2%(v/v) NP-40,0.02 mol/L 氯化鎂,0.001 mmol/L 乙二胺四乙酸(ethylene diamine tetraacetic acid,EDTA),0.02 mol/L DTT,0.001 mol/L苯甲基磺酰氟(phenylmethanesulfonyl fluoride,PMSF)][5],在冰上研磨10 min。4℃,12000 r/min離心30 min后取上清液,棄沉淀。上清液分為4組,分別加入適量50%(w/v)PEG4000至終濃度為 0,15%,17.5%,20%,充分混勻后冰浴30 min。以PEG終濃度 0為對照(Control,CK),其余3組4℃,12000 r/min離心1 h,分別取上清記為S1、S2、S3,相對應沉淀記為P1、P2、P3,加入3倍體積量-20℃預冷的10% (w/v)三氯乙酸/丙酮,混勻,-20℃過夜。4℃,12000 r/min離心1 h,棄上清,沉淀中加入3倍體積量-20℃預冷丙酮,重懸后置于-20℃ 1 h。 4℃,12000 r/min離心30 min,棄上清,沉淀凍干成粉末。
1.2.2聚丙烯酰胺凝膠(SDS-PAGE)電泳檢測蛋白質干粉中加入裂解液[7 mol/L尿素,2 mol/L硫脲,4%(w/v) 3-[3-(膽酰胺丙基)二甲氨基]丙磺酸內鹽(3-[(3-cholamidopropyl) dimethylammonio] propanesulfonate,CHAPS),0.04 mol/L DTT],振蕩均勻后置于搖床上持續振蕩1 h,使蛋白完全溶解。2D-Quant 試劑盒進行蛋白質定量,留取相應體積蛋白溶液,其余-80℃冰箱凍存。配制12%分離膠和5%濃縮膠用于SDS-PAGE電泳檢測。電泳結束后,考馬斯亮藍R-250染色,乙醇冰乙酸脫色至條帶清晰。白光掃描儀掃描凝膠,分析蛋白質分離情況。
1.2.3雙向電泳檢測選用13 cm IPG(pH 4~7)膠條,蛋白質上樣量為650 μg,加入水化液[8 mol/L尿素,2%(w/v)CHAPS,0.3%(w/v)DTT,1%(v/v)IPG buffer]稀釋至250 μL。第一向等電聚焦程序為:30 V,12 h;200 V,1 h;500 V,1 h;1000 V,2 h;8000 V,3 h;8000 V,45000 Vh;1000 V,任意。第二向12.5%聚丙烯酰胺凝膠電泳程序為:10 mA/膠條,0.5 h;40 mA/膠條,2.5~4 h。雙向電泳后,考馬斯亮藍R-250染色,10%(v/v)冰醋酸脫色后,白光掃描儀掃描凝膠,使用ImageMaster 2D Platinum凝膠分析軟件進行比對分析。
1.3 PEG沉淀法的應用性驗證
1.3.1蛋白質提取葉片全蛋白提取用傳統方法三氯乙酸/丙酮提取法。取凍存葉片(低溫4℃處理0,12 h)各0.5 g置于預冷的研缽中,加入液氮充分研磨后轉移至50 mL離心管中并加入三倍體積-20℃預冷的蛋白提取液[含10%(w/v)三氯乙酸、0.07%(v/v)β-巰基乙醇、1%(v/v)蛋白酶抑制劑的丙酮溶液],-20℃沉淀過夜。4℃,12000 r/min離心1 h,棄去上清,保留沉淀。用三倍于沉淀體積的含有0.07%(v/v)的β-巰基乙醇的冷丙酮將沉淀重懸,-20℃沉淀1 h以上。4℃,12000 r/min離心1 h,棄去上清,保留沉淀。將沉淀凍干成粉末,-80℃保存備用。
低豐度蛋白提取用Mg/NP-40+PEG法,PEG濃度為17.5% (詳見1.2.1)。
1.3.2雙向電泳選用24 cm IPG(pH 4~7)膠條,蛋白質上樣量為1000 μg,上樣體積為450 μL。第一向等電聚焦程序為:30 V,12 h;200 V,2 h;500 V,2 h;1000 V,2 h;8000 V,3 h;8000 V,65000 Vh;1000 V,任意。第二向12.5%聚丙烯酰胺凝膠電泳程序為:1 W/膠條,1 h;13 W/膠條,3.5~4 h。雙向電泳后,考馬斯亮藍R-250染色,10%(v/v)冰醋酸脫色后,白光掃描儀掃描凝膠,使用ImageMaster 2D Platinum凝膠分析軟件進行比對分析。
1.3.3差異蛋白質點的鑒定篩選出的差異點通過MADLI_TOF/TOF進行鑒定,所得結果利用MASCOT軟件搜索NCBIMedicago、NCBI Viridiplantae 和Uniprot數據庫。
1.4 統計分析
本試驗中,所有處理3次重復,相關數據通過軟件DPS進行差異顯著度分析。
2結果與分析
2.1 不同濃度PEG處理下SDS-PAGE電泳分析結果
為探究不同濃度PEG對紫花苜蓿葉片高豐度蛋白的分離情況,對各處理下蛋白質的沉淀和上清部分進行了SDS-PAGE檢測。圖1是以PEG濃度0%為對照組(control),不同濃度PEG處理蛋白樣品后沉淀和上清部分的SDS-PAGE電泳分析對比圖。其中P1~P3與S1~S3分別對應PEG濃度15%,17.5%,20% 蛋白樣品的沉淀和上清部分。圖中對照組高豐度蛋白條帶清晰,聚集在55 kD左右處。而PEG處理后,沉淀樣P1~P3可見清晰的高豐度蛋白條帶,并且Rubisco分散為55 kD左右大亞基(large subunit of Rubisco, LSR)和14.4 kD左右小亞基(small subunit of Rubisco, SSR);上清樣S1~S3高豐度蛋白條帶減弱,其中S2和S3中高豐度蛋白下降更明顯,低豐度蛋白條帶清晰穩定。可見,PEG可以沉淀富集紫花苜蓿葉片中的Rubisco,17.5%,20%的PEG均可用于去除高豐度。
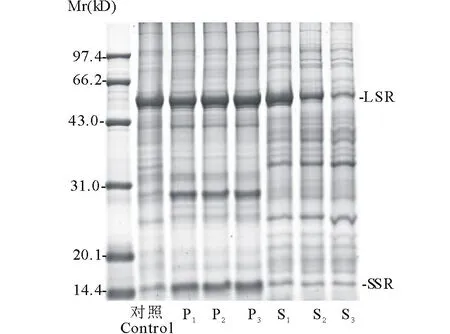
圖1 不同濃度PEG處理下蛋白質樣品SDS-PAGE電泳圖Fig.1 SDS-PAGE analysis of proteins from the PEG fractionation P1~P3為PEG濃度15%,17.5%,20% 蛋白樣品的沉淀部分;S1~S3為PEG濃度15%,17.5%,20% 蛋白樣品的上清部分。 P1-P3 stand for protein precipitation of treatment with 15%,17.5% and 20% PEG, respectively. S1-S3 stand for protein supernate of treatment with 15%,17.5% and 20% PEG, respectively.
2.2 不同濃度PEG處理下雙向電泳圖像分析結果
圖2顯示不同濃度PEG處理下蛋白質樣品上清部分的雙向電泳圖像。圖中紅框內為高豐度蛋白點,用3D圖像顯示豐度。對照和S1可見大量高豐度蛋白點,S2和S3中高豐度蛋白明顯減少。而隨著PEG濃度的升高可檢測出的蛋白質點數目增多(表1),與對照組重疊的點數減少,17.5%和20%的PEG處理均可獲得較多蛋白質點,且二者沒有顯著差異(P<0.05);與對照組相比差異點數變化較大,差異率分別為(33.33±0.72)%, (56.62±0.22)%,(52.28±0.72)%,差異顯著(P<0.05)。可見,17.5%和20%的PEG均可用于提取低豐度蛋白,與SDS-PAGE分析結果一致,但17.5%PEG處理下可檢測出更多差異點(表1)。在圖2的3D圖像中,S2的蛋白質點形成相對分離的單峰圖像,可觀察到清晰的蛋白點和豐度;S3則蛋白損失過多,呈現細小尖峰圖像。可見,17.5% PEG更適宜于紫花苜蓿的高豐度蛋白的去除。
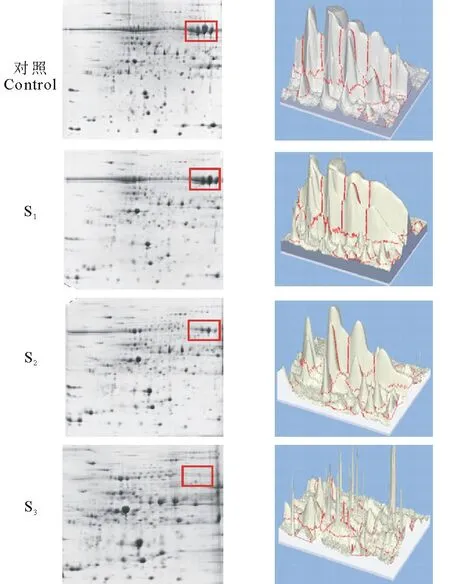
圖2 不同濃度PEG處理下2-DE對比圖像Fig.2 Comparation of 2-DE gels from the PEG fractionation 右側圖像為雙向電泳圖中紅框內蛋白質點的3D圖像3D images on the right showing protein spots in the red box of 2-DE gels.
2.3 PEG沉淀法在響應低溫蛋白質組學研究中的應用
為了驗證PEG沉淀法在紫花苜蓿響應低溫蛋白質組學研究中的可應用性,紫花苜蓿葉片的蛋白分別用三氯乙酸/丙酮法和17.5% PEG沉淀法進行提取,雙向電泳后經ImageMaster 2D Platinum軟件分析確定響應低溫的差異蛋白質點。圖3為4℃處理12 h下紫花苜蓿葉片的雙向電泳圖譜(A為三氯乙酸/丙酮法,B為17.5% PEG沉淀法),數字分別標注了全蛋白和低豐度蛋白響應低溫的差異蛋白質點。與未處理的對照組相比,經低溫后紫花苜蓿葉片蛋白(A)中有14個點蛋白質豐度變化在1.5倍以上(圖3),而PEG去除高豐度蛋白Rubisco后,低豐度蛋白(B)中發現8個新的差異點(圖3)。經MADLI_TOF/TOF鑒定,所得結果利用MASCOT軟件搜索相關數據庫確定其蛋白種類“L”表示低豐度蛋白。“L” represents low-abundance protein.
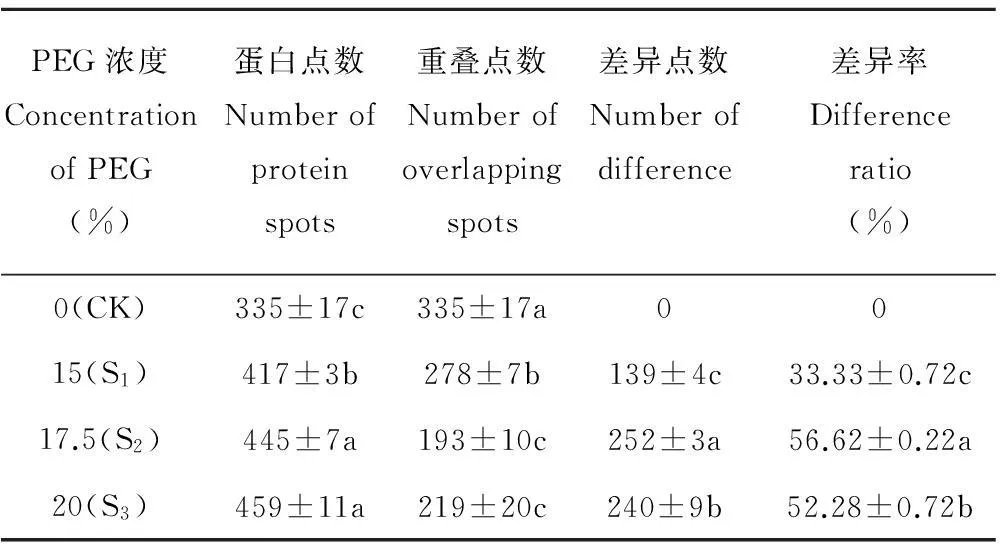
表1 不同處理下2-DE圖像蛋白點統計分析
注:重疊點數為PEG處理后上清液與對照組相重疊點數。同列不同小寫字母表示差異顯著(P<0.05)。
Note: Overlapping spots represent spots of supernatant from PEG fractionation overlapping with spots of control.The different letters in the same column mean significant differences atP<0.05.

圖3 紫花苜蓿葉片響應低溫的全蛋白(A)和低豐度蛋白(B)雙向電泳圖Fig.3 2-DE gels of holoprotein (A) and low-abundance protein (B) in response to low temperature in alfalfa數字表示差異點編號 Digits represent spots numbers.
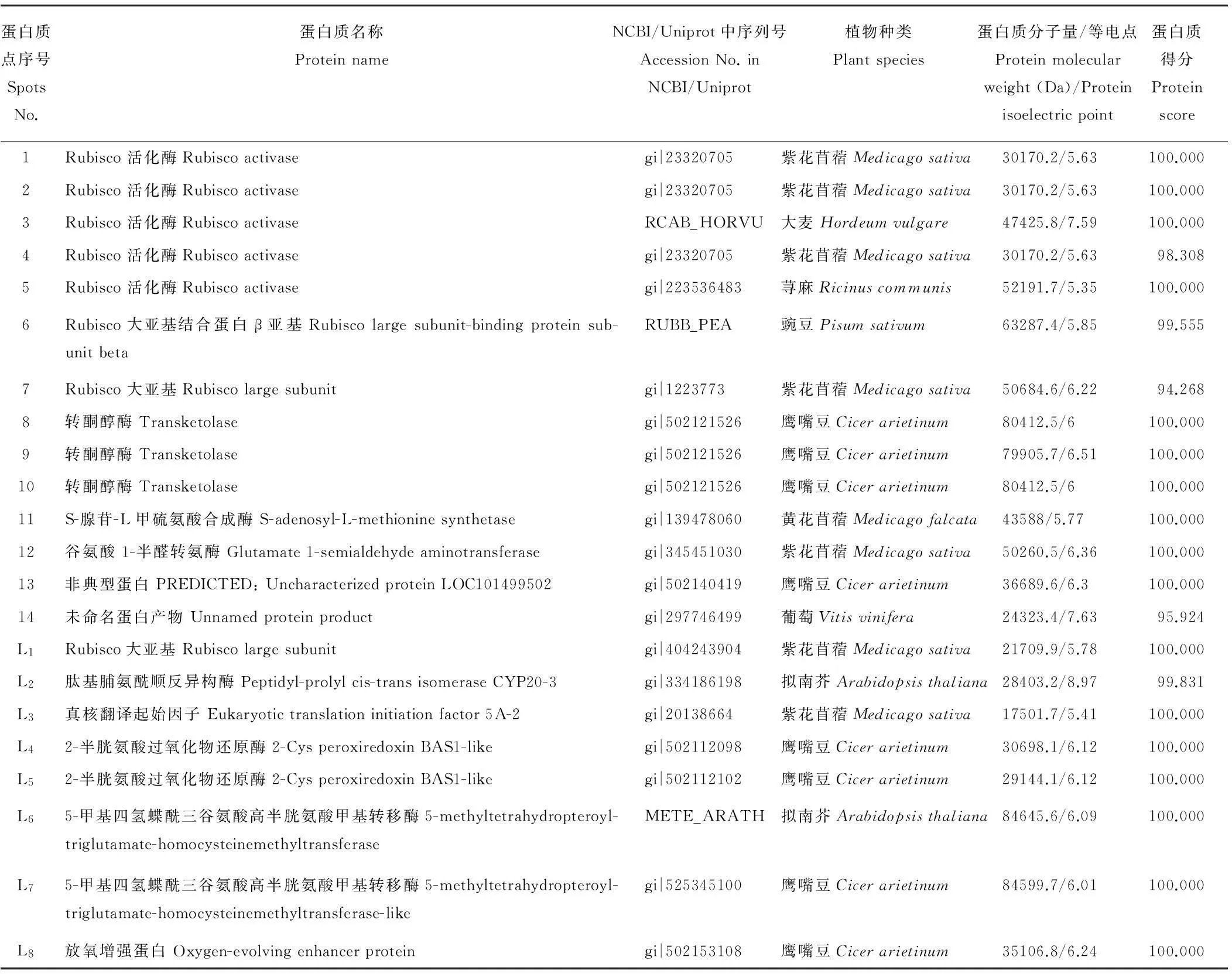
蛋白質點序號SpotsNo.蛋白質名稱ProteinnameNCBI/Uniprot中序列號AccessionNo.inNCBI/Uniprot植物種類Plantspecies蛋白質分子量/等電點Proteinmolecularweight(Da)/Proteinisoelectricpoint蛋白質得分Proteinscore1Rubisco活化酶Rubiscoactivasegi|23320705紫花苜蓿Medicagosativa30170.2/5.63100.0002Rubisco活化酶Rubiscoactivasegi|23320705紫花苜蓿Medicagosativa30170.2/5.63100.0003Rubisco活化酶RubiscoactivaseRCAB_HORVU大麥Hordeumvulgare47425.8/7.59100.0004Rubisco活化酶Rubiscoactivasegi|23320705紫花苜蓿Medicagosativa30170.2/5.6398.3085Rubisco活化酶Rubiscoactivasegi|223536483蕁麻Ricinuscommunis52191.7/5.35100.0006Rubisco大亞基結合蛋白β亞基Rubiscolargesubunit-bindingproteinsub-unitbetaRUBB_PEA豌豆Pisumsativum63287.4/5.8599.5557Rubisco大亞基Rubiscolargesubunitgi|1223773紫花苜蓿Medicagosativa50684.6/6.2294.2688轉酮醇酶Transketolasegi|502121526鷹嘴豆Cicerarietinum80412.5/6100.0009轉酮醇酶Transketolasegi|502121526鷹嘴豆Cicerarietinum79905.7/6.51100.00010轉酮醇酶Transketolasegi|502121526鷹嘴豆Cicerarietinum80412.5/6100.00011S-腺苷-L甲硫氨酸合成酶S-adenosyl-L-methioninesynthetasegi|139478060黃花苜蓿Medicagofalcata43588/5.77100.00012谷氨酸1-半醛轉氨酶Glutamate1-semialdehydeaminotransferasegi|345451030紫花苜蓿Medicagosativa50260.5/6.36100.00013非典型蛋白PREDICTED:UncharacterizedproteinLOC101499502gi|502140419鷹嘴豆Cicerarietinum36689.6/6.3100.00014未命名蛋白產物Unnamedproteinproductgi|297746499葡萄Vitisvinifera24323.4/7.6395.924L1Rubisco大亞基Rubiscolargesubunitgi|404243904紫花苜蓿Medicagosativa21709.9/5.78100.000L2肽基脯氨酰順反異構酶Peptidyl-prolylcis-transisomeraseCYP20-3gi|334186198擬南芥Arabidopsisthaliana28403.2/8.9799.831L3真核翻譯起始因子Eukaryotictranslationinitiationfactor5A-2gi|20138664紫花苜蓿Medicagosativa17501.7/5.41100.000L42-半胱氨酸過氧化物還原酶2-CysperoxiredoxinBAS1-likegi|502112098鷹嘴豆Cicerarietinum30698.1/6.12100.000L52-半胱氨酸過氧化物還原酶2-CysperoxiredoxinBAS1-likegi|502112102鷹嘴豆Cicerarietinum29144.1/6.12100.000L65-甲基四氫蝶酰三谷氨酸高半胱氨酸甲基轉移酶5-methyltetrahydropteroyl-triglutamate-homocysteinemethyltransferaseMETE_ARATH擬南芥Arabidopsisthaliana84645.6/6.09100.000L75-甲基四氫蝶酰三谷氨酸高半胱氨酸甲基轉移酶5-methyltetrahydropteroyl-triglutamate-homocysteinemethyltransferase-likegi|525345100鷹嘴豆Cicerarietinum84599.7/6.01100.000L8放氧增強蛋白Oxygen-evolvingenhancerproteingi|502153108鷹嘴豆Cicerarietinum35106.8/6.24100.000
(表2)。由鑒定結果可知,紫花苜蓿葉片蛋白和低豐度蛋白中Rubisco及其變體分別占差異蛋白的50%和12.5%。去除高豐度蛋白后,可檢測出原本被遮蓋或因共遷移影響的蛋白質,其蛋白種類可見表2。可見,17.5%的PEG可以清除大部分Rubisco,并可應用于紫花苜蓿響應低溫的蛋白質組學研究中。
3討論
植物組織中常含有拷貝數眾多的高豐度蛋白,例如,Rubisco作為光合作用中固定CO2關鍵酶,一般占蛋白總量的30%~60%,嚴重影響鄰近蛋白點的識別和其他蛋白點在凝膠電泳中的共同遷移,從而降低了低豐度蛋白的分辨率[17-19]。同樣的,由于高豐度蛋白占據了樣品中的一大部分,使得在雙向電泳IPG膠條上樣容量一定的情況下,低豐度蛋白的含量受到限制而不易被檢測到[20]。而低豐度蛋白作為細胞或組織中的受體分子、信號分子等參與基因表達調控,往往具有十分重要的功能[19]。因此,如何去除高豐度蛋白以便分離鑒定低豐度蛋白一直是蛋白質組學研究中亟待解決的問題之一。在以往的文獻中發現,15%~20%PEG可以有效去除植物蛋白中大部分高豐度蛋白,尤其是Rubisco的大亞基,但在不同物種或組織中用于預分離的PEG濃度是不同的。在一定范圍內,高濃度PEG對蛋白質的沉淀效率高。Xi等[13]利用16%的PEG去除了擬南芥中大部分高豐度蛋白;王瑩等[21]利用20%PEG提取了水稻葉鞘中的低豐度蛋白;鐘俐等[16]研究發現15%PEG更適宜于甜瓜葉片的低豐度蛋白提取。本研究表明,適宜的PEG濃度可以去除紫花苜蓿葉片的高豐度蛋白,且這種篩選是十分必要的。PEG濃度過低無法有效沉淀高豐度蛋白(圖2中S1);濃度過高則易造成蛋白質過度沉淀,蛋白質點損失(圖2中S3)。本研究中,17.5%PEG能夠清除大部分高豐度蛋白,使原本被遮蓋的蛋白質點更易于被檢測到;同時避免了蛋白的過度損失。可見,該濃度最適宜紫花苜蓿葉片高豐度蛋白的去除。
為了驗證PEG沉淀法在紫花苜蓿蛋白質組學研究中的可行性,比較了三氯乙酸/丙酮法與PEG去除葉片高豐度蛋白后響應低溫的差異蛋白變化。三氯乙酸/丙酮法是提取植物蛋白質的常用方法之一[22-25],具有降低次生代謝物質的干擾、減少蛋白降解等優點[26-27]。與其提取的蛋白相比,17.5% PEG去除了大部分Rubisco,并檢測到8個響應低溫的新蛋白質。其中Rubisco大亞基(點L1)主要參與光合作用過程中CO2的固定;肽酰脯氨酰-順反式異構酶(點L2)和真核翻譯起始因子(點L3)參與蛋白質的翻譯和折疊加工[25,28-30];2-半胱氨酸過氧化物還原酶(點L4和L5)響應低溫脅迫下機體內部的氧化還原平衡[31];5-甲基四氫蝶酰三谷氨酸高半胱氨酸甲基轉移酶(點L6和L7)是一類甲基轉移酶,可為S-甲硫氨酸腺苷途徑提供底物;而放氧增強蛋白(點L8)作為獲光復合體的組成元件,能通過激發葉綠素而獲得更多的光能,并把能量轉移到光化反應中心,有助于增強低溫逆境下的光合作用效率[32]。這些新發現的蛋白點在紫花苜蓿葉片中由于高豐度蛋白遮蓋而無法檢測到。可見,利用17.5%PEG去除高豐度蛋白在紫花苜蓿蛋白質組學研究中有實際的應用意義,有助于低豐度蛋白的檢測。該方法也可用于紫花苜蓿生長發育或逆境應答的蛋白質組學研究,有助于功能蛋白的分析與鑒定。
4結論
高豐度蛋白影響了紫花苜蓿葉片中低豐度蛋白的鑒定。本研究采用PEG沉淀法,比較了0,15%,17.5%和20%的PEG對高豐度蛋白的分離情況,結果顯示,17.5%的PEG最適宜用于紫花苜蓿葉片高豐度蛋白的去除。此法應用于紫花苜蓿低溫應答的差異蛋白鑒定中,獲得8個新差異蛋白,證明其適用于紫花苜蓿的蛋白質組學研究。
References:
[1]Andrew W C, Hiren J J, Joshua L H. Managing the green proteomes for the next decade of plant research. Frontiers in Plant Science, 2013, (4): 501.
[2]Jenny R, Jean-Francois H, Michael E W. Proteomics and low-temperature studies: bridging the gap between gene expression and metabolism. Physiologia Plantarum, 2006, 126: 97-109.
[3]Kang J M, Zhang T J, Wang M Y,etal. Research process in the quantitative trait loci (QTL) and genomic selection of alfalfa. Acta Prataculturae Sinica, 2014, 23(5): 304-312.
[4]Giavalisco P, Nordhoff E, Kreitler T,etal. Proteome analysis ofArabidopsisthalianaby two-dimensional gel electrophoresis and matrix-assisted laser desorption/ionisation-time of flight mass. Proteomics, 2005, 5: 1902-1913.
[5]Yan S P, Zhang Q Y, Tang Z C,etal. Comparative proteomic analysis provides new insights into chilling stress responses in rice. Molecular & Cellular, 2006, 5: 484-496.
[6]Cellar N A, Kuppannan K, Langhorst M L,etal. Cross species applicability of abundant protein depletion columns for ribulose-1,5-bisphosphate carboxylase/oxygenase. Journal of Chromatography, 2008, 861: 29-39.
[7]Cho J H, Wang H, Cho M H,etal. The effect of DTT in protein preparations for proteomic analysis: Removal of a highly abundant plant enzyme,ribulose bisphosphate arboxylase/oxygenase. Journal of Plant Biology, 2008, 51: 297-301.
[8]Li H B, Kang Z S. Study of a rapid method for depletion of highly abundant protein Rubisco enzyme from wheat (T.aestivum) leaf for proteome analysis. Journal of Northwest A&F University (Natural Science Edition), 2011, 39(6): 223-228.
[9]Krishnan H B, Natarajan S S. A rapid method for depletion of Rubisco from soybean (Glycinemax) leaf for proteomic analysis of lower abundance proteins.Phytochem, 2009, 70: 1958-1964.
[10]Atha D H, Ingham K C. Mechanism of precipitation of proteins by polyethylene glycols. The Journal of Biology Chemistry, 1981, 256: 12108-12117.
[11]Juckes I R. Fractionation of proteins and viruses with polyethylene glycol. Biochimica et Biophysica Acta, 1971, 229: 535-546.
[12]Kim S T, Cho K S, Jang Y S,etal. Two-dimensional electrophoretic analysis of rice proteins by polyethylene glycol fractionation for protein arrays. Electrophoresis, 2001, 22: 2103-2109.
[13]Xi J H, Wang X, Li S Y,etal. Polyethylene glycol fractionation improved detection of low-abundant proteins by two-dimensional electrophoresis analysis of plant proteome. Phytochemistry, 2006, 67: 2341-2348.
[14]Sun T K, Kyu S C, Yu S J,etal. Two-dimensional electrophoresis analysis of rice proteins by polyethylene glycol fractionation for protein arrays. Electrophoresis, 2001, 22: 2103-2109.
[15]Lee D G, Ahsan N, Lee S H,etal. An approach to identify cold-induced low-abundant proteins in rice leaf. Comptes Rendus Biologies, 2007, 330: 215-225.
[16]Zhong L, Ma C M, Liang Y Z,etal. Depletion of Rubisco from cucumis melon leaf and optimization of two-dimensional electrophoresis for proteome. Plant Physiology Journal, 2012, 48(3): 303-309.
[17]G?rg A, Weiss W, Dunn M J. Current two-dimensional electrophoresis technology for proteomics. Proteomics, 2004, 4: 3665-3685.
[18]Lilley K S, Razzaq A, Dupree P. Two-dimensional gel electrophoresis: recent advances in sample preparation, detection and quantitation. Current Opinion in Chemical Biology, 2002, 6: 46-50.
[19]Shaw M M, Riederer B M. Sample preparation for two-dimensional gel electrophoresis. Proteomics, 2003, 3: 1408-1417.
[20]Li H M, Shao M, Tan H Y,etal. Establishment and evaluation of the method for removing the high-abundance proteins in cerebrospinal fluid proteomics. Journal of Modern Clinical Medical Bioengineering, 2007, 13(2): 94-96.
[21]Wang Y, Cui W T, Yang M F,etal. An approach to detect the low-abundant proteins in rice leaf sheath. Acta Prataculturae Sinica, 2011, 20(3): 192-197.
[22]Tai F J, Li Y, Chen L,etal. Analysis of two-dimensional electrophoresis for the expressed proteins in cotton cotyledons under cold stress. Journal of Huazhong Normal University (Natural Science edition), 2008, 42(2): 262-266.
[23]Ning S J, Zhao M, Xiang X L,etal.Proteomics of rice and grain at late growth stage under different nitrogen fertilization levels. Chinese Journal of Applied Ecology, 2010, 21(10): 2573-2579.
[24]Iker A, Gemma M, Gorka E,etal. Plant physiology and proteomics reveals the leaf response to drought in alfalfa (MedicagosativaL.). Journal of Experimental Botany, 2010, 62(1): 111-123.
[25]Zhang S, Feng L H, Jiang H,etal. Biochemical and proteomic analyses reveal that populus cathayana males and females have different metabolic activities under chilling stress. Journal of Proteome Research, 2012, 11: 5815-5826.
[26]Granier F. Extraction of plant proteins for two-dimensional electrophoresis. Electrophoresis, 1988, 9: 712-718.
[27]Tsugita A, Kamo M. 2-D electrophoresis of plant proteins. Methods in Molecular Biology, 1999, 112: 95-97.
[28]Schatz O, Oft M, Dascher C,etal. Interaction of the HIV-1 Rev cofactor eukaryotic initiation factor 5A with ribosomal protein L5. Proceedings of National Academy of Sciences, 1998, 95: 1607-1612.
[29]Jao D L E, Chen K Y. Tandem affinity purification revealed the hypusine-dependent binding of eukaryotic initiation factor 5A to the translating 80S ribosomal complex. Journal of Cell Biochemistry, 2006, 97: 583-598.
[30]Thompson J E, Hopkins M T, Taylor C,etal. Regulation of senescence by eukaryotic translation initiation factor 5A: implications for plant growth and development. Trends Plant Science, 2004, 9: 174-179.
[31]Kim M D, Kim Y H, Kwon S Y,etal. Overexpression of 2-cysteine peroxiredoxin enhances tolerance to methyl viologen-mediated oxidative stress and high temperature in potato plants. Plant Physiology and Biochemistry, 2011, 49: 891-897.
[32]Green B R, Durnford D G. The chlorophyll-carotenoid proteins of oxygenic photosynthesis. Annual Review of Plant Physiology and Plant Molecular Biology, 1996, 47: 685-714.
參考文獻:
[3]康俊梅, 張鐵軍, 王夢穎, 等. 紫花苜蓿QTL與全基因組選擇研究進展及其應用. 草業學報, 2014, 23(5): 304-312.
[8]李紅兵, 康振生. 小麥葉片蛋白質組分析中高豐度蛋白Rubisco酶的快速去除方法研究. 西北農林科技大學學報(自然科學版), 2011, 39(6): 223-228.
[16]鐘俐, 馬成梅, 梁永增, 等. 甜瓜葉片中Rubisco的去除及雙向電泳體系的優化. 植物生理學報, 2012, 48(3): 303-309.
[20]李煥敏, 邵明, 譚紅愉, 等. 腦脊液蛋白質組學高豐度蛋白去除方法的建立與評價. 現代臨床醫學生物工程學雜志, 2007, 13(2): 94-96.
[21]王瑩, 崔為同, 楊明峰, 等. 一種有效檢測水稻葉鞘低豐度蛋白的方法. 草業學報, 2011, 20(3): 192-197.
[22]邰付菊, 李揚, 陳良, 等. 低溫脅迫下棉花子葉蛋白質差異表達的雙向電泳分. 華中師范大學學報(自然科學版), 2008, 42(2): 262-266.
[23]寧書菊, 趙敏, 向小亮, 等. 不同氮素水平下水稻生育后期葉片和籽粒的蛋白質組學. 應用生態學報, 2010, 21(10): 2573-2579.

