動物皮張基因組DNA三種提取方法比較
劉艷艷++東野升金++王想想++卞如如++陳杰++范陽陽++張全芳++步迅

摘要:以干燥的驢皮張樣品為試驗材料,將SDS-蛋白酶K法與Chelex-100法相結合,創新性地引入玻璃奶DNA純化技術,提取全基因組DNA,并將此方法與前人發表的陳舊皮張DNA提取方法和試劑盒提取方法進行比較分析。結果表明,本研究發明方法提取的基因組DNA純度較高,雜質較少,且操作簡單,所用試劑、儀器費用較低,能更好地應用于分子生物學實驗。
關鍵詞:動物皮張;DNA提取;玻璃奶
中圖分類號:Q78 文獻標識號:A 文章編號:1001-4942(2015)12-0006-04
DNA為分子生物學研究的基礎,隨著生物領域尤其是遺傳學方面研究的日漸深入,從分子水平上對動物進行物種鑒定、物種起源和多樣性評估及其親緣關系研究已獲得廣泛應用。20世紀80年代就已開始動物皮張標本DNA提取的研究,盡管提取成功的報道很多,但提取的基因組DNA含量較低,片段較短,且難以進行PCR擴增;提取方法雖然不斷改善,但得到的DNA質量仍舊不是很高。
當前皮張源性鑒定方法滯后,國內外仍然沒有統一的技術標準和較成熟的鑒別方法,傳統的宏觀觀察法、顯微鏡觀察法具有一定的主觀性,無法準確和快速地區分皮張源性。隨著分子生物學技術的發展,基于DNA的生物學鑒定手段逐漸豐富起來。DNA分子鑒定法主要是利用顯示生物特征的各生物物種所具有的不同DNA序列信息進行源性鑒別,它可以突破依據動物纖維結構形態進行鑒定的局限性,與傳統分析方法相比,更加客觀和準確。因此,獲得高質量的動物皮張基因組DNA已成為進行皮張源性鑒定研究的迫切要求。
本試驗以SDS法為基礎,將蛋白酶K、Chelex -100與Glass Milk DNA提取技術相結合,并將此方法與前人發表的陳舊皮張DNA提取新方法和DNA提取試劑盒法進行比較分析,旨在研建一種操作簡便、DNA提取效率高、花費較為低廉的優化方法,為后續分子生物學實驗提供必要條件。
1 材料與方法
1.1 試驗材料
9份干燥的驢皮樣品,分別編號為1~9。每份樣品平均分為三份,分別用于A、B、C三種方法。材料由山東省農業科學院生物技術研究中心測序中心提供。
1.2 試驗試劑
(1)消化緩沖液A(10mmol/L Tris-HCl,25mmol/L EDTA, 100mmol/L NaCl,0.5%SDS, pH8.0)、Chelex-100、20mg/mL蛋白酶K、氯仿、Glass Milk、DNA Binding Buffer、70%酒精、無水乙醇、TE緩沖液、超純水。
(2)消化緩沖液B(10mmol/L Tris-HCl,0.5% SDS,1mmol/L CaCl2,0.2mg/mL蛋白酶K, pH 8.0)、1μmol/L EDTA、10% Chelex溶液、酚、氯仿、異戊醇、3mol/L NaAc、無水乙醇、75%酒精、TE緩沖液、超純水。
(3) DNA提取試劑盒(E.Z.N.A.TissueDNA Kit)。
(4) HiFi Buffer: TransTaq DNA PolymoraseHigh Fidelity購自北京全式金生物技術有限公司。
1.3 試驗方法
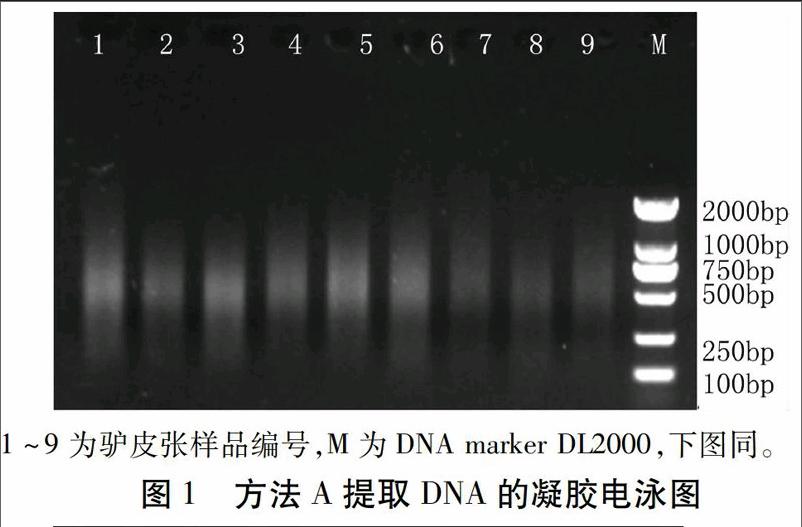
1.3.1 基因組DNA提取 方法A:取約30mg驢皮樣品研磨至微粒,分別裝到編號為A1~A9的2mL EP管中,加1mL去離子水洗滌2次;加入400μL消化緩沖液A以及380μL5%的Chel-ex-100和20μL 20mg/mL的蛋白酶K,50~600C水浴保溫,期間攪拌混勻,至全部溶解;取出冷卻至室溫,振蕩5~10s,100℃沸水浴8min;結束后取出樣品,10000r/min離心1min,取上清轉至新的EP管(記錄上清體積),加等體積氯仿,充分混勻,12000r/min離心5min,重復此步驟兩次;向收集液中加入5μL Glass Milk,并加600μL DNA Binding Buffer.充分混勻,65℃水浴15min;后室溫放置5min,8000r/min離心1min,棄上清;加入70%酒精500μL進行洗滌,8000r/min離心1min,重復此步驟兩次;加入500μL無水乙醇,吹打懸浮,8000r/min離心1min,棄上清;8000r/min離心30s,用10μL槍頭吸走殘余液體,室溫干燥10min;加入50μL TE緩沖液(pH 7.0),70℃水浴5min,快速離心,移至新離心管,- 200C保存。
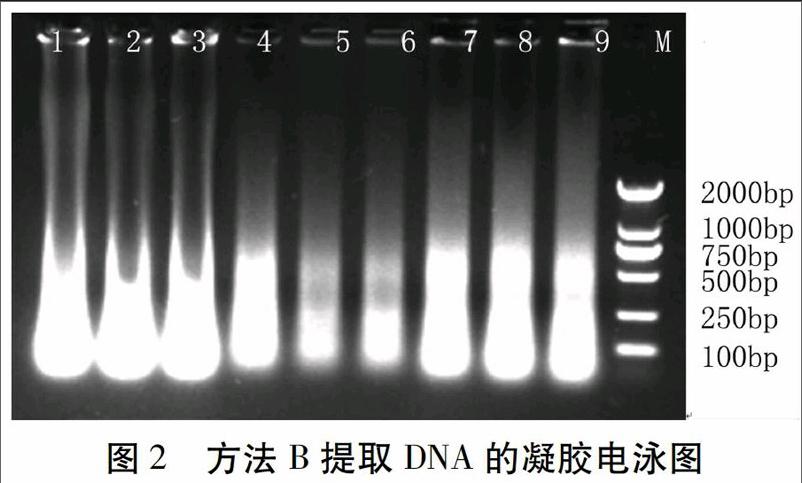
方法B參考陳舊皮張DNA提取新方法[12]。
方法C參考DNA提取試劑盒(E.Z.N.A.Tissue DNA Kit)。
1.3.2 基因組DNA檢測 (1)紫外分光光度計檢測:每份DNA樣品分別取2μL測定其濃度及A260和A280。每個樣品做三個生物學重復,取均值。
(2)瓊脂糖凝膠電泳檢測:配置2.0%的瓊脂糖凝膠,取1×Loading Buffer 10μL,加入2μL基因組DNA樣品,混合均勻后加入點樣孔中,同時加入DNA Marker,180V電泳10min。電泳結束后,利用凝膠成像儀進行拍攝,保存圖像,根據樣品的條帶亮度、整齊度及位置,粗略估計其純度及片段大小。
(3) PCR擴增檢測:根據驢線粒體基因組(16S rRNA)細胞色素b特異序列,利用PrimerPremier 5.0設計PCR引物,上游引物MtUF:5' -ATAAGACGAGAAGACCCTATCCAGC -3',下游引物MtUR:5-ACCAITITCTGITCTCCGAGGTCAC -3',擴增目的片段250 bp。在擴增之前分別將三種方法提取的共27份基因組DNA稀釋到100 ng/yL待用。PCR反應總體積為25μL,其中ExTaq聚合酶0.2μL,HiFi Buffer 2.5μL,dNTP 2.0μL,上游引物1.0μL,下游引物1.0μL,模板2.0μL,ddH20 16.3μL。endprint
2 結果與分析
2.1 三種方法提取的基因組DNA紫外分光光度計檢測
由表1可知,A、B、C三種方法獲得的DNA質量在A260和A280值上有明顯區別,B方法的A260值最高達100以上,A280值最高也達到50以上;C方法雖然A260和A280值比B方法稍低,但總體上數值仍然偏高。原因可能是B和C兩方法提取的DNA純度不高,含有較多苯酚、鹽酸胍、聚乙二醇等雜質。過多的雜質污染進而導致樣品濃度過高,兩方法獲得DNA濃度均大于500ng/μL,遠高于方法A的DNA濃度。利用方法A提取的DNA濃度范圍為74.8~177.5ng/μL,A260/A280在2.0左右,雖仍有部分RNA污染,但DNA純度相對B、C兩方法要好。
2.2 三種方法提取的基因組DNA瓊脂糖凝膠電泳分析
從瓊脂糖凝膠電泳圖可以看出,三種方法均可從驢皮張中提取出基因組DNA,但獲得的DNA量有顯著差別。其中A方法的基因組DNA瓊脂糖凝膠電泳圖條帶最弱,出現拖帶現象,提取量最少(圖1);B方法的基因組DNA提取量相對A方法稍多,但DNA降解較為嚴重,且雜質較多,不夠純凈(圖2);C方法的基因組DNA提取量相對于A、B兩方法最多,條帶最為明亮,提取效果最好,但存在降解現象及RNA等雜質污染(圖3)。
2.3 三種方法提取的基因組DNA的PCR擴增檢測
以三種方法提取的基因組DNA為模板,設計合成的引物擴增目的片段。由圖4~6可知,三種方法均能很好地進行目的片段的擴增,且條帶清晰,帶型整齊,說明三種方法均能從干燥的驢皮張中提取較好質量的基因組DNA,且都能夠應用于后續的分子生物學實驗。
3 結論與討論
玻璃奶是一種特殊制備的以二氧化硅作為懸浮物的水溶液,具備快速、定量、特異性結合DNA的能力,可以取代有毒的有機物苯酚、氯仿等用于分離和純化DNA。在高鹽溶液中,核酸是不溶的,可被吸附至玻璃基質上,而蛋白質在高鹽溶液中是高度溶解的,脂類物質懸浮于溶液表面,從而起到純化分離DNA的作用。將二氧化硅用于DNA的純化最早見于Engelstein等的報道,他們采用此法純化出了測序級的模板DNA。該法不僅杜絕了如酚、氯仿等對身體有毒害藥品的使用,而且可達到高效、快速、批量提取DNA的目的,具有經濟、實用、高效、無毒的特點。本研究利用SDS-蛋白酶K結合Chelex-100法提取動物皮張中的微量DNA,能獲取較高濃度的DNA,進一步利用玻璃奶DNA純化技術能顯著去除殘留的蛋白和金屬離子等污染,顯著提高了DNA純度。
將本研究使用Chelex-100結合玻璃奶提取驢皮張全基因組DNA的方法與前人報道方法和試劑盒法提取所得基因組DNA分別利用紫外分光光度計、瓊脂糖凝膠電泳以及PCR擴增技術進行評估,綜合分析可知,A方法所提基因組DNA雖然量較少但純度較高,電泳均能顯示出條帶,并且能夠很好地擴增目的片段,滿足分子生物學實驗要求,此外,所用試劑、儀器費用較低且操作較為簡單,耗時14小時,相對B方法時間較短。B方法獲取的基因組DNA濃度較高,也能用于PCR擴增目的條帶,雖然操作以及儀器試劑和A方法相似,但基因組DNA電泳條帶及吸光值顯示其存在蛋白質、RNA以及其他雜質污染,且電泳條帶也不是很整齊,耗時20小時,時間相對較長。C方法所提基因組DNA濃度居中,條帶清晰整齊,能很好地進行目的片段PCR擴增,但從其吸光值來看,可能存在較多的酚、聚乙二醇、鹽酸胍等雜質污染,且試劑盒價格相對較為昂貴。
綜上所述,本研究三種方法均可用于干燥動物皮張基因組DNA的提取,通過對比分析,方法A即本研究發明的SDS -蛋白酶K結合Chelex-100法和玻璃奶純化技術提取獲得的基因組DNA純度較高,雜質較少,且操作簡單,所用儀器、試劑均價格低廉,相對于方法B和C能更好地應用于分子生物學實驗。endprint

