中藥材北沙參種子DNA條形碼鑒定研究*
張改霞,金 鉞,賈 靜,石林春**,孫稚穎
(1.山東中醫藥大學藥學院 濟南 250355;2.中國醫學科學院 北京協和醫學院藥用植物研究所 北京 100193)
中藥材北沙參種子DNA條形碼鑒定研究*
張改霞1,2,金 鉞2,賈 靜2,石林春2**,孫稚穎1**
(1.山東中醫藥大學藥學院 濟南 250355;2.中國醫學科學院 北京協和醫學院藥用植物研究所 北京 100193)
目的:本研究旨在建立鑒定北沙參種子的新方法,確保藥材生產種植中種質基原準確。方法:對北沙參不同產地種子經初步形態特征鑒定后,取單粒種子為一份樣品,提取基因組DNA、PCR擴增、雙向測序拼接獲得ITS2序列,結合序列比對、變異位點分析,基于中藥材DNA條形碼鑒定系統(http://www. tcmbarcode.cn),系統地完成種子樣品的分子鑒定。結果:研究表明66份種子樣品中,64份與《中國藥典中藥材DNA條形碼標準序列》中北沙參序列特征一致,2份樣品序列中各有一處SNP位點,為北沙參暫未發現過的遺傳多樣性信息,鑒定結果顯示為北沙參。結論:DNA條形碼技術能準確鑒定北沙參種子,并為中藥材種子鑒定提供新方法,同時SNP位點的發現為后續研究北沙參品種多樣性問題提供參考。
北沙參 中藥材種子 ITS2條形碼 分子鑒定
北沙參為傘形科植物珊瑚菜Glehnia littoralis Fr. Schmidt ex Miq. 的干燥根,是我國常用大宗中藥材,具養陰清肺,益胃生津的功效,用于肺熱燥咳,勞嗽痰血,胃陰不足,熱病津傷,咽干口渴等癥狀[1]。目前北沙參野生資源極少,為國家三級保護瀕危植物[2],被《國家重點保護野生植物名錄》列為二級保護植物。據市場調查,流通北沙參藥材現均為栽培品,引種區域從山東煙臺、日照等沿海縣市不斷擴展到菏澤、濟寧、聊城等內陸地區,并在河北安國、內蒙赤峰等地形成規模化產業化栽培種植[3]。北沙參藥材的大規模人工培植,使得北沙參種源鑒定、良種選育問題尤為重要。
中藥材種子是中藥材產業化的基礎,其質量與真偽直接影響中藥材的種植、加工、流通、臨床使用等后續各個環節。準確鑒定中藥材種子,保證中藥材種源的純正優良,對后續種質優選、良種培育、規模化種植等各階段具有重要意義。近年來,條形碼技術廣泛地應用于植物和動物的鑒定中,本文依據《中藥材DNA條形碼分子鑒定指導原則》[4],采用ITS2為主體序列,基于條形碼技術,以中藥材北沙參種子為研究對象,對收集到的22批66份北沙參種子樣品鑒定,并從分子鑒定角度分析不同產地種源的可靠性,確保種子來源準確,為北沙參良種鑒定選育提供參考依據。
1 材料和方法
1.1 材料
為使實驗結果具有代表性[4],通過產地調查和文獻檢索,從藥材的主要分布區河北、安徽、內蒙古、山東等省份共收集22批次優質北沙參種子,每個批次種子取樣3次,共66份樣品。樣品信息見表1。
1.2 方法
1.2.1 形態學考察
取北沙參種子,確保取樣均勻性,于LY-WN-HPCCD型動態顯微成像系統(成都勵揚精密機電有限公司)下觀察種子形狀、大小、顏色等表面性狀,并拍照記錄,用游標卡尺(上海九量五金工具有限公司)精確測量長度、寬度,并用百粒法測其千粒重[5],準確記錄各項結果。
1.2.2 DNA提取
從22批樣品中,每批樣品取樣3次,分3批于不同時間、不同儀器實驗,每批每次輕取一粒種子(約15 mg,避免帶出它粒種子碎屑),使用75%乙醇擦拭表面、揮干,用DNA提取研磨儀MM400(德國Retsch公司)加2粒鋼珠研磨2 min(30次/s),使用植物DNA提取試劑盒[天根生化科技(北京)有限公司]提取總DNA,加入裂解液后,56℃水浴過夜。其后加入等體積氯仿∶異戊醇(24∶1)抽提,取上清,吸附柱過濾并用緩沖液、漂洗液洗滌,最后用洗脫緩沖液(Buffer TE)洗脫。提取到的基因組DNA,采用NanoDrop 2000 (美國Thermo Scientific公司)測其濃度,并根據A260/A280,A260/A230的值判斷DNA提取質量。

表1 實驗材料編號及產地

圖1 北沙參種子群體圖、個體圖
1.2.3 PCR擴增及測序
PCR反應為25 μL體系,反應體系的配置、擴增引物的選擇及反應條件的設置依照Chen等[6]的研究方法操作,PCR產物純化后使用ABI3730XL測序儀進行雙向測序。
1.2.4 數據處理
測序峰圖利用CodonCode Aligner V4.2.7(美國CodonCode公司)去除低質量區域、校對拼接、切除正反引物序列。基于隱馬爾可夫模型(Hidden Markov Model)去除5.8S rRNA和28S rRNA區段獲得ITS2序列[7]。利用Mega 5.05進行序列比對、變異位點分析,基于中藥材DNA條形碼鑒定系統,在物種鑒定項下植物類藥材鑒定 (ITS2)項,將實驗序列粘貼到鑒定框中,提交并查詢,系統地完成實驗種子樣品的鑒定。
2 結果
2.1 形態特征
北沙參藥材的“種子”實際上為珊瑚菜的雙懸果,呈棕色,氣香,灰黃色,近球形或橢圓形,分生果半橢圓形,合生面平坦棕色,長6-13 mm,寬6-10 mm,背部棱3,兩邊各1,稍延展,表面被棕色絨毛。將分生果果皮剝開現胚乳,腹面略凹陷。千粒重約15 g,群體圖、個體圖見圖1。
2.2 DNA提取、PCR擴增和測序
DNA提取時,直接取單粒種子實驗,在加入等體積冷凍異丙醇沉淀DNA過程中,會有個別樣品底部出現白色團塊膠狀物,為避免對后續PCR擴增、測序造成影響,應不考慮部分DNA損失,只吸取上清過濾,舍棄沉淀物。NanoDrop 2000檢測DNA濃度均大于10 ng·μL-1,OD260/280比值2.00左右,OD260/230比值2.00-3.00,擴增條帶整齊明亮,約500 bp,測序峰圖底峰干凈,無雜峰套峰的出現。不同時間、使用不同儀器實驗的3批樣品結果一致,可見實驗中各因素對結果影響較小,ITS2序列獲取較為容易。
2.3 北沙參種子物種鑒定
本研究共收集22批樣品,每批樣品取單粒種子3次,經實驗獲得66條ITS2序列,長度均為227 bp。共出現3種單倍型,單倍型A1 64條序列,單倍型A2、A3各1條序列。如圖2(正向測序峰圖),峰圖整體質量較高,單倍型A2 108位、單倍型A3124位各有1套峰,單倍型A1相應位點處并未出現套峰,且經反向序列驗證,結果與之相對應,可知套峰處即為多拷貝位點處,即單倍型A2代表序列BESS0059-2在108 bp處G(T)多拷貝,單倍型A3代表序列BESS0029-1在124 bp處G(A)多拷貝,主導單倍型A1與《中國藥典中藥材DNA條形碼標準序列》[8]中北沙參主導單倍型序列特征一致,可以確定其基原為北沙參。單倍型A2、A3有多拷貝位點出現,為新的單倍型,利用中藥材DNA條形碼鑒定系統網站,在物種鑒定項下植物類藥材鑒定(ITS2)項,將所得樣品ITS2序列粘貼到鑒定框中,提交并查詢,結果顯示種子樣品主導單倍型A1與數據庫中北沙參序列完全一致,單倍型A2、A3與數據庫中北沙參序列相似性高達99.6%,比對結果顯示為北沙參,表明所有種子樣品均為北沙參種子,從而系統的完成對北沙參種子樣品的鑒定。多拷貝位點分析峰圖見圖2。
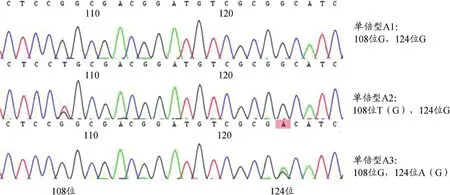
圖2 北沙參種子ITS2序列種內多拷貝位點峰圖
3 討論
3.1 DNA條形碼技術可準確鑒定中藥材北沙參種子
北沙參種子傳統鑒定法主要從種子表面形態、大小、形狀及內部特征如油管等方面鑒別,傳統鑒定方法一般需要豐富的專業經驗,多方面、多角度分析辨別,不便于數字化和自動化。本研究采用DNA條形碼技術對北沙參種子鑒定,采用單粒種子實驗的方法,有效避免不同種粒遺傳物質之間的相互干擾,66條序列,每條均源自一粒種子的DNA,從DNA 提取至ITS2序列獲得,各步進行均較順暢,沒有出現未能獲得序列的樣品,結果較好。在對種子物種鑒定時,單倍型A1的64條序列與《中國藥典中藥材DNA條形碼標準序列》中北沙參主導單倍型序列特征一致,可以確定其基原為北沙參,而單倍型A2、A3各有一個多拷貝位點,為北沙參ITS2序列中暫未出現的單倍型,可結合中藥材DNA條形碼鑒定系統網站鑒定,發現實驗樣品中并沒有北沙參種子混偽品出現,從而系統地完成各序列的鑒定。《中國藥典中藥材DNA條形碼標準序列》與中藥材DNA條形碼鑒定系統網站的結合應用簡便準確,給中藥材種子鑒定提供了新方法。序列比對中66條序列中只有2條序列分別各有1處多拷貝位點,可見ITS2條形碼鑒定北沙參種子種內變異小,遺傳穩定性高,新增變異位點的出現在一定程度上豐富了北沙參條形碼序列數據庫,為后續北沙參遺傳多樣性研究提供參考數據。
3.2 DNA條形碼技術可用作中藥材種子鑒定的通用方法
研究發現,目前對中藥材種子的鑒定,主要有性狀、顯微、理化和現代生物學鑒定法等[9-13]。這些方法在長期中藥材種子鑒定工作中發揮了重要作用,但各自又有一定的局限性,如易受研究者專業經驗、實驗設備條件、生物物種特異性等因素的影響。DNA條形碼技術以中藥材DNA條形碼鑒定系統為核心,無過高的專業經驗要求,所用實驗設備簡單且不受物種形態、性狀、成分等的限制,鑒定準確,通用性高,關于DNA條形碼在相關領域的應用,辛天怡等[14]做了詳細綜述。此外,《中藥DNA條形碼分子鑒定》、《中國藥典中藥材DNA條形碼標準序列》等基于條形碼技術對物種鑒定參考書目的出現,也充分說明DNA條形碼鑒定的可靠性與通用性[15,8]。本研究是將條形碼技術運用到中藥材種子鑒定中,目前,我國中藥材種子主要依托于已批準的17個中藥材專業市場作為收售渠道,缺少對應的監管部門,大多數中藥材種子質量檢驗以農作物種子標準作為參照。這種無質量標準可依、缺乏正規監管的種子市場勢必給中藥材生產及臨床用藥安全帶來隱患,影響中藥材在國內外市場的聲譽[16,17]。本研究運用DNA條形碼成功鑒定北沙參種子,是中藥材種子鑒定新方法的成功應用,可以為今后條形碼技術作為中藥材種子鑒定的標準技術,以及全國中藥材種質資源管理提供參考依據。
3.3 北沙參基因組內多拷貝問題的探討
利用DNA條形碼技術,經雙向測序分別在兩粒樣品中各發現一處多拷貝位點(T/G、A/G),經驗證發現并非由實驗或測序等過程中的錯誤造成,初步確定為北沙參基因組內多拷貝,為北沙參基因組內遺傳變異。在大多數物種中,由于經歷了一致性進化, 具有主導拷貝變異類型,但也有例外情況,例如:Ma等[18]對同一居群內29個樣品的ITS序列進行克隆研究,在獲得的102個克隆中共發現773變異位點分布 在ITS2區段,并指出這種基因水平的多態性與自身生理特征、自身進化、環境因素等有關。Song等[19]利用高通量測序方法對178個物種基因組內ITS2序列的多拷貝情況進行研究,發現平均每個物種有35個變異類型,3個主導變異類型,這3個主導變異類型的拷貝數占全部拷貝數的91%。進一步研究表明,盡管ITS2基因組內變異頻繁,多數情況下,僅依靠主導變異即可完成系統發育和物種水平的鑒定,次要和稀有變異可提高分辨能力。本研究中的北沙參種子為不同地區收集種子,遺傳變異位點的出現是否與產地的環境差異、長期不同區域間的引種栽培有關還需進一步研究。在后期進化中,是否會有北沙參變種出現,這種變異會對北沙參物種產生什么樣的影響等問題都值得深入探討。
1 國家藥典委員會.中華人民共和國藥典(一部).北京:中國醫藥科技出版社, 2015∶ 100.
2 傅立國.中國植物紅皮書——稀有瀕危植物.北京:科學出版社1993∶ 698.
3 徐祝封,張欽德,李慶典,等.北沙參道地產區種子生產、種苗培育現狀調查與分析.山東中醫藥大學學報, 2006, 30(6)∶ 493-496.
4 陳士林,姚輝,韓建萍,等.中藥材DNA條形碼分子鑒定指導原則.中國中藥雜志, 2013(2)∶ 141-148.
5 國家技術監督局. GB/T 3543.7-1995農作物種子檢驗規程:其他項目檢驗,北京:中國標準出版社, 1996∶ 95-96.
6 Chen S L, Yao H, Han J P, et al. Validation of the ITS2 region as a novel DNA barcode for identifying medicinal plant species. PLoS ONE, 2010, 5 (1)∶ e8613.
7 Keller A, Schleicher T, Schultz J, et al. 5.8S-28S rRNA interaction and HMM-based ITS2 annotation. Gene, 2009, 430∶ 50-57.
8 陳士林.中國藥典中藥材DNA條形碼標準序列.北京:科學出版社, 2015∶ 163-164.
9 韋穎. 105種常用藥用植物果實、種子性狀與顯微鑒別特征研究.北京:中國中醫科學院, 2012∶ 33-271.
10 高飛燕.種子類中藥微性狀鑒定法研究.合肥:安徽中醫藥大學碩士學位論文, 2013∶ 11-63.
11 張玉杰,張建軍,閆興麗,等.不同產地正偽品沙苑子HPLC指紋圖譜鑒別研究.中國中藥雜志, 2003, 28(9)∶ 30-32, 71.
12 張瓊光,陳科力,王克勤.厚樸兩種類型種子的電泳鑒別.時珍國醫國藥, 2003, 28(9)∶ 541-542.
13 鄭海燕.利用RAPD、ISSR和SRAP標記技術構建紅麻(Hibiscus cannabinus L.)種質資源分子身份證.北京:中國農業科學院碩士學位論文, 2010∶ 10-40.
14 辛天怡,雷美艷,宋經元.中藥材DNA條形碼鑒定研究進展.中國現代中藥, 2015, 17(2)∶ 170-176.
15 陳士林.中藥DNA條形碼分子鑒定.北京:人民衛生出版社, 2012∶ 1-568.
16 寧書菊,魏道智.關于我國中藥材種子產業的思考.中國種業, 2013, 3∶ 3-4.
17 邵長勇,尤泳,王光輝,等.安國中藥材種子種苗產業發展中的現代物理技術應用.種子, 2013, 32(12)∶ 70-72,75.
18 Ma X C, Xie C X, Guan M, et al. High levels of genetic diversity within one population of Rheum tanguticum on the Qinghai-Tibet Plateau have implications for germplasm conservation. Pharm Crops, 2014, 5∶ 1.
19 Song J Y, Shi L C, Li D Z, et al. Extensive pyrosequencing reveals frequent intragenomic variations of internal transcribedspacer regions of nuclear ribosomal DNA. PLoS ONE, 2012, 7(8)∶ e43971.
Identification of the Seeds of Glehniae Radix with DNA Barcoding Method
Zhang Gaixia1,2, Jin Yue2, Jia Jing2, Shi Linchun2, Sun Zhiying1
(1. College of Chinese Medicine, Shandong University of Traditional Chinese Medicine, Jinan 250355, China; 2. Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences and Peking Union Medical College, Beijing 100193, China)
In this study, a new technique was adopted to discriminate and ensure the pure sources of the seeds of Glehniae Radix. The morphological characteristics of the seeds from different habitats had been first surveyed. Then the DNA extraction, PCR amplification, DNA sequencing and assembly were carried out. Species identification for each sample was implemented through the DNA barcoding system of traditional Chinese medicine (http://www.tcmbarcode.cn). The results showed that sixty-four sequences were genuinely based on “Standard DNA Barcodes of Chinese Materia Medica in Chinese Pharmacopoeia”, and two samples contained SNP loci. The identification results referred to Glehniae Radix of all sequences according to the DNA barcoding system. In conclusion, DNA barcoding technique was suitable for the identification of the seeds of Glehniae Radix, which provided a new method for identification of the seeds of Chinese herbal medicines. The finding of SNP loci also furnished with a reference for studies on the variety diversity of Glehniae Radix.
Glehniae Radix, Chinese herbal seeds, ITS2, molecular identification
10.11842/wst.2016.02.006
R282.5
A
(責任編輯:朱黎婷 張志華,責任譯審:朱黎婷 王 晶)
2015-11-29
修回日期:2015-12-01
* 科學技術部國家科技支撐計劃子課題(2011BAI07B08):中藥種子種苗 DNA條形碼鑒定及流通管理平臺的建立,負責人:陳士林;山東省教育廳高等學校科技計劃項目(J12LM05):基于DNA條形碼技術的山東省珍稀瀕危特有野生藥用植物資源研究,負責人:孫稚穎。
** 通訊作者:石林春,副研究員,主要研究方向:中藥鑒定學;孫稚穎,副教授,主要研究方向:藥用植物資源與分類鑒定。

