預擴增qPCR方法檢測少量小鼠早期胚胎細胞中DNA甲基化相關基因的表達
程 琳,孫平楠,3,謝慶東,2,周小玲,2,3,*(. 汕 頭大學醫學院干細胞P2實驗室,廣東 汕頭 5504;2. 汕 頭大學醫學院生殖醫學中心,廣東 汕頭 5504;3. 廣 東省感染病與分子免疫病理重點實驗室,廣東 汕頭 5504)
?
預擴增qPCR方法檢測少量小鼠早期胚胎細胞中DNA甲基化相關基因的表達
程 琳1,2,3,孫平楠1,3,謝慶東1,2,周小玲1,2,3,*
(1. 汕 頭大學醫學院干細胞P2實驗室,廣東 汕頭 515041;2. 汕 頭大學醫學院生殖醫學中心,廣東 汕頭 515041;3. 廣 東省感染病與分子免疫病理重點實驗室,廣東 汕頭 515041)
【摘要】目的: 比較3種實時定量PCR(qPCR)方法對少量小鼠早期胚胎細胞中甲基化相關基因表達水平的檢測效果,以期獲得適合日常檢測的方法。方法:分別應用常規qPCR方法、基于等溫預擴增的qPCR方法、基于PCR預擴增的qPCR方法3種方案對少量小鼠早期胚胎細胞樣本中甲基化相關基因TET1、TET2、TET3、DNMT3A的mRNA表達進行檢測。結果:3種方法的靈敏度由高到低依次為基于等溫預擴增的qPCR方法、基于PCR預擴增qPCR方法、常規qPCR方法。前兩種方法均能在合適的循環數下對少量小鼠胚胎細胞中的多個甲基化相關基因表達進行定量。而基于PCR預擴增的qPCR方法比基于等溫預擴增的qPCR方法更經濟,適合日常檢測。因此,我們選擇應用基于PCR預擴增的qPCR方法,檢測了小鼠卵細胞以及卵細胞受精后22 h甲基化相關基因表達的變化,結果發現該過程中TET1表達量很低,不易檢測到;TET2表達呈降低趨勢;TET3和DNMT3A表達顯著升高(P<0.01),與文獻報道基本一致。結論:基于等溫預擴增的qPCR方法檢測少量細胞樣品中甲基化相關基因的表達的靈敏度在3種qPCR方法中最高。而基于PCR預擴增的qPCR方法操作簡單、靈敏度較高、成本相對低,適合少量細胞樣本中多個相關基因表達的實時定量日常檢測。
【關鍵詞】預擴增;實時定量PCR;小鼠;早期胚胎細胞;甲基化相關基因
作者信息: 程 琳,E-mail:470751814@qq.com。*通信作者,周小玲,
Detection of expression of methylation-related genes in mouse early embryonic cells using pre-amplification-based real time quantitative PCR
CHENG Lin1,2,3,SUN Pingnan1,3,XIE Qingdong1,2,ZHOU Xiaoling1,2,3,*
(1. Stem Cell P2 Laboratory, Shantou University Medical College, Shantou 515041; 2. Reproductive Medicine Center, Shantou University Medical College, Shantou 515041; 3. Guangdong Provincial Key Laboratory of Infectious Diseases and Molecular Immunopathology, Shantou 515041, Guangdong, China)
【ABSTRACT】OBJECTIVE: To identify a proper qPCR method for examining expression of methylation-related genes in mouse early embryonic cells. METHEDS:Normal qPCR,isothermal pre-amplification-based qPCR,and PCR pre-amplification-based qPCR methods were applied in quantitative assay of methylation-related genes (TET1,TET2,TET3 and DNMT3A) in mouse early embryonic cells. RESULTS:The sensitivity of these three methods decreased from isothermal pre-amplification-based qPCR method,PCR pre-amplification-based qPCR method to normal qPCR method. The former two methods detected expression of methylation-related genes in a few mouse embryonic cells within a proper PCR cycle number but the PCR pre-amplification-based qPCR method has lower cost than isothermal pre-amplification-based qPCR method. Therefore,we used PCR pre-amplification-based qPCR method to examine methylation-related genes in mouse oocytes and early embryonic cells after 22 h fertilization. The results showed that the expression level of TET1 mRNA was very low and not detectable,the expression of TET2 decreased,and the expression of TET3 and DNMT3A significantly increased (P<0.01) in this process,which was consistent with reported results.
CONCLUSION:The sensitivity of isothermal pre-amplification-based qPCR method was the highest among the three qPCR methods. However,the PCR pre-amplification-based qPCR method is the most suitable one for examining multigene expression in a few cells in daily practice due to the simple procedure, relatively high sensitivity and lowexpenditure.
【KEY W ORDS】pre-amplification;real t ime q uantitative P CR;mouse;early e mbryonic c ell;methylation-related g enes
在哺乳動物合子以及早期胚胎細胞發育過程中,甲基化相關基因和其他早期發育相關的基因起到重要作用,且具有特定的時空表達譜,因此其表達變化情況成為研究中關注的重點[1-3]。行使甲基化功能的甲基轉移酶主要包括DNA甲基轉移酶1(DNA methyltransferase 1 ,DNMT1)、DNA甲基轉移酶3A(DNMT3A)、DNA甲基化轉移酶3B(DNMT3B)。DNMT1可催化一條鏈甲基化的DNA雙鏈中的未甲基化鏈,使其完全甲基化;而DNMT3A、DNMT3B則催化未甲基化的DNA雙鏈從頭甲基化。DNA胞嘧啶甲基化后形成5-甲基胞嘧啶(5-mC)。行使去甲基化功能的酶類有Tet家族成員,包括甲基胞嘧啶雙加氧酶-1( ten-eleven translocation methyl cytosine dioxygenase 1,TET1)、甲基胞嘧啶雙加氧酶-2(TET2)、甲基胞嘧啶雙加氧酶-3(TET3)。TET蛋白主要通過催化5-mC轉化為5-羥甲基胞嘧啶(5-hmC),還能進一步氧化5-hmC生成一系列衍生物,最終形成無甲基化修飾的胞嘧啶,是DNA去甲基化過程中的一種重要的酶,對于維持干細胞的多能性有重要作用。
現階段,由于在研究中獲得的哺乳動物胚胎細胞的數量有限,應用常規實時定量PCR(quantitative realtime PCR,qPCR)對少量胚胎細胞中甲基化或發育相關基因表達進行定量檢測往往未能檢測到信號,或者即使檢測到信號,qPCR循環數也過多,從而導致處理組與對照組之間比較時可信度并不高。此外,由于起始細胞量少、所含的總RNA量也少,因此反轉錄獲得的cDNA量不能滿足下游檢測需要。這種情況下采用常規qPCR往往只能對1~2個基因同時進行檢測(每個基因包括內參均還需要有重復組),從而無法同時對5~10個基因甚至更多的基因進行實時定量檢測。
近年來,對于少量細胞樣本甚至是單細胞樣本的RNA深度測序技術有了很大的提高。有研究人員采用PCR預擴增或者基于Phi-29酶的等溫預擴增技術從少量樣本中預先擴增獲得了足量的cDNA來完成后續測序工作[4-5]。PCR預擴增方法主要是在對少量樣本進行反轉錄獲得cDNA后進行少量循環(6~12個循環)的PCR預擴增以獲得足量的相關基因表達產物進行后續定量檢測[5];基于Phi-29酶的等溫擴增技術是在對少量樣本進行反轉錄獲得cDNA后,對獲得的cDNA加接頭連接環化后,再用Phi-29酶對其進行等溫線型擴增[4]。后者相對前者而言,由于能線性擴增較長片段的cDNA,故能夠更加準確真實的反映少量細胞樣品中各個基因的表達量[4]。然而,現階段后者中用到的相關試劑價格較昂貴,這也限制了實際研究中對細胞數目有限的材料中多個目的基因的表達同時進行實時定量日常檢測。
因此,我們針對少量哺乳動物早期胚胎細胞樣本中與甲基化相關的多個基因(TET1、TET2、TET3、DNMT3A等)的定量檢測,比較了常規qPCR方法、基于等溫預擴增的qPCR方法和基于PCR預擴增的qPCR方法3種方案的檢測結果,為今后相關研究工作的開展提供重要參考。
1 材料與方法
1.1 主要試劑
PMSG和HCG購自寧波三生藥業有限公司。反轉錄試劑盒(ReverTra Ace qPCR RT Kit,FSQ-101)購自Toyobo公司。qPCR試劑盒(Quantifast SYBR Green PCR Kit)和純化試劑盒(QIAquick PCR purification Kit)均購自Qiagen公司。PCR試劑盒(Premix Taq? Hot Start Version,R028A)購自Takara公司。
經回顧性分析可知,引發胎兒先天性心臟病的獨立危險因素與孕早期感冒、負性生活事件和生殖系統感染存在相關性,同時與居住環境有著直接關系,見表2。
1.2 實驗動物
采用汕頭大學醫學院實驗動物研究中心飼養培育的清潔級昆明品系小鼠,雌鼠為8~10周齡,雄鼠10~12周齡。
1.3 體外授精
處死雄鼠,取附睪置于含有0.3% BSA的TYH培養基中,將附睪剪碎,加入1 mL培養基,于37 ℃、CO2體積分數為5%的培養箱中上游5 min。上游后600 g離心5 min,棄上清,加入1.5 mL獲能液(3.5% BSA)重懸精子后置于37 ℃、CO2體積分數為5%的培養箱獲能1 h。600 g離心5 min,棄去部分培養基,用剩余培養基重懸精子制作精子液滴,按每滴50 μL,覆以甘油,置于37 ℃、CO2體積分數為5%的培養箱中。雌鼠皮下注射10 IU PMSG,48 h后腹腔注射10 IU HCG誘導超排。HCG注射12~16 h后取出輸卵管,將輸卵管置于含有0.3% BSA的TYH培養基中,鏡下刺破壺腹部并擠壓,使卵丘細胞浸在培養基中將其轉入含有0.1%透明質酸酶的TYH中,洗滌卵細胞2次將卵母細胞移入受精液滴,37 ℃、CO2體積分數為5%的培養箱中培養22 h以獲得早期胚胎細胞。
1.4 卵細胞、胚胎細胞的收集
分別收集30個卵細胞或30個早期胚胎細胞,用PBS洗滌2次,把洗滌后的細胞轉移至裝有20 μL細胞裂 解緩沖液(cells-to-cDNATMⅡ Kit,Ambion)的EP管中,75 ℃ ,5 m in,放置于冰上5 m in,加入1.5 μ L D Nase 1 ,短暫離心后,37 ℃,30 min,置于冰上10 min, 然后75 ℃加熱5 min。
1.5 qPCR的3種方案
1.5.1 常規qPCR方法 用反轉錄試劑盒,將裂解后的產物反轉錄成cDNA。反應條件為:37 ℃、15 min;98 ℃、5 min。產物稀釋10倍后進行qPCR。
1.5.2 基于等溫預擴增的qPCR方法 具體步驟見參考文獻[4]。簡而言之,先將細胞裂解產物去除基因組DNA,反轉錄成cDNA,再將其進行平端連接,然后用phi-29酶對其進行擴增。最終產物純化后稀釋100倍進行qPCR。
1.5.3 基于PCR預擴增的qPCR方法 步驟見參考文獻[5]。用反轉錄試劑盒,將裂解后的產物反轉錄成cDNA。反應條件為:37 ℃、15 min;98 ℃、5 min。用PCR試劑盒對反轉錄產物進行以下處理:Premix 12.5 μL,目的正、反向引物各1 μL,DNA模板10 μL,最后加水至25 μL。PCR反應條件為:98 ℃、5 min;98 ℃、10 s,55 ℃、30 s,72 ℃、1 min,10個循環;72 ℃、7 min擴增。然后用純化試劑盒對上述RTPCR的產物進行純化。獲得的純化產物稀釋10倍后進行qPCR。
將以上3種不同方案的cDNA產物按照qPCR試劑盒進行反應。反應體系如下:SYBR(2×) 10 μL,目的正、反向引物各1 μL,cDNA模板8 μL,總量20 μL。PCR反應條件:95 ℃、5 min,95 ℃、10 s,60 ℃、30 s,共40個循環。引物序列為:TET1正向引物5′-CCAT TCTCACAAGGACATTCACA-3′,反向引物5′-GCAGG ACGTGGAGTTGTTCA-3′,跨內含子611 bp,擴增產物116 bp;TET2正向引物5′-GCCATTCTCAGGAGTCACT GC-3′,反向引物5′-ACTTCTCGATTGTCTTCTCTATTG AGG-3′,跨內含子1 700 bp,擴增產物120 bp。TET3正向引物5′-GGTCACAGCCTGCATGGACT-3′,反向引物5′-AGCGATTGTCTTCCTTGGTCAG-3′,跨內含子2 474 bp,擴增產物104 bp;DNMT3A正向引物5′-ATGT GGTTCGGAGATGGCAAG-3′,反向引物5′-AGATGGC TTTGCGGTACATGG-3′,跨內含子429 bp,擴增產物124 bp; RSP6(正向引物5′-AGGAGAGAAGGATATTCCTGG AC-3′,反向引物5′-GCTTCCTGACAACATACTG GC-3′)作為內參[6]。目的基因的表達水平與內參相比后采用2-ΔΔCT方法計算。實驗進行3次重復。
1.6 瓊脂糖凝膠電泳
1%的凝膠,電壓至120 V,電泳30 min后,取出凝膠,在數碼凝膠成像系統下照相記錄電泳圖譜。
1.7 統計學方法
小鼠卵細胞和早期胚胎細胞中甲基化相關基因的表達水平差異分析采用t檢驗進行統計學分析。
2 結 果
2.1 3種qPCR方案擴增的標準曲線圖
采用3種qPCR方案分別對30個受精后22 h的小鼠早期胚胎細胞中甲基化和去甲基化相關的基因TET1、TET2、TET3、DNMT3A進行擴增獲得擴增曲線(圖1)和循環閾值(CT值 )(表1)。從擴增曲線以及CT值 來看,基于PCR預擴增的qPCR方法的擴增效果優于常規qPCR方法,更接近于基于等溫預擴增的qPCR方法。

表1 3種qPCR方案擴增小鼠早期胚胎細胞中DNA甲基化相關基因的CT值
2.2 3種方案的擴增產物的瓊脂糖凝膠電泳
分別將3種方案擴增的qPCR產物進行瓊脂糖凝膠電泳(圖2)。各方案的電泳圖中擴增產物亮度與各方案在qPCR結果中呈現的靈敏度相一致,常規qPCR方法中TET1基因未擴增出來,TET2、TET3、DNMT3A和RSP6有擴增條帶,但是比基于等溫預擴增的qPCR方法和基于PCR預擴增的qPCR方法的條帶明顯要弱。就基因擴增效果而言,基于等溫預擴增的qPCR方法>基于PCR預擴增的qPCR方法>常規qPCR方法,而就實驗費用而言,基于PCR預擴增的qPCR方法要遠低于基于等溫預擴增的qPCR方法。因此,我們選擇基于PCR預擴增的qPCR方法進行下一步實驗。
2.3 基于PCR預擴增的qPCR方法定量檢測小鼠卵細胞受精后TET1、TET2、TET3和DNMT3A的基因表達變化
我們采用基于PCR預擴增的qPCR方法對小鼠卵細胞及受精后22 h的早期胚胎中去甲基化基因TET1、TET2和TET3和甲基化基因DNMT3A的表達變化進行了快速定量檢測。結果顯示小鼠卵細胞中去甲基化基因TET2和TET3和甲基化基因DNMT3A相對于內參RSP6的表達量分別是0.02±0.00、3.30±0.16、0.25±0.02;受精后22 h的早期胚胎細胞中TET2和TET3和甲基化基因DNMT3A相對于內參RSP6的表達量分別是0.01± 0.00、4.30±0.35、1.90±0.23。TET1表達量太低未檢測出(圖3)。統計分析結果表明,小鼠卵細胞體外受精后22 h的過程中TET2表達水平略下降、TET3和DNMT3A在該過程中表達則顯著增加(P<0.05)。獲得的結果和變化趨勢與Yan等[6]的深度測序結果、Iqbal等[3]以及Wossidlo等[7]報道的qPCR結果基本一致。
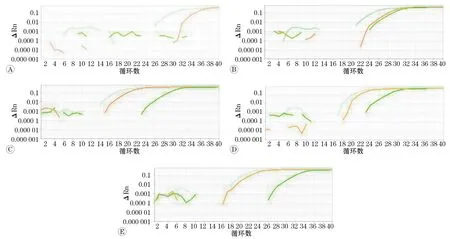
A:TET1的擴增曲線;B:TET2的擴增曲線;C:TET3的擴增曲線;D:DNMT3A的擴增曲線;E:內參RPS6的擴增曲線. 藍色線代表基于等溫預擴增的qPCR方法,橙色線代表基于PCR預擴增的qPCR方法,綠色線代表常規qPCR方法.圖1 少量小鼠胚胎細胞樣本分別采用3種qPCR方案的基因擴增曲線

M1、M2、M3分別代表常規qPCR方法、基于等溫預擴增的qPCR方法和基于PCR預擴增的qPCR方法.圖2 3種方案的qPCR擴增產物瓊脂糖凝膠電泳圖
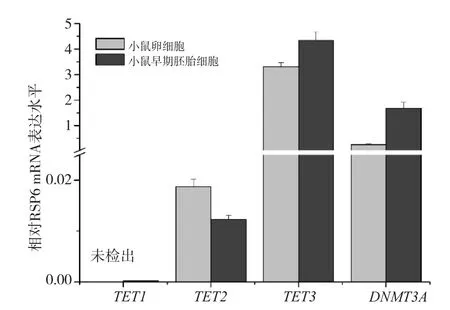
圖3 小鼠卵細胞和早期胚胎細胞TET1、TET2、TET3和DNMT3A的相對表達水平變化
3 討 論
研究中基于少量細胞樣本的基因表達水平檢測,往往難以實現同時對多個基因的檢測。近年發展起來的基于PCR預擴增或者基于等溫預擴增的對少量細胞或單細胞基因表達檢測的方法,為該問題的解決提供了有利的工具[4-5]。雖然后者的實驗結果更準確,但因為受實驗成本較高等因素影響,其在日常檢測中的應用受到限制。我們研究發現,基于PCR預擴增的qPCR方法定量檢測少量樣本細胞中基因表達獲得的qPCR擴增曲線與基于等溫預擴增的qPCR方法更接近,而采用常規qPCR方法對于少量細胞樣本中的基因進行檢測獲得的CT值 與其他兩種方法相比偏高,不利于少量樣本的準確定量檢測。我們進一步采用建立的基于PCR預擴增的qPCR方法對小鼠卵細胞到卵細胞受精后22 h的早期胚胎過程中重編程相關基因TET1、TET2、TET3和DNMT3A的mRNA表達水平變化情況。結果顯示在該過程中TET1表達量很低,不易檢測到;TET2表達呈下降趨勢;TET3和DNMT3A表達升高,這與文獻報道基本一致[3,6-7]。TET1是胚胎發育后期的關鍵調控因子在合子和二細胞中表達量少,而TET3僅在合子和二細胞胚胎階段高表達[8]。因此,與常規qPCR流程相比,基于PCR預擴增的qPCR方法更接近基于等溫預擴增的qPCR方法,亦即更準確,同樣的起始樣本量可以同時檢測更多的基因;與基于等溫預擴增的qPCR方法相比,更易于在日常檢測中使用。
參考文獻
[1] Kohli RM,Zhang Y. TET enzymes,TDG and the dynamics of DNA demethylation[J]. Nature,2013,502(7472):472-479.
[2] Li E. Chromatin modification and epigenetic reprogramming in mammalian development[J]. Nat Rev Genet,2002,3(9):662-673.
[3] Iqbal K,Jin SG,Pfeifer GP,et al. Reprogramming of the paternal genome upon fertilization involves genome-wide oxidation of 5-methylcytosine[J]. Proc Natl Acad Sci USA,2011,108(9):3642-3647.
[4] Pan X,Durrett RE,Zhu H,et al. Two methods for full-length RNA sequencing for low quantities of cells and single cells[J]. Proc Natl Acad Sci USA,2013,110(2):594-599.
[5] Tang F,Barbacioru C,Nordman E,et al. RNA-Seq analysis to capture the transcriptome landscape of a single cell[J]. Nat Protoc,2010,5(3):516-535.
[6] Yan L,Yang M,Guo H,et al. Single-cell RNA-Seq profiling of human preimplantation embryos and embryonic stem cells[J]. Nat Struct Mol Biol,2013,20(9):1131-1139.
[7] Wossidlo M,Nakamura T,Lepikhov K,et al. 5-Hydroxymethylcytosine in the mammalian zygote is linked with epigenetic reprogramming[J]. Nat Commun,2011,2:241.
[8] Lee K,Hamm J,Whitworth K,et al. Dynamics of TET family expression in porcine preimplantation embryos is related to zygotic genome activation and required for the maintenance of NANOG[J]. Dev Biol,2014,386(1):86-95.
基金項目:國家自然科學基金(81570567,81571994);廣東省自然科學基金(2014A030313483,2015A030313447);教育部留學回國人員科研啟動基金(第49批);汕頭大學醫學院李嘉誠基金
收稿日期:2015-05-05;
修訂日期:2015-12-24
中圖分類號:Q781
文獻標志碼:A
文章編號:1004-616X(2016)01-0051-05
doi:1 0.3969/j.issn.1004-616x.2016.01.011

