基于葉綠體全基因組的貝母屬特異性DNA條形碼的篩選*
李 瀅,姚 輝,宋經元,任風鳴,李西文,孫 超**
(1.中國醫學科學院 北京協和醫學院 藥用植物研究所 北京 100193;2.中國中醫科學院中藥研究所 北京 100700)
基于葉綠體全基因組的貝母屬特異性DNA條形碼的篩選*
李 瀅1,姚 輝1,宋經元1,任風鳴1,李西文2,孫 超1**
(1.中國醫學科學院 北京協和醫學院 藥用植物研究所 北京 100193;2.中國中醫科學院中藥研究所 北京 100700)
本研究通過對貝母屬4個物種葉綠體基因組進行全局分析,分別查找基因區域和基因間區的高變異區域,篩選用于高效鑒別貝母屬植物的新DNA條形碼序列。相關研究發現貝母屬植物的基因區域序列相似度極高,不適用于DNA條形碼鑒定研究;共有7個基因間區可以作為潛在的貝母屬植物鑒定的特異性DNA條形碼。本研究所構建的DNA條形碼篩選方法,為篩選用于難鑒定科屬的新的DNA條形碼提供了通用的方法體系。
貝母屬 葉綠體基因組 DNA條形碼 分子鑒定
貝母屬Fritillaria是百合科Liliaceae中的一個大屬,據電子植物志***記載,貝母屬共有約130個物種,主要分布在亞洲和地中海等北半球溫帶地區。中國有貝母屬植物24種及其4個變種,其中15個種為中國的特有物種。貝母屬藥材以干燥鱗莖入藥,具有“清熱潤肺、化痰止咳”的功效,藥用歷史悠久[1]。2010版《中國藥典》收錄的貝母屬藥材包括川貝母、平貝母、浙貝母、伊貝母、湖北貝母[1]。貝母是典型的多基源藥材,以川貝母FRITILLARIAE CIRRHOSAE BULBUS為例,藥典共收錄的6個物種分別為川貝母F. cirrhosa,暗紫貝母F. unibracteata,甘肅貝母F. przewalskii,梭砂貝母F. delavayi,太白貝母F. taipaiensis,和瓦布貝母F. unibracteata var. wabuensis。據統計,《中國藥典》涉及貝母屬物種共11個。目前,已有研究表明,貝母屬植物可能是處于激烈分化的物種形成階段,部分物種的分類存在很多爭議,同一物種在不同地區因植物形態和花部特征出現差異,而被劃分為不同物種[2,3]。由于貝母屬植物的這些特性,其形態學鑒定和現有分子鑒定都存在一定的困難。另外,由于貝母屬藥材市場需求量大,川貝母的價格昂貴等原因,貝母屬藥材的使用長期存在混偽品濫用的情況,這直接影響了貝母屬植物臨床用藥的安全和療效。因此,開展貝母屬藥材的鑒定研究,篩選高效鑒定貝母屬植物的DNA條形碼序列,對于貝母屬藥材的臨床用藥、質量控制以及合理開發利用具有重要意義。
DNA條形碼(DNA Barcode)是指生物體內能夠代表該物種的、標準的、有足夠變異的、易擴增且相對較短的DNA片段[4-6]。目前,公認較為高效的植物DNA條形碼序列包括核基因組的ITS和ITS2序列與葉綠體基因組的psbA-trnH,rbcL,matK,psbK-psbI,atpF-atpH等[7,8]。已有研究表明,在藥用植物豐富的多個大科中,DNA 條形碼具有良好鑒定效果的有菊科[9]、薔薇科[10]、豆科[11]等。但對貝母屬植物DNA條形碼的研究顯示,ITS2序列不能將貝母屬植物準確鑒定到“種”[3]。因此,篩選高效的貝母屬特異性DNA條形碼對貝母屬藥材的鑒定具有重要的應用價值。
國際條形碼協會認可核基因組序列ITS和ITS2在植物DNA條形碼鑒定中的潛力,但同時指出要警惕操作過程中的真菌污染問題[12]。采用葉綠體基因組片段作為植物DNA條形碼可以很好的避免此問題。因此,篩選葉綠體基因片段作為新的DNA條形碼仍然是植物分子鑒定的研究熱點之一。隨著新一代測序技術不斷發展,獲得葉綠體全基因組序列的技術不斷改進[13]。本文從葉綠體全基因組角度,篩選貝母屬潛在的特異性DNA條碼序列,該研究結果可較大提高篩選效率。
1 材料與方法
1.1 葉綠體基因組前期研究數據
本課題組前期工作獲得4條貝母屬植物葉綠體全基因組數據,分別為川貝母F. cirrhosa(KF769143)[13]、湖北貝母F. hupehensis(KF712486)[13]、太白貝母 F. taipaiensis(KF769144)[13]和瓦 布貝 母 F. unibracteata var. wabuensis(KF769142)[14]。后續的分析采用GenBank格式序列及注釋文件。
1.2 數據分析
編寫本地程序,提取4條貝母屬植物葉綠體基因組的基因和基因間區序列,比較4個物種葉綠體基因在葉綠體基因組上的排列順序是否一致,應用clustalw2對每個相同的基因區域和基因間區分別做多序列比對(Multiple Sequence Alignment),分別計算每個基因區域和基因間區的序列相似度,篩選序列相似度較低的區域,即高變異區域作為潛在的新DNA條形碼,人工檢查新DNA條形碼比對結果,最終確定適合貝母屬鑒定的特異性DNA條形碼。
2 結果與分析
2.1 葉綠體基因組基因區域和基因間區序列的獲取
4條貝母屬葉綠體基因組上基因順序是一致的,每個葉綠體基因組編碼136個基因,包括編碼基因90個、tRNA基因38個和rRNA基因8個,基因區總長約110 kb(包含內含子序列)。
根據基因在葉綠體基因組上的排列順序,獲取基因間區序列。序列提取過程中可發現兩種特例:①一些包含多個外顯子的基因,其內含子區域包含其他基因,如trnK基因的兩個外顯子被matK基因間隔成兩段;rps12基因的兩個區段分別位于葉綠體基因組的大拷貝區域和反向重復區,中間包含了多個其他的基因。此種情況需提取外顯子區域間的序列做后續比對分析;②兩個相鄰的基因中間沒有間隔或者有部分重疊,這種情況下就會缺少該位點的基因間區。例如ycf1和ndhF兩個基因之間為0 bp。最后,每個葉綠體基因組各獲得130個基因間區域序列片段,總長約為42 kb。
2.2 葉綠體基因組基因區域比對結果
應用clustalw2軟件對4個物種同一基因序列進行多序列比對分析,共得到136個比對結果。其中,56個基因對應的比對結果相似性為100%,21個基因對應的比對結果只有一個位點的差異,無法用于篩選DNA條形碼。將剩余比對結果按照序列相似性由小到大排列,表1展示的是貝母屬植物變異度最高的20個葉綠體基因,由表中數據可以看出,除了ycf15基因外,貝母屬物種的葉綠體基因區序列高度相似。ycf15基因差異較大的原因是瓦布貝母的ycf15(231 bp)基因有一個140 bp片段的丟失,剩余91 bp片段在4個物種中的相似度為100%。綜上所述,在葉綠體基因區域很難篩選到鑒定貝母屬物種的特異性DNA條形碼。
2.3 葉綠體基因組基因間區比對結果
應用clustalw2分別對130個基因間區序列做多序列比對分析,去除相似性為100%、只有1個位點差異的比對結果,剩余79個比對結果可用于篩選DNA條形碼。將備選基因間區序列按照序列的相似性排列,發現基因間區的序列相似性比基因區域序列相似性低,但可能由于貝母屬各物種間親緣關系很近,或者由于貝母屬植物葉綠體進化緩慢,整體比對結果的相似性仍較高(表2)。
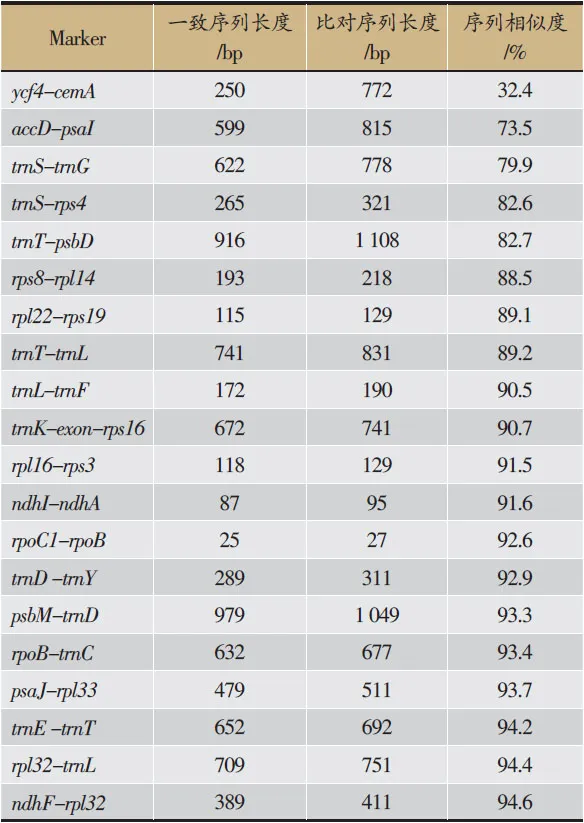
表2 貝母屬植物變異度最高的20個葉綠體基因間區

表3 貝母屬特異性DNA條形碼候選片段
2.4 根據比對結果篩選貝母屬特異性DNA條形碼
對于一個高效的DNA條形碼序列,除了需要滿足變異度足以區分不同物種的需求之外,還需要滿足PCR擴增和PCR產物測序的實驗要求。因此,序列長度也是需要考慮的篩選因素之一。本研究篩選出7個比對長度在300-700 bp之間,序列相似性< 97%的基因間區作為貝母屬特異性DNA條形碼的候選片段(表3)。對這7個區域的多序列比對結果逐一人工查看,檢測是否存在其中2條序列相似性為100%的情況。如圖1所示,trnP-psaJ基因間區比對結果顯示每個物種的該段序列都存在其特異性變異位點,僅從生物信息分析的角度,該序列片段能很好的區分已有4種貝母屬植物。因基因區序列保守性非常高,可在基因間區兩段的保守序列上設計擴增引物。
3 討論
貝母屬植物是清熱潤肺的傳統名貴藥材,具有十分重要的臨床應用價值和商業價值。雖然,電子植物志efloras.org目前收錄貝母屬植物“種”數和“變種”僅為28種。但是,最近的研究發現了貝母屬眾多新種和新變種,中國的國產貝母屬植物種數已達80個,“變種”數已達52個[2]。由于多種貝母屬植物藥材含多個基源且貝母屬物種基數龐大,使得貝母類藥材在使用、質量控制乃至生產流通等諸多領域面臨困難。貝母屬植物形態學鑒定和DNA條形碼鑒定研究表明,目前的鑒定方法還不能將貝母屬植物準確鑒定到“種”[3]。
本研究從葉綠體基因組的全局角度分析,篩選葉綠體上的高變異區段作為DNA條形碼篩選的候選區段。在圖2展示了根據4個貝母屬植物葉綠體基因組多序列比對結果獲得的高變異區域。可以發現,葉綠體基因組上存在多個高變異區域。對于篩選DNA條形碼,不僅需要考慮序列的變異程度,還需要綜合多方面因素,如序列長度、PCR擴增成功率、測序成功率、序列中是否含有poly結構等。為了驗證本研究所篩選的DNA條形碼在貝母屬物種中的鑒定效率,還需收集更多的貝母屬物種,再通過常規DNA條形碼研究方法,確定最終的貝母屬特異性DNA條形碼。常規DNA條形碼研究方法包括引物設計、DNA提取、PCR擴增、測序、計算種內種間變異、構建系統聚類樹等一系列的步驟。本研究所建立的方法體系,不僅適用于貝母屬植物的DNA條形碼篩選研究,對于其他難鑒定科屬也同樣適用。

圖1 使用clustalw 2對trnP-psaJ基因間區進行多序列比對

圖2 4個貝母屬葉綠體基因組高變異區域分布
1 國家藥典委員會.中華人民共和國藥典(一部). 北京:中國醫藥科技出版社, 2010: 72-73.
2 肖培根,姜艷,李萍,等.中藥貝母的基原植物和藥用親緣學的研究.植物分類學報, 2007, 45(4): 473-487.
3 羅焜,馬培,姚輝,等.基于ITS2序列鑒定川貝母及其混偽品基原植物.世界科學技術-中醫藥現代化, 2012, 14(1): 1153-1158.
4 陳士林,姚輝,宋經元,等.基于DNA barcoding(條形碼)技術的中藥材鑒定.世界科學技術-中醫藥現代化, 2007, 9(3): 7-12.
5 Li D Z, Liu J Q, Chen Z D, et al. Plant DNA barcoding in China. J Syst Evol, 2011, 49(3): 165-168.
6 Li M, Cao H, But P P, et al. Identification of herbal medicinal materials using DNA barcodes. J Syst Evol, 2011, 49(3): 271-283.
7 CBOL Plant Working Group. A DNA barcode for land plants. Proc Natl Acad Sci USA, 2009, 106(31): 12794-12797.
8 陳士林,龐曉慧,姚輝,等.中藥DNA條形碼鑒定體系及研究方向.世界科學技術-中醫藥現代化, 2011, 13(5): 747-754.
9 Gao T, Yao H, Song J Y, et al. Evaluating the feasibility of using candidate DNA barcodes in discriminating species of the large Asteraceae family. BMC Evol Biol, 2010, 10: 324.
10 Pang X H, Song J Y, Zhu Y J, et al. Applying plant DNA barcodes for Rosaceae species identification. Cladistics, 2011, 27: 165-170.
11 Gao T, Yao H, Song J Y, et al. Identification of medicinal plants in the family Fabaceae using a potential DNA Barcode ITS2. J Ethnopharmacol, 2010, 130(1): 116-121.
12 劉宇婧,劉越,黃耀江,等.植物DNA條形碼技術的發展及應用.植物資源與環境學報, 2011, 20(1): 74-82,93.
13 Li Q, Li Y, Song J, et al. High-accuracy de novo assembly and SNP detection of chloroplast genomes using a SMRT circular consensus sequencing strategy. New Phytol, 2014, 204(4): 1041-1049.
14 Li Y, Li Q, Li X, et al. Complete chloroplast genome sequence of Fritillaria unibracteata var. wabuensis based on SMRT Sequencing Technology. Mitochondrial DNA, 2015, 15: 1-2.
參考文獻
Screening Fritillaria Genus-specific DNA Barcodes Based on Complete Chloroplast Genome Sequences
Li Ying1, Yao Hui1, Song Jingyuan1, Ren Fengming1, Li Xiwen2, Sun Chao1
(1. Institute of Medicinal Plant Development, Chinese Academy of Medical Sciences & Peking Union Medical College, Beijing 100193, China; 2. Institute of Chinese Materia Medica, China Academy of Chinese Medical Sciences, Beijing 100700, China)
This study was aimed to find the Fritillaria genus-specific DNA barcodes through the comparison of the complete chloroplast genomes of 4 Fritillaria species. According to the results of multiple sequencing alignment (MSA), the high variation regions were located, which could be used as the potential DNA barcodes. The study showed that there were high similarities on the identities of gene regions, which were not applicable to the DNA barcoding identification. There were 7 intergenic regions, which can be used as the potential Fritillaria genus-s pecific DNA barcodes. The method established in this study for screening new DNA barcodes was a universal method. It can be used in the screening of other new DNA barcoding for the genera and families containing species which were difficult to be identified.
Fritillaria, chloroplast genome, DNA barcoding, molecular identification
10.11842/wst.2016.01.004
R931.5
A
(責任編輯:馬雅靜 張志華,責任譯審:王 晶)
2015-06-16
修回日期:2015-12-01
* 國家自然科學基金委青年科學基金項目(81503199):基于第三代單分子測序技術的科屬特異性DNA條形碼研究,負責人:李瀅;中國醫學科學院協和青年基金中央高校基本科研業務費專項資金項目(3332015144):基于單分子測序技術的增強型DNA條形碼研究,負責人:李瀅。
** 通訊作者:孫超,研究員,博士生導師,主要研究方向:藥用植物功能基因組學。
*** http://efloras.org/

