耐熱普魯蘭酶CBM68結(jié)構(gòu)域中關(guān)鍵位點(diǎn)對其酶學(xué)性質(zhì)的影響
申瑩瑩,鄭宏臣,李樹芳,付曉平,徐健勇,宋詼*
1(天津科技大學(xué) 生物工程學(xué)院,天津, 300457) 2(中國科學(xué)院 天津工業(yè)生物技術(shù)研究所,工業(yè)酶國家工程實驗室,天津, 300308) 3(中國科學(xué)院 天津工業(yè)生物技術(shù)研究所,天津工業(yè)生物系統(tǒng)與過程重點(diǎn)實驗室,天津, 300308)
?
耐熱普魯蘭酶CBM68結(jié)構(gòu)域中關(guān)鍵位點(diǎn)對其酶學(xué)性質(zhì)的影響
申瑩瑩1,2,3,鄭宏臣2,3,李樹芳2,3,付曉平2,3,徐健勇2,3,宋詼2,3*
1(天津科技大學(xué) 生物工程學(xué)院,天津, 300457)2(中國科學(xué)院 天津工業(yè)生物技術(shù)研究所,工業(yè)酶國家工程實驗室,天津, 300308)3(中國科學(xué)院 天津工業(yè)生物技術(shù)研究所,天津工業(yè)生物系統(tǒng)與過程重點(diǎn)實驗室,天津, 300308)
摘要通過對Anoxybacillus sp. LM18-11來源的耐熱普魯蘭酶(PulA)與麥芽三糖共結(jié)晶的晶體結(jié)構(gòu)分析,在新命名的底物結(jié)合域CBM68中預(yù)測出4個與底物結(jié)合有關(guān)的氨基酸位點(diǎn)(Y14、D16、K62和R96),分別將4個氨基酸位點(diǎn)突變?yōu)楸彼幔玫酵蛔凅wPulA(Y14A)、PulA(D16A)、PulA(K62A)和PulA(R96A)。其中,突變酶PulA(Y14A) 和PulA(R96A)的底物結(jié)合力及催化效率與野生酶PulA相比均有明顯降低,其Km值分別比PulA提高了4.2倍和2.5倍,k(cat)/Km 分別為PulA的29%和37%。同時,PulA(Y14A) 和PulA(R96A)的最適作用溫度均降低了10 ℃,PulA(R96A)的最適作用pH也降低了0.5個單位。說明Y14和R96是CBM68結(jié)構(gòu)域中的關(guān)鍵氨基酸位點(diǎn),這兩個位點(diǎn)的發(fā)現(xiàn)為進(jìn)一步探究CBM68結(jié)構(gòu)域的作用機(jī)制奠定了基礎(chǔ)。
關(guān)鍵詞耐熱普魯蘭酶;CBM68;定點(diǎn)突變;底物親和力;催化效率
普魯蘭酶是淀粉水解過程中一個重要的脫支酶,能夠?qū)R恍缘厮庵ф湹矸鄯种c(diǎn)中的α-1, 6糖苷鍵[1]。與α-淀粉酶、β-淀粉酶或葡糖淀粉酶共同作用,可使淀粉充分水解為小分子糖,提高淀粉原料的利用率,廣泛應(yīng)用于以淀粉為原料的食品深加工和新生物質(zhì)能源等工業(yè)領(lǐng)域[2-3]。普魯蘭酶屬于糖苷水解酶類,其分子結(jié)構(gòu)一般由多個結(jié)構(gòu)域組成,除了具有催化作用的催化結(jié)構(gòu)域外,還包含功能各異的其他非催化結(jié)構(gòu)域。其中,糖基結(jié)合模塊 (CBM) 是廣泛存在于糖基水解酶中的非催化性結(jié)構(gòu)域,該結(jié)構(gòu)域?qū)μ擒账饷傅牡孜锾禺愋浴⒌孜锝Y(jié)合能力及結(jié)構(gòu)穩(wěn)定性起到重要作用[4]。目前,在普魯蘭酶分子中出現(xiàn)頻率較高的CBM結(jié)構(gòu)域家族包括CBM41、CBM48和CBM68等[5-6]。
近年來對CBM結(jié)構(gòu)域的分子結(jié)構(gòu)與功能的研究越來越深入,也取得了一些成果,發(fā)現(xiàn)不同的CBM結(jié)構(gòu)域?qū)γ傅谋磉_(dá)、酶學(xué)性質(zhì)、動力學(xué)參數(shù)以及極端酶(高溫酶、耐酸酶和耐堿酶等)的催化性能等顯示了不同程度的影響。例如,VENDITTO 等人將嗜堿芽孢桿菌Bh Cel5B的內(nèi)切葡聚糖酶C端的CBM46結(jié)構(gòu)域去除后,發(fā)現(xiàn)CBM46缺失酶的底物親和力明顯下降[7];STROBEL 等人探究了里氏木霉Cel7A的CBM結(jié)構(gòu)域?qū)δ举|(zhì)素的親和力,通過定點(diǎn)突變將找到的31個氨基酸位點(diǎn)統(tǒng)一突變?yōu)楸彼幔C明了提高酶對底物的選擇性是增加酶催化能力的必要條件[8];此外,日本科研人員對幾丁質(zhì)酶A1(ChiA1) CBM12結(jié)構(gòu)域進(jìn)行了結(jié)合活性的研究,采用定點(diǎn)突變的方法對選擇的14個氨基酸同樣突變?yōu)楸彼幔?87位的芳香族氨基酸與結(jié)合活性有關(guān)外,非保守性的679位谷氨酰胺的突變也導(dǎo)致了結(jié)合活性的顯著降低,但核磁共振結(jié)果顯示該位點(diǎn)突變并沒有改變蛋白的整體結(jié)構(gòu)[9];江南大學(xué)生物技術(shù)重點(diǎn)實驗室研究了分別截掉普魯蘭酶N端CBM41結(jié)構(gòu)域、CBM41+X25結(jié)構(gòu)域和CBM41+X45+X25結(jié)構(gòu)域,所得到的突變酶在酶學(xué)性質(zhì)及分別表達(dá)的變化,從而發(fā)現(xiàn)CBM41和 X25對該酶的熱穩(wěn)定性有一定的不利影響,X45對該酶活性的保持是必須的[1, 10]。目前對于CBM結(jié)構(gòu)域的關(guān)鍵結(jié)合位點(diǎn)及其的催化功能的影響機(jī)制還有待進(jìn)一步研究。
本實驗室在前期的研究中獲得了1株耐熱普魯蘭酶產(chǎn)生菌Anoxybacillussp.LM18-11[11],并從中獲得了耐熱I型普魯蘭酶PluA,晶體結(jié)構(gòu)分析顯示PluA具有4個相對獨(dú)立的結(jié)構(gòu)域 (N1、N2、A、C),N末端存在兩個連續(xù)的CBM,N2 區(qū)屬于CBM48,而N1區(qū)不屬于已知的任何CBM家族,被CAZy首次認(rèn)定為一個新的CBM家族,即CBM68[12]。前期對CBM68結(jié)構(gòu)域的缺失實驗表明突變體的底物親和力、最適溫度以及熱穩(wěn)定性均較野生型有明顯降低[11],可見CBM68對PulA的催化性能起到了重要作用,因此本論文在PulA與小分子麥芽三糖共結(jié)晶的基礎(chǔ)上,根據(jù)序列比對及酶與小分子相互作用的結(jié)構(gòu)分析,在CBM68結(jié)構(gòu)域中選取4個可能與底物結(jié)合相關(guān)的氨基酸位點(diǎn)(Y14、D16、K62和R96),并對其分別進(jìn)行定點(diǎn)突變研究,揭示CBM68中的關(guān)鍵作用位點(diǎn)及其對普魯蘭酶催化性質(zhì)的影響,為進(jìn)一步探究新結(jié)構(gòu)域CBM68的作用機(jī)制奠定一定理論基礎(chǔ)。
1材料與方法
1.1材料
1.1.1菌種和質(zhì)粒
大腸桿菌DH5α和BL21trxB(DE3)、載體pET32a為本實驗室收藏。
1.1.2酶和主要試劑
Phusion熱啟動II高保真DNA聚合酶,ThermoFisher公司; 各種抗生素、普魯蘭糖、IPTG,Sigma公司;蛋白質(zhì)純化用HisTrap柱、XK16/70凝膠柱,GE公司;引物由中國科學(xué)院天津工業(yè)生物技術(shù)研究所合成。
1.1.3培養(yǎng)基
LB培養(yǎng)基(g/L):酵母粉 5,胰蛋白胨 10,NaCl10;添加 2% 瓊脂為固體培養(yǎng)基。
1.1.4定點(diǎn)突變引物

引物名稱引物序列(5’-3’)Y14AFGTCGTTTGAAGCTGCATTAGATGAAT-TGACRTGCAGCTTCAAACGACTGTTGTTTT-GD16AFGAAGCTTATTTAGCGGAATTGA-CAATGRCGCTAAATAAGCTTCAAACGACTGT-TGK62AFCATCATTCATTTATAGCGTATGAGT-GTCTCRCGCTATAAATGAATGATGCTTCT-CACTTGAR96AFGTAGGAGCTGTGATGGC-CACGAAAGCGTTTRGGCCATCACAGCTCCTACTTGCAATG-
1.2方法
1.2.1突變反應(yīng)
以pET32a-pulA為模板,采用Phusion熱啟動II高保真DNA聚合酶進(jìn)行PCR突變反應(yīng)。反應(yīng)體系(50 μL):10 μL 5× Phusion HF緩沖液,4 μL dNTPs(各2.5 mmol/L),1 μL 模板DNA,1.5 μL 正向引物,1.5 μL 反向引物,1.5 μL DMSO,0.5 μL Phusion熱啟動II高保真DNA聚合酶(2 U/μL),補(bǔ)加雙蒸水至50 μL。反應(yīng)條件:98 ℃預(yù)變性 3 min;98 ℃變性 10 s,55 ℃退火 30 s,72 ℃延伸 4 min,共30個循環(huán);72 ℃再延伸10 min。PCR產(chǎn)物經(jīng)1%瓊脂糖凝膠電泳進(jìn)行鑒定。
1.2.2篩選陽性克隆
電泳目的條帶位置正確的PCR產(chǎn)物在37 ℃經(jīng)Dpn I消化模板質(zhì)粒1 h后,轉(zhuǎn)DH5α感受態(tài),37 ℃培養(yǎng)12 h,挑選單克隆,提質(zhì)粒,經(jīng)1%瓊脂糖凝膠電泳進(jìn)行鑒定,將電泳目的條帶位置正確的質(zhì)粒送北京六合華大基因科技股份有限公司測序。
1.2.3蛋白表達(dá)與純化
基因位點(diǎn)突變正確的重組質(zhì)粒轉(zhuǎn)化到大腸桿菌BL21trxB(DE3),將含有突變重組質(zhì)粒的大腸桿菌BL21trxB(DE3)單菌落接種于4 mL液體LB(卡那霉素50 μg/mL、氨芐青霉素100 μg/mL)培養(yǎng)基中,于37 ℃、220 r/min下振蕩培養(yǎng)12 h,然后以1%的接種量轉(zhuǎn)接到200 mL含卡那霉素50 μg/mL和氨芐青霉素100 μg/mL的液體LB培養(yǎng)基中,37 ℃、220 r/min培養(yǎng)至OD600=0.6~0.8,加IPTG終濃度至0.05 mmol/L,在16 ℃、160 r/min下進(jìn)行蛋白誘導(dǎo)表達(dá),16 h后于4 ℃、5 000 r/min離心20 min收集菌體,高壓均質(zhì)破碎儀破碎細(xì)胞,蛋白經(jīng)His-Trap(5 mL)柱、XK16/70凝膠柱純化,純化后的蛋白經(jīng)適當(dāng)稀釋用SDS-PAGE進(jìn)行純度分析。
1.2.4普魯蘭酶酶活力測定
DNS方法[13]:在相應(yīng)的溫度下,取450 μL反應(yīng)緩沖液 (5%的普魯蘭糖溶液和相應(yīng)的pH緩沖液以1∶8的比例混合均勻),加入50 μL適當(dāng)稀釋的普魯蘭酶酶液混合均勻,反應(yīng)30 min,加入500 μL DNS溶液終止反應(yīng),100 ℃水浴10 min,測定OD540值。
酶活力單位定義為:在特定的反應(yīng)條件下,每分鐘水解普魯蘭糖產(chǎn)生相當(dāng)于1 μmol葡萄糖的還原糖,所需的酶量為1個活力單位(U)。
1.2.5突變酶最適pH值及pH穩(wěn)定性測定
采用pH 4.0~5.8乙酸-乙酸鈉緩沖液和pH 5.8~9.0磷酸氫二鈉-磷酸二氫鈉緩沖液分別配制pH 4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0緩沖液,取450 μL反應(yīng)緩沖液(5%的普魯蘭糖溶液和相應(yīng)的pH值緩沖液以1∶8的比例混合均勻),加入50 μL適當(dāng)稀釋的酶液,混合均勻后55 ℃保溫30 min,然后加入500 μL DNS溶液終止反應(yīng),100 ℃水浴10 min,測定OD540值。將普魯蘭酶液在以上不同pH值緩沖液中于4 ℃處理4 h,在最適條件下按以上方法測定殘余酶活力。
1.2.6突變酶最適溫度及耐熱穩(wěn)定性測
在35~80 ℃范圍內(nèi)測定突變酶最適溫度,取450 μL反應(yīng)緩沖液(5%的普魯蘭糖溶液和相應(yīng)的pH 6.0磷酸緩沖液以1∶8的比例混合均勻),加入50 μL適當(dāng)稀釋后的酶液,混合均勻后分別在35、40、45、50、55、60、65、70、75、80 ℃下保溫反應(yīng)30 min,然后加入500 μL DNS溶液終止反應(yīng),100 ℃水浴10 min,測定OD540值。另外,取適當(dāng)稀釋的突變酶500 μL加入10 μL石蠟油覆蓋酶液,置于60 ℃水浴鍋溫育不同時間,在最適條件下按以上方法測定殘余酶活力。
1.2.7突變酶底物特異性分析[14-15]
取50 μL適當(dāng)稀釋的酶液與400 μL磷酸緩沖液(20 mmol/L 、pH 6.0)混合,分別以5%的普魯蘭糖、可溶性淀粉、支鏈淀粉、糖原做反應(yīng)底物,反應(yīng)體系底物終濃度為5 mg/mL,55 ℃保溫30 min,然后加入500 μL DNS溶液終止反應(yīng),100 ℃水浴10 min,測定OD540值。以最高酶活力為100%,計算不同底物的相對酶活力。
1.2. 8突變酶動力學(xué)分析
取450 μL用pH 6.0磷酸緩沖液配制的不同濃度的普魯蘭糖溶液(反應(yīng)緩沖液中普魯蘭糖的終質(zhì)量濃度為1.0,1.25,1.5,2.0,2.5,3和5.0 mg/mL),在55 ℃預(yù)熱5 min后加入50 μL適當(dāng)稀釋的突變蛋白酶液,混合均勻后繼續(xù)保溫10 min,然后加入500 μL DNS溶液終止反應(yīng),100 ℃水浴10 min,測定OD540值。建立1/V-1/[S]的雙倒數(shù)曲線,計算其Km值、Vmax值、kcat值及kcat/Km值。
2結(jié)果與分析
2.1普魯蘭酶突變體表達(dá)菌株的構(gòu)建
以pET32a-PulA質(zhì)粒為模板,應(yīng)用1.1.4所示的定點(diǎn)突變引物,經(jīng)Phusion熱啟動II高保真DNA聚合酶進(jìn)行PCR突變反應(yīng),得到相應(yīng)的突變質(zhì)粒,質(zhì)粒全長8kb左右 (圖1)。經(jīng)基因測序,得到突變正確的重組質(zhì)粒分別命名為pET32a-PulAY14A、pET32a-PulAD16A、pET32a -PulAK62A和pET32a-PulAR96A(圖2)。然后將測序正確的突變質(zhì)粒分別轉(zhuǎn)化到E.coliBL21trixB(DE3)中,成功構(gòu)建了突變酶重組表達(dá)菌株E.coliBL21trixB(pET32a-PulAY14A)、E.coliBL21trixB(pET32a-PulAD16A)、E.coliBL21trixB(pET32a-PulAK62A)和E.coliBL21trixB(pET32a-PulAR96A)。
M1、M2、M3、M4: DNA marker 1: pET32a-PulAY14A 2: pET32a-Pu-lAD16A3: pET32a-PulAK62A 4: pET32a- PulAR96A圖1 Phusion DNA聚合酶定點(diǎn)突變PCR反應(yīng)結(jié)果Fig.1 Gel electrophoresis of mutant products using Phusion DNA polymerase
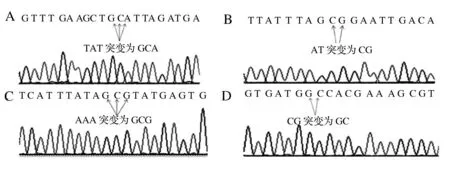
圖2 陽性克隆突變位點(diǎn)的測序圖 Fig.2 The DNA sequences of pullulanase mutants surrounding mutagenesis position圖示為正義鏈序列, 箭頭所指為定點(diǎn)突變核苷酸;A:pET32a-PulAY14A中PulA的14位酪氨酸TAT突變?yōu)楸彼酖CA;B:pET32a-PulAD16A中PulA的16位天冬氨酸GAT突變?yōu)楸彼酖CG;C:pET32a-PulAK62A中PulA的62位賴氨酸AAA突變?yōu)楸彼酖CG;D:pET32a-PulAR96A中PulA的96位精氨酸CGC突變?yōu)楸彼酖CC。
2.2普魯蘭酶PulA及其突變酶的誘導(dǎo)表達(dá)與純化
將得到的大腸桿菌重組工程菌分別進(jìn)行搖瓶培養(yǎng),應(yīng)用IPTG誘導(dǎo)重組普魯蘭酶的表達(dá),然后應(yīng)用Ni2+親和柱、XK16/70凝膠柱對重組普魯蘭酶進(jìn)行純化。純化后的蛋白經(jīng)SDS-PAGE蛋白電泳,結(jié)果如圖3所示,PulA與各點(diǎn)突變普魯蘭酶均得到了較好的純化,其蛋白分子量均為100 kDa左右。將點(diǎn)突變后的普魯蘭酶分別命名為PulAY14A、PulAD16A、PulAK62A和PulAR96A。
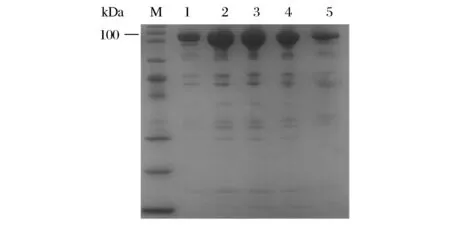
M-protein marker;1-PulA;2-PulAY14A;3-PulAD16A;4-PulAK62A;5-PulAR96A圖3 純化后普魯蘭酶PulA及其突變體的SDS-PAGE分析Fig.3 The SDS-PAGE analysis of PulA and its mutants
2.3氨基酸突變位點(diǎn)對普魯蘭酶最適pH值及其pH穩(wěn)定性的影響
將純化后的普魯蘭酶PulA和4個點(diǎn)突變的普魯蘭酶分別在不同溫度下測定酶活,結(jié)果如圖4a所示。突變酶PulAY14A、PulAD16A和PulAK62A在pH 4.5~8.0范圍內(nèi)顯示了與PulA相似的趨勢曲線,最適作用pH均為6.0,而突變酶PulAR96A的最適作用pH為5.5,并且在pH 5.5條件下,4個突變酶的相對酶活均高于野生型普魯蘭酶PulA。同時,4個突變普魯蘭酶在不同pH下的穩(wěn)定性曲線均與野生型普魯蘭酶PulA相似 (圖4b)。可見,除了R96A突變位點(diǎn)外,其余3個氨基酸位點(diǎn)的突變對PulA的最適pH及pH穩(wěn)定性的影響并不明顯,而R96A突變使得PulA的最適作用pH向酸性偏移了0.5個單位,說明R96位點(diǎn)對PulA的酸堿催化過程具有重要影響。
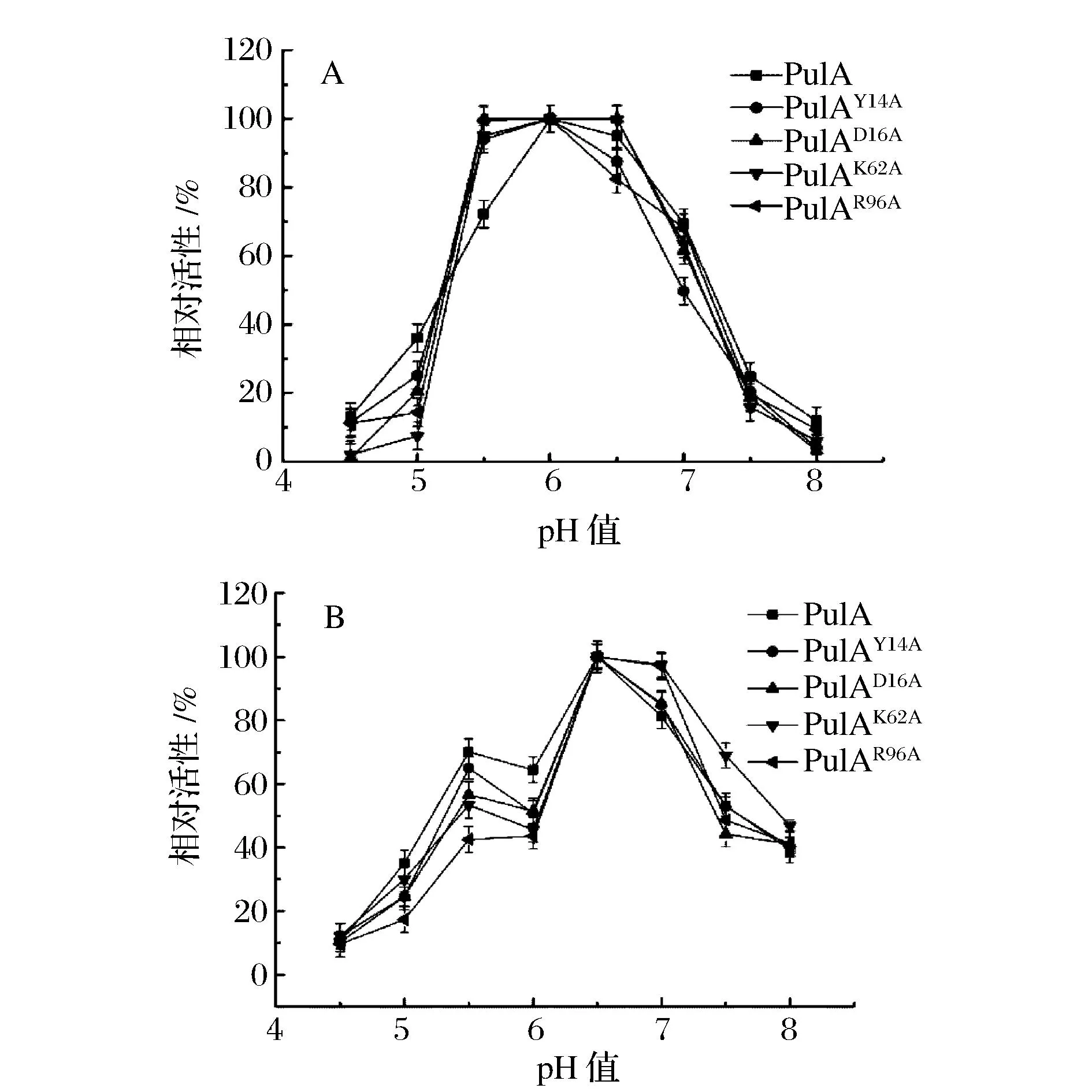
圖4 重組普魯蘭酶最適作用pH及pH穩(wěn)定性Fig.4 The optimum pH and pH stability of the recombinant pullulanases
2.4氨基酸突變位點(diǎn)對普魯蘭酶最適溫度及溫度穩(wěn)定性的影響
重組普魯蘭酶在不同溫度下測定酶活力,結(jié)果如圖5a所示,PulAD16A和PulAK62A的最適作用溫度為55~60 ℃,和PulA的最適作用溫度相同,而PulAY14A和PulAR96A的最適作用溫度為50 ℃,并且PulAR96A在60 ℃時相對酶活降到50%。另外,PulAR96A在60 ℃的半衰期為8 h,比PulA的半衰期48 h降低了5倍 (圖5b)。當(dāng)保溫時間到達(dá)25 h時,PulAR96A幾乎完全失活。可見,Y14A和R96A的點(diǎn)突變可使PulA的耐熱性降低,尤其是R96A的作用最為明顯。經(jīng)分析,將PulA96位側(cè)鏈為長鏈的精氨酸突變?yōu)槎替湹谋彼幔赡芨淖兞说鞍椎慕Y(jié)構(gòu)柔性,使其在高溫下維持蛋白三級結(jié)構(gòu)的能力降低,蛋白分子更容易變性,從而導(dǎo)致該突變酶的最適溫度和熱穩(wěn)定性都顯著降低[16]。
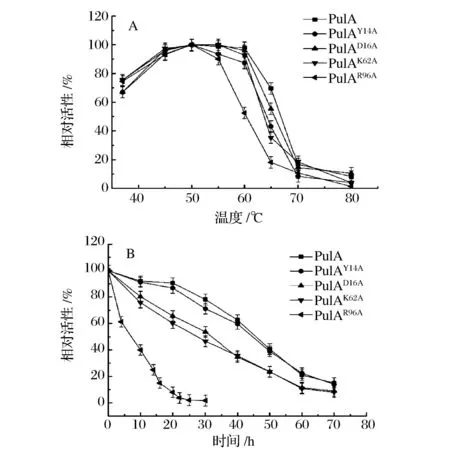
圖5 重組普魯蘭酶的最適作用溫度及耐熱穩(wěn)定性Fig.5 The optimum temperature and thermostability of the recombinant pullulanases
2.5氨基酸突變位點(diǎn)對普魯蘭酶底物特異性的影響
分別選取普魯蘭糖、支鏈淀粉、可溶性淀粉以及糖原為底物在普魯蘭酶的最適作用條件下測定酶活 ,結(jié)果如圖6所示,4個突變普魯蘭酶與PulA一樣,均對普魯蘭糖顯示了最高催化活性,對支鏈淀粉和可溶性淀粉催化活性相對較低,對糖原幾乎沒有催化作用。其中,突變體PulAY14A對支鏈淀粉和可溶性淀粉的催化活性完全喪失,可見14位上這種高保守型的芳香族氨基酸(Tyr)對底物特異性具有重要影響。另外,PulAD16A與PulA相比,其對支鏈淀粉的催化活性幾乎降低為零,而對可溶性淀粉的催化作用卻明顯增強(qiáng)了。而PulAK62A和PulAR96A對支鏈淀粉的催化活性完全喪失,對可溶性淀粉的催化作用影響不大。綜上,Y14A和R96A定點(diǎn)突變不但影響該酶的作用溫度及pH,對其底物特異性也具有顯著影響,而D16A和K62A定點(diǎn)突變雖然促使該酶的底物特性發(fā)生了一定變化,但對該酶其他性質(zhì)影響不大,可見,4個位點(diǎn)的突變對該酶酶學(xué)性質(zhì)和底物特異性的影響機(jī)制可能是不同的。
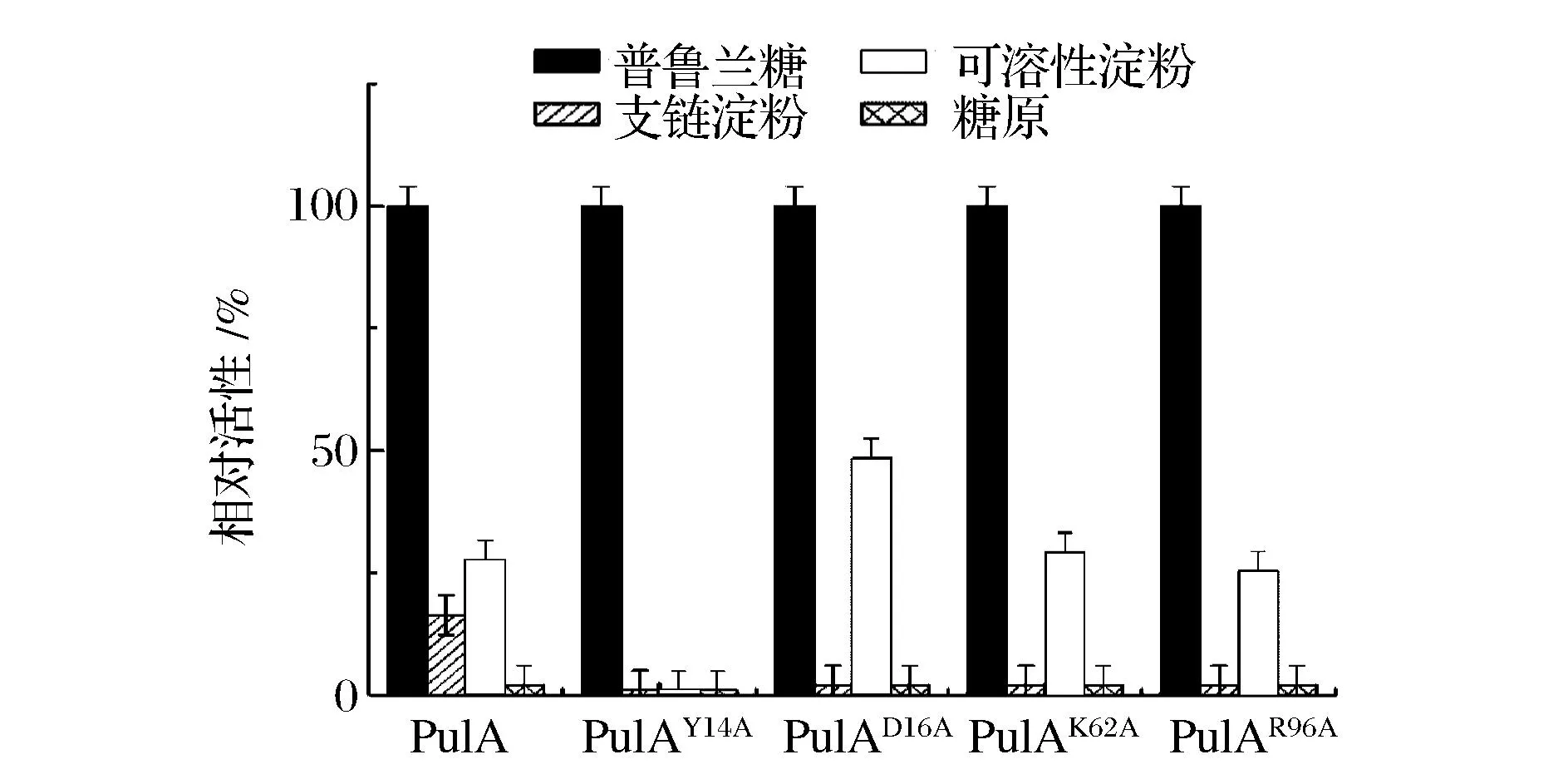
圖6 普魯蘭酶PulA及其突變體的底物特異性Fig.6 Substrate specificity of PulA and its mutants
2.6氨基酸突變位點(diǎn)對普魯蘭酶動力學(xué)參數(shù)的影響
以普魯蘭糖為底物分別對重組普魯蘭酶動力學(xué)參數(shù)進(jìn)行測定和比較,結(jié)果如表1所示,PulAD16A和PulAK62A的動力學(xué)參數(shù)與PulA非常接近,說明D16A和 K62A突變對PulA底物親和力及催化效率沒有影響。而PulAY14A和PulAR96A的Km值分別比PulA提高了4.2倍和2.5倍,kcat/Km分別為PulA的29%和37%。可見,Y14A和R96A突變嚴(yán)重降低了PulA的底物親和力及催化效率。說明Y14和R96是PulA中CBM68結(jié)構(gòu)域的關(guān)鍵氨基酸位點(diǎn)。將CBM68結(jié)構(gòu)域的保守芳香族氨基酸Y突變成A,對底物的親和力性影響較大,Y的苯環(huán)與環(huán)狀糖分子之間靠范德華力形成堆疊作用,可能決定酶與底物結(jié)合的方式及親和力[16]。而PulAR96A親和力顯著降低可能是因氨基酸側(cè)鏈變短,致使蛋白結(jié)構(gòu)柔性增大[10],從而降低其底物親和力。至于這兩個突變體催化效率的明顯降低,很可能是親和力的降低促使底物被運(yùn)送到活性中心的速率降低所致。
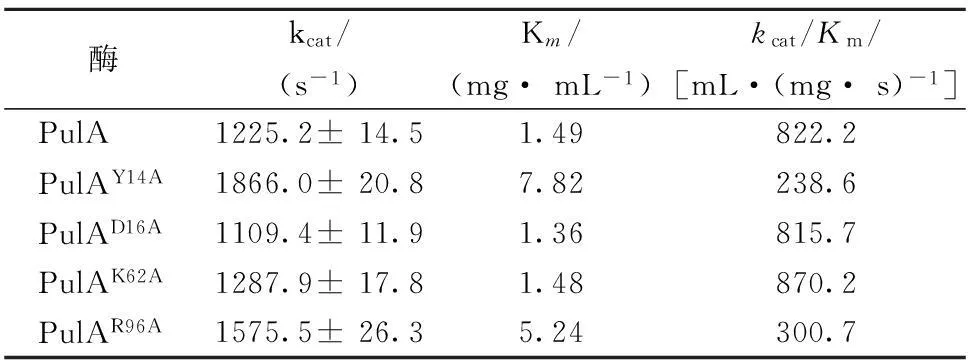
表1 普魯蘭酶PulA及其突變體動力學(xué)參數(shù)
3結(jié)論
本論文為研究耐熱普魯蘭酶PulA中新鑒定的底物結(jié)合結(jié)構(gòu)域CBM68的作用機(jī)制,成功構(gòu)建了4個單點(diǎn)突變的普魯蘭酶突變體PulAY14A、PulAD16A、PulAK62A和PulAR96A,經(jīng)底物特異性、動力學(xué)參數(shù)及其他酶學(xué)性質(zhì)的測定與對比分析,發(fā)現(xiàn)Y14和R96是CBM68中的兩個關(guān)鍵氨基酸位點(diǎn),對這兩個關(guān)鍵氨基酸位點(diǎn)的突變不僅顯著影響普魯蘭酶PulA的底物親和力及催化效率,也對其作用溫度、pH及穩(wěn)定性具有一定的影響,然而具體的影響機(jī)制仍需進(jìn)一步揭示。
參考文獻(xiàn)
[1]DUAN Xu-guo,WU Jin.Enhancing the secretion efficiency and thermostability of a Bacillus deramificans pullulanase mutant (D437H/D503Y) by N-terminal domain truncation[J].Appl Environ Microbiol,2015,81(6):1 926-1 931.
[2]姜楠,宋詼,王萍.普魯蘭酶及其分泌相關(guān)蛋白的研究進(jìn)展[J].微生物學(xué)報,2011,51(6):725-731.
[3]SIEW Ling Hii,JOO Shun TAN,TAU Chuan Ling,et al. Pullulanase:Role in starch hydrolysis and potential industrial applications[J].Enzyme Res,2012,2012:921 362.
[4]BORASTON A B,HEALEY M,KLASSEN J,et al. A structural and functional analysis of alpha-glucan recognition by family 25 and 26 carbohydrate-binding modules reveals a conserved mode of starch recognition[J].Biol Chem, 2006,281(1):587-98.
[5]MIKAMI B,IWAMOTO H,MALLE D,et al. Crystal structure of pullulanase:evidence for parallel binding of oligosaccharides in the active site[J].Mol Biol,2006,359(3):690-707.
[6]TURKENBURG J P,BRZOZOWSKI A M,SVENDSEN A,et al. Structure of a pullulanase from Bacillus acidopullulyticus[J].Proteins,2009,76(2):516-519.
[7]VENDITTO I, NAJMUDIN S,Lus A S,et al. Family 46 carbohydrate-binding modules contribute to the enzymatic hydrolysis of xyloglucan and β-1,3-1,4-Glucans through distinct mechanisms[J]. Journal of Biological Chemistry,2015,290(17):10 572-10 586.
[8]STROBEL K L,PFEIFFER K A,HARVEY W B,et al. Structural insights into the affinity of Cel7A carbohydrate-binding module for lignin[J].Journal of Biological Chemistry,2015,290(37):22 818-22 826.
[9]HARA M,SUGIMOTO H,UEMURA M,et al. Involvement of Gln679,in addition to Trp687,in chitin-binding activity of the chitin-binding domain of chitinase A1 from Bacillus circulans WL-12[J].Journal of Biochemistry, 2013,154(2):185-193.
[10]DUAN Xu-guo,CHEN Jian,WU Jing.Improving the thermostability and catalytic efficiency of Bacillus deramificans pullulanase by site-directed mutagenesis[J].Applied and Environmental Microbiology,2013, 79(13):4 072-4 077.
[11]甄杰,胡政,李樹芳,等.一個新型耐熱普魯蘭酶的結(jié)構(gòu)與功能[J].生物工程學(xué)報,2014,30(1):119-128.
[12]XU Jiang-yong,REN Fei-fei,HUANG Chun-hsiang,et al. Functional and structural studies of pullulanase from Anoxybacillus sp. LM18-11[J].Proteins,2013,82(9):1 685-1 693.
[13]李紅,孫紅軍.國內(nèi)外普魯蘭酶市場分析及預(yù)測報告(2003、2004年度)[R].北京:理德斯普企業(yè)管理咨詢有限公司,2004.
[14]ZHOU Cheng,XUE Yan-feng,ZHANG Yue-ling,et al. Recombinant expression and characterization of Thermoanaerobacter tengcongensis MB4 thermostable α-glucosidase with regioselectivity for high yield isomaltooligosaccharides synthesis[J].Journal of Microbiology and Bchnology,2009,19(12):1 547-1 556.
[15]CHEN C,XUE Yan-fen,MA Yan-he.Enhancing the thermostability of α-glucosidase fromThermoanaerobactertengcongensisMB4 by single proline substitution[J].Bioscience and Bioengineering,2010,110(1):12-17.
[16]GUILLEN D,S SANCHEZ,RODRIGUEZ-SANOJA R.Carbohydrate-binding domains: multiplicity of biological roles[J]. Appl Microbiol Biotechnol,2010,85(5):1 241-1 249.
Key amino acid sites in the CBM68 structure of thermostable pullulanase and their effects on the enzymatic properties
SHEN Ying-ying1,2,3,ZHENG Hong-chen2,3,LI Shu-fang2,3,F(xiàn)U Xiao-ping2,3,XU Jian-yong2,3,SONG Hui2,3*
1(College of Biotechnology, Tianjin University of Science and Technology, Tianjin 300457, China)2(Industrial Enzymes National Engineering Laboratory, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China)3(Key Laboratory of Tianjin Industrial Biosystem and Process Engineering, Tianjin Institute of Industrial Biotechnology, Chinese Academy of Sciences, Tianjin 300308, China)
ABSTRACTBased on the analysis of the crystalline structure of pullulananse (PulA)-maltotetraose complex, four key amino acid sites (Y14, D16, K62 and R96) in a new carbohydrate-binding module CBM68 closely related to substrate binding were discovered. The thermostable pullulanase (PulA) was derived from Anoxybacillus sp. LM18-11. Four mutants of PulA(Y14A), PulA(D16A), PulA(K62A) and PulA(R96A) were obtained respectively by site-directed mutation. Among them, PulA(Y14A) and PulA(R96A) showed lower affinity for substrate and lower catalytic efficiency compared with the wild-type pullulanase (PulA). The Km values of PulA(Y14A) and PulA(R96A) were 4.2 times and 2.5 times higher than that of PulA, respectively. The k(cat)/Km values of PulA(Y14A) and PulA(R96A) only remained 29% and 37% of the original k(cat)/Km value (PulA), respectively. Moreover, the optimum temperatures for both PulA(Y14A) and PulA(R96A) were 10 ℃ lower than that for the wild-type pullulanase (PulA). And PulA(R96A) also showed decreasing of the optimum pH by 0.5 units. All the results in this work have proved the significance of the key amino acid sites Y14 and R96 on the structure CBM68. Further study on the function of the two key amino acid sites could enhance the understanding of the functional mechanism of CBM68.
Key wordsthermostable pullulanase; CBM68; site-directed mutation; substrate affinity; catalytic effeciency
收稿日期:2015-11-06,改回日期:2015-12-04
基金項目:國家863計劃(2012AA022205);天津市科學(xué)基金(13JCYBJC39500)
DOI:10.13995/j.cnki.11-1802/ts.201603003
第一作者:碩士研究生(宋詼研究員為通訊作者,E-mail:song_h@tib.cas.cn)。

