濃香型白酒窖泥變質前后古菌群落差異分析
于春濤,項 明,王 蕾,許鄭林,劉振江
(1.滄州醫學高等專科學校,河北滄州061001; 2.河北省寧晉縣泥坑酒業有限責任公司,河北石家莊054000)
濃香型白酒窖泥變質前后古菌群落差異分析
于春濤1,項明1,王蕾1,許鄭林1,劉振江2
(1.滄州醫學高等專科學校,河北滄州061001; 2.河北省寧晉縣泥坑酒業有限責任公司,河北石家莊054000)
窖泥是濃香型白酒的生命線,窖泥變質則為酒廠帶來巨大損失。采用Illumina Miseq高通量測序DNA技術對窖泥變質前后古菌群落結構進行分析。通過構建古菌16S rDNA基因文庫,結果表明,變質后窖泥共擴增出52876條序列,變質前窖泥擴增出11703條序列,古菌豐度有明顯提高。對序列進行聚類、測序、分析,窖泥變質前優勢古菌為:甲烷短桿菌屬(35.66%)、甲烷桿菌屬(10.25%)、類芽孢桿菌屬(4.27%)、芽孢桿菌屬(3.39%)、甲烷八疊球菌屬(3.27%)等;窖泥變質后優勢菌為:產甲烷袋菌屬(38.18%)、甲烷桿菌屬(34.21%)、甲烷袋狀菌屬(6.90%)等。結果揭示了甲烷菌是窖泥變質前后古菌的優勢菌,為進一步研究窖泥微生物全貌及窖泥培養、養護提供依據。
窖泥; 變質; 古菌群落; 差異分析; 高通量測序; 白酒
窖泥是濃香型白酒生產的基礎,窖泥微生物通過復雜的物質能量代謝,產生具有獨特風味和品質的白酒,因此窖泥的好壞直接影響產酒的質量。窖泥微生態中的微生物多樣性十分復雜,包括細菌、真菌及古菌,這些微生物群落會隨著生長環境的變化(如溫度、濕度、pH值、C/N等)而變化[1]。較早開展濃香型白酒微生物研究的是法國人A.Calmette的探索性實驗——“阿米露法”[2]。目前我國學者在窖泥功能菌研究方面取得了不少可喜成績,主要以細菌為主,真菌和古菌研究較少,如鄧依[3]等通過細菌rDNA ITS-AFLP結合聚類分析技術比較了多糧發酵新老窖池間的細菌群落差異,羅惠波[4]通過PCR-SSCP技術研究窖池微生物群落等。
由于北方四季溫差大,氣候干燥,窖泥中微生物菌群演替快,再加上管理不善,致使窖泥中微生態平衡被破壞,代謝出現異常,導致窖泥出現板結變硬、腐敗氣味等變質現象[5],用于生產致使酒質下降,給酒廠帶來了巨大損失。已有研究表明,一些古菌在濃香型白酒“產香”過程中起著重要作用[6]。因此弄清窖泥古菌群落的動態變化,檢測出窖泥變質前后的主要優勢古菌,對人工窖泥培養和提高濃香型白酒質量尤為重要。但由于古菌獨特的生長方式,窖泥中大部分古菌是未培養的,即使得到了純培養,其形態和生理也可能發生了很多變化[7],因此,采用傳統的分離培養方法研究窖泥古菌存在很大的局限性[8-9]。
近幾年發展起來一項全新的高通量DNA測序技術——Illumina Solexa合成測序,能檢測出土壤中80%以上的優勢菌群,具有高準確性、高通量、高靈敏度和低運行成本等突出優勢,可以同時分析樣本內菌群豐度和樣本間菌群豐度差異,已廣泛應用于探索土壤、海洋和濕地,以及食品發酵和廢水處理反應器等微生物區系研究,并取得了豐碩的研究成果[10-11]。目前,利用Illumina Solexa高通量DNA測序技術對窖泥中古菌群落結構的研究還未見文獻報道。鑒于此,本實驗通過Illumina Solexa高通量DNA測序技術,分析了濃香型白酒窖泥變質前后古菌群落差異,以期全面、系統認識窖泥微生物群落組成與窖泥質量之間的關系,為揭示窖泥微生態全貌提供幫助。
1 材料與方法
1.1樣品采集
窖泥樣品取自河北某知名酒廠釀酒車間優質窖泥(HQ1)和變質窖泥(HU1)各100 g。分裝后密封冷凍保存。
1.2實驗方法
1.2.1窖泥預處理及總DNA提取[12]
稱取200 mg的樣品,放入滅菌的2 mL離心管中,加入1 mL70%vol乙醇,振蕩混勻,10000 r/min室溫離心3 min,棄置上層液體。加入1 xPBS溶液,振蕩混勻,10000 r/min室溫離心3 min,棄置上層液體。倒置2 mL管于吸水紙上1 min,直至沒有液體流出。將樣品管放入55℃烘箱中維持10 min,使殘留酒精完全揮發,保證后續實驗操作。
DNA提取步驟按OMEGA試劑盒E.Z.N.ATM Mag-Bind Soil DNAKit的試劑盒使用說明書。
1.2.2PCR擴增及對產物進行瓊脂糖凝膠電泳
利用Qubit2.0 DNA檢測試劑盒對基因組DNA精確定量,以確定PCR反應應加入的DNA量。古菌引用槽式PCR擴增3輪,第1輪使用M-340F,GU1ST-1000R引物擴增:古1st-340F CCCTAYGGGGYGCASCAG,古1st-1000R GGCCATGCACYWCYTCTC,PCR體系按照如下進行:10×PCR buffer 5 μL,dNTP(10 mM each)0.5 μL,Genomic DNA 10 ng,Bar PCR primer F(50 μM)0.5 μL,Primer R(50 μM)0.5 μL,Plantium Taq(5 U/μL)0.5 μL,H20 add to 50 μL。配制好的PCR體系按照如下反應條件進行擴增:94℃變性3 min,接下來5個循環,分別是94℃變性30 s,45℃退火20 s,65℃延伸30 s;再接著20個循環,分別是94℃變性20 s,55℃退火20 s,72℃延伸30 s,72℃延伸5 min,然后進行第2輪擴增,使用第1輪PCR產物進行擴增,PCR所用的引物已經融合了Miseq測序平臺的V3—V4通用引物,349F引物:CCCTACACGACGCTCTTCCGATCTN(barcode)GYGCASCAGKCGMGAAW,806R引物GACTGGAGTTCCTTGGCACCCGAGAATTCCAGGACTACVSGGGTATCTAAT,PCR體系和擴增條件均同第1輪。第3輪擴增引入Illumina橋式PCR兼容引物。PCR結束后,產物進行瓊脂糖凝膠電泳,對DNA進行回收。
1.2.316S rDNA擴增片段的測序、質控和聚類
采用Misep測序平臺對擴增片段進行雙端測序,測得的基因序列經質量控制軟件Prinsep處理,去除barcode、兩端primer以及部分低質量基因序列,經質控后的基因序列長度大部分分布在400~600 bp之間,基本滿足分析需要。
對測得的基因序列采用uclust軟件進行OUT聚類,通常域值的序列相似性定位0.97,操作分類單元被認為可能屬于屬。
水壓試驗應注意事項:試壓時管道內不應有氣泡,否則當管道發生漏水時,會導致壓力下降現象,不易從壓力表上反映出來;管道升壓過程中,壓力表搖擺不定,擺幅較大,讀數不穩,且升壓又較慢時,示意管道內氣體沒有排盡,應重新排氣再升壓;管道內注水后,必須讓其充分浸泡,才能保證管道試壓準確;管道頂部土方回填時,宜留出接口位置,以方便檢查和修理;當管道有壓力時,檢查管道不得用手錘敲打管壁和接口,嚴禁修理管道缺陷,遇有缺陷,應做出標志,卸壓后再修補;水壓試驗過程中,管道危險區域,例如后背、支撐、管端等嚴禁站人,確保人員安全。
1.2.4對處理后序列進行物種分類,比較窖泥變質前后菌群結構差異
物種分類采用的軟件為RDP classifier,基于OUT聚類的結果,獲取每一個OUT聚類的代表序列,分別是長度最長序列(length)、豐度最大序列(abundance)和所有序列(ALL)形成3份結果,并對各類RDP分析[13-14]。本文所有的展示,均使用OUT_ALL中的數據,genus水平進行展示,采用柱形圖及表格的形式對比窖泥變質前后群落結構差異。
1.2.5MEGAN分析
使用MEGAN軟件,通過交互式搜索NCBI中的分類數據庫信息,以樹狀圖形式表現物質豐度情況與菌落結構,反應窖泥變質前后真菌的組成情況。
2 結果與分析
2.1窖泥變質前后古菌16S rDNA的PCR產物電泳圖譜(見圖1)
結合圖1中的3條泳道對比可知,泳道1和泳道2有較清晰的條帶,條帶大小位于400~600 bp之間,基本能夠滿足分析需要。DNA提取時一般都通過試劑盒過柱提取,這種柱子有固定孔徑,從而決定了基因組片段的大小。
2.2窖泥變質前后16S rDNA多樣性分析
擴增后的PCR產物去除嵌合體和靶區域序列,得到的基因序列數見表1。
將多條序列按其序列間的距離對它們進行聚類,后根據序列之間的相似性作為域值分成操作分類單元(OTU),通常域值的序列相似性定為0.97,操作分類單元被認為屬于屬[15-16]。采用OUT VENN分析圖統計樣本中共有的和獨有的OUT數目,直觀展示出窖泥變質前后OUT數目的差異(圖2)。HU1有1218個OTUs,HQ1有783個OTUs,兩者共有192個OTUs。窖泥變質后,古菌的種類和豐度有所提高。說明窖泥變質后的生境更適合一些古菌的生長。
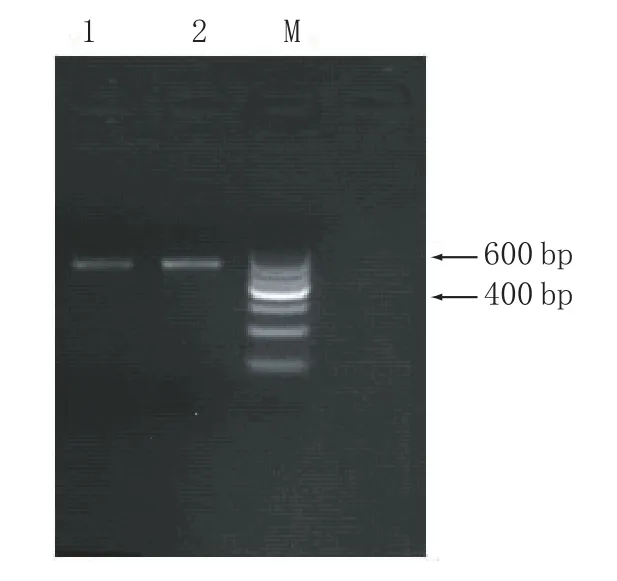
圖1 古菌16S rDNA電泳圖譜

表1 處理后基因序列統計表
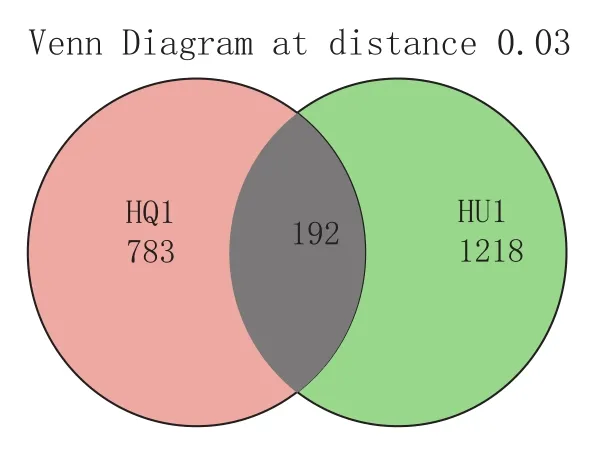
圖2 窖泥變質前后OUT數
2.3古菌群落結構分析
對處理后序列,采用RDP classifier軟件對每條序列在genus水平上計算其分配到此rank中的概率值,一般概率值大于0.8,即RDP分類域值。根據分類學分析結果,可以得知樣品在屬分類水平的數據,樣本中菌群的reads數目,也就是菌群的豐度值(表2、表3)。
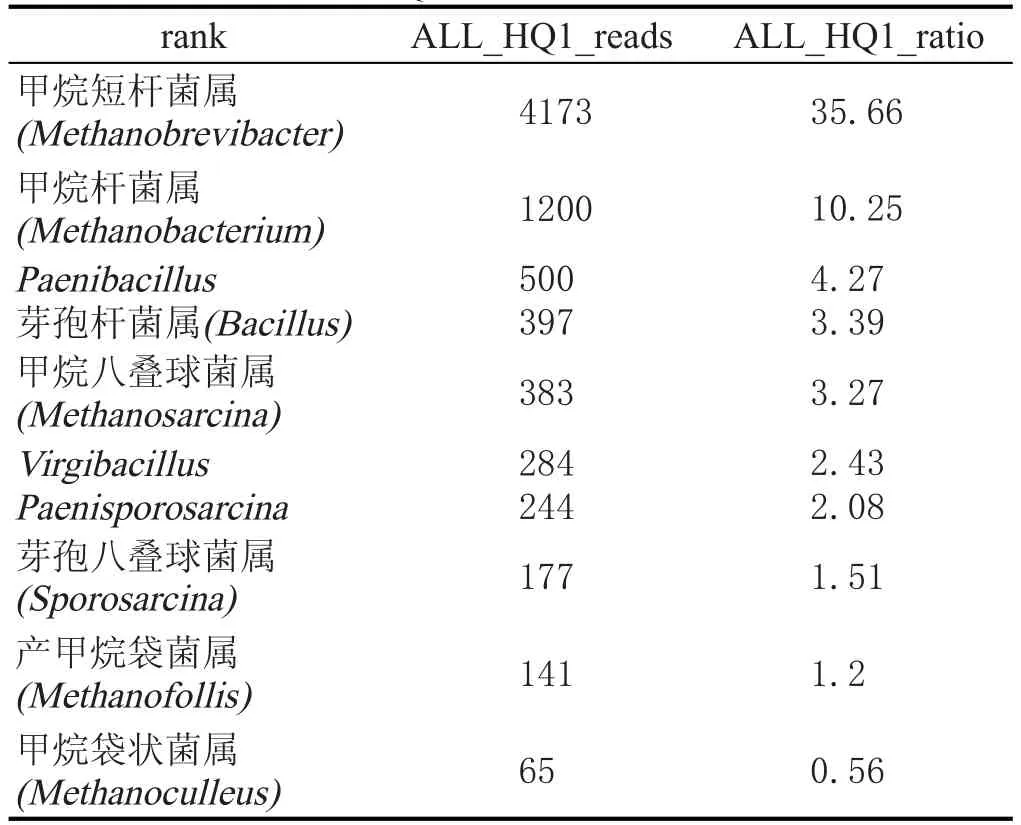
表2 HQ1古菌種類及豐度值
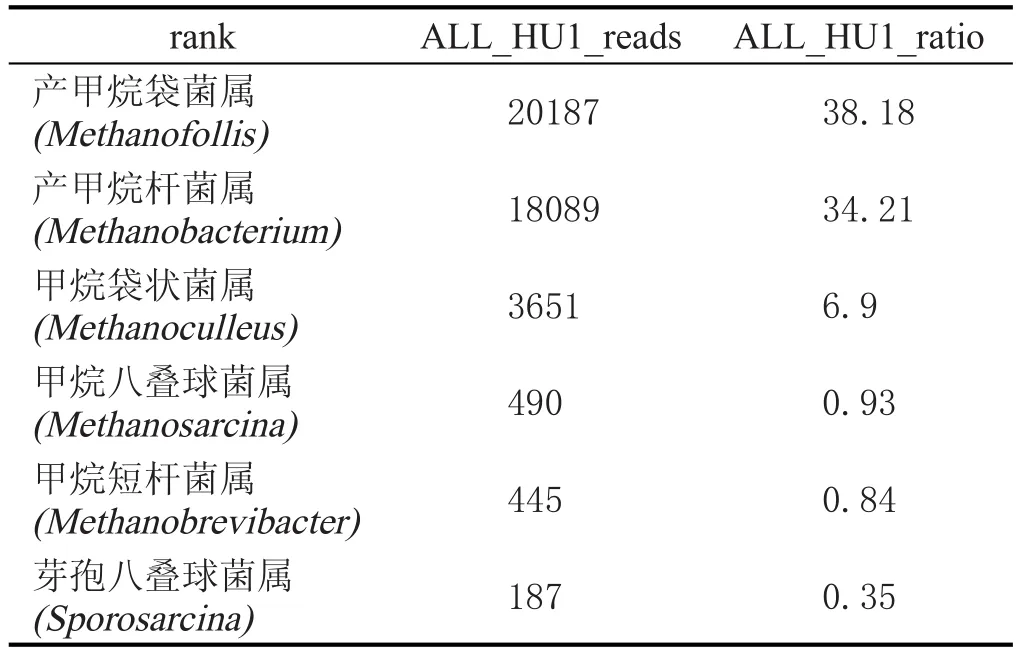
表3 HU1古菌種類及豐度值
由表2、表3可知,窖泥變質前后的古菌群落結構、豐度差異顯著,選擇優勢菌群進行比較。窖泥變質前的絕對優勢菌屬為甲烷短桿菌屬(35.66%)和甲烷桿菌屬(10.25%),窖泥變質后產甲烷袋菌屬(38.18%)和產甲烷桿菌屬(34.21%)成為絕對優勢菌屬。Thomton[17]認為:在原始土壤中,部分優勢菌種的存在都是有特定的有利于其生長環境的結果,這個結論普遍被人們所接受。窖泥中古菌群落結構發生變化,歸根結底是由于窖泥生境的變化導致。Widden[18]認為,土壤生境的變化很復雜,其中包括生物因素和非生物因素,非生物因子如Ca2+、溫度、濕度、K+、窖泥的形成過程等,可能是少數或幾個主要真菌種出現的首要調節者。但有更多的證據證實生物因子有可能是主要的調節者,Gochenaur[19]研究后得出結論:窖泥中微生物種間競爭,將會導致微生態群落發生演替,適者生存。窖泥變質后產甲烷袋菌屬及產甲烷桿菌屬更顯示出對環境的適應性。
已有研究表明[20],古菌發酵有促進己酸發酵進程多產己酸的作用,從而增加己酸乙酯含量,提高酒質。隨著釀造周期的延長,窖泥中的古菌經歷了一個長期馴化、變化的過程,古菌的群落結構會隨著窖泥生境的改變而發生改變。甲烷菌作為窖泥古菌域的優勢菌,其以乙酸、H2、CO2作為營養,這幾種物質窖泥中很豐富,因此,甲烷菌群落結構變化與營養關系不大。許寶孝等[21]研究認為,甲烷短桿菌生長和產甲烷的最適溫度為40℃,最適pH值為7.6。窖泥在變質后pH值小于5.5,抑制甲烷短桿菌的生長,而產甲烷袋狀菌和產甲烷桿菌則表現出適應這種酸性環境。
2.4窖泥變質前后古菌分類樹
使用MEGAN軟件,通過交互式搜集NCBI中的分類數據庫信息,以樹狀圖形式表現窖泥變質前后古菌群落結構(圖3、圖4)。
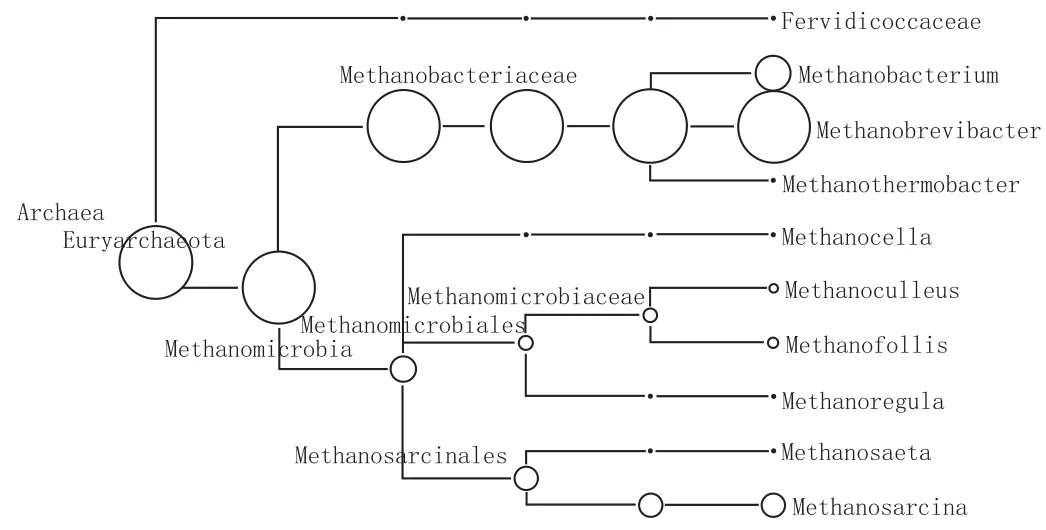
圖3 HQ1屬水平MEGANF豐度圖
如圖3所示,窖泥變質前古菌群落有廣古菌門(Euryarchaeota)和泉古菌門(Crenarchaeota)的各級菌屬,主要是甲烷菌。甲烷菌在自然界中分布很廣,如人類消化系統、水稻田、湖泊、濕地污泥等[22]。目前大多數產甲烷古菌依然沒有被分離,關于這些菌在窖泥中的生理、微生態之間的關系不得而知。甲烷菌在厭氧條件下合成甲烷,形成維持細胞生存所需的能量,合成甲烷的底物有3種:乙酸占60%以上、H2和CO2占30%,甲基化合物占10%,這些底物在窖泥中很豐富,因此窖泥中還有數量眾多、種類多樣的甲烷菌。
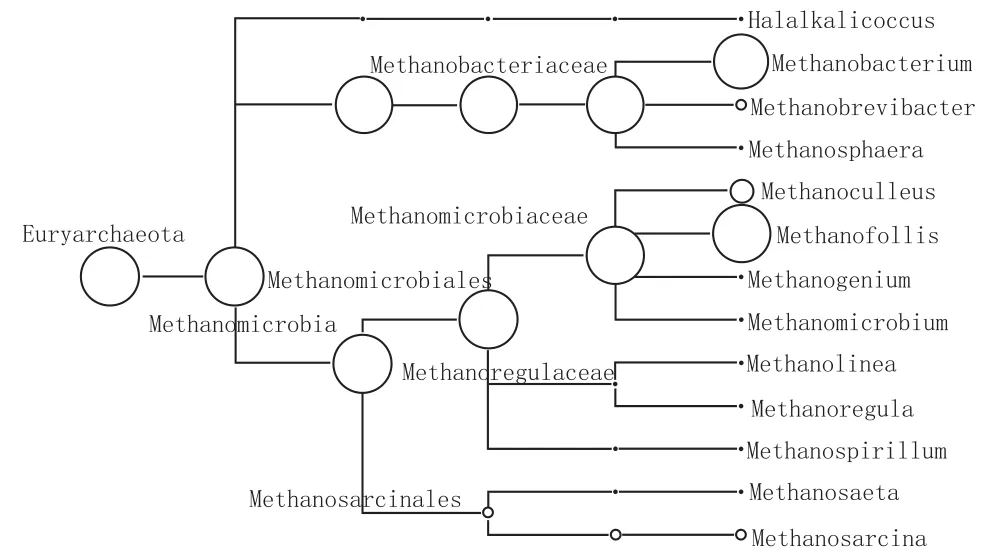
圖4 HU1屬水平MEGANF豐度圖
如圖4所示,窖泥變質后古菌群落主要由廣古菌門(Euryarchaeota)構成,甲烷菌依然是優勢菌屬,只是種屬間豐度發生了較大的變化。由于古菌獨特的代謝方式和極強的環境適應能力,使得同一地域的古菌群落種屬多樣性差異較小,但由于溫度、pH值、C/N等因素的影響,種屬之間豐度差異比較顯著。窖泥變質前后甲烷微球菌目(Methanomicrobiales)的含量分別為1.78%和45.15%,說明甲烷微球菌目更適應窖泥變質后的環境。Brauer等研究表明,甲烷微球菌目的一個種屬最佳生長pH值在4.5~5之間[23],而窖泥變質后的pH值也在這個區間,由此可以得知,pH值可能是引起甲烷菌群落結構變化的主因。
3 討論
窖泥被許多酒廠視為具有獨特風格和酒質的基礎,對窖泥功能菌的研究有助于認識窖泥微生物的代謝過程。窖泥變質后,微生態群落發生變化,酒質下降,給酒廠帶來巨大損失。古菌域的產甲烷菌與己酸菌互利共生,對發酵濃香型白酒具有重要的作用[24]。因此,了解窖泥變質前后古菌群落差異,對于研究窖泥變質有重要意義。由于傳統培養技術和顯微鏡觀察方法的局限性,對窖泥微生物區系古菌群落組成研究還遠遠不夠。采用Illumina Miseq測序平臺的高通量DNA測序技術,對窖泥變質前后古菌群落組成、結構、動態方面有了更深入的了解,為研究窖泥功能古菌及窖泥變質的古菌影響提供有利支持。
通過16S rDNA序列分析表明,窖泥變質前后16S rDNA擴增序列為11703條和52876條,聚類結果為783個OTUs和1218個OTUs,說明古菌的種類和豐度在窖泥變質后均有較大的提高。窖泥的物理環境條件對古菌群落產生一定的影響(如土壤質地、pH值、C/N、光照、濕度、溫度等),它們或是單個因子或是多個因子共同起作用影響著窖泥古菌的群落組成[25]。從擴增序列和聚類OUT數量分析,窖泥變質后生境更適合一些古菌的生長。
采用RDP classifier軟件對16S rDNA序列分析得知,窖泥變質前優勢菌屬甲烷短桿菌屬(35.66%)和甲烷桿菌屬(10.25%),窖泥變質后優勢菌屬為產甲烷袋菌屬(38.18%)和產甲烷桿菌屬(34.21%)。產甲烷袋菌屬和產甲烷桿菌屬是否在窖泥變質過程中起到直接或間接作用,有待于認識窖泥微生物全貌后再作綜合分析。
濃香型白酒窖泥微生物的研究有很高的理論價值和應用價值。通過高通量DNA測序技術可以快速直觀地得到窖泥古菌的多樣性并且獲得大量未知種類,為我們了解窖泥古菌群落組成提供技術保障。然而,要真正了解窖泥古菌群落及其生理生化特性、形態結構必須將傳統培養方法和現代分子生物學方法結合起來。
[1]Widden P.Fungal communities in soils along an elevationgradient in northern England[J].Mycologia,1987,79:298-309.
[2]陳憲儀.生物與中國釀造文化[J].中國釀造,2001(3):33-35.
[3]鄧依,唐云容,張文學.16S-23S rRNAITS-AFLP指紋圖譜分析在白酒窖泥原核微生物多樣性分析中的應用[J].釀酒科技,2010(3):46-48.
[4]羅惠波,甄攀,黃治國.濃香型白酒窖池細菌群落[J].微生物學通報,2010,37(11):1621-1627.
[5]吳三多,賴凳燡,溫寬和,等.北方地區窖泥退化原因及管理養護的研究[J].釀酒科技,2014(9):71-74.
[6]吳衍庸,盧世珩.利用窖泥甲烷菌與己酸菌二元發酵技術提高瀘型曲酒質量的研究[J].釀酒科技,2011(5):38-41.
[7]Amann R I,Ludwig W,Schleifer K H.Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].Microbiol Rev,1995,59(1):143-169.
[8]易彬,任道群,唐玉明,等.不同窖齡窖泥微生態變化研究[J].釀酒科技,2011(6):32-34.
[9]陶勇,芮俊鵬,李家寶,等.濃香型白酒窖泥中細菌和古菌的組成與多樣性[J].化工學報,2014,65(5):1800-1804.
[10]Lauber C L,Hamady M,Knight R,et al.Pyrosequencingbased assessment of soil pH as a predictor of soil bacterial community structure at the continental scale[J].Appl Environ Microbio,2009,75(15):5111-5120.
[11]曾祥勇,董雅舒,胡貝,等.不同年份窖泥細菌16S rDNA系統發育分析[J].四川大學學報,2014(9):71-74.
[12]黃永光,黃平,涂華彬.窖泥微生物總DNA的提取純化研究[J].釀酒科技,2004(3):41-42.
[13]Lee S H,Kang H J,Lee Y H,et al.Monitoring bacterial community structure and variability in time scale in full-scale anaerobic digesters[J].Environ Monit,2012,14(7):1893-1905.
[14]YannarellAC,Triplett E W.Geographic and environmental sources of variation in lake bacterial community composition [J].Applied and Environmental Microbiology,2005,71(1):227-239.
[15]Sun Y Z,Mao S Y,Zhu WY.Rumen chemical and bacterial changes during stepwise adaptation to a high-concentrate diet in goats[J].Animal,2010,4(2):210-217.
[16]Good I J.The population frequencies of species and the estimation of population parameters[J].Biometrika,1953,40:237-264.
[17]Thomton R H.Fungi occurring in mixed oakwood and heath soil profiles[J].Trans Brit Mycol Soc,1956,39:484--494.
[18]Gochenaur S E.Fungi of a Long Island oak-birch forest II. Population dynamics and hydrolase patterns for the soil penicillia[J].Mycologia,1984,76:218-231.
[19]吳衍庸.論甲烷發酵在傳統釀制瀘型曲酒中的特殊意義[J].釀酒科技,2000(5):33-34.
[20]許寶孝,奚明權,金秀其.甲烷短桿菌屬HX菌株的分離和特性[J].微生物學報,1985,25(4):283-288.
[21]朱晨光,許政暟,宋任濤.產甲烷古菌[J].生命的化學,2009,29 (1):129-133.
[22]Brauer S L,Cadillo-Quiroz H,Yashiro E,et al.Isolation of a novel acidiphilic methanogen from an acidic peat bog[J]. Nature,2006,442:192-194.
[23]羅青春,劉超蘭.不同年份窖泥中主要產甲烷菌的熒光定量PCR研究[J].釀酒科技,2013(12):17-19.
[24]Kobayashi T,Yan F,Takahashi S,et al.Effect of starch addition on the biological conversion and microbial community in a methanol-fed UASB reactor during long-term continuous operation[J].Bioresource technol,2011,102:7713-7719.
Analysis of the Difference in Archaea Communities in Pit Mud of Nongxiang Baijiu before and after Mud Deterioration
YU Chuntao1,XIANG Ming1,WANG Lei1,XU Zhenglin1and LIU Zhenjiang2
(1.Cangzhou Medical College,Cangzhou,Hebei 061001;2.Nikeng Distillery Co.Ltd.,Shijiazhuang,Hebei 054000,China)
Pit mud is the lifeline of Nongxiang Baijiu and its deterioration might bring huge loss for distilleries.The structure of archaea communities in pit mud before and after mud deterioration was analyzed by Illumina Miseq high throughput sequencing DNA technology.By constructing 16S rDNA gene bank of archaea,the results showed that there were 52876 sequences amplified in pit mud after the deterioration,11703 sequences had amplified in pit mud before the deterioration,and the abundance of archaea increased evidently.The clustering,sequencing and analysis of the sequence suggested that the dominant archaea before mud deterioration included Methanobrevibacter(35.66%),Methanobacterium(10.25%),Paenibacillus(4.27%),Bacillus(3.39%),Methanosarcina(3.27%),etc;and the dominant archaea after mud deterioration included Methanofollis(38.18%),Methanobacterium(34.21%),Methanoculleus(6.90%),etc.The results revealed that methane-producing bacteria was the dominant bacteria of archaea before and after mud deterioration.This study provided scientific evidence for pit mud culture/ maintenance and further research on pit mud microbes.
pit mud;deterioration;archaea community;analysis of the difference;high throughput sequencing;Baijiu
TS262.3;TS261.4;TS261.1;Q93-3
A
1001-9286(2016)08-0060-05
10.13746/j.njkj.2016114
2016-04-06
于春濤(1979-),男,研究生,研究方向為微生物發酵,E-mail:aimiao818@163.com。
優先數字出版時間:2016-05-26;地址:http://www.cnki.net/kcms/detail/52.1051.TS.20160526.1007.003.html。

