基于數(shù)字PCR的轉(zhuǎn)基因水稻LL62品系精準(zhǔn)定量檢測(cè)方法建立
任怡菲高琴鄧婷婷李想黃文勝陳舜勝陳穎
(1. 上海海洋大學(xué)食品學(xué)院,上海 201306;2. 上海出入境檢驗(yàn)檢疫局,上海 200135;3. 中國(guó)檢驗(yàn)檢疫科學(xué)研究院,北京 100176)
基于數(shù)字PCR的轉(zhuǎn)基因水稻LL62品系精準(zhǔn)定量檢測(cè)方法建立
任怡菲1,2高琴2鄧婷婷3李想2黃文勝3陳舜勝1陳穎3
(1. 上海海洋大學(xué)食品學(xué)院,上海 201306;2. 上海出入境檢驗(yàn)檢疫局,上海 200135;3. 中國(guó)檢驗(yàn)檢疫科學(xué)研究院,北京 100176)
為了促進(jìn)轉(zhuǎn)基因產(chǎn)品標(biāo)識(shí)制度的順利實(shí)施,對(duì)未獲我國(guó)農(nóng)業(yè)部批準(zhǔn)的轉(zhuǎn)基因水稻LL62品系進(jìn)行精準(zhǔn)定量檢測(cè)方法研究,以保障我國(guó)進(jìn)出口轉(zhuǎn)基因農(nóng)產(chǎn)品的檢測(cè)監(jiān)管。基于轉(zhuǎn)基因水稻LL62品系外源插入片段和水稻基因組DNA的3'端旁側(cè)序列設(shè)計(jì)數(shù)字PCR檢測(cè)體系,建立了精準(zhǔn)定量檢測(cè)方法。結(jié)果顯示,設(shè)計(jì)的引物探針?lè)磻?yīng)效率高,基于此建立的數(shù)字PCR方法高度特異于轉(zhuǎn)基因水稻LL62品系檢測(cè);方法可重復(fù)性好,生成微滴數(shù)相對(duì)標(biāo)準(zhǔn)偏差(RSD)值介于0.60%-11.11%;準(zhǔn)確度高,3個(gè)混合樣品(10%、1%和0.1%)測(cè)定值與真實(shí)值之間的偏差(bias)分別為0.12、0.09和0.10,相對(duì)標(biāo)準(zhǔn)偏差(RSD)介于0.09%-10.31%。建立的轉(zhuǎn)基因水稻LL62品系微滴數(shù)字PCR定量檢測(cè)方法簡(jiǎn)便快捷、特異性強(qiáng)、重復(fù)性好、準(zhǔn)確度高,可準(zhǔn)確有效地對(duì)農(nóng)產(chǎn)品和食品中轉(zhuǎn)基因水稻LL62成分進(jìn)行精準(zhǔn)定量分析。
轉(zhuǎn)基因水稻LL62品系;微滴數(shù)字PCR;品系特異性;定量
自1996年國(guó)際上首次種植轉(zhuǎn)基因作物至今已有19個(gè)年頭,轉(zhuǎn)基因作物的發(fā)展可謂突飛猛進(jìn),截至2014年全球轉(zhuǎn)基因作物種植面積已達(dá)1.815億hm2[1]。由于公眾對(duì)轉(zhuǎn)基因作物食用安全和環(huán)境安全性的擔(dān)憂(yōu),很多國(guó)家和地區(qū)均實(shí)施了標(biāo)識(shí)管理措施,并設(shè)定了閾值。如歐盟等國(guó)實(shí)施強(qiáng)制性的轉(zhuǎn)基因標(biāo)簽制度,歐盟和俄羅斯設(shè)定的閾值為0.9%[2,3],日本和中國(guó)臺(tái)灣為5.0%[4,5],韓國(guó)為3.0%[6]等;美國(guó)和加拿大實(shí)施自愿性的轉(zhuǎn)基因標(biāo)簽制度;我國(guó)目前對(duì)轉(zhuǎn)基因產(chǎn)品實(shí)施無(wú)閾值的強(qiáng)制性標(biāo)識(shí)制度[7]。為了促進(jìn)標(biāo)識(shí)管理制度的順利實(shí)施,與之相對(duì)應(yīng)的轉(zhuǎn)基因產(chǎn)品精準(zhǔn)定量檢測(cè)方法的研制至關(guān)重要。
目前食品中轉(zhuǎn)基因成分的定量檢測(cè)方法多采用實(shí)時(shí)熒光PCR方法[8-11],是通過(guò)標(biāo)準(zhǔn)物質(zhì)制備的標(biāo)準(zhǔn)曲線(xiàn)對(duì)未知樣品進(jìn)行定量分析的方法[12]。然而,該方法由于影響因素較多,如標(biāo)準(zhǔn)物質(zhì)和樣品間背景不同,標(biāo)準(zhǔn)曲線(xiàn)線(xiàn)性范圍有限,低拷貝DNA擴(kuò)增可重復(fù)性差等,常造成定量結(jié)果準(zhǔn)確性差,偏差大[13-16]。
數(shù)字PCR(digital polymerase chain reaction,dPCR)是近年來(lái)在實(shí)時(shí)熒光PCR基礎(chǔ)上發(fā)展起來(lái)的微量DNA 分子定量檢測(cè)新技術(shù)。dPCR是將PCR體系分配到足夠小的反應(yīng)單元中,實(shí)現(xiàn)每個(gè)反應(yīng)單元只有單個(gè)模板分子進(jìn)行PCR擴(kuò)增,再采用泊松分布原理,根據(jù)陽(yáng)性微滴與陰性微滴數(shù)的比例計(jì)算目標(biāo)分子拷貝數(shù),實(shí)現(xiàn)絕對(duì)定量[17-19]。該方法降低了標(biāo)準(zhǔn)曲線(xiàn)對(duì)測(cè)量結(jié)果產(chǎn)生影響等問(wèn)題,降低了基體效應(yīng),實(shí)現(xiàn)了PCR 擴(kuò)增的樣品分離,消除了本底信號(hào)的影響,提高了低拷貝DNA的擴(kuò)增靈敏度[20]。相比實(shí)時(shí)熒光PCR,數(shù)字PCR具有更好的測(cè)量獨(dú)立性,且無(wú)需任何校準(zhǔn)物,具有更高的特異性、靈敏度、精確性和穩(wěn)定性。
轉(zhuǎn)基因水稻LL62品系由拜耳公司研發(fā),由于在基因組上重組bar基因從而耐受草銨磷除草劑。目前該水稻品系已在6個(gè)國(guó)家獲得了種植、食用、飼用等不同方面的批準(zhǔn)[21]。但我國(guó)迄今還未對(duì)境外任何轉(zhuǎn)基因水稻品系頒發(fā)農(nóng)業(yè)轉(zhuǎn)基因生物安全證書(shū)。因此在水稻進(jìn)境檢測(cè)中,口岸重點(diǎn)關(guān)注我國(guó)未批準(zhǔn)品系的檢測(cè)監(jiān)管,包括LL62品系。為此,本研究基于外源插入片段和水稻基因組DNA的3'端旁側(cè)序列,建立了轉(zhuǎn)基因水稻LL62品系特異性微滴式數(shù)字PCR定量檢測(cè)方法。
1 材料與方法
1.1材料
轉(zhuǎn)基因玉米(Zea mays L.)Bt11、MON98140、NK603和MON810品系,轉(zhuǎn)基因棉花(Gossypium)T304-40、GHB119和281-24-236×3006-210-23品系標(biāo)準(zhǔn)物質(zhì)購(gòu)于歐盟聯(lián)合研究中心標(biāo)準(zhǔn)物質(zhì)與測(cè)量研究所(Institute for Reference Materials and Measurements,IRMM)。轉(zhuǎn)基因棉花MON88913、MON531、MON15985和 MON1445品系,轉(zhuǎn)基因大豆(Glycine max)GTS40-3-2和MON89788品系以及轉(zhuǎn)基因水稻(Oryza sativa)LL62品系(0306-I5)標(biāo)準(zhǔn)物質(zhì)購(gòu)于美國(guó)油脂化學(xué)家協(xié)會(huì)(American Oil Chemists' Society,AOCS)。轉(zhuǎn)基因水稻TT51-1、KF6和KMD以及非轉(zhuǎn)基因水稻、棉花、大豆、玉米樣品由中國(guó)檢驗(yàn)檢疫科學(xué)研究院提供或本實(shí)驗(yàn)室儲(chǔ)備。
1.2方法
1.2.1DNA提取 所有轉(zhuǎn)基因與非轉(zhuǎn)基因植物材料均根據(jù)植物基因組DNA提取試劑盒(Cat:#DP305-02,TianGen,北京)的操作方法進(jìn)行基因組DNA提取。利用微量分光光度計(jì)(GE公司Nanovue Plus,美國(guó))測(cè)定提取DNA溶液的濃度并通過(guò)測(cè)定A260/A280和A260/A230值來(lái)衡量提取的基因組DNA質(zhì)量。DNA稀釋至10 ng/μL保存?zhèn)溆谩?/p>
1.2.2混合樣品制備 為了驗(yàn)證建立的轉(zhuǎn)基因水稻LL62 品系特異性微滴數(shù)字PCR定量檢測(cè)方法的精確度和準(zhǔn)確度,本研究分別制備了含有10%、1%和0.1% 3個(gè)百分比含量的轉(zhuǎn)基因水稻LL62品系成分混合樣品,標(biāo)記為S1、S2和S3。制備過(guò)程為:首先分別吸取20 μL 100%的LL62 DNA標(biāo)準(zhǔn)溶液(10 ng/μL)和180 μL非轉(zhuǎn)基因水稻DNA溶液(10 ng/μL)置于1.5 mL離心管中,使用渦旋震蕩儀將其充分混合均勻,制成10.0%混合樣品S1;然后分別吸取10 μL混合樣品S1和90 μL非轉(zhuǎn)基因水稻DNA溶液(10 ng/μL)置于1.5 mL離心管中,使用渦旋震蕩儀將其充分混勻,得到1.0%混合樣品S2;最后分別吸取10 μL混合樣品S2和90 μL非轉(zhuǎn)基因水稻DNA溶液(10 ng/μL)置于1.5 mL離心管中,充分混勻后得到0.1%混合樣品S3。
1.2.3引物和探針設(shè)計(jì)及篩選 在NCBI數(shù)據(jù)庫(kù)中查找轉(zhuǎn)基因水稻LL62品系的外源插入基因和基因組DNA鄰接區(qū)序列信息,得到3'端旁側(cè)序列(GenBank號(hào)為NC_008399.2)。由于數(shù)字PCR擴(kuò)增對(duì)引物和探針的特殊要求,如擴(kuò)增效率高、熒光信號(hào)強(qiáng)、本底低等,采用PrimerExpress2.0軟件設(shè)計(jì)轉(zhuǎn)基因水稻LL62品系以及水稻內(nèi)標(biāo)準(zhǔn)基因SPS的特異性引物和探針序列,以混合樣品S1(10.0%)DNA為模板,分別對(duì)多套轉(zhuǎn)基因水稻LL62品系特異性檢測(cè)體系進(jìn)行測(cè)試,對(duì)比其反應(yīng)效率的高低、微滴生成的穩(wěn)定性等因素,篩選適宜的引物和探針,并與歐盟發(fā)布的轉(zhuǎn)基因水稻LL62品系特異性檢測(cè)方法[19]進(jìn)行比較。同時(shí)對(duì)水稻內(nèi)源標(biāo)準(zhǔn)基因SPS的擴(kuò)增體系也進(jìn)行了篩選。引物及探針由上海輝睿生物科技有限公司合成并純化,引物和探針序列見(jiàn)表1。

表1 本研究使用的檢測(cè)引物、探針序列
1.2.4微滴式數(shù)字PCR反應(yīng)體系及條件 微滴式數(shù)字PCR反應(yīng)體系總體積為20 μL,其中1×ddPCRTMSupermix for Probes(no dUTP)(美國(guó)Bio-Rad公司,Cat#186-3024)10 μL;上下游引物(10 μmol/L)各1.5 μL;探針(10 μmol/L)0.75 μL;超純水4.25 μL;DNA模板2 μL。
配制反應(yīng)混合液,并充分混勻后加入微滴發(fā)生板(美國(guó)Bio-Rad公司,Cat:#951020362),使用微滴發(fā)生器將反應(yīng)混合液制成約 2×104個(gè)微滴。為防止微滴破裂,用排槍緩慢吸取微滴轉(zhuǎn)移入96 孔PCR反應(yīng)板中進(jìn)行PCR。反應(yīng)條件為:95℃ 10 min;95℃ 30 s,60℃ 1 min,40 個(gè)循環(huán);98℃ 10 min。將反應(yīng)后的96孔板放入微滴分析儀(美國(guó)Bio-Rad公司)進(jìn)行熒光測(cè)定和數(shù)據(jù)讀取,后采用Quantasoft分析軟件(美國(guó)Bio-Rad公司)計(jì)算微滴生成的個(gè)數(shù),并計(jì)算出陽(yáng)性微滴數(shù),換算得到樣品濃度(拷貝/μL PCR產(chǎn)物)。
1.2.5特異性實(shí)驗(yàn) 采用22種轉(zhuǎn)基因及非轉(zhuǎn)基因植物材料,包括常規(guī)水稻、棉花、大豆和玉米樣品,轉(zhuǎn)基因水稻LL62、KMD、KF6和TT51-1品系,轉(zhuǎn)基因大豆GTS40-3-2和MON89788品系,轉(zhuǎn)基因玉米Bt11、MON98140、NK603和MON810品系以及轉(zhuǎn)基因棉花T304-40、GHB119、281-24-236×3006-210-23、MON88913、MON531、MON1445 和MON15985品系基因組DNA為模板,進(jìn)行PCR擴(kuò)增。每個(gè)樣品重復(fù)2次,采用微滴式數(shù)字PCR方法進(jìn)行檢測(cè),測(cè)試建立方法的特異性。
1.2.6可重復(fù)性實(shí)驗(yàn) 為測(cè)試建立的轉(zhuǎn)基因水稻LL62品系微滴式數(shù)字PCR檢測(cè)方法的可重復(fù)性,取含有10%轉(zhuǎn)基因水稻LL62品系的樣品DNA溶液(100 ng/μL),用0.1×TE(Tris-HCl,EDTA·Na,pH 8.0)緩沖液連續(xù)梯度稀釋至6個(gè)濃度:50、10、2、0.4、0.08和0.001 6 ng/μL,即121 000、24 200、4 840、968、193.6和38.72拷貝/μL水稻基因組DNA(根據(jù)1 ng≈2 420拷貝水稻單倍體基因組DNA計(jì)算[20]),分別擴(kuò)增LL62品系特異性序列和內(nèi)源標(biāo)準(zhǔn)基因SPS,每個(gè)濃度每個(gè)目標(biāo)設(shè)置3次平行重復(fù),實(shí)驗(yàn)共重復(fù)3次,共得到9個(gè)數(shù)值。然后分別計(jì)算標(biāo)準(zhǔn)偏差(standard deviation,SD)和相對(duì)標(biāo)準(zhǔn)偏差(relative standard deviation,RSD)來(lái)判斷方法的可重復(fù)性。
1.2.7準(zhǔn)確度、精確度測(cè)定 將含有不同比例轉(zhuǎn)基因水稻LL62成分的混合樣品S1(10%)、S2(1%)、S3(0.1%)的DNA,用微滴式數(shù)字PCR定量方法分別擴(kuò)增LL62品系特異性序列和內(nèi)源標(biāo)準(zhǔn)基因SPS,每個(gè)樣品重復(fù)測(cè)定3次,整個(gè)實(shí)驗(yàn)重復(fù)3次。根據(jù)內(nèi)外源基因的拷貝數(shù)計(jì)算轉(zhuǎn)基因含量,并計(jì)算測(cè)定值與真實(shí)值間的偏差(bias)、各平行重復(fù)之間的標(biāo)準(zhǔn)偏差(SD)、相對(duì)標(biāo)準(zhǔn)偏差(RSD)判定定量的準(zhǔn)確度和精確度。
通過(guò)數(shù)字PCR儀運(yùn)行后自動(dòng)給出的內(nèi)源標(biāo)準(zhǔn)基因SPS拷貝數(shù)(copies/μL)和LL62品系特異性序列的拷貝數(shù)(copies/μL),按照下列公式計(jì)算樣品中轉(zhuǎn)基因水稻LL62的相對(duì)含量[25]。

2 結(jié)果
2.1核酸提取結(jié)果
所有提取的植物基因組DNA經(jīng)微量分光光度計(jì)測(cè)定,DNA濃度在100-200 ng/μL之間,OD260/OD280值在1.8-2.0之間。結(jié)果表明,所提取的基因組DNA符合進(jìn)一步定量檢測(cè)的要求。
2.2引物探針檢測(cè)體系比較
微滴生成是影響微滴式數(shù)字PCR(droplet digital PCR,ddPCR)擴(kuò)增的關(guān)鍵步驟,只有微滴數(shù)大于10 000時(shí),DNA分子的分布才能符合泊松分布的統(tǒng)計(jì)學(xué)原理,系統(tǒng)才可以對(duì)陽(yáng)性和陰性微滴進(jìn)行有效計(jì)數(shù)。首先對(duì)歐盟發(fā)布的方法和本實(shí)驗(yàn)設(shè)計(jì)的方法進(jìn)行擴(kuò)增效果比對(duì)。兩個(gè)方法的上下游引物一致,但探針序列不同,分別為L(zhǎng)L62-P1和LL62-P2。采用LL62-P1探針的測(cè)試結(jié)果表明生成的微滴數(shù)處于15 264-18 319之間,采用LL62-P2探針測(cè)試生成的微滴數(shù)處于15 833-18 964之間,均大于10 000,滿(mǎn)足ddPCR微滴的分析要求。ddPCR擴(kuò)增形成的微滴如圖1所示。由圖1-A可見(jiàn),采用LL62-P1進(jìn)行的ddPCR反應(yīng)中,本底熒光值達(dá)到7 851.8-8 331.8,區(qū)間較寬,而陽(yáng)性擴(kuò)增獲得的熒光值介于14 197-14 488之間,陽(yáng)性熒光擴(kuò)增值與本底間差異約為6 000,差異偏小,陽(yáng)性微滴和陰性微滴區(qū)分界限不明晰,對(duì)微滴的計(jì)數(shù)有一定影響;采用LL62-P2實(shí)施的反應(yīng)中,ddPCR本底熒光值介于3 514.5-3 556.1之間,變化范圍較小,且較LL62-P1擴(kuò)增的本底熒光均值低4 500左右,生成的陽(yáng)性微滴熒光值處于12 336-12 563之間,二者差異達(dá)到9 000左右,陽(yáng)性微滴和陰性微滴界限分明,系統(tǒng)可更準(zhǔn)確判讀出陽(yáng)性微滴和陰性微滴數(shù)目(圖1-B)。因此,確定采用本實(shí)驗(yàn)設(shè)計(jì)的LL62-P2擴(kuò)增體系進(jìn)一步進(jìn)行轉(zhuǎn)基因水稻LL62品系ddPCR定量檢測(cè)方法建立。
2.3特異性實(shí)驗(yàn)
采用20個(gè)常見(jiàn)轉(zhuǎn)基因植物和常規(guī)植物基因組DNA為模板,對(duì)建立的轉(zhuǎn)基因水稻LL62品系數(shù)字PCR定量檢測(cè)方法的特異性進(jìn)行測(cè)試。當(dāng)以真核生物18S rRNA引物/探針進(jìn)行PCR擴(kuò)增時(shí),所有植物來(lái)源材料基因組DNA均有陽(yáng)性微滴生成,微滴數(shù)在12 023-14 792之間,而陰性對(duì)照和空白對(duì)照無(wú)陽(yáng)性微滴生成,表明提取的 DNA 適于用作微滴數(shù)字PCR 擴(kuò)增。當(dāng)以水稻內(nèi)源基因SPS引物/探針進(jìn)行PCR擴(kuò)增時(shí),只有以水稻材料來(lái)源的基因組DNA有陽(yáng)性微滴生成,其他植物來(lái)源材料則無(wú)陽(yáng)性微滴生成。當(dāng)以L(fǎng)L62品系特異性引物/探針進(jìn)行PCR擴(kuò)增時(shí),只有LL62品系樣品有陽(yáng)性微滴生成,而其他轉(zhuǎn)基因水稻、轉(zhuǎn)基因大豆、轉(zhuǎn)基因棉花、轉(zhuǎn)基因玉米和非轉(zhuǎn)基因植物樣品則沒(méi)有產(chǎn)生陽(yáng)性微滴(表2)。因此,設(shè)計(jì)的水稻內(nèi)源SPS和 LL62 品系特異性擴(kuò)增體系特異性好,可以進(jìn)一步用于微滴式數(shù)字PCR定量檢測(cè)轉(zhuǎn)基因水稻LL62品系。

表2 建立的轉(zhuǎn)基因水稻LL62品系數(shù)字PCR檢測(cè)方法特異性檢測(cè)結(jié)果
2.4可重復(fù)性實(shí)驗(yàn)
定量PCR反應(yīng)的重復(fù)性反映所建立的數(shù)字PCR定量體系的穩(wěn)定性和可靠性。為此,本實(shí)驗(yàn)取6個(gè)濃度的10%含量轉(zhuǎn)基因水稻LL62樣品DNA溶液(50、10、2、0.4、0.08 和 0.016 ng/μL)進(jìn)行數(shù)字PCR反應(yīng)。根據(jù)軟件計(jì)算的陽(yáng)性微滴數(shù)分析方法的可重復(fù)性。結(jié)果(表3)顯示,3次重復(fù)擴(kuò)增生成微滴數(shù)的SD值介于1.00-264.49之間,RSD值介于0.60%-11.11%之間,DNA模板濃度越高,RSD值越小,RSD值均在可接受范圍內(nèi)(小于25%)[26]。因此,建立的轉(zhuǎn)基因水稻LL62品系ddPCR定量檢測(cè)方法微滴生成穩(wěn)定,可重復(fù)性好。

表 3 建立的轉(zhuǎn)基因水稻LL62品系數(shù)字PCR定量檢測(cè)方法可重復(fù)性測(cè)試
2.5定量的準(zhǔn)確度、精確度實(shí)驗(yàn)
配制3個(gè)百分比含量混合樣品S1(10.0%)、S2 (1.0%)和S3(0.1%),對(duì)混合樣品中LL62轉(zhuǎn)基因水稻成分的含量進(jìn)行了定量測(cè)試。結(jié)果如表4所示,3個(gè)混合樣品S1(10.0%)、S2(1.0%)和S3(0.1%)的百分比含量測(cè)定值分別為11.23%、1.09%和0.09%;測(cè)定值與真實(shí)值之間的偏差(bias)分別為0.12、0.09 和0.10;每個(gè)混合樣品的3次重復(fù)之間的標(biāo)準(zhǔn)偏差(SD)值介于0.01-0.02,相對(duì)標(biāo)準(zhǔn)偏差(RSD)值介于0.09%-10.31%,均在可接受的范圍之內(nèi),而且偏差較小。結(jié)果表明,建立的轉(zhuǎn)基因水稻LL62品系特異性微滴式數(shù)字定量PCR檢測(cè)方法具有較好的特異性、可重復(fù)性、準(zhǔn)確度和精確度,能夠有效的對(duì)混合樣品中LL62轉(zhuǎn)基因成分進(jìn)行定性和定量檢測(cè)分析。
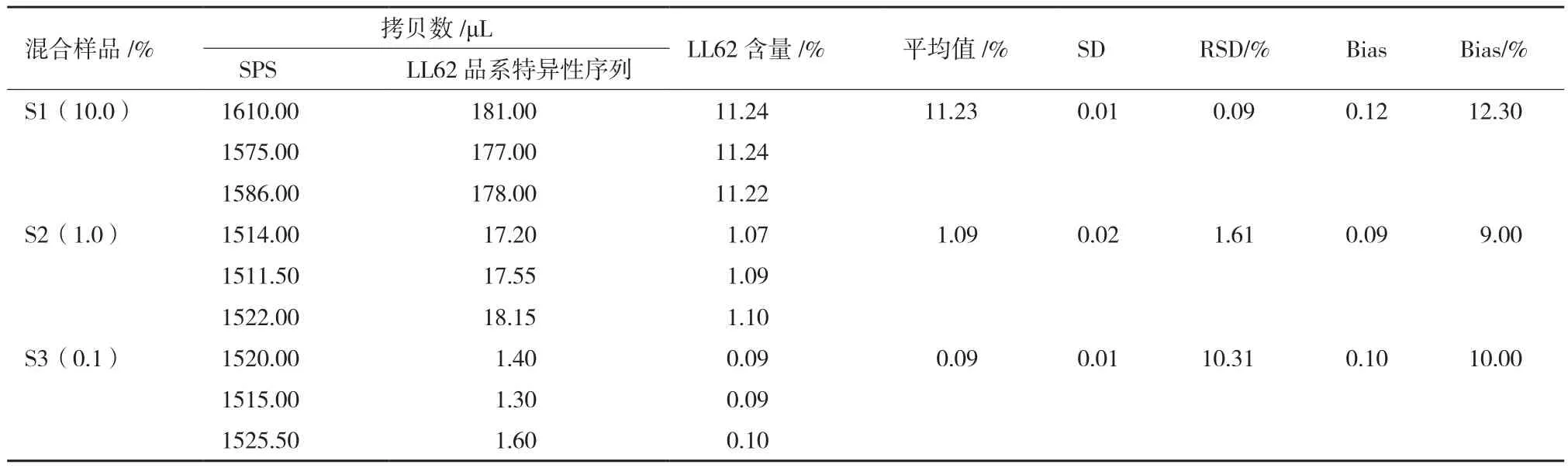
表4 轉(zhuǎn)基因水稻LL62盲樣測(cè)試結(jié)果
3 討論
近年來(lái),隨著轉(zhuǎn)基因作物商業(yè)化的不斷發(fā)展,轉(zhuǎn)基因產(chǎn)品的標(biāo)識(shí)閾值制度和低水平混雜(low level presence,LLP)政策受到各國(guó)的關(guān)注,政策的實(shí)施很大程度上依靠轉(zhuǎn)基因成分的精準(zhǔn)定量。目前對(duì)轉(zhuǎn)基因作物及其產(chǎn)品的定量檢測(cè)方法一直采用基于標(biāo)準(zhǔn)曲線(xiàn)的相對(duì)定量,但由于技術(shù)的固有問(wèn)題,很難達(dá)到較高的精確度,尤其是轉(zhuǎn)基因成分含量低于1%水平時(shí)。近年發(fā)展起來(lái)的數(shù)字PCR定量檢測(cè)技術(shù),雖然也是基于實(shí)時(shí)熒光PCR擴(kuò)增,但由于擴(kuò)增是在足夠小的僅容納一個(gè)反應(yīng)的微單元中進(jìn)行,因此極大降低了反應(yīng)間的相互抑制,提高了反應(yīng)效率。
目前食品中轉(zhuǎn)基因成分的定量檢測(cè)方法多采用實(shí)時(shí)熒光PCR方法。如王渭霞等[9]建立了轉(zhuǎn)基因水稻科豐6號(hào)定量檢測(cè)方法;汪秀秀等[22]建立了轉(zhuǎn)基因棉花GHB119品系特異性定量檢測(cè)方法;蘇長(zhǎng)青等[27]建立了Bt63 實(shí)時(shí)熒光定量方法;吳永彬等[28]建立了轉(zhuǎn)基因大豆 GTS40-3-2、玉米 NK603、油菜 RT73和水稻 TT51-1的定量檢測(cè)方法等。而這些方法定量下限多在0.5%及以上,有些方法定量下限雖能低至0.1%,但多基于質(zhì)粒標(biāo)準(zhǔn)分子的定量結(jié)果為依據(jù)而非植物材料,因此檢測(cè)下限的局限性也使方法的應(yīng)用受到了限制。
與實(shí)時(shí)熒光PCR定量檢測(cè)方法相比,由于數(shù)字PCR是在足夠小的反應(yīng)單元中進(jìn)行實(shí)時(shí)熒光PCR擴(kuò)增,降低了反應(yīng)間的相互影響和相互抑制,可重復(fù)性好,穩(wěn)定性高;而且數(shù)字PCR方法采用直接計(jì)數(shù)的方法實(shí)現(xiàn)絕對(duì)定量,使定量結(jié)果更加準(zhǔn)確,定量下限可低至0.1%或以下,而且由于數(shù)字PCR無(wú)需制備標(biāo)準(zhǔn)曲線(xiàn),節(jié)省人力物力。數(shù)字PCR方法的應(yīng)用對(duì)應(yīng)對(duì)轉(zhuǎn)基因產(chǎn)品閾值制度和低水平混雜措施的實(shí)施更加有利。
目前國(guó)內(nèi)外采用數(shù)字PCR技術(shù)對(duì)轉(zhuǎn)基因成分含量進(jìn)行定量的研究還剛剛起步。Corbisier等[15]比較了數(shù)字PCR和實(shí)時(shí)熒光PCR兩種技術(shù)應(yīng)用于轉(zhuǎn)基因玉米MON810品系定量檢測(cè)上的優(yōu)劣,認(rèn)為數(shù)字PCR具有計(jì)量特性,在定量上有更大的優(yōu)勢(shì)。Burns等[29]采用經(jīng)驗(yàn)證的應(yīng)用于轉(zhuǎn)基因成分檢測(cè)的qPCR反應(yīng)體系,評(píng)估了dPCR技術(shù)的絕對(duì)檢測(cè)限和定量限,并表明dPCR 可以更精確地測(cè)量拷貝數(shù)。另外,基于數(shù)字PCR技術(shù),國(guó)內(nèi)外專(zhuān)家學(xué)者也進(jìn)行了單細(xì)胞基因表達(dá)、臨床診斷、生物樣品種屬鑒定和基因拷貝數(shù)分析等領(lǐng)域研究[30-33]。
數(shù)字PCR作為更精確和更靈敏的DNA定量檢測(cè)新技術(shù),實(shí)現(xiàn)了單分子DNA的絕對(duì)定量,為轉(zhuǎn)基因產(chǎn)品的定量分析提供了新的技術(shù)手段,也為轉(zhuǎn)基因產(chǎn)品中低含量成分的檢測(cè)指出了一個(gè)新的潛在發(fā)展方向。我們期望在不遠(yuǎn)的將來(lái),這項(xiàng)技術(shù)的發(fā)展在分子生物學(xué)基礎(chǔ)研究和應(yīng)用方面發(fā)揮更大的作用。
4 結(jié)論
本研究基于3'端旁側(cè)序列,建立了轉(zhuǎn)基因水稻LL62品系特異性數(shù)字PCR定量檢測(cè)方法。對(duì)方法的適用性鑒定結(jié)果表明,建立的數(shù)字PCR方法特異于轉(zhuǎn)基因水稻LL62品系檢測(cè);方法可重復(fù)性好,生成微滴數(shù)的RSD值介于0.60%-11.11%之間,小于歐盟定量檢測(cè)限25%的要求;準(zhǔn)確度高,3個(gè)混合樣品(10%、1%和0.1%)測(cè)定值與真實(shí)值之間的偏差(bias)分別為0.12、0.09和0.10,相對(duì)標(biāo)準(zhǔn)偏差(RSD)值介于0.09%-10.31%。因此,本研究建立的基因水稻LL62 品系特異性微滴數(shù)字PCR方法可以用于我國(guó)口岸農(nóng)產(chǎn)品中轉(zhuǎn)基因水稻LL62成分的定性定量分析,加強(qiáng)相關(guān)部門(mén)對(duì)轉(zhuǎn)基因水稻及其制品的監(jiān)控、安全評(píng)價(jià)和風(fēng)險(xiǎn)預(yù)警,同時(shí)也為我國(guó)轉(zhuǎn)基因產(chǎn)品標(biāo)識(shí)制度的順利實(shí)施提供技術(shù)支持。
[1] Clive J. 2014年全球生物技術(shù)/轉(zhuǎn)基因作物商業(yè)化發(fā)展態(tài)勢(shì)[J].中國(guó)生物工程雜志, 2015, 35(1):1-14.
[2] European Commission. Regulation(EC)No. 1829/2003 of the European Parliament and of the Council of 22 September 2003 on genetically modified food and feed[J]. Official Journal of the European Union, 2003, L268:1-23.
[3] Russian Federation. SanPiN 2. 3. 2. 2227- 07“Additions and Changes #5 to the Sanitary- Epidemiological Rules SanPiN 2. 3. 2. 1078- 01 “Hygiene Requirements to Safety and Nutrition Value of Food Products”. 2007.
[4] Ministry of Agriculture, Forestry and Fisheries of Japan. Notification 1775. Food and Marketing Nureau[S]. Tokyo, Japan:2000.
[5] Chiueh LC, Chen YL, Shih DY. Study on the detection method of six varieties of genetically modified maize and processed foods[J]. Journal of Food and Drug Analysis, 2002, 10(1):25-33.
[6] Ministry of Agriculture and Forestry of Korea(MAF). Notification No. 2000- 31, Guidelines for Labeling of Genetically Modified Agricultural Products[S]. Seoul, Korea:2000.
[7]中華人民共和國(guó)農(nóng)業(yè)部. 農(nóng)業(yè)部第10號(hào)令《農(nóng)業(yè)轉(zhuǎn)基因生物標(biāo)識(shí)管理辦法》[S]. 2002.
[8]宋君, 雷紹榮, 劉勇, 等. 轉(zhuǎn)基因玉米NK603品系特異定量PCR檢測(cè)方法的建立[J]. 生物技術(shù)通訊, 2012, 23(2):238-241.
[9]王渭霞, 賴(lài)?guó)P香, 洪利英, 等. 實(shí)時(shí)定量PCR檢測(cè)轉(zhuǎn)基因水稻科豐6號(hào)插入拷貝數(shù)和轉(zhuǎn)基因含量[J]. 農(nóng)業(yè)生物技術(shù)學(xué)報(bào),2012, 20(1):9-15.
[10]敖金霞, 高學(xué)軍, 仇有文, 等. 實(shí)時(shí)熒光定量PCR技術(shù)在轉(zhuǎn)基因檢測(cè)中的應(yīng)用[J]. 東北農(nóng)業(yè)大學(xué)學(xué)報(bào), 2009, 40(6):141-144.
[11]Li X, Pan LW, Li JY, et al. Establishment and application of eventspecific polymerase chain reaction methods for two genetically modified soybean events, A2704-12 and A5547-127[J]. Journal of Agricultural and Food Chemistry, 2011, 59(24):13188-13194.
[12]潘良文, 田風(fēng)華, 張舒亞. 轉(zhuǎn)基因抗草丁膦油菜籽中Barnase基因的實(shí)時(shí)熒光定量PCR檢測(cè)[J]. 中國(guó)油料作物學(xué)報(bào),2006, 28(2):194-198.
[13]Burns M, Burrell A, Foy C. The applicability of digital PCR for the assessment of detection limits in GMO analysis[J]. European Food Research and Technology, 2010, 231(3):353-362.
[14]Heyries KA, Carolina T, Michael C, et al. Megapixel digital PCR[J]. Nat Methods, 2011, 8(8):649-651.
[15]Corbisier P, Somanath B, Lina P, et al. Absolute quantification of genetically modified MON810 maize(Zea mays L. )by digital polymerase chain reaction[J]. Anal Bioanal Chem, 2010, 396(6):2143-2150.
[16]Terry CF, Shanahan DJ, Ballam LD, et al. Real-time detection of genetically modified soya using Lightcycler and ABI 7700 platforms with TaqMan, Scorpion, and SYBR GreenⅠchemistries[J]. J AOAC International, 2002, 85(4):938-944.
[17]Bhat S, Herrmann J, Armishaw P, et al. Single molecule detection in nanofluidic digital array enables accurate measurement of DNA copy number[J]. Anal Bioanal Chem, 2009, 394(2):457-467.
[18]李春勇. 數(shù)字PCR技術(shù)原理及應(yīng)用[J]. 生物技術(shù)與世界,2014:9-13.
[19]Sanders R, Huggett JF, Bushell CA, et al. Evaluation of digital PCR fo rabsolute DNA quantification[J]. Anal Chem, 2011, 83(17):6474-6484.
[20]李亮, 隋志偉, 王晶, 等. 基于數(shù)字PCR的單分子DNA定量技術(shù)研究進(jìn)展[J]. 生物化學(xué)與生物物理進(jìn)展, 2012, 39(10):1017-1023.
[21]趙衛(wèi)東, 鄭文杰, 賀艷. 熒光 PCR 方法定性和定量檢測(cè)BT63轉(zhuǎn)基因大米[J]. 食品研究與開(kāi)發(fā), 2009, 30(3):133-135.
[22]汪秀秀, 楊捷琳, 宋青, 等. 轉(zhuǎn)基因棉花GHB119品系特異性定量PCR檢測(cè)方法的建立[J]. 農(nóng)業(yè)生物技術(shù)學(xué)報(bào), 2014, 22 (3):380-388.
[23]Joint Research Centre Institute for Health and Consumer Protection Biotechnology & GMOs Unit. Event-specific method for the quantification of rice line LLRICE62 using Real-time PCR (CRLVL05/04VP). 2006.
[24]National Center for Biotechnology Information. Oryza sativa(rice). [OL]http://www. ncbi. nlm. nih. gov/ genome?term=txid4530%5Borgn%5D
[25]Liu J, Guo JC, Zhang HB, et al. Development and in-house validation of the event-specific polymerase chain reaction detection methods for genetically modified soybean MON89788 based on the cloned integration flanking sequence[J]. Journal of Agricultural and Food Chemistry, 2009, 57(22):10524-10530.
[26]European Network of GMO Laboratories(ENGL). Definition of minimum performance requirements for analytical methods of GMO testing. 2008.
[27]蘇長(zhǎng)青, 謝家建, 王奕海, 等. 轉(zhuǎn)基因水稻Bt汕優(yōu)63的整合結(jié)構(gòu)和品系特異性定量PCR方法[J]. 農(nóng)業(yè)生物技術(shù)學(xué)報(bào),2011, 19(3):434-441.
[28]吳永彬, 肖維威, 張寶, 等. 轉(zhuǎn)基因大豆、玉米、油菜和水稻品系特異性檢測(cè)質(zhì)粒標(biāo)準(zhǔn)分子的快速構(gòu)建及應(yīng)用[J]. 中國(guó)生物化學(xué)與分子生物學(xué)報(bào), 2011, 27(8):775-782.
[29]Burns M, Burrell A, Foy C. The applicability of digital PCR for the assessment of detection limits in GMO analysis[J]. European Food Research and Technology, 2010, 231(3):353-362.
[30] White AK, Michael V, Oleh IP, et al. High-throughput micro fluidic single-cell RT-qPCR[J]. Proc Natl Acad Sci USA, 2011, 108 (34):13999-14004.
[31]Jones MA, Bhide S, Chin E, et al. Targeted polymerase chain reaction-based enrichment and next generation sequencing for diagnostic testing of congenital disorders of glycosylation[J]. Genet Med, 2011, 13(11):921-932.
[32]胡偉, 陳榮華, 張晨, 等. 微滴式數(shù)字PCR技術(shù)用于生物樣品種屬鑒定和絕對(duì)定量[J]. 法醫(yī)學(xué)雜志, 2014, 30(5):342-345.
[33]姜羽, 胡佳瑩, 楊立桃, 等. 利用微滴數(shù)字PCR分析轉(zhuǎn)基因生物外源基因拷貝數(shù)[J]. 農(nóng)業(yè)生物技術(shù)學(xué)報(bào), 2014, 22(10):1298-1305.
(責(zé)任編輯 狄艷紅)
Establishment of Precisely Quantitative Method of Genetically Modified Rice LL62 Based on Digital PCR
REN Yi-fei1,2GAO Qin2DENG Ting-ting3LI Xiang2HUANG Wen-sheng3CHEN Shun-sheng1CHEN Ying3
(1. School of Food Science,Shanghai Ocean University,Shanghai 201306;2. Shanghai Entry-Exit Inspection and Quarantine Bureau,Shanghai 200135;3. Chinese Academy of Inspection and Quarantine,Beijing 100176)
In order to promote the smooth implementation of labeling policy for genetically modified(GM)products,we study an precise quantitative detection method for the GM rice LL62 that has not been approved by the Ministry of Agriculture in China,aiming at guarantee the detection and supervision of GM agricultural products in export and import. A digital PCR(dPCR)detection system were designed based on the inserted exogenous fragment of LL62 and 3' flanking sequence of DNA of rice genome,and a precisely quantitative method was established. The results showed that the designed probes presented high efficiency,the developed dPCR method was highly specific for GM rice LL62 detection. The method was highly repeated,and the relative standard deviation(RSD)values of droplets' number ranged from 0.60%-11.11%. Results of the quantification of three blind samples showed that the bias between the true value and the measured value was 0.12,0.09 and 0.10,the RSD was 0.09%-10.31%,indicating that the accuracy was high. In conclusion,the established droplet dPCR method for GM rice LL62 detection is simple and convenient with high specificity,solid repeatability and high accuracy,and is suitable for precisely and effectively quantitative analysis of ingredients of GM rice or peeled rice LL62 in agricultural products and foods.
genetically modified rice LL62;droplet digital PCR;event-specific;quantification
10.13560/j.cnki.biotech.bull.1985.2016.08.011
2015-12-01
科技部轉(zhuǎn)基因生物新品種培育重大專(zhuān)項(xiàng)(2016ZX08012001-007)
任怡菲,女,碩士研究生,研究方向:食品科學(xué)與工程;E-mail:yuki_ryf@163.com
李想,女,博士,高級(jí)工程師,研究方向:分子生物學(xué);E-mail:idealne@163.com

