茶樹丙氨酸氨基轉移酶基因的克隆與表達分析
白培賢,王麗鴛,韋康,阮麗,成浩,張芬,張成才
1.中國農業科學院茶葉研究所/國家茶樹改良中心/農業部茶樹生物學與資源利用重點實驗室,浙江 杭州 310008;2.中國農業科學院研究生院,北京 100081
茶樹丙氨酸氨基轉移酶基因的克隆與表達分析
白培賢1,2,王麗鴛1*,韋康1,阮麗1,成浩1,張芬1,2,張成才1,2
1.中國農業科學院茶葉研究所/國家茶樹改良中心/農業部茶樹生物學與資源利用重點實驗室,浙江 杭州 310008;2.中國農業科學院研究生院,北京 100081
丙氨酸氨基轉移酶(Alanine Aminotransferase,AlaAT)是與碳氮代謝相關的一種重要酶類。采用反轉錄PCR的方法克隆了茶樹CsAlaAT1的cDNA序列,該序列全長1 747 bp,包含一個完整的ORF(1 626 bp),編碼 541個氨基酸,推導的蛋白質分子量為 59.4 kD,理論等電點(pI)為 5.82。同源比對結果表明,CsAlaAT1含有丙氨酸氨基轉移酶亞家族保守的輔酶磷酸吡哆醛結合位點,其氨基酸序列與擬南芥 AtAlaAT1蛋白的相似性為84%。二級結構預測顯示該蛋白由α-螺旋(40.67%)、無規則卷曲(29.57%)、β-折疊(13.68%)和延伸鏈(16.08%)組成,定位于線粒體,不含信號肽與跨膜結構。實時熒光定量PCR(RT-PCR)檢測發現CsAlaAT1在茶樹各組織中均有表達,在根中的表達量最高;CsAlaAT1基因表達對氮素的響應研究表明,成熟葉中CsAlaAT1受氮素誘導上調表達,高濃度(1 mmol·L-1NH4NO3)氮素的誘導效應比低濃度(0.1 mmol·L-1NH4NO3)氮素誘導效應更強烈;在根中,處理24 h后,高氮誘導CsAlaAT1上調表達,低氮誘導CsAlaAT1下調表達。
茶樹;丙氨酸氨基轉移酶;克隆;表達;氮素誘導
氮素是植物生長發育過程中需求量最大的礦質元素,氮素供應不足會嚴重影響農作物的產量。為了保證產量,在現代農業生產過程中往往施用大量氮肥。但是大多數農作物對氮肥的利用率只有三分之一,其余因流失而造成環境污染[1]。因此借助基因工程手段培育氮高效利用品種,成為降低氮肥施用量、減少環境污染的有效途徑之一[2]。
茶樹作為葉用作物,其氮素利用率只有18.4%~44.7%[3],選育氮素利用效率高的品種一直是茶樹育種工作者的目標之一。茶樹的氮素吸收利用率主要取決于其吸收、同化以及運輸分配到收獲器官中的能力。目前茶樹中氮素吸收同化相關基因NR[4](Nitrate reductase)、NRT[5-6](Nitrate transporters)、GS[7](Glutamine synthetase)、GOGAT(Glutamine-α-oxoglutarate aminotransferase )、GDH[8](Glutamate dehydrogenase)以及相關的 DOF轉錄因子[9]已經被克隆。近來研究發現,氮素同化的下游途徑基因也可以調控植物對氮素的吸收利用[10]。
丙氨酸氨基轉移酶(Alanine aminotransferase,AlaAT)催化丙氨酸和α-酮戊二酸形成丙酮酸和谷氨酸[11],是植物體內與碳氮代謝相關的一種重要酶類[12]。丙氨酸氨基轉移酶家族包含 4個成員,其中AlaAT1 和AlaAT2編碼AlaAT蛋白;GGAT1和GGAT2具有谷氨酸-乙醛酸氨基轉移酶(Glutamateglyoxylate aminotransferase,GGAT)活性[13-14]。在擬南芥的葉、根和花器官[13],水稻的果實[15]、內胚乳[16]中,均可檢測到AlaAT酶的活性。有研究顯示,AlaAT參與了光合作用的C4途徑[17-18]和 L-丙氨酸的轉氨降解途徑。植物AlaAT酶活性受到光照刺激的調控。例如,黃化的黍稷幼苗受到光刺激后,葉片中的AlaAT-2基因上調表達[18]。AlaAT酶活性還受到低氧脅迫的調控,參與解除低氧脅迫后丙氨酸的降解過程[2]。有研究將大麥[10-19]的HvAlaAT基因轉入水稻和歐洲油菜中,轉基因植株不僅產量得到顯著提高,其生物量也顯著增加,這暗示著AlaAT基因在以采葉為主的茶樹中,可能具有同樣的增產和改善氮素利用效率的作用,而茶樹上尚無關于AlaAT基因的報道。本研究根據茶樹根轉錄組測序獲得的AlaAT的 cDNA 全長序列,克隆了茶樹CsAlaAT1基因,并分析該基因的組織表達特性及氮素對該基因表達的影響,為進一步研究該基因在氮素吸收利用及低氧脅迫中的作用提供研究基礎。
1 材料與方法
1.1茶樹CsAlaAT1基因的克隆
1.1.1總RNA提取和第一鏈cDNA的合成
采用天根多糖多酚植物總RNA提取試劑盒(離心柱型),提取龍井43葉片中的總RNA,并用 1%的瓊脂糖凝膠檢測完整性,采用Takara PrimeScriptTM 1st Strand cDNA Synthesis Kit將RNA反轉錄為cDNA。
1.1.2目的基因的全長驗證
根據轉錄組中獲得的CsAlaAT1的核酸序列,用DNAStar軟件包中的EditSeq軟件查找該基因的開放閱讀框(ORF),并根據ORF兩側的序列設計引物,正向引物:GTGCAAAATGTGGAAATTCGTAGC;反向引物:CAAGGGGCAAGGTTGGATTTG。用該引物做PCR對CsAlaAT1基因的ORF進行擴增,以 0.5 μL第一鏈 cDNA為模板,加入10×LA Taq bufferⅡ(Mg2+,plus)5 μL,dNTPMix(2.5 mmol·L-1each)8 μL,正反向引物各 1.5 μL,LA Taq(50 U·μL-1)0.5 μL,加滅菌的ddH2O至20 μL。反應程序為:94℃預變性,94℃變性30 s,60℃退火30 s,72℃延伸2 min,30個循環,72℃延伸10 min。PCR產物經瓊脂糖凝膠電泳驗證目標條帶,用AxyPrep DNA Gel Extraction Kit進行回收,回收產物鏈接pMD18-T載體后,轉化至大腸桿菌DH5α感受態細胞,菌落PCR挑選陽性克隆送往上海華津生物科技有限公司測序。測序結果用 MEGA 6.0軟件比對后發現克隆的cDNA序列具有CsAlaAT1基因完整的ORF。
1.2生物信息學分析
采用BioEdit軟件對本研究獲得的CsAlaAT1基因的cDNA序列進行ORF查詢后輸出氨基酸序列。在ExPASy網站(http://www.expasy.org/resources)用ProtParam軟件推導蛋白質的分子量和理論等電點,SignalP預測信號肽,TMHMM預測跨膜螺旋,PredictProtein進行蛋白質的二級結構預測和亞細胞定位。在NCBI網站上swiss-prot數據庫中搜索CsAlaAT1的同源蛋白,用MEGA 6.0鄰接法構建系統發生樹,并在ClustalW軟件上進行多序列比對,在 GeneDoc中編輯輸出。SWISS-MODEL模擬 CsAlaAT1蛋白質三維結構,并用Swiss-PdbViewer編輯輸出。
1.3氮素響應試驗
參試材料為龍井43、中茶108和中茶302 等3個品種的2年生扦插苗,水培于溫室中。水培溶液的配方參照Ruan等[20]稍作調整,包括大量元素N(0.2 mmol·L-1或2 mmol·L-1),P(0.07 mmol·L-1),K(0.67 mmol·L-1),Ca(0.53 mmol·L-1),Mg(0.67 mmol·L-1)和微量元素Zn(0.67 μmol·L-1),Cu(0.13 μmol·L-1),Mn(1.0 μmol·L-1),B(7.0 μmol·L-1),Mo(0.33 μmol·L-1),Fe(4.2 μmol·L-1),Al(70 μmol·L-1)及EDTA鹽(4.2 μmol·L-1)。
長勢良好的茶苗氮饑餓預培養兩周后,分別在低氮(0.1 mmol·L-1NH4NO3)和高氮(1 mmol·L-1NH4NO3)濃度的營養液中進行處理,每隔2 d換一次營養液。于氮素處理后0、24、72、120、168 h時取樣,第一次取樣時間為上午10:00,每次取3株茶苗的一芽二葉,成熟葉和嫩根組織,用蒸餾水清洗后轉入液氮,-80℃保存備用。
1.4茶樹CsAlaAT1基因的表達分析
利用在線軟件Primer-blast設計熒光定量PCR引物,正向引物:TCCCGGTCTATG GGGTATGG,反向引物:CTCCAACCTT GGGAGGGTTC,反應體系參照胡娟等[9]。反應程序:95℃ 30 s,95℃ 5 s,60℃ 34 s,40個循環;95℃ 15 s,60℃ 1 min,95℃ 15 s。反應在 ABI 7500實時熒光定量PCR儀上進行,4次技術性重復,以GAPDH為內參基因,用法計算結果。
2 結果與分析
2.1CsAlaAT1基因的克隆與序列分析
反轉錄PCR擴增得到CsAlaAT1的cDNA(圖1)。經測序,獲得的cDNA全長1 747 bp,編碼序列區長度為1 626 bp,編碼541個氨基酸殘基。預測的蛋白質相對分子質量為 59.4 kD,理論等電點(pI)為5.82,總平均親水性:-0.174,為親水性蛋白,不穩定指數:47.40,表示該蛋白在植物體內可能不穩定。
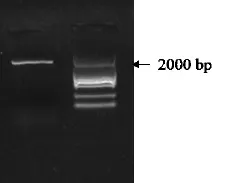
圖1 CsAlaAT1基因RT-PCR電泳結果Fig.1 Electrophoresis results of CsAlaAT1 cloning by RT-PCR

注:a:磷酸吡哆醛結合位點;b:同源二聚體接觸面;c:催化位點。AtAlaAT2:擬南芥AlaAT2(sp_Q9LDV4.1);AtAlaAT1:擬南芥AlaAT1(sp_F4I7I0.1);CsAlaAT1:茶樹AlaAT1;HvAlaAT2:大麥AlaAT2(sp_P52894.1);PmAlaAT2:黍稷AlaAT2(sp_P34106.1)。Note:a:pyridoxal 5′-phosphate binding site.b:homodimer interface.c:catalytic residue.AtAlaAT2:Arabidopsis thaliana AlaAT2(sp_Q9LDV4.1).AtAlaAT1:Arabidopsis thaliana AlaAT1(sp_F4I7I0.1).CsAlaAT1:Camellia sinensis AlaAT1.HvAlaAT2:Hordeum vulgare AlaAT2(sp_P52894.1).PmAlaAT2:Panicum miliaceum AlaAT2(sp_P34106.1).圖2 CsAlaAT1與其他植物AlaAT蛋白序列同源比對Fig.2 Alignment of the protein sequences of CsAlaAT1 and AlaATs from other species
2.2同源比對和系統發生樹的構建
在NCBI網站上,將推測的CsAlaAT1氨基酸序列于swiss-prot數據庫中進行BLASTP同源序列搜索并進行多序列比對(圖 2),結果顯示,CsAlaAT1與擬南芥(Arabidopsis thaliana)AlaAT1、AlaAT2,大麥(Hordeum vulgare)和黍稷(Panicum miliaceum)AlaAT2的相似性分別為 78%,84%,72%,79%,CsAlaAT1具有丙氨酸氨基轉移酶亞家族保守的輔酶磷酸吡哆醛結合位點,屬于丙氨酸氨基轉移酶亞家族。將茶樹CsAlaAT1的氨基酸序列與擬南芥、水稻(Oryza sativa)、亞麻薺(Camelina sativa)、大豆(Glycine max)、黃瓜(Cucumis sativus)、玉米(Zea mays)、大麥7個物種的14條AlaAT蛋白序列在MEGA 6.0中進行比對分析,并用鄰接法構建系統發生樹(圖3)。結果表明,不同物種的AlaAT1 與AlaAT2蛋白并沒有聚在一起,表明AlaAT基因的演化情況比較復雜;茶樹AlaAT1與黃瓜 AlaAT2、大豆 AlaAT1聚在一起,推測CsAlaAT1與之具有相似的生物學功能。
2.3CsAlaAT1的結構預測
二級結構預測顯示CsAlaAT1蛋白主要由α-螺旋(40.67%)和無規則卷曲(29.57%)組成,β折疊(13.68%)和延伸鏈(16.08%)僅占一小部分,不含信號肽(圖4左),無跨膜結構(圖 4右),定位于線粒體(圖 5)。在SWISS-MODEL中進行三維結構預測(圖6),結果表明,CsAlaAT1蛋白為同源二聚體,其推測的催化位點為第358位的賴氨酸殘基。
2.4CsAlaAT1基因的表達分析
2.4.1組織表達特性
將龍井43、中茶108和中茶302 3個品種的 2年生無性系茶苗在正常氮素濃度的營養液中培養7天后,對其一芽二葉、成熟葉和嫩根中CsAlaAT1基因的表達量進行檢測,結果(圖7)顯示,CsAlaAT1基因在3個組織中均有表達,且在根中的表達量高于一芽二葉和成熟葉。CsAlaAT1基因表達的組織特異性在品種間存在差異。龍井 43和中茶 108的CsAlaAT1基因在一芽二葉、成熟葉和嫩根中的表達量依次遞增,中茶302的CsAlaAT1基因表達量在嫩根、一芽二葉和成熟葉中依次遞減。
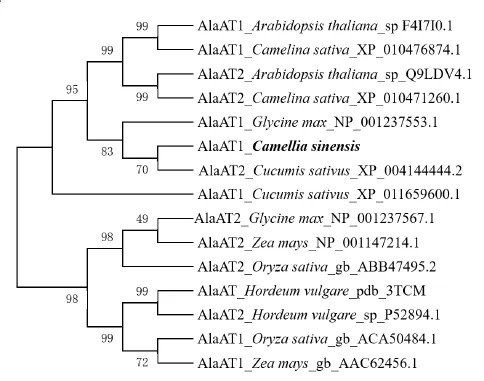
圖3 不同物種AlaAT蛋白的系統發生樹Fig.3 Phylogenetic tree of AlaAT proteins from various species
2.4.2CsAlaAT1基因時空表達對不同氮素水平的響應
氮饑餓2周后,高氮處理后茶苗的一芽二葉、成熟葉和根中CsAlaAT1基因的表達量均高于低氮處理(圖8)。表明CsAlaAT1基因表達對高氮素水平的響應更強烈。
高氮培養24 h內,CsAlaAT1基因在各組織的表達量均上調,一芽二葉、成熟葉中的表達量分別增加到原始表達量的1.7和2.2倍,24 h后呈下降趨勢;一芽二葉中的表達量在120 h降至最低,之后又迅速增加;根中的表達量在 72 h達到最大,之后緩慢下降。在低氮水平下,一芽二葉中CsAlaAT1基因的表達量呈現略微上調趨勢;成熟葉中的表達量在72 h內上調,120 h之后逐漸下降;根中的表達量在24 h內增長,之后呈下降趨勢,120 h達到最低,隨后又呈增加趨勢。總體而言,高氮條件下,CsAlaAT1基因上調表達;在低氮處理24 h之后,CsAlaAT1基因在一芽二葉和成熟葉中上調表達,在根系中下調表達。
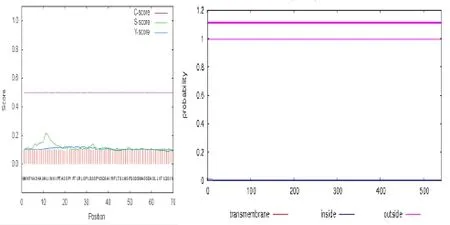
圖4 SignalP和TMHMM軟件預測CsAlaAT1蛋白信號肽和跨膜螺旋Fig.4 Prediction of signal peptide and transmembrane helix in CsAlaAT1 by SignalP and TMHMM respectively
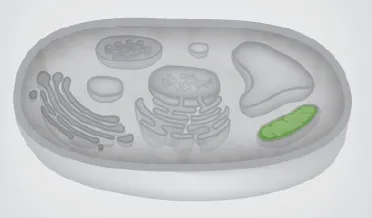
圖5 PredictProtein預測CsAlaAT1的亞細胞位置Fig.5 Subcellular location prediction of CsAlaAT1 by PredictProtein
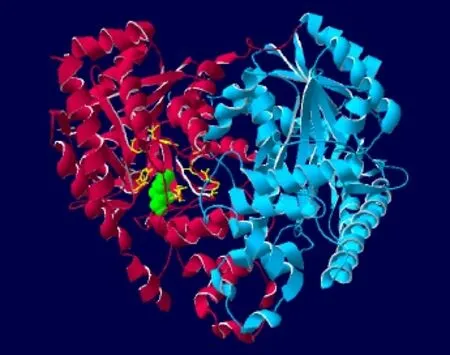
圖6 通過在線軟件SWISS-MODEL預測的CsAlaAT1蛋白三維結構Fig.6 Three-dimensional structure prediction of protein CsAlaAT1 by online software SWISS-MODEL
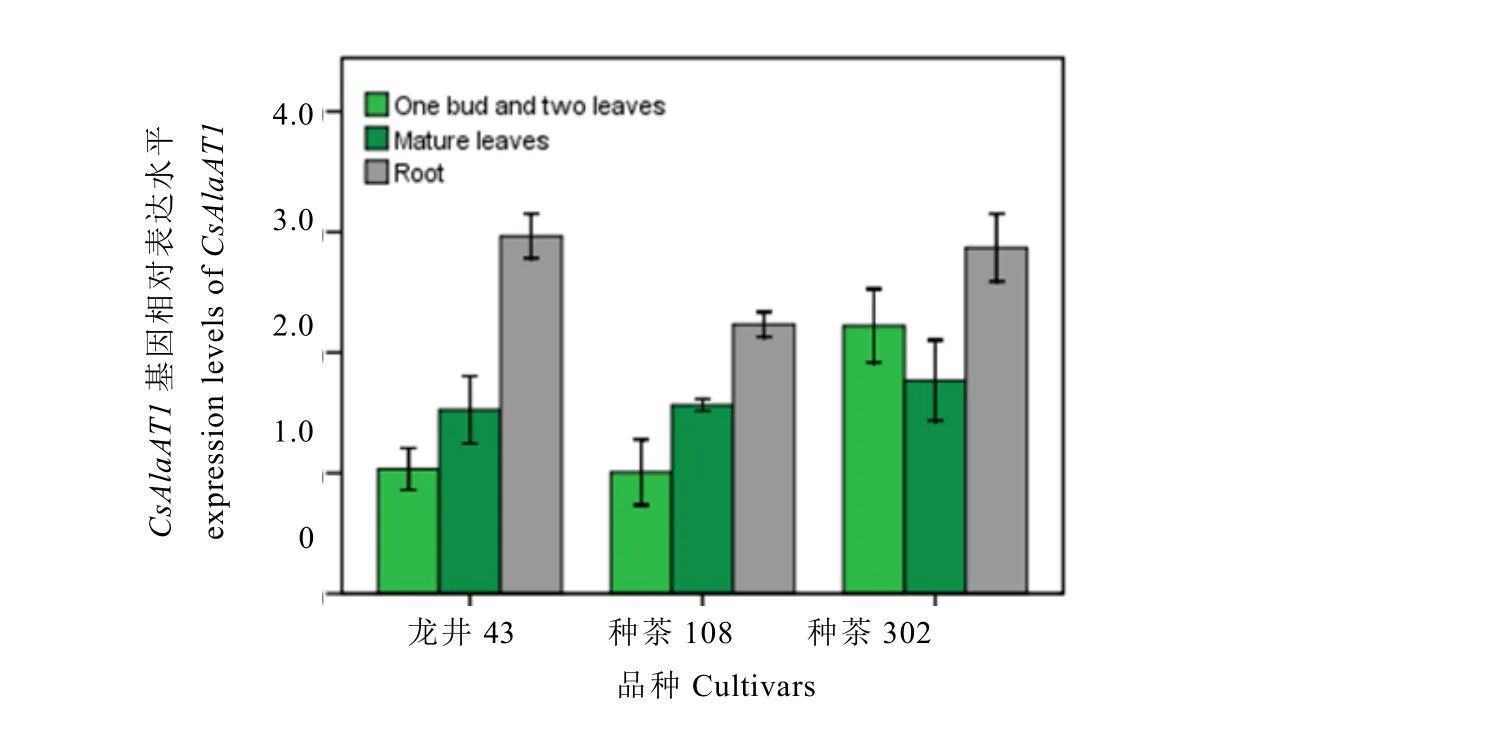
圖7 CsAlaAT1基因在不同組織的表達水平Fig.7 Relative expression levels of CsAlaAT1 in different tissues of tea plants
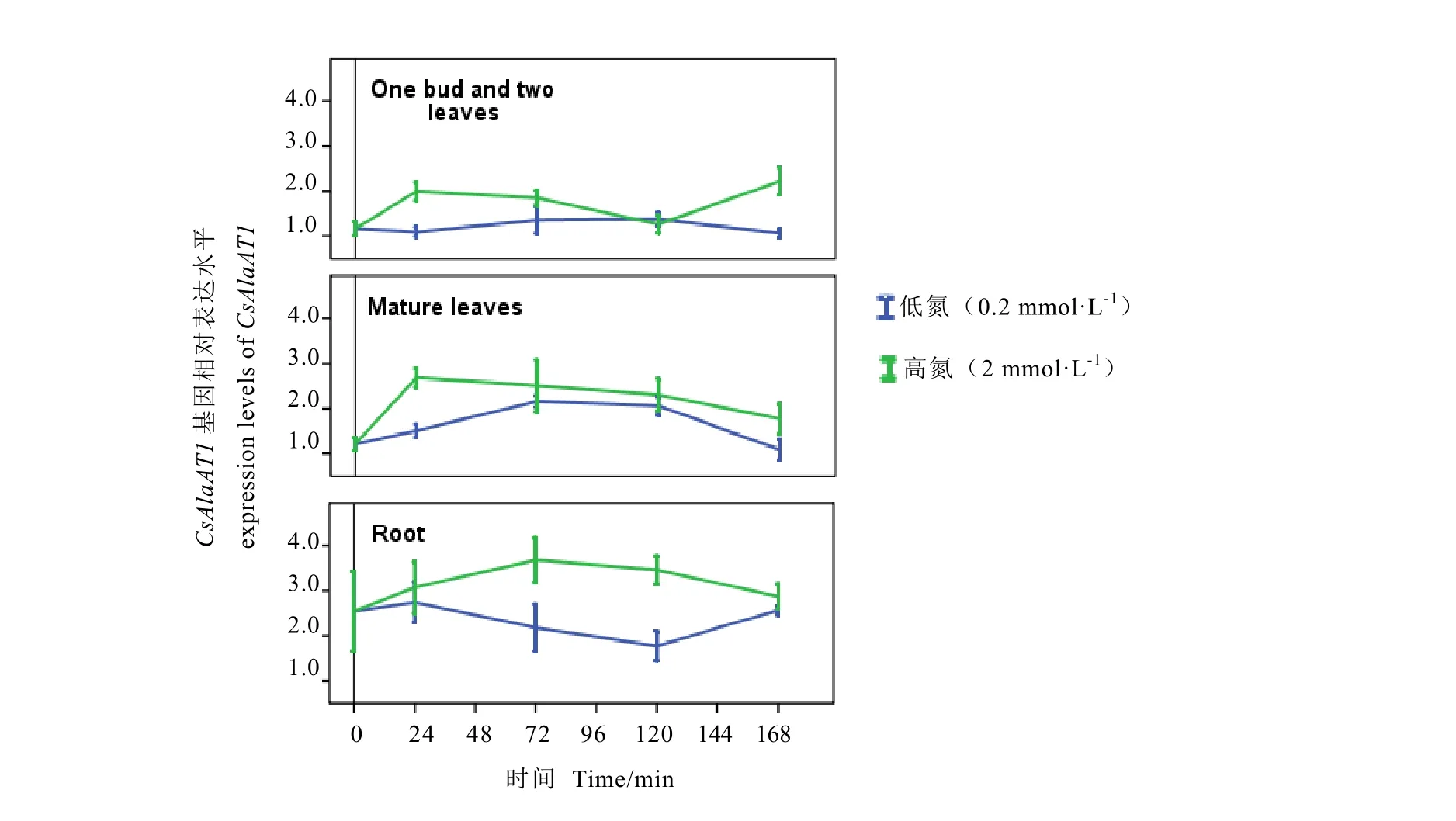
圖8 在低氮和高氮處理下茶樹CsAlaAT1基因不同時間的表達水平Fig.8 Time course of relative expression levels of CsAlaAT1 under low and high nitrogen levels
3 討論
3.1茶樹CsAlaAT1基因的克隆及其序列分析
茶樹葉片中克隆出的CsAlaAT1基因編碼541個氨基酸,與擬南芥、大麥和黍稷的AlaAT相似性達70%以上,屬于丙氨酸氨基轉移酶亞家族。預測CsAlaAT1不含信號肽,無跨膜結構,定位于線粒體。這與擬南芥AtAlaAT2同樣位于線粒體的預測結果相一致[14,21]。AtAlaAT位于線粒體可能與其代謝功能直接相關[22],線粒體 AlaAT似乎可以通過增加丙酮酸含量而激活線粒體中的交替氧化酶,從而調節植物的呼吸氧耗[23]。
3.2CsAlaAT1組織表達特性及其對氮素響應的時空表達差異
Rocha等[12]研究表明 AlaAT-A類基因在根系和結節中表達量最高,因此推測,AlaAT基因功能的發揮可能更多地依賴于根系。Miyashita等[11]發現擬南芥 AlaAT基因主要在葉片和根系的維管組織中表達,推測 AlaAT酶可能在丙酮酸或者丙氨酸的轉運過程中發揮重要作用,有利于維持整株植物的碳氮平衡[23]。本研究組織表達特性檢測表明,CsAlaAT1基因在各組織中均有表達,在根中表達量最高,這與其他植物中的研究結果一致。
CsAlaAT1在不同品種間的表達特性也存在差異。中茶302的一芽二葉中,該基因的表達量高于成熟葉,為其他兩個品種一芽二葉中的2倍以上。有研究顯示,在相同栽培條件下,中茶 302茶苗的生長勢以及鮮葉產量均高于中茶108和龍井43[24],因此推測該基因表達量的品種間差異與生長勢差異相關。AlaAT是與氮代謝相關的一種酶,中茶302一芽二葉中CsAlaAT1的高表達,可能有利于茶苗對氮素的利用,進而促進其生物量的增加。
AlaAT基因的表達受氮素調控。Kendziorek等[22]研究表明,小麥在缺氮培養基上培養5 d后,芽和根中的AlaAT酶活性降低了一半以上,而在正常培養基上培養的小麥中,AlaAT酶活性保持不變。氮素脅迫解除后恢復供氮短期內,黍稷[17]葉片中AlaAT2基因的表達量增加了3倍,玉米[25]根系中的AlaAT基因的表達量增加了14倍。本研究對茶苗在氮饑餓2周后,分別用低氮和高氮進行處理,恢復供氮24 h內,CsAlaAT1基因均上調表達,表明茶樹CsAlaAT1基因對氮素誘導有響應。高氮處理下,CsAlaAT1基因的表達量更高,表明CsAlaAT1基因的時空表達對高氮水平的響應更強烈;其一芽二葉和成熟葉中表達量均增加了2倍左右,根系中增加了約1.2倍,較之黍稷葉片(3倍)和玉米根系(14倍),CsAlaAT1基因的表達增量較低。本研究推測茶樹作為木本植物,調控機制較為復雜,多途徑參與對氮素的響應是造成該差異的原因。在根中,高氮處理24 h后,CsAlaAT1上調表達,而低氮處理24 h后,CsAlaAT1表達出現下調,其他作物中并沒有關于不同濃度氮素對CsAlaAT1基因表達調控的報道。本研究發現,CsAlaAT1在不同氮素濃度下表達模式不同,推測CsAlaAT1基因受氮素有效性的調節,但是該基因與茶樹氮素吸收和同化的關系有待進一步研究。
[1]Raun WR,Solie JB,Johnson GV,et al.Improving nitrogen use efficiency in cereal grain production with optical sensing and variable rate application [J].Agron J,2002,94(4):815-820.
[2]de Sousa CAF,Sodek L.Alanine metabolism and alanine aminotransferase activity in soybean(Glycine max)during hypoxia of the root system and subsequent return to normoxia [J].Environ Exp Bot,2003,50(1):1-8.
[3]吳洵,茹國敏.茶樹對氮肥的吸收和利用[J].茶葉科學,1986,6(2):15-24.
[4]周月琴,龐磊,李葉云,等.茶樹硝酸還原酶基因克隆及表達分析[J].西北植物學報,2013,33(7):1292-1297.
[5]馮素花.茶樹硝酸根轉運蛋白 NRT1.2、NRT1.5、NRT2.5基因的克隆與表達[D].杭州:中國農業科學院茶葉研究所,2014.
[6]汪進,添先鳳,江昌俊.茶樹硝酸鹽轉運蛋白基因的克隆與表達分析[J].植物生理學報,2014,50(7):983-988.
[7]徐乾.茶樹中與氨基酸合成相關的三個基因的克隆與分析[D].合肥:安徽農業大學,2012.
[8]林鄭和,鐘秋生,陳常頌.茶樹葉片 GDH、GS、GOGAT基因的克隆及熒光定量 PCR分析[J].茶葉科學,2012,32(6):523-529.
[9]胡娟,王麗鴛,韋康.茶樹 CsCDF1基因克隆及表達分析[J].茶葉科學,2015,35(5):501-511.
[10]Good AG,Johnson SJ,De Pauw M.Engineering nitrogen use efficiency with alanine aminotransferase [J].Can J Bot,2007,85(3):252-262.
[11]Miyashita Y,Dolferus R,Ismond KP,et al.Alanine aminotransferase catalyses the breakdown of alanine after hypoxia in Arabidopsis thaliana [J].Plant J,2007,49(6):1108-1121.
[12]Rocha M,Sodek L,Licausi F.Alalysis of alanine aminotransferase in various organs of soybean(Glycine max)and in dependence of different nitrogen fertilisers during hypoxic stress [J].Amino Acids,2010,39(4):1043-1053.
[13]Igarashi D,Miwa T,Seki M,et al.Identification of photorespiratory glutamate:glyoxylate aminotransferase(GGAT)gene in Arabidopsis [J].Plant J,2003,33(6):975-987.
[14]Liepman AH,Olsen LJ.Alanine aminotransferase homologs catalyze the glutamate:glyoxylate aminotransferase reaction in peroxisomes of Arabidopsis [J].Plant Physiol,2003,131(1):215-227.
[15]Rech J,Crouzet J.Partial purification and initial studies of the tomato L-alanine:2-oxoglutarate aminotransferase [J].Biochim Biophys Acta,1974,350(2):392-399.
[16]Kikuchi H,Hirose S,Toki S,et al.Molecular characterization of a gene for alanine aminotransferase from rice(Oryza sativa)[J].Plant Mol Biol,1999,39(1):149-159.
[17]Son D,Kobe A,Sugiyama T.Nitrogen-dependent regulation of the gene for alanine aminotransferase which is involved in the C4 pathway of panicum miliaceum [J].Plant Cell Physiol,1992,33(8):507-509.
[18]Son D,Sugiyama T.Molecular cloning of an alanine aminotransferase from NAD-malic enzyme type C4plant Panicum miliaceum [J].Plant Mol Biol,1992,20(4):705-713.
[19]Shrawat AK,Carroll RT,DePauw M.Genetic engineering of improved nitroge use efficiency in rice by the tissue-specific expression of alanine aminotransferase [J].Plant Biotechnology Jorunal,2008,6(7):722-732.
[20]Ruan J,Gerendas J,Hardter R,et al.Effect of nitrogen form and root-zone pH on growth and nitrogen uptake of tea(Camellia sinensis)plants [J].Ann Bot,2007,99(2):301-310.
[21]Ricoult C,Echeverria LO,Cliquet JB,et al.Characterization of alanine aminotransferase(AlaAT)multigene family and hypoxic response in young seedlings of the model legume Medicago truncatula [J].J Exp Bot,2006,57(2):3079-3089.
[22]Kendziorek M,Paszkowski A,Zagdanska B.Differential regulation of alanine aminotransferase homologues by abiotic stresses in wheat(Triticum aestivum L.)seedlings [J].Plant Cell Rep,2012,31(6):1105-1117.
[23]Gupta KJ,Zabalza A,van Dongen JT.Regulation of respiration when the oxygen availability changes [J].Physiol Plant,2009,137(4):383-391.
[24]闞君杰,丁仕波.山東日照市茶樹無性系良種引種試驗初報[J].茶業通報,2015,37(2):66-70.
[25]Muench DG,Good AG.Hypoxically inducible barley alanine aminotransferase:cDNA cloning and expression analysis [J].Plant Mol Biol,1994,24(3):417-427.
Cloning and Expression Analysis of Alanine Aminotransferase Gene in Camellia sinensis
BAI Peixian1,2,WANG Liyuan1*,WEI Kang1,RUAN Li1,CHENG Hao1,ZHANG Fen1,2,ZHANG Chengcai1,2
1.Tea Research Institute,Chinese Academy of Agricultural Sciences,National Center for Tea Improvement,Key Laboratory of Tea Biology and Resource Utilization,Ministry of Agriculture,Hangzhou 310008,China;
2.Graduate School of Chinese Academy of Agricultural Sciences,Beijing 100081,China
Alanine Aminotransferase(AlaAT)is a critical enzyme involved in carbohydrate and nitrogen metabolisms.In this study,a cDNA(1 747 bp)with a complete ORF(1 626 bp)of AlaAT1 was isolated from tea plant(Camellia sinensis).The cDNA encodes a protein with 541 amino acids,which has a molecular mass of 59.4 kD and a theoretical isoelectric point(pI)of 5.82.The deduced sequence of protein CsAlaAT1 shared 84% similarity with AlaAT1 in Arabidopsis thaliana,which contains a highly-conserved pyridoxal 5′-phosphate binding site.Secondary structure prediction showed that the CsAlaAT1 was comprised of alpha helix(40.67%),random coil(29.57%),beta turn(13.68%)and extended strand(16.08%),localized in mitochondrion and had no signal peptide or transmembrane structure.The expression levels of CsAlaAT1 in various tissues and its responses to different N concentration were investigated by real-time fluorescent quantitative RT-PCR.The results of RT-PCR showed that CsAlaAT1 expressed in all tissues of tea plant and the highest transcript level was observed in roots.The transcript abundanceof CsAlaAT1 was up-regulated by N in both shoots and mature leaves,especially under high N condition.Interestingly,the expression of CsAlaAT1 in roots was highly induced high N condition,but showed an opposite trend under low N treatment for 24 h.
tea plant(Camellia sinensis),Alanine aminotransferase,cloning,expression,nitrogen induction
S571.1;Q52
A
1000-369X(2016)04-405-09
2016-03-24
2016-04-21
國家自然科學基金(31570695)、國家現代農業產業技術體系(nycytx-23)、浙江省農業新品種選育重點專項(2012C2905-4)。
白培賢,女,碩士研究生,主要從事茶樹種質資源與育種研究。*通訊作者:wangly@tricaas.com

