生物乙烯研究進展
孫夢婷范曉蕾郭榮波邱艷玲趙曉嫻
(1.山東省沼氣工業化生產與利用工程實驗室 中國科學院青島生物能源與過程研究所生物燃料重點實驗室,青島 266101;2.中國科學院大學,北京 100049)
生物乙烯研究進展
孫夢婷1,2范曉蕾1郭榮波1邱艷玲1趙曉嫻1
(1.山東省沼氣工業化生產與利用工程實驗室 中國科學院青島生物能源與過程研究所生物燃料重點實驗室,青島 266101;2.中國科學院大學,北京 100049)
乙烯是世界需求量最大的化工原料,被廣泛用于制造塑料制品、紡織品及其他化工產品。目前,最常用的生產乙烯的方式是通過石油裂解制備乙烯,但其存在生產成本高、環境不友好等弊端,這為生物乙烯的發展帶來契機。生物乙烯合成以CO2或生物質等可再生能源為原料,具資源節約、環境友好的優勢。描述了生物乙烯合成的兩種途徑──間接和直接合成途徑,著重論述微生物通過乙烯合成酶(EFE)直接合成乙烯的原理,描述了EFE的結構、序列及催化機制,列舉了數個工程菌異源合成乙烯的成功案例,提出提高產量的策略,并對微生物制乙烯的發展前景進行展望。
生物乙烯;直接合成;微生物;乙烯合成酶;異源表達
乙烯是重要的化工原料,主要用于生產聚乙烯、環氧乙烯、二氯乙烷等,其生產規模、技術水平和產量可衡量一個國家的化工工業水平[1]。目前,多數國家通過石腦油、瓦斯油等裂解制備乙烯。近年,中國乙烯生產量和自給率不斷提高,但仍不及需求的增長速度,并且,隨石油資源的日益枯竭和價格攀升,乙烯的傳統生產成本日益增加,這為生物乙烯提供了發展契機[2]。
乙烯生物合成以生物質為原料,利用生物體生長代謝活動,通過直接或間接方法獲得乙烯[3,4]。間接合成以可再生生物質為原料,通過微生物發酵生成乙醇,催化劑作用于乙醇脫水生成乙烯。間接合成經多年發展已實現產業化,并有效緩解了能源緊缺問題,但其存在弊端,如產力低、工藝不合理、規模小等[5]。直接合成是生物體以有機物或CO2為原料在體內直接合成乙烯,通過生物工程手段將其引入微生物來生產乙烯,投資小、環境友好、產品純度高,是切實可行的新型合成路線[6]。
目前,國內外生物乙烯研究多集中在間接合成的成本降低和優化生產工藝上,并未足夠重視直接合成。本文在之前研究基礎上,綜述了生物制乙烯技術現狀,著重論述直接合成法生物學基礎和異源表達案例,并對生物乙烯發展前景進行展望。
1 生物乙烯合成
1.1 生物乙烯間接合成
間接合成起步較早,已具備較成熟的生產工藝和流程,具體如圖1-A所示,蔗糖、淀粉和纖維素等水解生成葡萄糖等還原糖,經微生物糖酵解生成乙醇,再在催化劑作用下脫水生成乙烯。生物乙醇來源于玉米、甘蔗、甜高粱和甜菜等作物,由此帶來成本高和糧食安全問題,因此,近年來國內外研究多集中于降低乙醇制造成本和非糧乙醇方面,將植物秸稈中的纖維素、半纖維素降解并供給微生物發酵生產乙醇,成為未來生物乙醇主要生產模式。目前,間接合成法最成熟的催化劑是活性氧化鋁,其穩定性好、壽命長,但存在反應溫度高、進料空速低等不足,經改良的HZSM-5、SAPO-34分子篩類催化劑及雜多酸催化劑等,性能優于前者,但仍無法滿足商業化需求。因此,開發高性能新型催化劑成為間接合成法的關鍵所在。乙醇脫水生成乙烯有絕熱床和等溫床兩種工藝,均存在轉化率低、能耗高等不足,有待優化。
與石油裂解制乙烯相比,間接合成具以下優點:(1)原料來源廣,為可再生生物質;(2)反應條件溫和;(3)工藝流程簡單;(4)能耗低;(5)投資小;(6)環境友好。但其仍存在生物乙醇生產成本高、生產規模小、催化劑性能有待提高、反應器設計不合理、生產工藝待優化等劣勢[7-9]。
1.2 生物乙烯直接合成
許多生物體具乙烯合成功能,包括一些植物、微生物和動物,其分別采取不同的乙烯合成途徑[10-12]。直接合成法將適當的乙烯合成途徑通過生物工程手段引入微生物,使微生物以有機物或CO2為原料合成乙烯,與間接合成法相比,其具有顯著優勢:(1)設備少,不需催化劑;(2)成本低,原料來源廣;(3)能耗低,CO2排放少,環境友好(光合自養菌可通過光合作用固定CO2轉化為乙烯);(4)產物純度高,副產物少;(5)產率受O2和營養條件影響,生產過程可實現優化控制。
2 生物乙烯直接合成原理
2.1 植物合成乙烯
2.1.1 植物合成乙烯途徑 在高等植物中,如圖1-b所示,乙烯合成前體為甲硫氨酸(Methionine,Met),關鍵酶為ACS(ACC合酶)和ACO(ACC氧化酶)。反應過程如下:Met在S-腺苷甲硫氨酸合成酶(S-Adenosylmethionine synthetase,SAMS)作用下生成S-腺苷甲硫氨酸(S-adenosylmethionine,SAM),ACS催化SAM生成5'-甲硫基核糖(5'-methylthioribose,MTR)和1-氨基-1-羧基環丙烷(1-aminocyclopropane-1-carboxylic acid,ACC),MTR通過甲硫氨酸循環參與Met再生,ACC則在ACO催化下生成氰化物并放出乙烯[13]。
2.1.2 植物合成乙烯的調控 植物合成乙烯關鍵酶是ACS和ACO。ACS催化SAM轉化為ACC,是限速步驟。ACS以磷酸吡哆醛為輔基,由多基因家族編碼,其基因表達和催化活性響應于內外部各種刺激,乙烯積累可抑制ACS基因表達,自身mRNAs積累增強ACS催化活性。ACO催化ACC生成乙烯,其需要抗壞血酸和氧作輔助底物,Fe2+和CO2作輔助因子,解偶聯劑和自由基清除劑能抑制乙烯生成。ACO由多基因家族編碼,乙烯、Cu2+、NaCl、ACC等可誘導ACO表達[14,15]。Hamilton等[16]使用番茄植株ACO1基因轉化釀酒酵母后,可將ACC轉化為乙烯。乙烯合成還受信號轉導通路調節,擬芥南中存在5個乙烯受體:ETR1、ERS1、ETR2、ERS2和EIN4,它們均是乙烯合成的負調控因子[17]。植物合成乙烯途徑調控機制復雜,易受干擾,為產業化生產帶來困難。
2.2 微生物合成乙烯
2.2.1 微生物合成乙烯途徑 微生物合成乙烯途徑分為兩條:一是KMBA途徑,發生在大腸桿菌(E. coli)、淺白隱球酵母(C. albidus)等細菌中,如圖1-C所示,Met通過轉氨作用生成中間產物2-酮-4-甲基硫代丁酸(2-keto-4-methylthiobutyric acid,KMBA),KMBA被氧化裂解放出乙烯[18]。二是EFE途徑,發生在丁香假單胞桿菌(P. syringae)、青霉菌(P. digitatum)等微生物中,如圖1-D所示,谷氨酸(Glu)脫氨生成 α-酮戊二酸(α-ketoglutarate,AKG),AKG在乙烯合成酶(ethylene-forming enzyme,EFE)作用下生成乙烯。由于后者的乙烯合成速率遠高于前者,因此EFE途徑是微生物直接合成乙烯的研究重點。

圖1 生物乙烯間接與直接合成途徑
2.2.2 EFE的發現、結構和efe基因 1940年,Yang和Biale等[19,20]首次報道青霉菌(P. digitatum)產乙烯,青霉菌成為首個被鑒定的產乙烯微生物。自青霉菌提純EFE,測得EFE是相對分子質量為42 kD的單體蛋白,在Fe2+和O2存在下,催化AKG和精氨酸(Arg)生成琥珀酸鹽、胍類、吡咯啉-5-羧酸(P5C)、CO2和乙烯。丁香假單胞菌(P. syringae)是目前已知的產乙烯效率最高的微生物。提取丁香假單胞菌的EFE進行檢測,其相對分子質量為42 kD且為單體蛋白,在Fe2+、O2、Arg和AKG存在下催化乙烯生成,這與青霉菌中EFE性質基本一致[21]。
實驗表明,青霉菌和丁香假單胞菌的EFE的N端序列不同,但兩者具一整段相似保守序列,推測這段序列是EFE催化功能所必需的。EFE功能域相對保守,定向誘變丁香假單胞菌efe基因,以Glu殘基取代His殘基,使其在大腸桿菌中表達,分離純化EFE后進行試管實驗,檢測其Km值、kcat值及熱穩定性。實驗結果表明,His305和His335位點發生突變的酶,活性分別減少至原活性水平的40%和60%,His189和His233位點發生突變的酶則完全失活,表明這兩個位點在絡合鐵離子時起關鍵作用,在His268位點突變的酶未完全失活,但活性急劇下降,表明此位點突變破壞了EFE活性部位。除此之外,EFE還含有兩個保守的氨基酸殘基Asp191和Arg277,對酶催化活性起重要作用[22]。
上述實驗將丁香假單胞菌efe基因在大腸桿菌中克隆并表達,觀測到乙烯生成,這表明在不同菌株中,單個efe基因是產生乙烯的充分條件,因此,是否具efe基因可確定待測菌株是否具產乙烯能力。Sato等[23]分離丁香假單胞菌突變株P. syringae pv. phaseolicola PK2(Kudzu菌株,產乙烯菌株)efe基因作引物,對P. syringae pv. cannabina 和P. syringae pv. sasemi 兩種菌株基因進行PCR擴增,它們的PCR產物均包含大小與efe基因相似的片段,以Kudzu菌株efe基因作DNA探針與其PCR產物雜交,結果顯示產物中均含efe同源基因,據此表明兩菌株均具產乙烯能力。
2.2.3 EFE催化機制和化學計量分析 對青霉菌和丁香假單胞菌的EFE進行試管實驗,測反應化學計量數,得化學方程式如下:
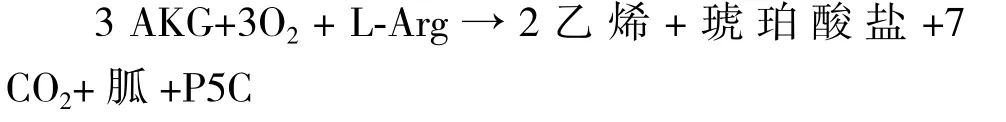
反應底物AKG和L-Arg比例為3∶1,產物乙烯、琥珀酸鹽和P5C比例為2∶1∶1。EFE同時催化兩個反應發生,主反應是2分子AKG被氧化生成2分子乙烯和6分子CO2,副反應是1分子AKG轉化為琥珀酸鹽和CO2各1分子的同時,Arg轉化為胍類和P5C。如果EFE是可催化兩個反應的復合酶,并且兩反應可分離,那么利用工程化手段改造EFE,使其僅催化主反應,不催化副反應,不生成副產物,將會大大提高產率和反應物利用率。因此,關于EFE功能結構及催化機制應當深入研究。
2.2.4 EFE反應與胞內碳流分析 對EFE反應所涉及的新陳代謝通路進行分析,優化反應途徑,使反應產物與底物摩爾比最大化,降低副產物量、能量和輔因子消耗,從而使EFE反應有序高效進行成為可能。
乙烯的碳來源大體可分為3類:一是CO2固定,存在于光合自養型生物中,CO2被核酮糖1,5-二磷酸加氧酶(Rubisco)固定,通過卡爾文循環進入EFE反應,消耗ATP和還原劑轉換為乙烯;二是糖酵解,己糖在NAD(P)+依賴性的甘油-3-磷酸脫氫酶、異檸檬酸脫氫酶、丙酮酸脫氫酶等催化下分解,其中3個碳進入EFE反應生成乙烯;三是磷酸戊糖途徑,木糖或混合糖通過磷酸戊糖途徑進入EFE反應產生乙烯,還原產物和碳產量與糖酵解相近。
3條途徑中,CO2固定途徑雖需大量ATP和還原劑,但為碳負過程,另兩種途徑雖所需輔因子較少,但代謝過程中存在碳丟失,因此,應當找到有效策略來挽救丟失的碳原子[24]。
除確定最佳代謝途徑外,阻礙途徑和競爭途徑也應當被納入研究范疇,通過流量平衡分析(FBA)和代謝流分析(MFA)找到影響乙烯產量的因素。Larsson等[25]對efe基因轉化的釀酒酵母進行FBA,研究表明添加外源脯氨酸(Pro)、使用NAD依賴型谷氨酸脫氫酶(NAD-GDH)代替NADH依賴型谷氨酸脫氫酶(NADH-GDH)、使用谷氨酸鹽代替銨鹽作氮源均可增加乙烯產量。另外,通過改變O2量來限制其呼吸作用,同樣會影響乙烯產量。
2.3 動物合成乙烯
哺乳動物可合成少量乙烯,有報道顯示,大鼠肝臟中存在KMBA途徑合成乙烯。由于動物合成乙烯量少,其對產業化生產乙烯并無太大價值。
3 生物乙烯異源合成案例
已有研究表明,細菌efe基因在大腸桿菌(E. coli)、釀酒酵母(Saccharomyces cerevisiae)、藍細菌聚球藻屬(Synechococcus)、藍細菌集胞藻屬(Synechocystis)和木霉(Trichoderma)等菌株中實現了異源表達,并使轉化后的工程菌株獲得乙烯合成能力。
3.1 假單胞菌合成乙烯
目前已知產乙烯微生物中,丁香假單胞菌產量最高,其中Kudzu菌株和丁香假單胞菌大豆致病突變株(P. syringae pv. glycinea)是研究最多的菌株。為進一步提高乙烯產量,Ishihara等[26]構建了高拷貝數、廣譜宿主質粒載體pMEFE1,其上攜帶來源于Kudzu菌株的efe基因,將pMEFE1質粒分別導入丁香假單胞菌(P. syringae)、惡臭假單胞菌(P. putida)、大腸桿菌JM109后,重組細胞均具EFE合成能力,并且乙烯產量比親代丁香假單胞菌分別高出41倍、20倍和5.3倍。Wang等[27]將丁香假單胞菌efe基因重組至惡臭假單胞菌KT2440的16S rRNA位點,PCR結果顯示轉化后菌株DC1、DC2、DC3分別具有3、4和5個efe基因拷貝,其乙烯產量分別為36.2、47.2、53.8 mg/(h·g),與親代丁香假單胞菌14.7 mg/(h·g)的產量相比大幅增加。將攜帶efe基因的pBBR1MCS2質粒載體轉入DC3菌株以繼續增加efe拷貝數,乙烯合成量達到80.2 mg/(h·g),進一步說明efe基因拷貝數與乙烯合成量呈正相關。對重組菌乙烯合成動力曲線進行分析,乙烯產量隨時間逐漸增高,7 h達到峰值,之后開始下降,繼續培養至17 h添加0.25%葡萄糖溶液,產力比7 h時提高40%,這說明碳源葡萄糖是乙烯合成的限制性因子。
3.2 大腸桿菌異源合成乙烯
efe基因最早在大腸桿菌中異源表達。來自丁香假單胞菌Kudzu菌株的efe基因被克隆,通過高拷貝pUC19載體在大腸桿菌中表達。實驗將轉化后的大腸桿菌分別在25℃和30℃下培養,結果表明,25℃下乙烯產量較高,推測37℃時EFE易形成包含體從而活性降低。另外,將轉化后的大腸桿菌在添加外源底物的試管內反應,測得乙烯生成量為相同條件下細胞內反應(即底物僅來自于胞內)的5倍,這表明底物利用率與乙烯產量呈正相關。
3.3 釀酒酵母異源合成乙烯
2008年,Pirkov等[28]將丁香假單胞菌efe基因轉入釀酒酵母中,使其利用葡萄糖為碳源合成乙烯,產量達到(320±90)μg/g葡萄糖。2011年,Larsson等[25]對釀酒酵母乙烯合成進行代謝流分析,結果表明,使用NAD-GDH代替NADH-GDH可有效提高乙烯產量,這是由于谷氨酸脫氫酶(GDH)催化谷氨酸鹽脫氨產生乙烯合成底物AKG,GDH分為NAD和NADP依賴型兩種,NADPH主要來源于磷酸戊糖途徑,該途徑存在碳丟失,使碳源減少,AKG水平下降,從而使乙烯產量減少。實驗以半乳糖替代葡萄糖作碳源,產量急劇下降,說明酵母合成乙烯的最佳碳源是葡萄糖。除此之外,由于乙烯合成副產物P5C是Pro合成的中間體,添加外源脯Pro可使P5C轉而合成Glu,提高乙烯產量。為確認影響乙烯產量因素,2013年,Johansson等[29]對釀酒酵母在不同條件下乙烯產量進行測定。分析表明,O2量影響乙烯產量,由于O2維持細胞呼吸作用,并重新氧化NADH用于乙烯合成,是重要限制性因子。實驗以谷氨酸鹽代替銨鹽作氮源,乙烯合成速率大大提高。2014年,Johansson等[30]繼續對酵母乙烯合成的氮代謝進行分析,以銨鹽、谷氨酸鹽、Arg、谷氨酸鹽/Arg分別作氮源。結果表明,以谷氨酸鹽作氮源乙烯產量最高,這是由于谷氨酸鹽在GDH作用下轉化為乙烯合成底物AKG;以Arg作氮源產量減少,但Arg缺陷型突變株完全沒有乙烯生成,表明雖然Arg作氮源產量較少,但卻是乙烯合成所必需。因此,高濃度AKG和一定數量Arg是乙烯產量最大化所必需。綜上可得,利用酵母合成乙烯,其產量受到碳源、氮源、O2量、底物濃度、輔因子NADP等影響。
3.4 木霉異源合成乙烯
木霉是一種絲狀真菌,其內富含豐富的纖維素酶系,能將纖維素降解為葡萄糖,這意味著,一旦木霉具乙烯合成能力,便能利用農業廢棄物中的纖維素為碳源生產乙烯。2008年,Tao等[31]首次將丁香假單胞菌efe基因轉入綠色木霉菌(Trichoderma viride)中,得到具乙烯合成能力的轉化菌株Y2,速率達1 059.7 nL/(L·h)。同樣,乙烯產量受碳源和氮源影響:以纖維素作碳源,Y2菌株乙烯合成速率最高,以羧甲基纖維素鈉或乳糖做碳源,產量減少;以蛋白胨作氮源,乙烯產量升高。另外,用包含小麥秸稈的固體培養基培養Y2菌株,可獲得較高乙烯產量。2011年,Chen等[32]將丁香假單胞菌efe基因轉入里氏木霉菌(Trichoderma reesei)中,觀察到乙烯合成量隨efe表達量增加而增加,以稻草秸稈為碳源培養轉化菌株,乙烯合成量達到4 012 nL/(L·h)。以上兩項研究均表明,efe基因轉化的木霉菌可利用植物廢棄物轉化成乙烯,這使乙烯低碳環保生產成為可能。
3.5 藍細菌異源合成乙烯
與大腸桿菌、酵母、木霉等產乙稀工程菌株相比,產乙稀工程藍細菌具有明顯的優勢:第一,乙烯合成速率較高;第二,藍細菌可通過光合作用將CO2轉化為乙烯,將乙烯生產與CO2減排有機結合,真正實現了乙烯的綠色合成;第三,可利用海水培養基對藍細菌進行培養,培養基廉價易得,資源友好。因此,我們認為藍細菌光合產乙稀將是最具發展前景的生物乙烯生產方式。早在1997年,Sakai和Fukuda等[33]將丁香假單胞菌Kudzu菌株的efe基因轉入藍細菌聚球藻Synechococcus sp. PCC 7942中,使其具有產乙烯能力,但隨著細胞分裂,由于質粒pEXE3和宿主染色體psb AI位點發生同源重組,efe基因拷貝數減少,使得菌株乙烯合成量急劇下降。2003年,Takahama等[34]將丁香假單胞菌efe基因直接插入藍細菌聚球藻Synechococcus elongatus PCC 7942的psb AI位點,使轉化菌株獲得451 nL/(mL·h·OD730)的乙烯合成能力,但聚球藻生長和代謝速率隨時間減慢,相應乙烯合成量也減少。2012年,Ungerer等[35]將Kudzu菌株efe基因修飾后轉入藍細菌集胞藻Synechocystis sp. PCC 6803中,EFE穩定性大幅提高,使菌株能夠持續合成乙烯,最高乙烯合成速率達5 650 μL/(L·h),并觀察到高達5.5%的碳進入乙烯合成途徑。代謝途徑優化是提高藍細菌產乙烯能力的另一重要途徑。本研究單位朱濤等[36]前期研究表明,經過TCA循環途徑的修飾和優化,在抑制α-酮戊二酸脫羧酶(OGDC)和琥珀酸半醛脫氫酶(SSADH)表達的同時,將大腸桿菌中編碼α-酮戊二酸透過酶(KgtP)的基因導入藍細菌phaAB位點,從而增加細胞內底物α-酮戊二酸的量,結合過量表達EFE,能夠大大提高藍細菌工程菌株的乙烯合成能力,合成速率達到了9.7 mL/(L·h)。
4 產物乙烯的分離與純化
產物分離與純化是乙烯生產的關鍵步驟。在傳統石油化工領域,乙烯分離技術有深冷分離法、吸附分離法、膜分離法、膨脹機法、金屬絡合分離法及聯合工藝等。生物制乙烯與石油裂解制乙烯的產物組分不同,通常,前者的產物中除乙烯外,還存在CO2,水蒸氣、N2、O2等,但不存在石油裂解過程中產生的金屬及其他污染物。另外,光合自養產乙烯微生物通過光合作用產生O2,乙烯在一定濃度O2中具可燃性,這也是分離生物乙烯時必須考慮的因素[6,27]。因此,在設計生物乙烯分離與純化工藝過程中,不僅要運用和借鑒石油裂解制乙烯的分離、純化工藝,還要充分考慮生物制乙烯的產物組分和性質,從而設計出安全、高效的分離與純化技術。
4.1 深冷分離法
該技術的分離流程包括氣體凈化系統、壓縮冷卻系統和精餾分離系統,具體過程是利用產物混合氣中各組分相對揮發度的差異,低溫下將各組分按工藝要求冷凝下來,再用精餾法將各類產物逐一分離,從而實現乙烯的分離與純化。此方法乙烯回收率較高,可用于大規模生產。
4.2 吸附分離法
該方法利用吸附劑對混合氣中各組分的吸附選擇性不同,通過改變壓力或溫度來實現分離,分為變壓吸附法、變溫吸附法和變溫變壓吸附法。按所用載體不同,吸附劑分為分子篩類、樹脂類、A1203類、SiO2類、活性炭類和粘土類等,對于生物乙烯來講,開發適用、高效的新型吸附劑是運用此方法分離乙烯的關鍵。
4.3 膜分離法
該方法利用混合氣各組分在膜中滲透速率的差異對乙烯進行分離。用于分離乙烯的膜主要有平片膜和中空纖維膜,膜中金屬離子有Na+、Ag+和Cu2+等,各組分與膜中離子形成絡合物,進行遷移從而得到分離。目前,膜分離法在乙烯分離中還處于研究階段,進一步的研究可根據生物合成乙烯方式與傳統石油生產方式的混合氣組分的差異變換膜材料,有望實現乙烯的高效分離。
4.4 膨脹機法
該法的核心是膨脹制冷技術,利用高壓氣體通過膨脹機在近似等熵膨脹的同時輸出外功,產生比節流更大的溫降,使混合氣中露點較高的組分冷凝,達到分離乙烯的目的。其優點是能耗小、操作靈活、對進料要求不太嚴格,但是膨脹制冷技術在國內尚未得到解決。
4.5 金屬絡合分離法
該法采用溶于芳烴溶劑中的一種雙金屬鹽類四氯化亞銅鋁絡合物,從混合氣中有選擇性地絡合吸附乙烯組份,乙烯分子與絡合物所形成較弱的鍵,可在緩和條件下進行汽提解吸,從而得到純度較高的聚合級乙烯。此方法的局限性是絡合物的制備難度較大,且國內目前尚無法掌握其關鍵技術。
4.6 聯合工藝
采用聯合工藝會改善分離效果以及經濟性,如變壓吸附法與蒸餾聯合、變壓吸附法與膜分離聯合等,均使乙烯的選擇性大大提高[37]。
5 展望
乙烯作為重要化工原料,其產量與國民生產生活息息相關。隨著化石能源枯竭,傳統合成成本升高,且產量已無法滿足我國對乙烯的需求,因此降低生產成本、提高產量、開辟新型合成途徑成為乙烯生產的重點研究方向。微生物合成乙烯具低成本、低能耗、環境友好、易控制等優勢,特別是藍細菌,可直接利用太陽能將二氧化碳合成乙烯,顯示出極大的發展潛力,因此我們認為藍藻乙烯是最具發展前景的生物乙烯生產方式之一。但受限于包括EFE的結構功能關系,以及轉基因微生物乙烯合成代謝機制等在內的許多基礎問題仍未清晰,生物乙烯的直接合成技術仍處于發展的萌芽期,面臨產量低、EFE不穩定、未實現產業化等問題。因此,對微生物合成乙烯的研究,依然任重道遠。鑒于此,筆者認為,今后微生物合成乙烯研究重點可集中于以下幾個方面:第一,EFE的結構功能關系研究。DNA改組技術是揭示其結構功能關系及提高催化效率的有效手段,通過非理性分子設計結合定向篩選,可賦予EFE特殊的結構與功能。第二,代謝通路研究。通過深入分析來得到各代謝物更為精確的化學計量關系,利用合成生物學代謝途徑改造手段設計出高產量通路,使乙烯高效合成成為可能。第三,工藝技術研究。通過培養系統條件優化以及發展低成本高效率的產物分離技術等,從系統集成上促進微生物合成乙烯技術體系的發展。
[1] 賈瑩瑩, 杜平, 杜風光, 等. 生物乙烯的發展狀況及展望[J].當代化工, 2011, 40(10):1071-1072.
[2] Haro P, Ollero P, Trippe F. Technoeconomic assessment of potential processes for bio-ethylene production[J]. Fuel Processing Technology, 2013, 114(4):35-48.
[3] Hu YC, Zhan NN, Dou C, et al. Selective dehydration of bio-ethanol to ethylene catalyzed by lanthanum-phosphorous-modified HZSM-5 Influence of the fusel[J]. Biotechnology Journal, 2010, 5(11):1186-1191.
[4] 孫芝蘭, 陳以峰. 乙烯的直接生物合成[J]. 生物工程學報,2013, 29(10):1431-1440.
[5] 胡燚, 李慧, 黃和, 等. 生物乙烯開發進展與產業化[J]. 現代化工, 2009, 29(1):6-9.
[6] Eckert C, Wu X, Wei X, et al. Ethylene-forming enzyme and bioethylene production[J]. Biotechnol Biofuels, 2014, 7(1):33.
[7] Fan D, Dai DJ, Wu HS. Ethylene formation by catalytic dehydration of ethanol with industrial considerations[J]. Materials, 2013, 6(1):101-115.
[8] 杜平, 賈瑩瑩, 孫沛勇, 等. 纖維乙醇制生物乙烯前景展望[J].廣東化工, 2013, 9(40):78-79.
[9] 胡鐵剛, 程可可, 張建安, 等. 生物乙醇催化制備乙烯的研究進展[J]. 現代化工, 2007(Z):96-99.
[10] Schlink K. Gene expression profiling in wounded and systemic leaves of Fagus sylvatica reveals up-regulation of ethylene and jasmonic acid signaling[J]. Plant Biology, 2011, 13(3):445-452.
[11] Li Q, Csanády GA, Kessler W, et al. Kinetics of ethylene and ethylene oxide in subcellular fractions of lungs and livers of male B6C3F1 mice and male fischer 344 rats and of human livers[J]. Toxicol Ogical Science, 2011, 123(2):384-398.
[12] Lin ZF, Zhong SL, Grierson D. Recent advances in ethylene research[J]. Journal of Experimental Botany, 2009, 60(12):3311-3336.
[13] 陳建新, 劉國順, 陳占寬, 等. 乙烯生物合成途徑及其相關基因工程的研究進展[J]. 熱帶亞熱帶植物學報, 2002, 10(1):83-98.
[14] 劉進平. 乙烯生物合成關鍵酶基因研究進展[J]. 熱帶農業科學, 2013, 33(1):51-57.
[15] 陳銀華, 黃偉, 王海. ACC氧化酶基因研究進展[J]. 海南大學學報:自然科學版, 2006, 24(2):194-200.
[16] Hamilton AJ, Bouzayen M, Grierson D. Identification of a tomato gene for the ethylene-forming enzyme by expression in yeast[J]. Plant Biology, 1991, 88(8):7434-7437.
[17] Chen YF, Gao ZY, Kerris RJ, et al. Ethylene receptors function as components of high-molecular-mass protein complexes in Arabidopsis[J]. PLoS One, 2010, 5(1):e8640.
[18] Qadir A, Hewett EW, Long PG, et al. A non-ACC pathway for ethylene biosynthesis in Botrytis cinerea[J]. Postharvest Biology and Technology, 2011, 62(3):314-318.
[19] Young RE, Pratt HK, Biale JB, et al. Identification of ethylene as a volatile product of the fungus Penicillium digitatum[J]. Plant Physiology, 1951, 26(7):304-310.
[20] Biale JB. Effect of emanations from several species of fungi on respiration and color development of citrus fruits[J]. Science,1940, 91(2367):458-459.
[21] Nagahama K, Ogawa T, Fujii T, et al. Purification and properties of an ethylene-forming enzyme from Pseudomonas syringae pv. phaseolicola PK2[J]. Journal of General Microbiology, 1991, 10(137):2281-2286.
[22] Nagahama K, Yoshino K, Matsuoka M, et al. Site-directed mutagenesis of histidine residues in the ethylene-forming enzyme from Pseudomonas syringae[J]. Journal of Fermentation Bioengineering, 1998, 85(3):255-258.
[23] Sato M, Watanabe K, Yazawa M, et al. Detection of new ethyleneproducing bacteria, Pseudomonas syringae pvs. cannabina and sesami, by PCR amplification of genes for the ethylene-forming enzyme[J]. Bacteriology, 1997, 87(12):1192-1196.
[24] Zhang C, Wei ZH, Ye BC. Quantitative monitoring of 2-oxoglutarate in Escherichia coli cells by a fluorescence resonance energy transfer-based biosensor[J]. Appl Microbiol Biotechnol, 2013,97(7):8307-8316.
[25] Larsson C, Snoep JL, Norbeck J, et al. Flux balance analysis for ethylene formation in genetically engineered saccharomyces cerevisiae[J]. IET Systems Biology, 2011, 5(4):245-251.
[26] Ishihara K, Matsuoka M, Ogawa T, et al. Ethylene production using a broad-host-range plasmid in Pseudomonas syringae and Pseudomonas putida[J]. Journal of Fermentation Bioengineering,1996, 82(5):509-511.
[27] Wang Jp, Wu Lx, Xu F, et al. Metabolic engineering for ethylene production by inserting the ethylene-forming enzyme gene(efe)at the 16S rDNA sites of Pseudomonas putida KT2440[J]. Bioresource Technology, 2010, 101(3):6404-6409.
[28] Pirkov I, Albers E, Norbeck J, et al. Ethylene production by metabolic engineering of the yeast Saccharomyces cerevisiae[J]. Metabolic Engineering, 2008, 10(5):276-280.
[29] Johansson N, Quehl P, Norbeck J, et al. Identification of factors for improved ethylene production via the ethylene forming enzyme in chemostat cultures of Saccharomyces cerevisiae[J]. Microbial Cell Factories, 2013, 12(1):89.
[30] Johansson N, Persson KO, Quehl P, et al. Ethylene production in relation to nitrogen metabolism in Saccharomyces cerevisiae[J]. FEMS Yeast Research, 2014, 14(7):1110-1118.
[31] Tao L, Dong HJ, Chen X, et al. Expression of ethylene-forming enzyme(EFE)of Pseudomonas syringae pv. glycinea in Trichoderma viride[J]. Appl Microbiol Biotechnol, 2008, 80(4):573-578.
[32] Chen SF, Qin WS, Chen X, et al. Overexpression of bacterial ethylene-forming enzyme gene in Trichoderma reesei enhanced the production of ethylene[J]. International Journal of Biological Sciences, 2010, 6(1):96-106.
[33] Sakai M, Ogawa T, Matsuoka M, et al. Photosynthetic conversion of carbon dioxide to ethylene by the recombinant cyanobacterium,Synechococcus sp. PCC 7942 which harbors a gene for the ethyleneforming enzyme of Pseudomonas syringae[J]. J Ferment Bioeng,1997, 84(5):434-443.
[34] Takahama K, Matsuoka M, Nagahama K, et al. Construction and analysis of a recombinant Cyanobacterium expressing a chromosomally inserted gene for an ethylene-forming enzyme at the psbAI locus[J]. Journal of Bioscience and Bioengineering, 2003,95(3):302-305.
[35] Ungerer J, Tao L, Davis M, et al. Sustained photosynthetic conversion of CO2to ethylene in recombinant Cyanobacterium Synechocystis 6803[J]. Energy & Environmental Science, 2012,5(10):8998-9006.
[36] Zhu T, Xie XM, Li ZM, et al. Enhancing photosynthetic production of ethylene in genetically engineered Synechocystis sp. PCC 6803[J]. Green Chemistry, 2015, 17(1):421-434.
[37] 趙光輝, 李景燕, 李小軍, 等. 煉廠干氣中乙烯的分離技術及綜合利用[J]. 化工中間體, 2008, 1(3):25-29.
(責任編輯 狄艷紅)
Research Progress on Bio-ethylene
SUN Meng-ting1,2FAN Xiao-lei1GUO Rong-bo1QIU Yan-ling1ZHAO Xiao-xian1
(1. Shandong Industrial Engineering Laboratory of Biogas Production & Utilization,Qingdao Institute of Bioenergy and Bioprocess Technology,Chinese Academy of Sciences,Qingdao 266101;2. University of Chinese Academy of Sciences,Beijing 100049)
Ethylene is the chemical raw material of largest demand in the world, and is widely utilized for producing plastics, textiles and other chemical products. Currently, the most common method of producing ethylene is steam cracking of fossil oil;however, there exist drawbacks of high cost and non-friendly to environment, which bring the opportunity for the development of bio-ethylene. Bio-ethylene is produced from renewable resources of CO2and biomass , which contributes to saving resources and protecting environment. In this review,we introduce two pathways of bio-ethylene production—direct and indirect synthesis. Here we focus on the direct synthesis pathway of microorganisms via an ethylene forming enzyme(EFE), describing the structure, sequence and catalytic mechanism of EFE, illustrating several successful cases of engineered heterologous hosts synthetizing ethylene, presenting the strategies for improving yields, and prospecting the development of producing ethylene by microorganisms.
bio-ethylene;direct synthesis;microorganism;ethylene forming enzyme;heterologous expression
10.13560/j.cnki.biotech.bull.1985.2016.02.004
2015-04-20
國家“863”計劃項目(2012AA052103),國家自然科學基金面上項目(41276143),國家科技支撐項目(2013BAD22B00)
孫夢婷,女,碩士,研究方向:生物化工;E-mail:374695153@qq.com
范曉蕾,女,博士,副研究員,研究方向:生物化工;E-mail:fanxl@qibebt.ac.cn

