魯西黑頭羊mtDNA D—loop區(qū)遺傳多樣性與遺傳分化研究
王金文 崔緒奎 陶衛(wèi)東 儲明星 張果平 王德芹 狄冉
摘要: 魯西黑頭羊是一個優(yōu)良的綿羊培育品種,具備高產(chǎn)肉力和高繁殖力特征。為了評估這個品種線粒體DNA的遺傳多樣性,同時獲得該新品種界定的分子證據(jù),本研究對魯西黑頭羊、杜泊羊和小尾寒羊mtDNA的高變異區(qū)進(jìn)行了測序,分析了它們的遺傳多樣性和遺傳分化,并計算了魯西黑頭羊與其它兩個品種間的遺傳距離。結(jié)果表明:三個品種的遺傳多樣性較為豐富,遺傳分化程度較高(Fst為 0.3240~0.5909);魯西黑頭羊與杜泊羊之間的遺傳距離為0.1187,達(dá)到了種間的遺傳距離,與小尾寒羊之間的遺傳距離為0.0654,達(dá)到了亞種間的水平。以上結(jié)果為魯西黑頭羊的品種界定提供了數(shù)據(jù)支持。
關(guān)鍵詞:魯西黑頭羊;mtDNA;D-loop區(qū);遺傳多樣性;遺傳距離
中圖分類號:S826.212文獻(xiàn)標(biāo)識號:A文章編號:1001-4942(2016)09-0115-04
AbstractLuxi black head sheep is an excellent cultivated breed with high yield of meat and high prolificacy. To assess the genetic diversity of Luxi black head sheep on mtDNA and obtain the molecular evidence on breed identification, the hypervariable regions of mtDNA in Luxi black head sheep, Dorper and small tail Han sheep were sequenced, and their genetic diversity, genetic differentiation and genetic distance were analyzed. The results indicated that the genetic diversity among three breeds was rich and the degree of genetic differentiation was high (Fst as 0.3240~0.5909). The genetic distance between Luxi black head sheep with Dorper was 0.1187, which reached the value between species; and that with small tail Han sheep was 0.0654, which reached the value between subspecies. The results provided data support for the breed identification of Luxi black head sheep.
KeywordsLuxi black head sheep; mtDNA; D-loop region; Genetic diversity; Genetic distance
魯西黑頭羊是利用杜泊羊作父本、小尾寒羊作母本,經(jīng)過級進(jìn)雜交、橫交固定、繼代選育和擴(kuò)繁而培育形成的肉用綿羊新品種。該品種頭頸部被毛黑色,體軀被毛白色,體格大、結(jié)構(gòu)勻稱、背腰平直、后軀豐滿,肉用體型明顯;6月齡公羔體重達(dá)到(45.75±3.70) kg,6月齡母羔體重達(dá)到(43.76±6.03) kg;平均產(chǎn)羔率220%,屠宰率55.1%,具有生長速度快、繁殖率高、耐粗飼、羊肉品質(zhì)好等優(yōu)良特性,頗受市場歡迎。
動物線粒體DNA(Mitochondrial DNA, mtDNA)是動物體內(nèi)唯一的核外遺傳物質(zhì),具有母系遺傳的特征,結(jié)構(gòu)簡單穩(wěn)定、進(jìn)化速度較快,而且能直觀保存群體突變的發(fā)生[1,2]。Fan等[3]對不同尾型的綿羊mtDNA基因組進(jìn)行了測序,分析了不同尾型品種間的系統(tǒng)進(jìn)化關(guān)系。Zhao等[4]利用mtDNA測序方法分析了中國綿羊品種的遺傳多樣性和品種間遺傳變異情況。本研究擬通過對魯西黑頭羊、杜泊羊和小尾寒羊mtDNA D-loop區(qū)進(jìn)行測序,分析它們的遺傳多樣性和遺傳變異,計算魯西黑頭羊與其它兩個品種間的遺傳距離,以期為魯西黑頭羊的品種界定提供數(shù)據(jù)支持。
1材料與方法
1.1樣品采集
選擇健康的無親緣關(guān)系的羊群,隨機(jī)抽測杜泊羊8只、小尾寒羊10只(后期有1只樣品不能用)、魯西黑頭羊10只,均為成年羊,公母各半。利用EDTA抗凝管頸靜脈采血5 mL,-20℃保存?zhèn)溆谩?/p>
1.2試驗方法
1.2.1基因組DNA提取采用生工生物工程(上海)股份有限公司的EZUP柱式動物基因組DNA抽提試劑盒提取血液基因組DNA。-20℃保存。
1.2.2PCR擴(kuò)增本試驗所使用的D-loop區(qū)引物序列,F(xiàn): 5′-AGCCCCACTATCAACACC-3′;R: 5′-AAATAGTTACCCCCACAGTTAG-3′。
PCR擴(kuò)增反應(yīng)體系為25 μL,反應(yīng)體系為:10×PCR buffer 2.5 μL、dNTPs(2.5 mmol/L) 2 μL、正向引物和反向引物(10 μmol/L)各為1 μL、DNA(10 ng/μL) 模板為1 μL、Taq酶(2.5 U/μL)0.5 μL、ddH2O 補(bǔ)足總體積到25 μL。PCR擴(kuò)增條件為:94℃預(yù)變性 5 min;94℃變性30 s,56℃退火 30 s,72℃ 延伸 30 s,30個循環(huán);72℃延伸5 min,4℃保存。
1.2.3序列比對和分析PCR產(chǎn)物經(jīng)1%瓊脂糖凝膠電泳檢測后,送生工生物工程(上海)股份有限公司純化和測序,測序結(jié)果用ChromasPro(version 1.7.4)進(jìn)行拼接,拼接好的序列在NCBI中進(jìn)行Blast比對,確保得到的序列是目標(biāo)序列;然后用Clustal X[5]進(jìn)行多重比對,并對比對結(jié)果進(jìn)行篩查與糾正;采用DnaSP v5(version 5.10.01)軟件[6]計算各堿基組成、單倍型多樣度(Hd)、核苷酸多樣性(Pi)、平均核苷酸差異數(shù)(k)、種群間核苷酸平均差異數(shù)(Kxy)、核苷酸歧義度(Dxy)、遺傳分化系數(shù)(Gst)和固定系數(shù)(Fst)等分子遺傳學(xué)參數(shù),并進(jìn)行中性檢驗;采用MEGA 4.0軟件[7],按照Kimura雙參數(shù)法(two-parameter method)[8],計算種間和種內(nèi)的平均遺傳距離,采用鄰接法(Neighbor-Joining)構(gòu)建系統(tǒng)發(fā)育樹,選用K2P替代模型,進(jìn)行1 000次自舉檢驗。
2結(jié)果與分析
2.1PCR擴(kuò)增結(jié)果
以杜泊羊、小尾寒羊、魯西黑頭羊基因組DNA為模板對其D-loop區(qū)進(jìn)行擴(kuò)增,經(jīng)電泳檢測,條帶與預(yù)期目標(biāo)片段大小一致,隨后進(jìn)行純化和測序。
2.2D-loop區(qū)序列堿基組成
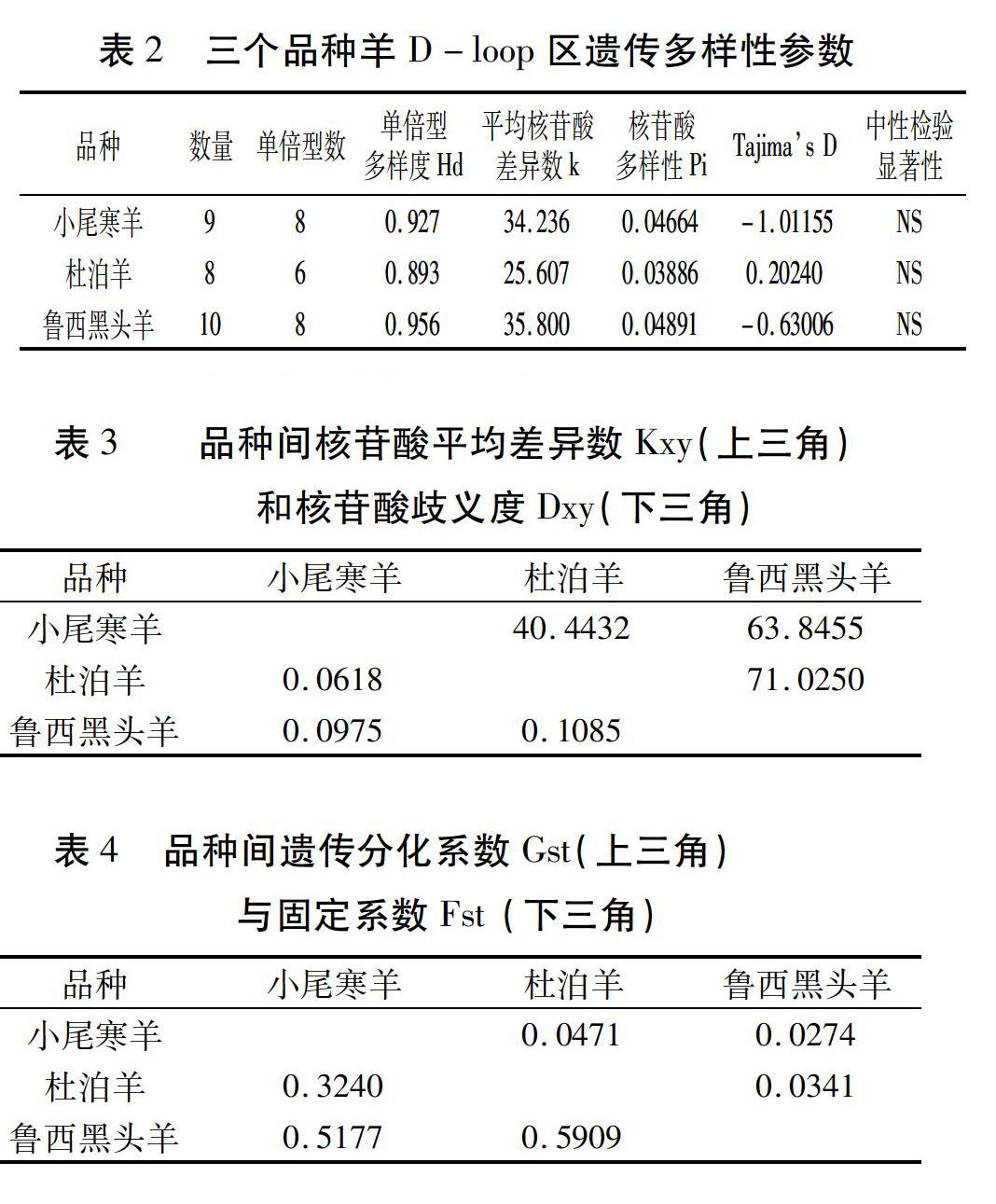
測序結(jié)果經(jīng)ChromasPro拼接后,除去引物部分,獲得的D-loop區(qū)序列為735 bp,經(jīng)在線Blast比較,證實所得序列為D-loop區(qū)片段。三個品種平均堿基含量見表1,G+C含量在35.1%~37.4%之間,A+T的含量明顯高于G+C的含量,C含量最低,明顯低于其它3種堿基的含量。
系統(tǒng)發(fā)生樹(NJ 法)
3討論與結(jié)論
線粒體基因組是母系遺傳,基因進(jìn)化速率較快,種群之間的遺傳差異易被檢出,線粒體基因組中的控制區(qū)D-loop區(qū)不編碼基因,受到的選擇壓力較編碼基因小,其進(jìn)化速率是其他區(qū)段的5倍[9],因此常用于研究種及以下水平的遺傳分化,以獲得較多的遺傳變異信息。四種堿基在線粒體基因組中的分布呈不均一性,本研究所測定的三個品種的D-loop序列,其T、C、A、G的平均含量分別為36.1%、13.1%、27.6%、23.1%,A+T含量(63.7%)高于G+C(36.3%),符合脊椎動物mtDNA堿基組成的特點。
物種的遺傳變異反映了該物種適應(yīng)環(huán)境變化能力的強(qiáng)弱,遺傳變異程度越高,說明群體的多樣性越豐富,對環(huán)境變化的適應(yīng)能力也就越強(qiáng);而低的遺傳多樣性對物種的生存有著不利影響,增加了物種滅絕的風(fēng)險。根據(jù)Grant等[10]對線粒體DNA序列遺傳變異的分類,三個品種都具有高的單倍型多樣度(Hd>0.500)和高的核苷酸多樣性指數(shù)(Pi>0.00500),說明這些群體遺傳變異較為豐富,可產(chǎn)生許多新的突變,有較強(qiáng)的適應(yīng)能力、生長能力和進(jìn)化潛力。李祥龍等[11,12]和金紅等[13]利用RFLP方法分析表明中國蒙古羊、小尾寒羊等家養(yǎng)綿羊品種線粒體DNA多態(tài)性比較貧乏;而本研究獲得的三個品種的mtDNA D-loop區(qū)核苷酸多樣度(Pi) 分別為0.04664、0.03886和0.04891,這些值均高于被認(rèn)為線粒體DNA多態(tài)程度較低的范圍(0.00150~0.00470)[14]。本研究所得核苷酸多樣度結(jié)果與利用測序法研究綿羊mtDNA D-loop區(qū)的研究結(jié)果基本一致[15-18]。另外,三個品種擁有較高的單倍型多樣度(平均值為0.9253),與成述儒等[19]的研究結(jié)果一致。就小尾寒羊這個品種而言,本研究計算所得單倍型多樣度和核苷酸多樣性指數(shù)與黃慶華等[20]報告的結(jié)果相近。中性檢驗顯示這三個品種都未經(jīng)歷過群體擴(kuò)張或瓶頸。
在群體遺傳學(xué)研究中,遺傳分化指數(shù)值常用來表示群體間的遺傳分化程度,數(shù)值越大表明群體間分化程度越顯著,F(xiàn)st值在0~0.05之間說明群體間的遺傳分化很小,在0.05~0.15之間說明群體間存在中等程度的遺傳分化,在0.15~0.25之間說明群體間的遺傳分化較大,F(xiàn)st值大于0.25說明群體間的遺傳分化很大。本研究中三個品種的Fst值在0.3240~0.5909之間,遠(yuǎn)大于0.25,說明群體間遺傳分化程度較高。
根據(jù)Nei估算,同一物種內(nèi)的種群間遺傳距離變動在0~0.05之間;亞種間的遺傳距離約為0.02~0.20;種間遺傳距離的變幅為0.10~2.00之間。從三個品種的遺傳距離來看,魯西黑頭羊與杜泊羊之間的遺傳距離為0.1187,達(dá)到了種間的遺傳距離,與小尾寒羊之間的遺傳距離為0.0654,達(dá)到了亞種間的水平,這可能是由于育種過程中采用小尾寒羊作為母本,而線粒體基因又是母系遺傳造成的。魯西黑頭羊以杜泊羊與小尾寒羊通過雜交選育和擴(kuò)繁育成,經(jīng)過10多年的育種,繼承了杜泊羊的高產(chǎn)肉性能和小尾寒羊的高繁殖性能,遺傳上已經(jīng)達(dá)到了品種的要求。
參考文獻(xiàn):
[1]Xu W, Jameson D, Tang B, et al. The relationship between the rate of molecular evolution and the rate of genome rearrangement in animal mitochondrial genomes[J]. J. Mol. Evol., 2006, 63(3): 375-392.
[2]張亞平, 施立明. 動物線粒體DNA多態(tài)性的研究概況[J]. 動物學(xué)研究, 1992, 13(3): 289-298.
[3]Fan H Y, Zhao F P, Zhu C Y, et al. Complete mitochondrial genome sequences of Chinese indigenous sheep with different tail types and an analysis of phylogenetic evolution in domestic sheep[J]. Asian Australas. J. Anim. Sci., 2016, 29(5): 631-639.
[4]Zhao E, Yu Q, Zhang N, et al. Mitochondrial DNA diversity and the origin of Chinese indigenous sheep[J]. Trop. Anim. Health Prod., 2013, 45(8): 1715-1722.
[5]Thompson J D, Gibson T J, Plewniak F, et al. The CLUSTAL_X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Res., 1997, 25(24): 4876-4882.
[6]Librado P, Rozas J. DnaSP v5: a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics, 2009, 25(11): 1451-1452.
[7]Tamura K, Dudley J, Nei M, et al. MEGA4: molecular evolutionary genetics analysis (MEGA) software version 4.0[J]. Mol. Biol. Evol., 2007, 24(8): 1596-1599.
[8]Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J. Mol. Evol., 1980, 16(2): 111-120.
[9]Rosel P E, Haygood M G, Perrin W F. Phylogenetic relationships among the true porpoises (Cetacea:Phocoenidae)[J]. Mol. Phylogenet. Evol., 1995, 4(4): 463-474.
[10]Grant W, Bowen B. Shallow population histories in deepevolutionary lineages of marine fishes: insights from sardines and anchovies and lessons for conservation [J]. J. Hered., 1998, 89 (5): 415-426.
[11]李祥龍, 田慶義, 劉錚鑄, 等. 幾個綿羊品種線粒體DNA限制性片斷長度多態(tài)性比較研究[J]. 畜牧獸醫(yī)學(xué)報, 2001, 32(4): 295-298.
[12]李祥龍, 張增利, 鞏元芳, 等. 我國主要地方綿羊品種mtDNA D-loop區(qū)PCR-RFLP研究[J]. 遺傳, 2006, 28(2): 165-170.
[13]金紅, 陳志成, 梁婧嫻, 等. 藏系綿羊紅原、賈洛、歐拉類群mtDNA D-loop區(qū)多態(tài)性分析[J]. 西南民族大學(xué)學(xué)報(自然科學(xué)版), 2012, 28(2): 235-241.
[14]Hiendleder S, Mainz K, Plante Y, et al. Analysis of mitochondrial DNA indicates that domestic sheep are derived from two different ancestral maternal sources: no evidence for contributions from urial and argali sheep[J]. Journal of Heredity, 1998, 89(2): 113-120.
[15]韓旭, 劉丑生, 劉剛, 等. 中國地方綿羊線粒體DNA D-loop區(qū)遺傳多樣性研究[J]. 中國畜牧雜志, 2015, 51(7): 10-15.

