基于線粒體16S rRNA基因的14種造礁石珊瑚系統(tǒng)發(fā)育關(guān)系分析
廖寶林,肖寶華,2,楊小東,謝子強
(1.廣東海洋大學(xué)深圳研究院,廣東 深圳 518108,2.廣東海洋大學(xué),廣東 湛江 524025,3.深圳市碧海藍(lán)天海洋科技有限公司,廣東 深圳 518108)
基于線粒體16S rRNA基因的14種造礁石珊瑚系統(tǒng)發(fā)育關(guān)系分析
廖寶林1,肖寶華1,2,楊小東1,謝子強3
(1.廣東海洋大學(xué)深圳研究院,廣東 深圳 518108,2.廣東海洋大學(xué),廣東 湛江 524025,3.深圳市碧海藍(lán)天海洋科技有限公司,廣東深圳 518108)
通過16S rRNA基因特異擴增測序,對惠州大亞灣大辣甲島和小辣甲島附近海域14種造礁石珊瑚16S rRNA基因片段序列進(jìn)行了比較分析。結(jié)果表明,該片段序列的平均 A+T含量為 63.5%,所有物種的序列堿基AT含量大于GC含量,序列都處于高度飽和的狀態(tài)。14種造礁石珊瑚的平均遺傳距離為0.179。采用NJ、ML和ME法構(gòu)建系統(tǒng)發(fā)育樹,結(jié)果顯示蜂巢珊瑚科單獨作為分支處于發(fā)育樹的基部,而第二支分支包括菌珊瑚科、鹿角珊瑚科、濱珊瑚科和枇杷珊瑚科。裸肋珊瑚科的腐蝕刺柄珊瑚聚到了蜂巢珊瑚科中,與傳統(tǒng)形態(tài)學(xué)分類存在差異,提示造礁石珊瑚表型的變異性可能對傳統(tǒng)分類存在影響。
造礁石珊瑚; 16S rRNA;分子系統(tǒng)學(xué)
造礁石珊瑚屬于刺胞動物門(Cnidaria)珊瑚綱(Anthozoa)六射珊瑚亞目(Hexocorallia)石珊瑚目(Scleractinia),主要分布于地球赤道南北緯30°以內(nèi)的海域[1]。由造礁石珊瑚組成的珊瑚礁生態(tài)系統(tǒng)是世界上第二大生態(tài)系統(tǒng)。過去造礁石珊瑚的分類主要通過其骨骼結(jié)構(gòu)作為標(biāo)準(zhǔn)。近30年來,通過分子生物學(xué)手段對造礁石珊瑚親緣關(guān)系鑒定已進(jìn)行了廣泛的研究,隨著研究的深入,很多結(jié)果都與傳統(tǒng)形態(tài)學(xué)鑒定結(jié)果存在一定的差異[2-3],顯示了全面研究造礁石珊瑚分類的必要性。
大辣甲島和小辣甲島位于大亞灣海域西側(cè),島嶼沿岸水深5m以內(nèi)的基巖石上均有珊瑚群落分布[4]。早在20世紀(jì)80年代,就有學(xué)者對大、小辣甲島嶼附近珊瑚分布進(jìn)行初步的調(diào)查[5],調(diào)查結(jié)果顯示大、小辣甲島嶼附近海域珊瑚礁覆蓋率達(dá)到78.1%和72.7%,霜鹿角珊瑚為這兩個地區(qū)的優(yōu)勢品種。2000年以后,陳天然等[6-7]對大亞灣石珊瑚群落的分布及動態(tài)變化進(jìn)行調(diào)查,調(diào)查結(jié)果顯示大、小辣甲的活珊瑚覆蓋率有明顯下降的趨勢,優(yōu)勢品種也從原有的分支狀的鹿角珊瑚轉(zhuǎn)變成團(tuán)塊狀的角蜂巢珊瑚。近幾年來,廣東珊瑚礁普查都在大、小辣甲島進(jìn)行調(diào)查,數(shù)據(jù)顯示大辣甲的覆蓋率僅在25%左右,而小辣甲的覆蓋率更低,僅為15%左右,大亞灣造礁石珊瑚退化情況十分嚴(yán)重。
16S rRNA基因具有線粒體基因的母系遺傳、變化速率快、高度保守性的共同特征,同時該基因還具有種間差異明顯而種群內(nèi)個體間變異小的特點,被認(rèn)為是科以下水平物種間親緣關(guān)系和系統(tǒng)演化研究的有效標(biāo)記[8-9],在水螅蟲綱(Hydrozoa)、對蝦屬(Penaeus)、珠母貝屬(Pinctada)等無脊椎動物中得到了廣泛應(yīng)用[10-12]。1996年Romano等[13]在世界上首次使用16S rRNA基因作為分子標(biāo)記對34種造礁石珊瑚的系統(tǒng)進(jìn)化關(guān)系進(jìn)行研究,此后國內(nèi)外都有應(yīng)用16S rRNA基因?qū)υ旖甘汉飨到y(tǒng)進(jìn)化和遺傳多樣性進(jìn)行研究的報道[14-17]。這些分子研究結(jié)果與傳統(tǒng)形態(tài)學(xué)系統(tǒng)進(jìn)化結(jié)果出現(xiàn)差異,傳統(tǒng)形態(tài)學(xué)結(jié)果存在受珊瑚骨骼生長變異性限制的可能性。
本研究通過聚合酶鏈?zhǔn)椒磻?yīng)技術(shù)(PCR),利用線粒體16S rRNA基因的部分序列片段作為分子標(biāo)記,對大、小辣甲海域的14種造礁石珊瑚的系統(tǒng)進(jìn)化關(guān)系進(jìn)行研究,為該地區(qū)造礁石珊瑚資源保護(hù)提供理論基礎(chǔ)。
1 材料與方法
1.1樣品采集和形態(tài)學(xué)鑒定
本研究的造礁石珊瑚樣品采自廣東省惠州市大亞灣地區(qū)的大辣甲島及小辣甲島附近海域,形態(tài)學(xué)鑒定依照文獻(xiàn) [1]和[18],將采集的樣品可分為6科10屬14種,樣品形態(tài)學(xué)鑒定結(jié)果見表1。樣品截取一小段保存于無水乙醇中以備后續(xù)DNA提取實驗使用。
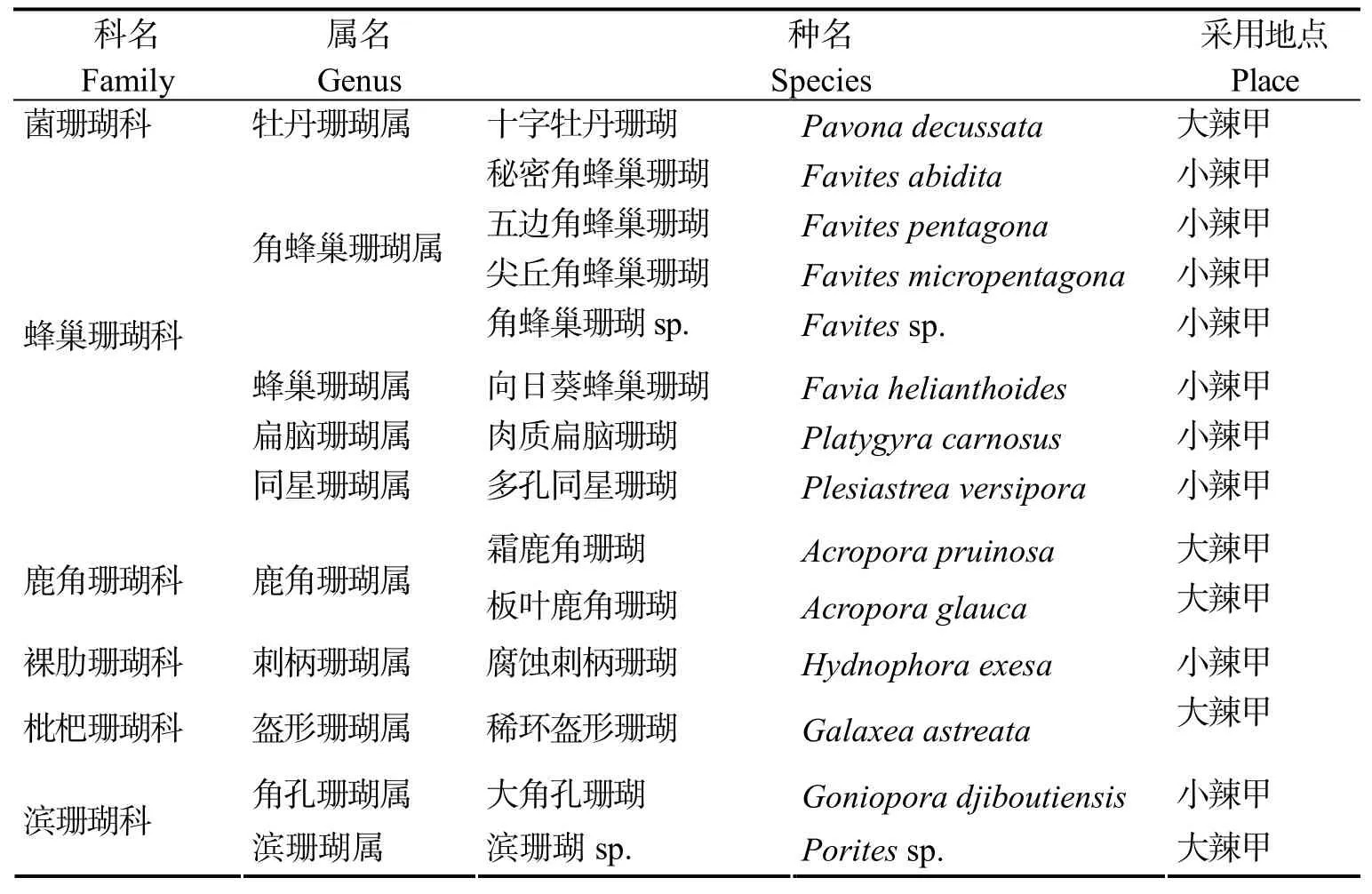
表1 珊瑚樣品Tab.1 Coral samples
1.2基因組總DNA的提取
使用北京天根生物有限公司生產(chǎn)的海洋動物組織基因組提取試劑盒行提取珊瑚樣品的DNA,所提取的DNA置于4℃保存?zhèn)溆谩?/p>
1.3引物設(shè)計合成
16S rRNA基因的PCR引物均參考Forsman等[14]的研究報道。引物由上海生工生物技術(shù)服務(wù)有限公司合成,引物信息見表2。
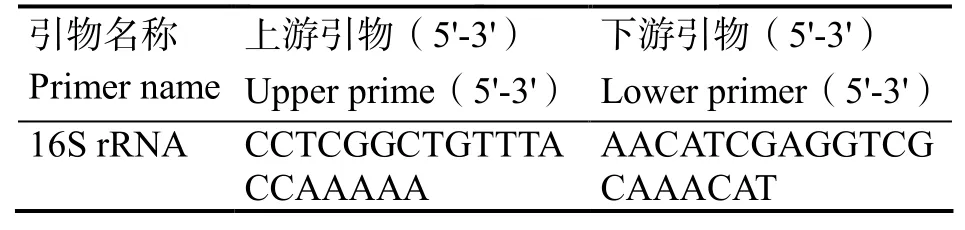
表2 16S rRNA基因的PCR引物Tab.2 List of PCR primers for 16S rRNA
1.3PCR擴增和DNA測序
PCR反應(yīng)體系反應(yīng)總體積25 μL:10 × Buffer 2.5 μL,dNTP 2 μL,上下游引物各1 μL,Taq DNA聚合酶1 U及DNA模板2 μL;其余用去離子水補至25 μL。PCR反應(yīng)條件:95℃預(yù)變性5min,95℃變性45 s,50℃退火45 s,72℃延伸1min 30 s,共35個循環(huán),最后72℃延伸10min。擴增產(chǎn)物在10 mg/mL的瓊脂糖凝膠中檢測,凝膠成像儀拍照記錄。選取擴增條帶單一、濃度較高的PCR產(chǎn)物送至上海生工生物技術(shù)公司進(jìn)行測序,獲取各基因片段的序列信息。
1.4序列分析及系統(tǒng)進(jìn)化樹的構(gòu)建
測序所得序列用DNASTAR中的Editseq軟件進(jìn)行編輯、剪切、校正,再將校正好的序列與NCBI的數(shù)據(jù)庫進(jìn)行比對,確認(rèn)樣品的分子結(jié)果。采用MEGA6.0軟件進(jìn)行基因序列分析,統(tǒng)計出16S rRNA基因組序列長度、堿基組成、簡約信息位點等相關(guān)信息。同時根據(jù)簡約信息位點,計算出種間遺傳距離。本研究3種16S rRNA的系統(tǒng)進(jìn)化樹使用MEGA6.0軟件基于Kimu-ra雙參數(shù)模型( Kimura 2-parameter,K2P),只計算顛換(Transversional substitutions only),采 用 鄰位 連 接 法(Neighbor-Joinning,NJ)和 最 小 進(jìn) 化 法(Minimum-Evolution,ME)構(gòu)建出來。兩種系統(tǒng)進(jìn)化樹中節(jié)點的自舉置信度水平由自引導(dǎo)值( Bootstrap value) 估計,重復(fù)次數(shù)為1 000。
2 結(jié)果與分析
2.1造礁石珊瑚16S rRNA基因堿基組成分析
本研究的14個造礁石珊瑚品種的16S rRNA基因部分序列長度在474~630 bp之間。從14個造礁石珊瑚品種的16S rRNA基因部分序列的堿基組成比例來看,全部序列堿基AT含量大于GC含量,與目前已知的無脊椎動物mtDNA序列中AT含量大于GC含量的情況相同。16S rRNA片段堿基中的A、T、C、G含量相差較大,序列基因都處于高度飽和狀態(tài)。堿基組成信息見表3。
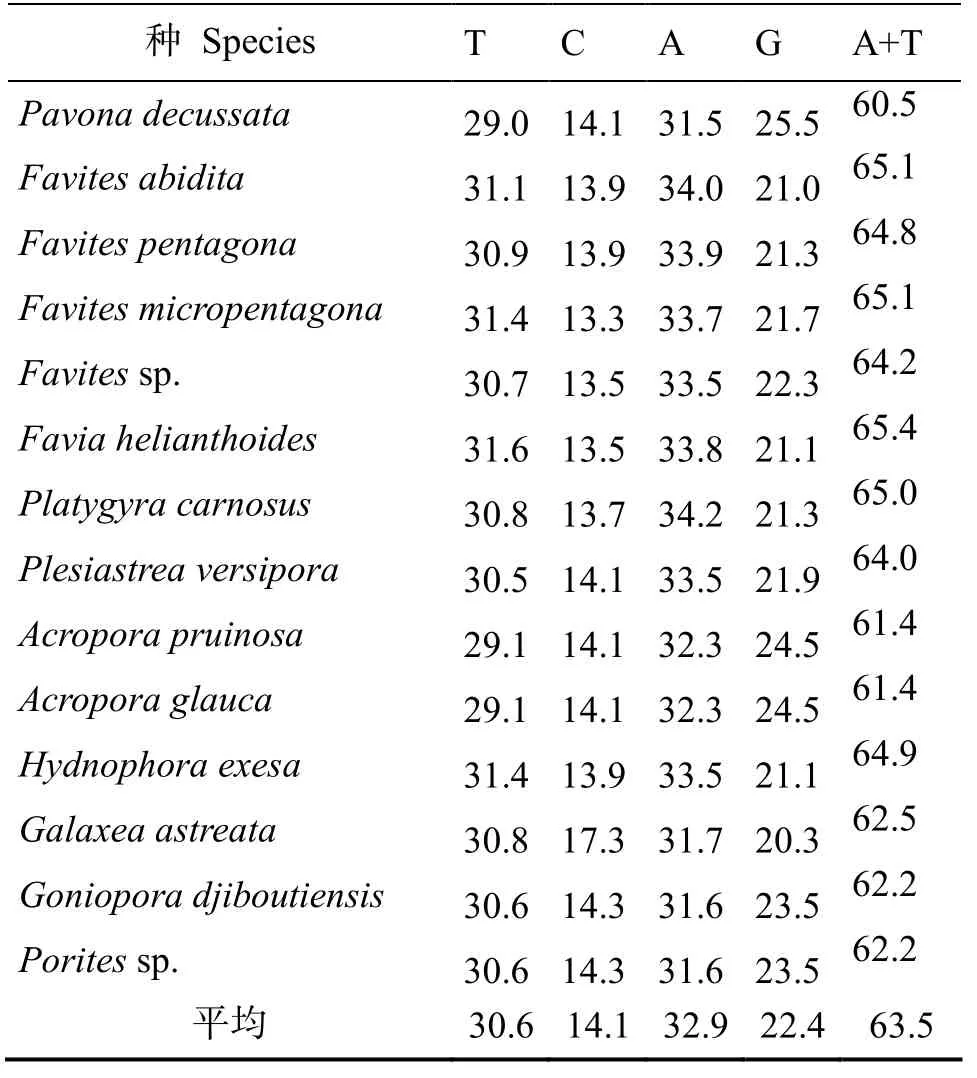
表3 14種造礁石珊瑚16S rRNA基因片段的堿基組成比例Tab.3 The nucleotide Percentage composition of the 16S rRNA fragments
2.2造礁石珊瑚16S rRNA基因序列變化情況
基于Kimura 2-parameter模式的最大似然法計算14種造礁石珊瑚16S rRNA基因部分序列的轉(zhuǎn)換顛換比值R為1.42,序列堿基突變未達(dá)到飽和。使用DAMEB軟件對15種石珊瑚的16S rRNA序列進(jìn)行堿基替換飽和分析(圖1),當(dāng)種間遺傳距離超過0.42時,堿基轉(zhuǎn)換(Ts)趨于飽和,堿基顛換(Tv)開始超過轉(zhuǎn)換(Ts)。
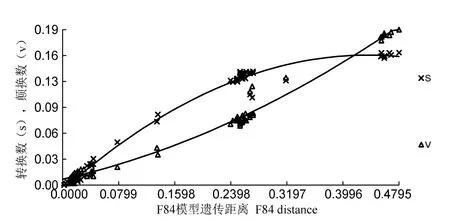
圖1 14種造礁石珊瑚16S rRNA基因堿基替換飽和分析Fig 1 Substitution saturation in the 16S rRNA gene of the 14 species of Scleractinian
2.314種造礁石珊瑚種間遺傳距離
通過MEGA6.0計算14種造礁石珊瑚的種間遺傳距離,結(jié)果顯示14種造礁石珊瑚的平均遺傳距離為0.179。其中稀環(huán)盔形珊瑚(Galaxea astreata)和向日葵蜂巢珊瑚(Favia helianthoides)的種間遺傳距離最大,為0.447,而鹿角珊瑚科的霜鹿角珊瑚(Acropora pruinosa)和板葉鹿角珊瑚(Acropora glauca)、濱珊瑚科的大角孔珊瑚(Goniopora djiboutiensis)和濱珊瑚sp.(Porites sp.)的遺傳距離最小,為0.000。種間遺傳距離信息見表4。
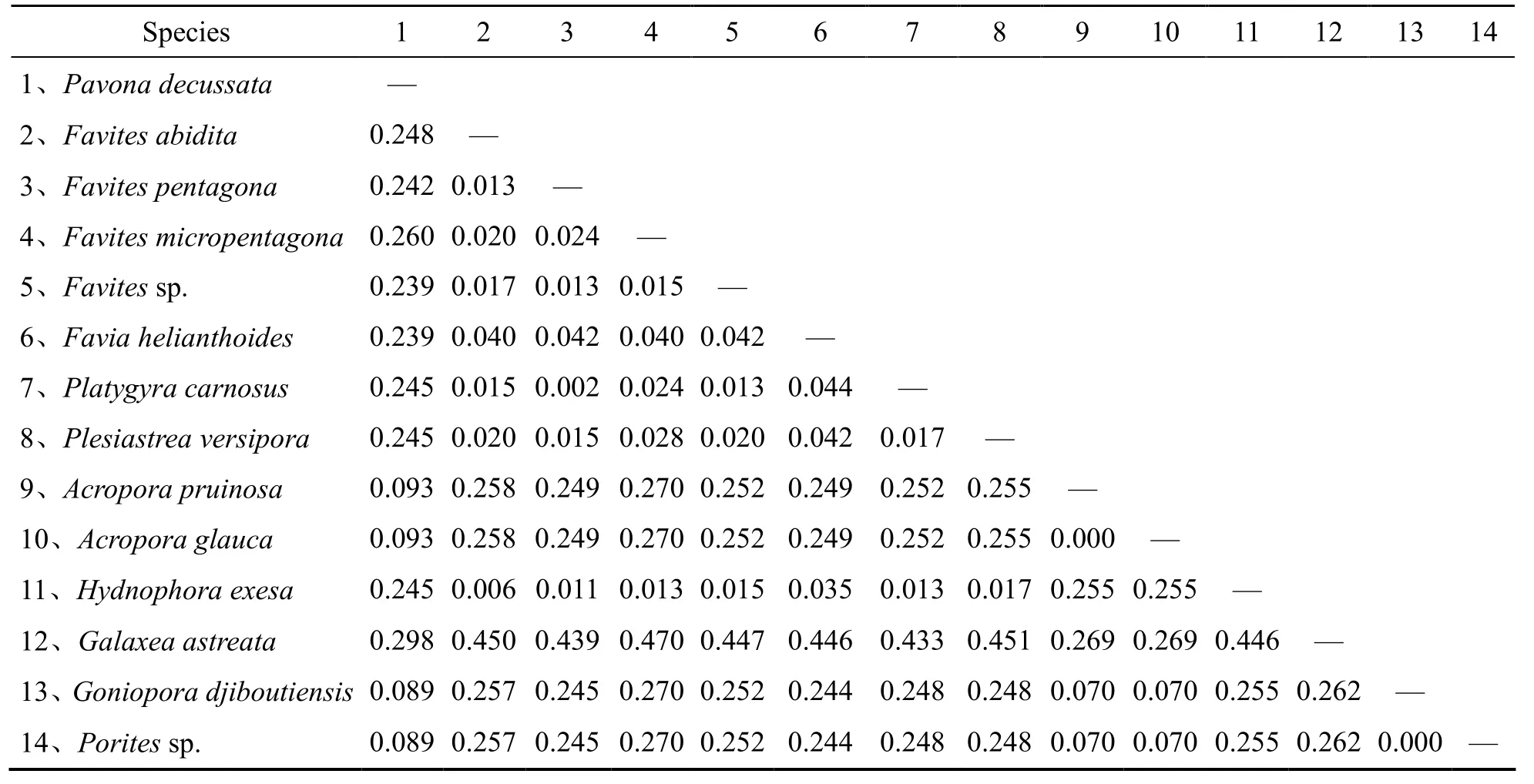
表4 基于16S rRNA的14種造礁石珊瑚種間遺傳距離Tab.4 The Pairwise Tamura-Nei genetic distances of nucleotide sequences based on 16S rRNA among the species of Scleractinian
2.4基于16S rRNA對14種造礁石珊瑚的的系統(tǒng)進(jìn)化分析
以多管水母科(Aequoreidae)的錐形多管水母(Aequorea conica)作為外源種(GenBank序列號:JQ715990.1),采用鄰位連接法(Neighbor-Joinning,NJ)、最大似然法(Minimum-Likelihood,ML)和最小進(jìn)化法(Minimum-Evolution,ME)構(gòu)建14種造礁石珊瑚的16S rRNA分子系統(tǒng)樹(圖2~4),結(jié)果顯示三種方法構(gòu)建的系統(tǒng)進(jìn)化樹拓?fù)浣Y(jié)構(gòu)完全一致,蜂巢珊瑚科(Faviidae)單獨作為分支處于發(fā)育樹的基部,而第二支分支包括菌珊瑚科(Agaricidae)、鹿角珊瑚科(Acroporidae)、濱珊瑚科(Portidae)和枇杷珊瑚科(Oculinidae)。各科的珊瑚品種各聚為一枝,但出現(xiàn)了裸肋珊瑚科(Merulinidae)的腐蝕刺柄珊瑚(H.exesa)與秘密角蜂巢珊瑚(F.abidita)聚為一枝的現(xiàn)象。
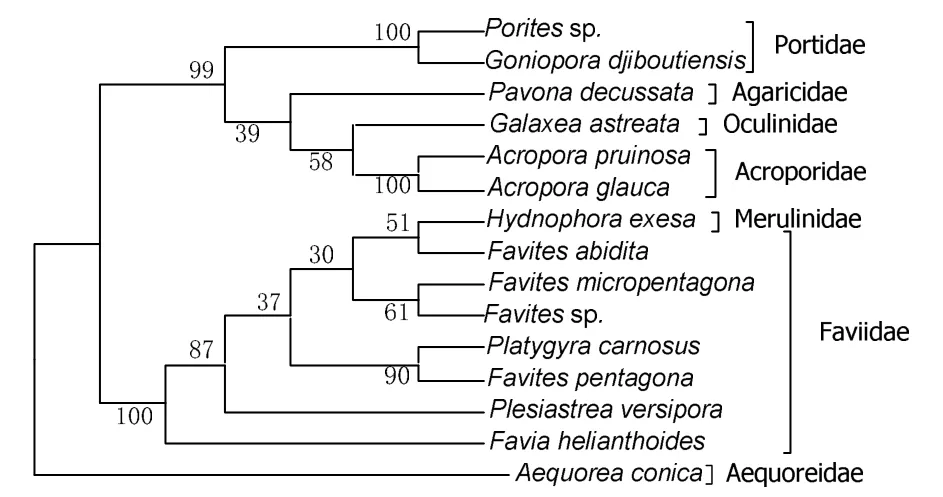
圖2 利用16S rRNA序列數(shù)據(jù)構(gòu)建的分子系統(tǒng)進(jìn)化NJ樹Fig.2 The Neighbor-Joining tree constructed with 16S RNA gene sequence
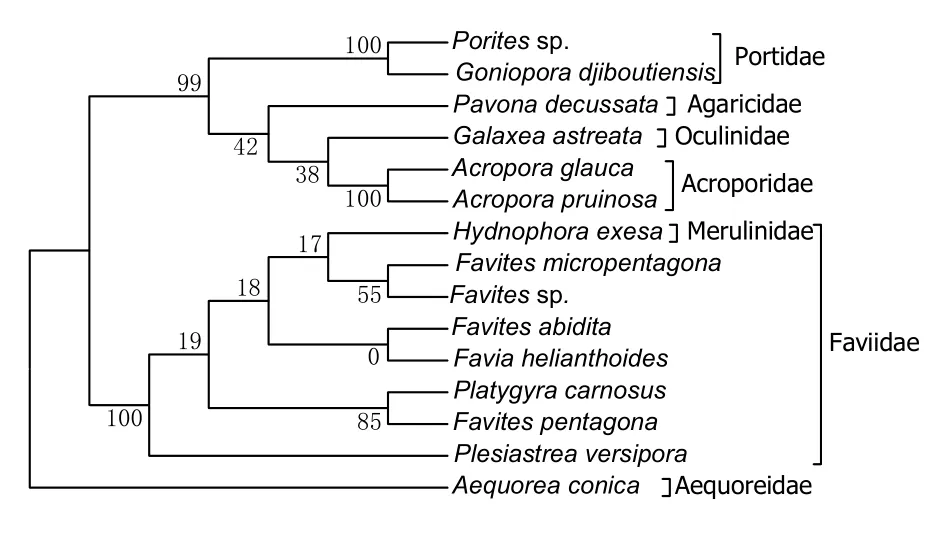
圖3 利用16S rRNA序列數(shù)據(jù)構(gòu)建的分子系統(tǒng)進(jìn)化ML樹Fig.3 The Minimum-Likelihood tree constructed with 16S RNA gene sequence
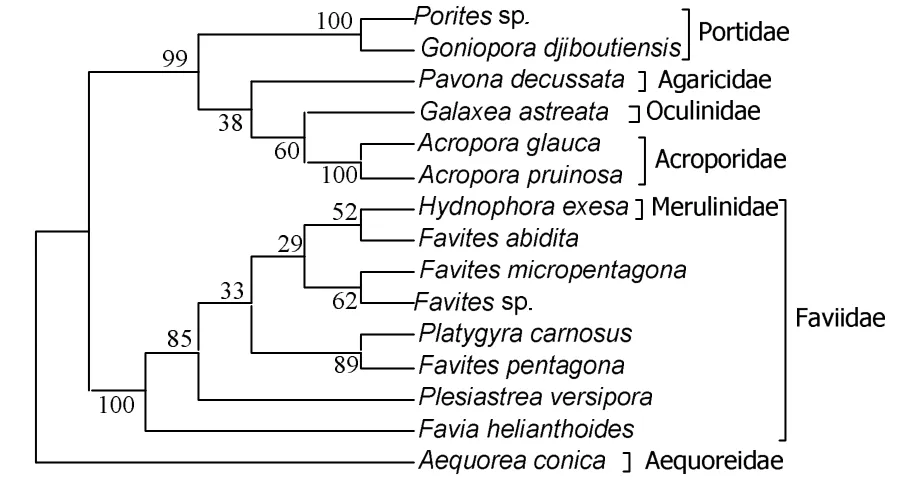
圖4 利用16S rRNA序列數(shù)據(jù)構(gòu)建的分子系統(tǒng)進(jìn)化ME樹Fig.4 The Minimum-Evolution tree constructed with 16S RNA gene sequence
3 討論
3.114種造礁石珊瑚16S rRNA基因序列特點
線粒體16S核糖體RNA基因相較其他造礁石珊瑚的線粒體基因進(jìn)化速率較慢,且較為保守,常用于種以上分子分類的標(biāo)記[19]。本研究的 14個造礁石珊瑚品種的 16S rRNA基因部分序列長度在474~630 bp之間。Romano等[15]對20科61屬68種造礁石珊瑚的16S rRNA片段序列進(jìn)行堿基組成比較,片段長度在407~566 bp之間。李曉娜[20]使用與本研究相同的引物對廣東徐聞 18種造礁石珊瑚的 16S rRNA片段序列進(jìn)行比較,序列長度在503~688 bp之間。而Forsman等[14]同樣使用與本研究相同的引物,對夏威夷的 7種薔薇珊瑚屬(Montipora)珊瑚16S rRNA片段序列進(jìn)行測序,片段序列長度均為790 bp。16S rRNA基因在造礁石珊瑚屬以上的分類階元中種間片段長度具有較大的差異,而屬內(nèi)差異片段長度差異不大。從序列的堿基組成、序列變化和遺傳距離上看,16S rRNA基因十分適宜作為科、屬之間的分子分類標(biāo)記。Romano等[13]發(fā)現(xiàn)造礁石珊瑚16s rRNA基因片段的多樣性較低于其他后口動物,16S rRNA 基因盡管可以用于屬之間的分類,但是由于多樣性的不高,不適合作為屬內(nèi)親緣關(guān)系較近的種間分類標(biāo)記。本研究中鹿角珊瑚屬和濱珊瑚科的4種珊瑚的確存在序列完全相似的情況,但也有可能是使用的16S rRNA片段長度較短和樣品采集數(shù)量較少造成。本研究結(jié)果支持16S rRNA作為科一級分類階元的分子標(biāo)記,而其是否適宜作為屬以下更具體的分類階元的分子標(biāo)記,仍需要更多的造礁石珊瑚種類的16S rRNA數(shù)據(jù)進(jìn)行驗證。
3.2造礁石珊瑚系統(tǒng)分析
早在20世紀(jì)90年代,Romano等[13]就首次應(yīng)用16S rRNA對34種造礁石珊瑚的系統(tǒng)進(jìn)化關(guān)系進(jìn)行研究,并推斷全球范圍內(nèi)現(xiàn)存的造礁石珊瑚在200萬年前分化為兩大類群,并將這兩大類群命名為堅實型類群(Robust)和復(fù)合型類群(Complex)。結(jié)合已有的形態(tài)學(xué)研究,Romano認(rèn)為堅實型珊瑚骨骼鈣化程度較高,具有堅實的骨骼結(jié)構(gòu),珊瑚外觀主要以碟片狀和團(tuán)塊狀為主,其代表種類包括蜂巢珊瑚科(Faviidae)、石芝珊瑚科(Fungiidae)、褶葉珊瑚科(Lobophyllidae)等。復(fù)合型珊瑚骨骼鈣化程度較低,具有較為復(fù)雜多變的生長形態(tài),其代表種類包括鹿角珊瑚科(Acroporidae)、菌珊瑚科(Agaricidae)、木珊瑚科(Dendrophlliidae)、濱珊瑚科(Portidae)等。經(jīng)過近20 a的造礁石珊瑚分子系統(tǒng)學(xué)研究發(fā)展,國內(nèi)外的許多學(xué)者積累的分子數(shù)據(jù)都極力地支持了這一學(xué)說。Fukami等[21]使用多種分子標(biāo)記對17科75屬127種造礁石珊瑚進(jìn)行了系統(tǒng)進(jìn)化分析,在堅實型和復(fù)合型兩大分支的基礎(chǔ)上將造礁石珊瑚細(xì)分成21個分類群,其中9個分支類群屬于復(fù)合型珊瑚,12個分支類群屬于堅實型珊瑚。本研究采用鄰位連接法(NJ)、最大似然法(ML)和最小進(jìn)化法(ME)對14種造礁石珊瑚16S rRNA基因片段構(gòu)建了系統(tǒng)發(fā)育樹,聚類結(jié)果符合兩大類群學(xué)說的系統(tǒng)進(jìn)化結(jié)果。濱珊瑚科、菌珊瑚科、枇杷珊瑚科和鹿角珊瑚科的品種聚為一大類,即為復(fù)合型類群。蜂巢珊瑚和裸肋珊瑚科的品種聚為另一類,即為堅實型類群。
相對來說,復(fù)合型珊瑚類群的分子系統(tǒng)進(jìn)化關(guān)系與傳統(tǒng)分類學(xué)的系統(tǒng)關(guān)系較為一致。而堅實型珊瑚類群的分子系統(tǒng)進(jìn)化關(guān)系則與傳統(tǒng)分類學(xué)存在很多不一致的地方,部分科屬之間關(guān)系較為混亂。Huang等[22]根據(jù)Fukami劃分的21個造礁石珊瑚分支類群進(jìn)行研究,發(fā)現(xiàn)屬于堅實型的第17個分支類群最為混亂,里面包含了的4個科:蜂巢珊瑚科(Faviidae)、裸肋珊瑚科(Merulinidae)、梳狀珊瑚科(Pectiniidae)、腦珊瑚科(Trachyphylliidae)。這個類群在分子系統(tǒng)進(jìn)化關(guān)系上存在相互并系現(xiàn)象,不同科屬之間的物種重新聚為新的分支。本研究從科一級聚類結(jié)果看,16S rRNA基因構(gòu)建的系統(tǒng)進(jìn)化關(guān)系與傳統(tǒng)形態(tài)學(xué)的構(gòu)建的系統(tǒng)進(jìn)化關(guān)系基本一致,各科的造礁石珊瑚品種都各自聚為一支,但裸肋珊瑚科的腐蝕刺柄珊瑚的系統(tǒng)進(jìn)化關(guān)系卻與傳統(tǒng)形態(tài)學(xué)存在一定的差異,聚類結(jié)果顯示腐蝕刺柄珊瑚聚到了蜂巢珊瑚科中并且與角蜂巢珊瑚關(guān)系最為接近。許多學(xué)者利用不同分子標(biāo)記對蜂巢珊瑚科和裸肋珊瑚科的珊瑚品種進(jìn)行系統(tǒng)進(jìn)化分析,都得到了與本研究相類似的聚類結(jié)果[13,21~23]。近年來在骨骼研究方面,我國臺灣學(xué)者戴昌鳳等[24]通過共骨結(jié)構(gòu)對比同樣認(rèn)為刺柄珊瑚屬也應(yīng)歸屬于蜂巢珊瑚科。因此,傳統(tǒng)分類對于刺柄珊瑚屬的歸類于裸肋珊瑚科的論斷值得商榷,本研究結(jié)果支持刺柄珊瑚屬歸屬于蜂巢珊瑚科。
蜂巢珊瑚科是造礁石珊瑚中種類第二豐富的一個類群,近年來許多學(xué)者通過分子系統(tǒng)進(jìn)化研究的結(jié)果都顯示蜂巢珊瑚科的多重起源造成了不同屬間復(fù)雜的親緣關(guān)系[21-23]。本研究中,同為角蜂巢珊瑚屬的秘密角蜂巢珊瑚(Favites.abidita)、小五邊角蜂巢珊瑚(Favites.micropentagona)和角蜂巢珊瑚sp(Favites.sp)都聚為一支,但五邊角蜂巢(Favites.pentagona)先與扁腦珊瑚屬(Platygyra)的肉質(zhì)扁腦珊瑚(Platygyra.carnosus)聚為一支,再與角蜂巢珊瑚屬聚到一起。這與傳統(tǒng)形態(tài)學(xué)系統(tǒng)分類存在一定的差異。Huang等[25]在對新加坡附近海域的蜂巢珊瑚科珊瑚系統(tǒng)進(jìn)化關(guān)系研究的過程中發(fā)現(xiàn)蜂巢珊瑚科的并系現(xiàn)象普遍存在,形態(tài)學(xué)分類為同一屬的品種往往是不同祖先的蜂巢珊瑚趨同進(jìn)化的結(jié)果。Arrigoni等[23]采用COI作為分子標(biāo)記對印度洋海域的蜂巢珊瑚科的珊瑚品種進(jìn)行系統(tǒng)進(jìn)化分析,結(jié)果同樣顯示五邊角蜂巢珊瑚與扁腦珊瑚屬關(guān)系更近,而與其他幾種角蜂巢珊瑚關(guān)系更遠(yuǎn)。從本研究結(jié)果來看,五邊角蜂巢珊瑚和肉質(zhì)扁腦珊瑚可能具有更近一個共同祖先。
近年來,許多學(xué)者通過顯微掃描技術(shù)對造礁石珊瑚骨骼的精細(xì)結(jié)構(gòu)進(jìn)行觀察,對20世紀(jì)Veron[18]經(jīng)典的傳統(tǒng)形態(tài)學(xué)研究結(jié)果進(jìn)行了校正和補充[26-27]。新的造礁石珊瑚骨骼研究正逐漸改變對造礁石珊瑚傳統(tǒng)分類的看法,同時也對分子系統(tǒng)進(jìn)化研究結(jié)果提供了有力的佐證。在當(dāng)今的造礁石珊瑚系統(tǒng)進(jìn)化研究中,無論是傳統(tǒng)的形態(tài)學(xué)方法還是利用分子技術(shù)手段,兩者都必須相互借鑒和補充。
[1]鄒仁林.中國動物志—造礁石珊瑚 [M].北京:科學(xué)出版杜,2001.
[2]尤豐,黃暉,董志軍,等.造礁石珊瑚的分子系統(tǒng)學(xué)研究進(jìn)展[J].海洋通報,2010,29(4):459-465.
[3]劉麗,陳育盛,李曉娜,等.基于線粒體 Cytb 基因的10 種石珊瑚的系統(tǒng)發(fā)育關(guān)系[J].廣東海洋大學(xué)學(xué)報,2011,31(1):6-11.
[4]余克服,王肇鼎,練建生,等.近50年來大亞灣及周邊地區(qū)氣候變化趨勢[M]//潘金培,王肇鼎,吳信忠.海灣生態(tài)環(huán)境與生物資源持續(xù)利用.北京:科學(xué)出版社,2001:1-9.
[5]張元林,鄒仁林.大亞灣淺水石珊瑚群落結(jié)構(gòu)的研究[J].熱帶海洋,1987,6(1):12-18.
[6]陳天然,余克服,施祺,等.大亞灣石珊瑚群落近25年的變化及其對2008年極端低溫事件的響應(yīng)[J].科學(xué)通報,2009(6):812-820.
[7]陳天然,余克服,施祺,等.廣東大亞灣石珊瑚群落的分布及動態(tài)變化[J].熱帶地理,2007,27(6):493-498.
[8]FOIGHIL D ó,GAFFNEY P M,HILBISH T J.Differences in mitochondrial 16S ribosomal gene sequences allow discrimination among American[Crassostrea virginica(Gmelin)] and Asian [C.gigas(Thunberg) C.ariakensis Wakiya] oyster species[J].Journal of Experimental Marine Biology and Ecology,1995,192(2):211-220.
[9]SCHNEIDER J A,F(xiàn)OIGHIL D ó.Phylogeny of giant clams( Cardiidae:Tridacninae) based on partial mitochondrial 16S rDNA gene sequences[J].MolecularPhylogenetics and Evolution,1999,13(1):59-66.
[10]張珰妮,鄭連明,何勁儒,等.基于線粒體 COI 和16S 片段序列的北部灣北部水螅水母 DNA 條形碼分析[J].生物多樣性,2015,23(1):50-60.
[11]CHAN T Y,TONG J,TAM Y K,et al.Phylogenetic relationships among the genera of the Penaeidae(Crustacea:Decapoda)revealed by mitochondrial 16S rRNA gene sequences[J].Zootaxa,2008,1694(3).
[12]THOMAS Y,BELLIARD C,GAREN P,et al.Development of in situ hybridisation using 16S rRNA gene to monitor black-lip pearl oyster,Pinctada margaritifera,larvae in plankton samples[J].Aquatic Living Resources,2011,24(01):27-34.
[13]ROMANO S L,PALUMBI S R.Molecular evolution of a portion of the mitochondrial 16S ribosomal gene region in scleractinian corals[J].Journal of Molecular Evolution,1997,45(4):397-411.
[14]FORSMAN Z H,CONCEPCION G T,HAVERKORT R D,et al.Genetic and Morphological Characterization of a Coral Species of Concern Montipora dilatata in Kaneohe Bay,Oahu,Hawaii[J].Pacific Islands Region Species of Concern,2008.
[15]ROMANO S L,CAIRNS S D.Molecular phylogenetic hypotheses for the evolution of scleractinian corals[J].Bulletin of Marine Science,2000,67(3):1043-1068.
[16]LE Goff-Vitry M C,ROGERS A D,BAGLOW D.A deep-sea slant on the molecular phylogeny of the Scleractinia[J].Molecular Phylogenetics and Evolution,2004,30(1):167-177.
[17]劉麗,李曉娜,陳育盛,等.基于線粒體基因的石珊瑚分子系統(tǒng)學(xué)研究[J].海洋與湖沼,2012,43(4):814-820.
[18]VERON J E N.Corals in space and time:the biogeography and evolution of the Scleractinia[M].Ithaca,New York:Comstock/Cornell,1995.
[19]FUKAMI H,KNOWLTON N.Analysis of complete mitochondrial DNA sequences of three members of the Montastraea annularis coral species complex(Cnidaria,Anthozoa,Scleractinia)[J].Coral Reefs,2005,24(3):410-417.
[20]李曉娜.基于線粒體基因的常見造礁石珊瑚系統(tǒng)進(jìn)化研究[D].廣東海洋大學(xué),2012.
[21]FUKAMI H,CHEN C A,BUDD A F,et al.Mitochondrial and nuclear genes suggest that stony corals are monophyletic but most families of stony corals are not(Order Scleractinia,Class Anthozoa,Phylum Cnidaria)[J].PloS one,2008,3(9):e3222.
[22]HUANG D,LICUANAN W Y,BAIRD A H,et al.Cleaning up the ‘Bigmessidae':Molecular phylogeny of scleractinian corals from Faviidae,Merulinidae,Pectiniidae and Trachyphylliidae[J].BMC Evolutionary Biology,2011,11(1):1.
[23]ARRIGONI R,STEFANI F,PICHON M,et al.Molecular phylogeny of the robust clade(Faviidae,Mussidae,Merulinidae,and Pectiniidae):an Indian Ocean perspective[J].Molecular Phylogenetics and Evolution,2012,65(1):183-193.
[24] 戴昌鳳,洪圣雯.臺灣珊瑚圖鑒[M].臺北:貓頭鷹出版杜,2009.
[25]HUANG D,MEIER R,TODD P A,et al.More evidence for pervasive paraphyly in scleractinian corals:Systematic study of Southeast Asian Faviidae(Cnidaria;Scleractinia)based on molecular and morphological data[J].Molecular Phylogenetics and Evolution,2009,50(1):102-116.
[26]STOLARSKI J,KITAHARA M V,MILLER D J,et al.The ancient evolutionary origins of Scleractinia revealed by azooxanthellate corals[J].BMC Evolutionary Biology,2011,11(1):316.
[27]HUANG D,BENZONI F,F(xiàn)UKAMI H,et al.Taxonomic classification of the reef coral families Merulinidae,Montastraeidae,and Diploastraeidae(Cnidaria:Anthozoa:Scleractinia)[J].Zoological Journal of the Linnean Society,2014,171(2):277-355.
(責(zé)任編輯:陳莊)
Phylogenetic Analysis for 14 Species of Reef-building Corals Based on 16S rRNA genes
LIAO Bao-Lin1,XIAO Bao-hua1,2,YANG Xiao-Dong1,XIE Zi-Qiang3
(1.Shenzhen Research Institute of Guangdong Ocean University,Shenzhen 518108,China;2.Guangdong Ocean University,Zhanjiang 524025,China;3.Shenzhen Ocean Hyaline Marine Science and Technology Co,Ltd.Shenzhen 518108,China)
Partial sequences of 16S rRNA from 14 species of reef-building corals in Dalajia Island and Xiaolajia Island were amplified and sequenced,and Base ratio and genetic distances were analyzed.The result indicated that the average A+T of this region accounted for 63.5% from 14 species; A+T contents of 16S rRNA fragments were higher than G+C obviously.The sequences of 16S rRNA fragments had a high degree of saturation.The average genetic distance was 0.179 from 14 species.According to the Neighbour-joining,Maximum-likelihood and Minimum-evolution trees,the separated Faviidae was the root clade,and the second clade was composed of Agariciidae,Acroporidae,Poritidae and Oculinidae.The Hydnophora exesa of Merulinidae merged into the Faviidae.The result suggested that morphological classification may be limited by coral skeletons plasticity.
Reef-building corals; 16SRNA; Molecular phylogenetics
S917.4
A
1673-9159(2016)04-0023-07
10.3969/j.issn.1673-9159.2016.04.005
2016-05-13
廣東省公益研究與能力建設(shè)專項 (K15216);廣東省海洋漁業(yè)科技推廣專項(A201308E02)
廖寶林(1984—),男,碩士,工程師,主要從事海洋生態(tài)學(xué)研究。 E-mail:13590010881@163.com
肖寶華(1978—),男,助理研究員,碩士,主要從事水產(chǎn)養(yǎng)殖及海洋生態(tài)學(xué)研究。電話:0759-2396216,E-mail:gdouxxhpaper@126.com

