長江中下游湖泊和云南撫仙湖黃顙魚群體遺傳結(jié)構(gòu)研究
劉紅艷, 陳校輝, 熊 飛
(1.江漢大學(xué) 生命科學(xué)學(xué)院, 武漢 430056; 2.江蘇省淡水水產(chǎn)研究所, 南京 210017)
?
長江中下游湖泊和云南撫仙湖黃顙魚群體遺傳結(jié)構(gòu)研究
劉紅艷1*, 陳校輝2, 熊 飛1
(1.江漢大學(xué) 生命科學(xué)學(xué)院, 武漢 430056; 2.江蘇省淡水水產(chǎn)研究所, 南京 210017)
采用10個(gè)微衛(wèi)星位點(diǎn)對(duì)長江中下游5個(gè)湖泊和云南撫仙湖黃顙魚群體遺傳結(jié)構(gòu)進(jìn)行分析.結(jié)果顯示,黃顙魚各群體平均等位基因數(shù)為3.5~4.6,平均有效等位基因數(shù)為2.240~3.041,平均觀測雜合度為0.343~0.499,平均期望雜合度為0.432~0.600,平均多態(tài)性信息指數(shù)為0.389~0.571.鄱陽湖、滆湖和洪澤湖群體的遺傳多樣性高于太湖、巢湖和撫仙湖群體.群體間的遺傳距離為0.089~0.524,其中,鄱陽湖與巢湖群體的遺傳距離最小,鄱陽湖與滆湖群體遺傳距離最大,IBD分析結(jié)果顯示黃顙魚群體不遵循地理距離模型.AMOVA顯示大多數(shù)遺傳變異存在于群體內(nèi)(84.51%),群體間的遺傳變異為15.49%(Fst=0.1549),表明黃顙魚群體存在明顯的遺傳分化,兩兩遺傳分化指數(shù)也證實(shí)了這一點(diǎn).UPGMA聚類和Structure分析結(jié)果均顯示6個(gè)黃顙魚群體可分為2組,鄱陽湖、巢湖和洪澤湖為一組,滆湖、太湖和撫仙湖群體為另一組.
黃顙魚; 微衛(wèi)星; 遺傳結(jié)構(gòu)
黃顙魚(Peltebagrusfulvidraco)屬鯰形目(Siluriformes)、鲿科(Bagridae)、黃顙魚屬(Pelteobagrus),是一種以動(dòng)物性餌料為主的雜食性魚類,常棲息于河流、水庫和湖泊的底層[1].黃顙魚的營養(yǎng)價(jià)值高,生存適應(yīng)能力強(qiáng),黃顙魚在我國各大水系均有分布[2-3].通過微衛(wèi)星對(duì)不同水系黃顙魚群體的遺傳多樣性和遺傳結(jié)構(gòu)分析已經(jīng)有一些報(bào)道,如吳勤超等[4]分析了長江中上游流域黃顙魚野生群體的遺傳多樣性,李大宇等[5]研究了東北、長江中上游和天津地區(qū)黃顙魚群體的遺傳多樣性,馬洪雨等[6]和郭金峰等[7]研究了山東黃顙魚群體的遺傳多樣性.而目前未見長江中下游的湖泊黃顙魚群體微衛(wèi)星遺傳變異研究.另外,黃顙魚作為一種廣適性魚類,成功建群于云南高原湖泊撫仙湖,成為撫仙湖的外來種,目前僅有黃顙魚作為外來種的年齡和生長特征報(bào)道[8],未見其遺傳多樣性研究.
本研究采集鄱陽湖、巢湖、洪澤湖、滆湖及太湖5個(gè)長江中下游湖泊群體和云南撫仙湖黃顙魚群體,通過對(duì)其微衛(wèi)星基因型的分析,探討6個(gè)湖泊黃顙魚群體遺傳多樣性和遺傳結(jié)構(gòu)差異,群體間遺傳關(guān)系和遺傳分化情況,以期了解長江中下游黃顙魚野生群體的遺傳背景和種質(zhì)資源現(xiàn)狀.同時(shí)對(duì)比分析長江中下游地區(qū)和撫仙湖群體遺傳多樣性差異,考察撫仙湖外來黃顙魚群體的遺傳多樣性情況,為探討外來種的遺傳適應(yīng)機(jī)制提供參考.
1 材料與方法
1.1 樣本采集和DNA提取
黃顙魚樣品采集于鄱陽湖、巢湖、洪澤湖、滆湖和太湖5個(gè)長江中下游湖泊及云南高原湖泊撫仙湖.鄱陽湖群體樣本數(shù)為21尾,其它群體的樣本數(shù)均為30尾.取黃顙魚背部肌肉組織置于95%乙醇中帶回實(shí)驗(yàn)室.取保存于乙醇中的樣品約20 mg,0.9%的生理鹽水浸泡約12 h(期間更換生理鹽水3~4次),用標(biāo)準(zhǔn)的酚-氯仿法提取DNA.獲得的DNA用核酸蛋白分析儀檢測其純度和濃度,最終DNA模板濃度定量在20 ng/μL,保存于冰箱備用.
1.2 微衛(wèi)星分析
根據(jù)已發(fā)表的文獻(xiàn)[4,6,9],選取10個(gè)黃顙魚微衛(wèi)星位點(diǎn)進(jìn)行研究,這些位點(diǎn)為:AG12、AG48、AG128、CT30、CT81、CT154、CT209、HSY208、HSY407及MFW30(Thermo Fisher公司合成),微衛(wèi)星特征如表1.PCR反應(yīng)總體積為10 μL,包括Taq DNA 聚合酶0.5 U(Thermo Fisher公司生產(chǎn)),dNTP 0.2 mmol/L,MgCl22.0 mmol/L,1×Buffer,引物0.2 μmol/L,DNA 模板約20 ng,補(bǔ)充滅菌蒸餾水到10 μL.PCR 擴(kuò)增程序?yàn)?5℃預(yù)變性5 min,接著進(jìn)行35個(gè)循環(huán):94℃變性45 s、適當(dāng)退火溫度退火30 s(表1)、72℃延伸45 s,最后72℃延伸10 min.反應(yīng)在PCR儀(PTC100型)上進(jìn)行.擴(kuò)增產(chǎn)物用非變性的8%聚丙烯酰胺凝膠電泳進(jìn)行分離,并通過銀染法進(jìn)行檢測.
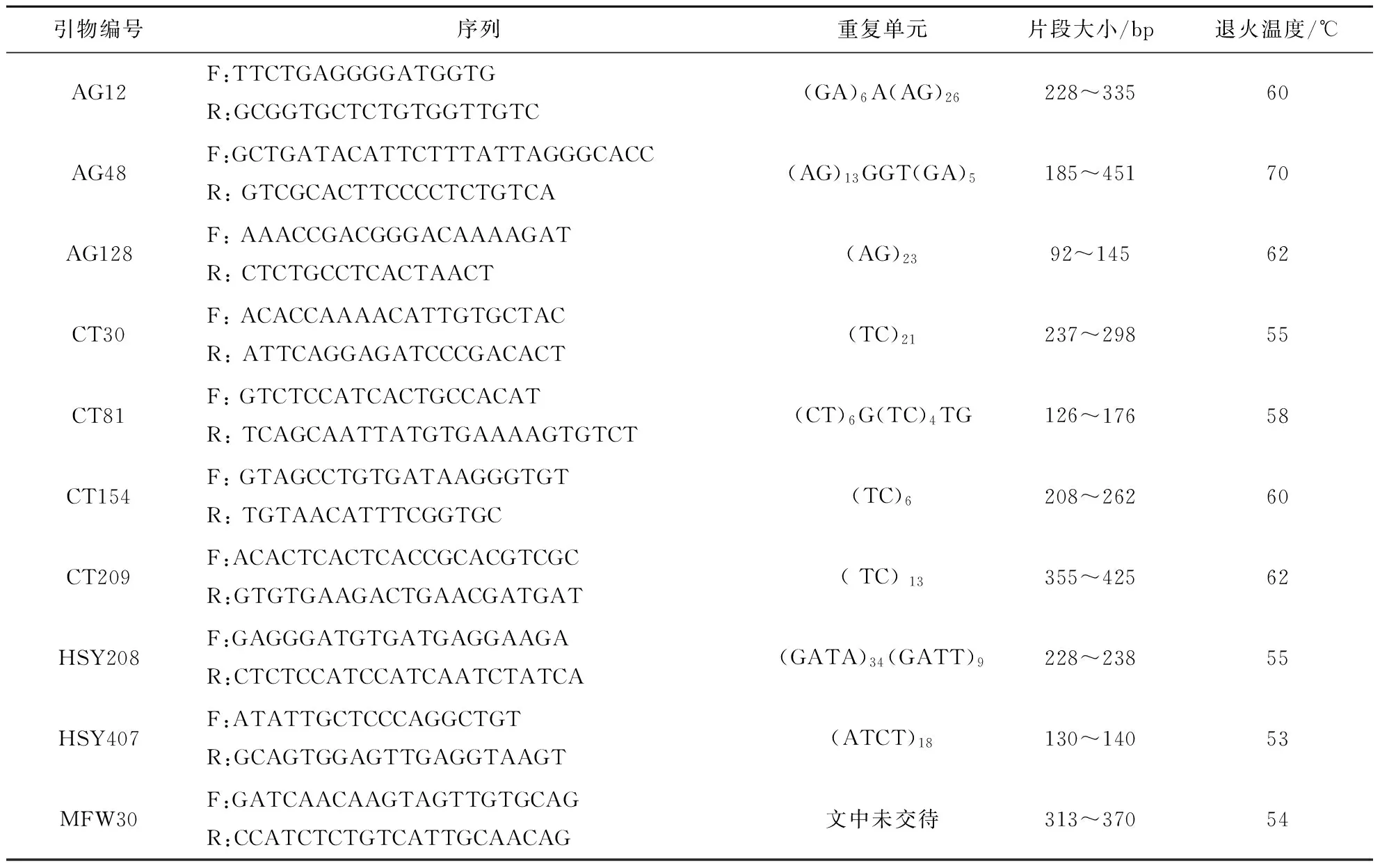
表1 10對(duì)微衛(wèi)星引物特征
1.3 數(shù)據(jù)分析
根據(jù)DNA Marker標(biāo)記和微衛(wèi)星DNA的遷移率,讀取各等位基因條帶.利用CONVERT1.31軟件[10]將各群體等位基因條帶矩陣,轉(zhuǎn)化相關(guān)分子遺傳學(xué)軟件所需的格式.用GenePop1.2軟件[11]計(jì)算各遺傳多樣性參數(shù),包括各位點(diǎn)的等位基因數(shù)目(Na)、有效等位基因數(shù)目(Ne)、觀測雜合度(Ho)、期望雜合度(He).用PIC-CALC 0.6軟件計(jì)算多態(tài)性信息指數(shù)(PIC).
群體間遺傳距離、遺傳分化指數(shù)用Arlequin 3.1軟件[12]計(jì)算.通過IBDWS 3.23軟件[13]里的Mantel檢驗(yàn)程序,對(duì)不同群體遺傳距離和地理距離的相關(guān)性進(jìn)行檢驗(yàn)(參數(shù)設(shè)定為10 000次重復(fù)).基于Nei’s遺傳距離,采用NTSYS-PC 2.1軟件[14]中的SAHN程序和非加權(quán)組平均法(UPGMA)進(jìn)行聚類,分析群體間的相互遺傳關(guān)系.
分子變異方差分析用AMOVA軟件[15]計(jì)算,檢測群體內(nèi)和群體間的遺傳變異情況,計(jì)算中考慮微衛(wèi)星等位基因的大小差異,所有多重比較中的P值都進(jìn)行泊松校正.群體遺傳結(jié)構(gòu)用Stucture2.3軟件[16]的Bayesian方法進(jìn)行分析,采用混合模型和等位變異發(fā)生相關(guān)頻率模型,參數(shù)“Burnin Period”設(shè)為200 000次,“after Burnin”設(shè)為 500 000 次,將所有個(gè)體設(shè)定為1~7個(gè)種群(即K=1~7),每個(gè)K值運(yùn)行10次.基于K值對(duì)應(yīng)的對(duì)數(shù)值變化率的ΔK作曲線圖,確定真正的K值.
2 結(jié)果
2.1 微衛(wèi)星遺傳多樣性
從表2可以看出,黃顙魚各群體平均等位基因數(shù)為3.5~4.6,平均有效等位基因數(shù)為2.240~3.041,平均觀測雜合度為0.343~0.499,平均期望雜合度為0.432~0.600,平均多態(tài)性信息指數(shù)為0.389~0.571.6個(gè)群體的總體Na=7.5,Ne=3.527,Ho=0.444,He=0.615,PIC=0.575.有效等位基因數(shù)為:鄱陽湖>滆湖>洪澤湖>太湖>撫仙湖>巢湖.觀測雜合度為:鄱陽湖>滆湖>洪澤湖>太湖>巢湖>撫仙湖.期望雜合度為:洪澤湖>鄱陽湖>滆湖>太湖>撫仙湖>巢湖;多態(tài)性信息含量指數(shù)為:鄱陽湖>洪澤湖>滆湖>太湖>撫仙湖>巢湖.總體來看,鄱陽湖、滆湖和洪澤湖的遺傳多樣性高于太湖、撫仙湖和巢湖.相對(duì)而言,巢湖群體和外來撫仙湖群體的遺傳多樣性較低.

表2 黃顙魚6個(gè)群體的遺傳多樣性參數(shù)
Na:等位基因數(shù);Ne:有效等位基因數(shù);Ho:觀測雜合度;He:期望雜合度;PIC: 多態(tài)性信息含量指數(shù)
Notes:Na: No. of alleles;Ne:No. of effective alleles;Ho: observed heterozygosity;He: expected heterozygosity;PIC: polymorphism information content
2.2 遺傳距離與聚類分析
群體間的兩兩遺傳距離為0.089~0.524,鄱陽湖與巢湖群體的遺傳距離最小,鄱陽湖與滆湖群體遺傳距離最大(表3).UPGMA聚類分析結(jié)果如圖1.鄱陽湖與巢湖群體首先聚在一起,然后再與洪澤湖群體聚為一分支,滆湖與太湖群體聚在一起,然后再與撫仙湖群體聚成另一分支.

表3 黃顙魚群體兩兩遺傳距離和遺傳分化指數(shù)

圖1 基于Nei’s遺傳距離的UPGMA聚類圖Fig.1 UPGMA Clustering result based on Nei’s genetic distance
IBD檢驗(yàn)6個(gè)群體的遺傳距離與地理距離相關(guān)性,表明黃顙魚遺傳距離與地理距離不相關(guān)(r=0.2230,p=0.5440,圖2A).因撫仙湖黃顙魚群體為外來種,為了排除干擾,去掉撫仙湖群體,只檢測長江中下游5個(gè)群體的遺傳距離與地理距離相關(guān)性,結(jié)果同樣顯示遺傳距離與地理距離沒有相關(guān)性(r=0.4611,p=0.0825,圖2B),表明黃顙魚群體不遵循地理距離模型.

A):6個(gè)黃顙魚群體的遺傳距離與地理距離相關(guān)性檢驗(yàn);B):去掉撫仙湖群體后,長江中下游5個(gè)群體遺傳距離與地理距離相關(guān)性檢驗(yàn)圖2 Mantel檢驗(yàn)遺傳距離與地理距離相關(guān)性Fig.2 Correlation of genetic distance and geographic distance by Mantel test
2.3 遺傳分化和遺傳結(jié)構(gòu)
分子方差分析(AMOVA)顯示(表4),大多數(shù)遺傳變異存在于黃顙魚的個(gè)體水平(81.41%),群體內(nèi)個(gè)體間的為3.10%,群體間的遺傳變異為15.49%(Fst=0.1549).從群體兩兩比較的遺傳分化指數(shù)來看(表3),撫仙湖、滆湖和太湖的兩兩遺傳分化指數(shù)在0.005~0.024之間,這3群體間的遺傳分化指數(shù)較小;同樣,鄱陽湖、巢湖和洪澤湖兩兩遺傳分化指數(shù)在0.069~0.107之間,這3群體間的遺傳分化指數(shù)也較小.但聚類關(guān)系中兩分支的兩兩群體間遺傳分化指數(shù)較大,為0.202~0.367.太湖與撫仙湖的遺傳分化指數(shù)最小(0.005),鄱陽湖與滆湖的遺傳分化指數(shù)最大(0.367).

表4 黃顙魚群體遺傳結(jié)構(gòu)AMOVA分析
通過Bayesian分析得出的ΔK值,構(gòu)建K與ΔK的值曲線圖(圖3),可以看出,雖然出現(xiàn)多峰,但當(dāng)ΔK值最大時(shí),對(duì)應(yīng)的K值為2,表明黃顙魚遺傳結(jié)構(gòu)可分為2組.我們進(jìn)一步對(duì)K=2時(shí)生成的Structure柱狀圖進(jìn)行了分析,結(jié)果顯示,撫仙湖、滆湖和太湖群體為一組,洪澤湖、巢湖和鄱陽湖群體為另一組(圖4).2組間的遺傳分化顯著,表明6個(gè)黃顙魚群體可分為2個(gè)明顯的遺傳分化群.

圖3 structure 混合模型分析的K與ΔK值曲線圖Fig.3 The model choice criterion ΔK of the Structure analysis for each K value

圖4 STRUCTURE 2.3混合模型分析黃顙魚的遺傳分化群Fig.4 Genetic clusters of Pelteobagrus fulvidraco performed by mixture model with STRUCTURE 2.3
3 討論
遺傳多樣性是評(píng)價(jià)物種資源狀況的一個(gè)重要依據(jù).它是物種適應(yīng)多變的環(huán)境條件、維持長期生存和進(jìn)化的基礎(chǔ)[17-18].Ne、Ho、He和PIC等都是反映群體遺傳多樣性的度量,其數(shù)值越大,說明基因豐富度越高.從本次研究結(jié)果來看,黃顙魚群體的遺傳多樣性水平較高(Ne=3.527,Ho=0.444,He=0.615,PIC=0.575).與吳勤超等[4]報(bào)道的長江中上游3個(gè)黃顙魚群體的平均值(Ne=2.568,He=0.543 8,PIC=0.465 7)相比,本研究結(jié)果要略高.與李大宇等[5]報(bào)道的東北、長江中上游和天津地區(qū)6個(gè)黃顙魚群體(Ne=1.96,Ho=0.49,He=0.40,PIC=0.34)和馬洪雨等[6]報(bào)道的東平湖黃顙魚群體(Ho=0.583 3,He=0.537 2,PIC=0.488)相比,本研究所檢測的觀測雜合度稍低,但有效等位基因、期望雜合度和多態(tài)性信息指數(shù)要高于馬洪雨和李大宇的結(jié)果.與郭金峰等[7]報(bào)道的3個(gè)山東黃顙魚群體(Ho=0.641 8,PIC=0.588 9)相比略低.DeWoody等[19]得出淡水魚類(He=0.46,Ne=7.50)的結(jié)論,本研究6個(gè)黃顙魚群體的He與之相差不大,而Ne偏低.Ne較低的原因可能是由于過度捕撈、棲息地環(huán)境的變化及產(chǎn)卵場的縮小等原因所導(dǎo)致的有效種群縮小使得有效等位基因更容易丟失.
根據(jù)Botstein等[20]的標(biāo)準(zhǔn),當(dāng)PIC>0.5時(shí),為高度多態(tài)性;當(dāng)0.25
在這6個(gè)群體中,撫仙湖外來黃顙魚群體的遺傳多樣性相對(duì)較低.Wares等[21]對(duì)29個(gè)有關(guān)外來動(dòng)物的遺傳多樣性的比較研究后發(fā)現(xiàn),外來種群的期望雜合度比原產(chǎn)地種群均有降低,本研究中,撫仙湖外來黃顙魚群體的觀測雜合度均低于原產(chǎn)地;期望雜合度除高于原產(chǎn)地巢湖外,相比其它原產(chǎn)地的期望雜合度均有降低.這可能是由于外來種群常常由原產(chǎn)地的少數(shù)個(gè)體繁衍而來,會(huì)受到奠基者效應(yīng)的影響,另外,外來種在引入地可能會(huì)因新環(huán)境的選擇壓力而長期處于小種群狀態(tài),因此遺傳多樣性水平往往會(huì)低于原產(chǎn)地.
6個(gè)黃顙魚群體間的遺傳距離為0.089~0.524,相對(duì)于李大宇等[5]用微衛(wèi)星對(duì)東北、長江中上游和天津地區(qū)黃顙魚的遺傳距離結(jié)果要小,但本研究的微衛(wèi)星遺傳距離結(jié)果要遠(yuǎn)大于線粒體Cyt b和線粒體D控制區(qū)的遺傳距離結(jié)果[22-23].6個(gè)群體當(dāng)中,僅考慮原產(chǎn)地的群體,滆湖與太湖在地理位置上距離最近,鄱陽湖與太湖的距離最遠(yuǎn),而群體間的遺傳距離表明:鄱陽湖與巢湖群體的遺傳距離最小,鄱陽湖與滆湖群體遺傳距離最大,IBD檢驗(yàn)結(jié)果顯示群體所分布的地理位置及群體間遺傳距離的遠(yuǎn)近并沒有一定的聯(lián)系.這可能與近年來長江水域人類活動(dòng)的干擾(如捕撈強(qiáng)度,引種、棲息地破壞等因素)相關(guān),而人類活動(dòng)的干擾具有一定的差異性和隨機(jī)性.
本研究中,群體間的Fst為0.154 9,顯示黃顙魚群體存在明顯的遺傳分化.聚類結(jié)果顯示鄱陽湖、巢湖和洪澤湖群體聚為一支,滆湖、太湖和撫仙湖群體聚成另一支,Structure分析結(jié)果也支持這6個(gè)黃顙魚群體分為2個(gè)明顯的遺傳分化群.鐘立強(qiáng)等[22]用線粒體Cyt b(Fst=0.068 4)研究長江中下游黃顙魚群體認(rèn)為群體間沒有遺傳分化,劉朋朋等[23]用線粒體D控制區(qū)(Fst=0.089 6)研究長江中下游黃顙魚群體認(rèn)為群體間的遺傳分化處于中等水平.本研究是用微衛(wèi)星分子標(biāo)記是對(duì)整個(gè)基因組的檢測,而線粒體Cyt b和線粒體D控制區(qū)僅對(duì)幾百個(gè)堿基的檢測,故而本研究能檢測到更多的遺傳變異,微衛(wèi)星與mtDNA技術(shù)比,信息含量更大,靈敏度更高,因此,微衛(wèi)星標(biāo)記檢測到長江中下游水系黃顙魚群體間存在2個(gè)明顯的遺傳分化群是較為可信的.外來物種的遺傳變異可用來推測其地理起源,聚類分析和Structure結(jié)果均支持,撫仙湖外來群體與太湖和滆湖群體聚在一起.20世紀(jì)80年代初,云貴高原湖泊相繼從長江流域太湖等湖泊引入太湖新銀魚等經(jīng)濟(jì)魚類,黃顙魚卵或幼魚可能隨太湖新銀魚等經(jīng)濟(jì)魚類而被引入,另外黃顙魚撫仙湖群體與太湖群體的遺傳距離和遺傳分化指數(shù)均較小,由此推測,撫仙湖黃顙魚可能從長江流域太湖等湖泊引入.
[1] 褚新洛, 莫天培. 中國動(dòng)物志[M].北京:科學(xué)出版社,1999:152-156.
[2] 代應(yīng)貴, 陳毅峰. 清水江的魚類區(qū)系及生態(tài)類型[J].生態(tài)學(xué)雜志, 2007, 26(5):682-687.
[3] 張國松, 侯新遠(yuǎn), 尹紹武, 等. 黃顙魚屬(Pelteobagrus)魚類遺傳多樣性的研究進(jìn)展[J].江蘇農(nóng)業(yè)科學(xué), 2014, 42(3):174-179.
[4] 吳勤超, 梁宏偉, 李 忠, 等. 黃顙魚微衛(wèi)星標(biāo)記的篩選及三個(gè)野生群體的遺傳結(jié)構(gòu)分析[J].生物技術(shù)通報(bào), 2010(3):154-159.
[5] 李大宇, 殷倩茜, 侯 寧, 等. 黃顙魚(Pelteobagruseupogon)不同生態(tài)地理分布群體遺傳多樣性的微衛(wèi)星分析[J].海洋與湖沼, 2009, 40(4):460-469.
[6] 馬洪雨, 姜運(yùn)良, 郭金峰, 等. 利用微衛(wèi)星標(biāo)記分析東平湖黃顙魚的遺傳多樣性[J].激光生物學(xué)報(bào), 2006, 15(2):136-139.
[7] 郭金峰, 王 玉, 馬洪雨, 等. 三個(gè)黃顙魚群體遺傳多樣性及親緣關(guān)系的微衛(wèi)星標(biāo)記分析[J].氨基酸和生物資源, 2006, 28(3):5-8.
[8] 李秀啟, 陳毅峰, 李 堃. 撫仙湖外來黃顙魚種群的年齡和生長特征[J].動(dòng)物學(xué)報(bào), 2006, 52(2):263-271.
[9] 劉 臻, 魯雙慶, 張建社, 等. 黃顙魚微衛(wèi)星標(biāo)記篩選及特征分析[J].農(nóng)業(yè)生物技術(shù)學(xué)報(bào), 2008, 16(4):604-609.
[10] GLAUBITZ J. CONVERT:A user-friendly program to reformat diploid genotypic data for commonly used population genetic software packages[J].Molecular Ecology Notes, 2004, 4(2):309-310.
[11] RAYMOND M,ROUSSET F. POPGENE (version 1. 2),population genetics software for exact test and ecumenicism[J]. Journal of Heredity, 1995, 86:248-249.
[12] SCHNEIDER S,KUEFFER J M,ROESSLI D,et al. Arlequin, version 2.0:a software for population genetic data analysis[D]. Geneva:University of Geneva,2000.
[13] JENSEN J L,BOHONAK A J,KELLEY S T. Isolation by distance,web service[J]. BMC Genet, 2005, 6:13.
[14] ROHLF F J. Ntsys-PC Version 2.1:Numerical taxonomy and multivariate analysis system[D]. New York:Stony Brook University,2000.
[15] EXCOFFIER L,SMOUSE P E,QUATTRO J M. Analysis of molecular variance inferred from metric distances among DNA haplotypes:application to human mitochondria DNA restriction sites[J]. Genetics, 1992, 131(2):479-491.
[16] PRITCHARD J K,WEN W,F(xiàn)ALUSH D. Documentation for structure software:Version 2. 3[M]. Pittsburgh:Software Engineering Institute,2009:369-371.
[17] 肖明松, 崔 峰, 康 健, 等. 淮河烏鱧線粒體DNA控制區(qū)結(jié)構(gòu)分析及遺傳多樣性研究[J]. 華中師范大學(xué)學(xué)報(bào)(自然科學(xué)版), 2013, 47(1):82-90.
[18] 劉紅艷, 熊 飛, 董元火, 等. 鱇浪白魚野生與養(yǎng)殖群體遺傳多樣性的ISSR分析[J]. 華中師范大學(xué)學(xué)報(bào)(自然科學(xué)版), 2012, 46(5):591-595.
[19] DEWOODY J A,AVISE J C. Microsatellite variation in marine,freshwater and anadromous fishes compared with other animals[J]. J Fish Biol, 2000, 56(3):461-473.
[20] BOTSTEIN D,WHITE R L,SKOLNICK M,et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms [J]. Am J Human Genet, 1980, 32:314-331.
[21] WARES J P,HUGHES A R,GROSBERG P K. Species Invasions:Insights into Ecology, Evolution and Biogeography[M].Sunderland:Sinauer Press, 2005: 229-257.
[22] 鐘立強(qiáng), 劉朋朋, 潘建林, 等. 長江中下游5個(gè)湖泊黃顙魚(Pelteobagrusfulvidraco)種群線粒體細(xì)胞色素b基因的遺傳變異分析[J]. 湖泊科學(xué), 2013, 25(2):302-308.
[23] 劉朋朋, 鐘立強(qiáng), 潘建林, 等. 基于線粒體D-loop區(qū)分析黃顙魚(Pelteobagrusfulvidraco)五個(gè)淡水湖泊群體的遺傳多樣性[J]. 海洋與湖沼, 2013, 44(3):728-733.
Population genetic structure ofPelteobagrusfulvidracoin the lakes of the middle and lower reaches of the Yangtze River and in Fuxian Lake of Yunnan
LIU Hongyan1, CHEN Xiaohui2, XIONG Fei1
(1.School of Life Sciences, Jianghan University, Wuhan 430056;2.Freshwater Fisheries Research Institute of Jiangsu Province, Nanjing 210017)
Ten microsatellite loci were used to analyze the population genetic structure of Pelteobagrus fulvidraco in five lakes of the middle and lower reaches of the Yangtze River and in Fuxian Lake of Yunnan. In P. fulvidraco populations, the average number of alleles was 3.5 to 4.6 with the mean number of valid alleles being 2.240 to 3.041. The average value of observed and expected heterozygosity ranged from 0.343 to 0.499 and 0.432 to 0.600, and the mean PIC was 0.389 to 0.571. Genetic diversity parameters indicated that the populations from Poyan Lake, Ge Lake, and Hongze Lake had higher levels of genetic diversity than those from Taihu Lake, Chao Lake and Fuxian Lake. The value of pairwise genetic distance was 0.0089 to 0.524. The genetic distance between Poyang Lake and Chao Lake was the smallest, while the largest one was that between Poyang Lake and Ge Lake. IBD results showed that P. fulvidraco populations did not match the geographic distance model. AMOVA analysis revealed that most of the total variation occurred within populations (84.51%). The amount of differentiation among populations was 15.49% (Fst=0.1549), which exhibited a clear population differentiation. PairwiseFstcomparisons of populations also confirmed this. Both UPGMA clustering and Structure analysis results showed that the six populations were able to be divided into two groups. One was assembled with the individuals from Hongze Lake,Poyang Lake and Chao Lake, while the other was consisted of the individuals from the rest lakes.
Pelteobagrusfulvidraco; microsatellite; genetic structure
2015-11-13.
國家自然科學(xué)基金項(xiàng)目(51109091);江漢大學(xué)出國留學(xué)基金項(xiàng)目.
1000-1190(2016)02-0269-07
Q347
A
*E-mail: lhy9603@126.com.
 華中師范大學(xué)學(xué)報(bào)(自然科學(xué)版)2016年2期
華中師范大學(xué)學(xué)報(bào)(自然科學(xué)版)2016年2期
- 華中師范大學(xué)學(xué)報(bào)(自然科學(xué)版)的其它文章
- 非自治Kuramoto-Sivashinsky方程一致吸引子的存在性、一致有界性和收斂性
- 基于隨機(jī)跳違約強(qiáng)度的可轉(zhuǎn)換債券的定價(jià)
- 基于超效率DEA的中原經(jīng)濟(jì)區(qū)農(nóng)業(yè)生產(chǎn)效率研究
- 魚腥藻PCC7120染色體基因?qū)sr0757/alr0758的表達(dá)優(yōu)化及其鑒定
- 汽車傳感器電極漿料制備及相關(guān)特性研究
- 戰(zhàn)略性新興產(chǎn)業(yè)技術(shù)異地協(xié)同共享機(jī)制研究
——演化博弈視角
