環狀RNA研究進展
張進威,龍科任,王 訊,李明洲,馬繼登
(四川農業大學動物科技學院,動物遺傳育種研究所,成都 611130)
?
環狀RNA研究進展
張進威,龍科任,王 訊,李明洲,馬繼登*
(四川農業大學動物科技學院,動物遺傳育種研究所,成都 611130)
環狀RNA(circRNAs)是一類不同于線性RNA的內源非編碼RNA,它是通過反向剪接形成的閉合環狀RNA分子。circRNA不具有5′端帽子和3′端poly(A)尾巴結構,能夠穩定存在于各種類型真核細胞中。對于circRNA的數量和豐度的檢測,傳統分子生物學方法的能效非常有限,因此一直以來circRNA被認為是RNA異常剪接的產物。隨著生物信息學的快速發展和高通量測序技術的不斷革新,目前已經在真核細胞中發現了大量內源性circRNA,其中一些circRNA表達豐度高并呈現出時空表達特異性和物種間保守性,說明circRNA可能在調節基因表達方面具有重要功能。本文綜述了circRNA的特征、形成機制及生物學功能,并對近年來circRNA研究進展進行歸納總結,以期為真核生物基因表達調控研究、疾病檢測等提供基礎資料,并對circRNA在動物分子育種方面的研究進行展望。
環狀RNA;特征;生成機制;生物學功能
環狀RNA(circRNAs)是一類不具有5′端帽子和3′端poly(A)尾巴結構的共價閉合環狀RNA分子[1]。早在1976年,H.L.Sanger等[2]就在植物類病毒中發現了circRNA。隨后的幾十年間,科學家又相繼在小鼠[3]、猴子[4]、豬[5]、人[6]等物種中發現了circRNA。由于傳統的分子生物學方法對circRNA數量和豐度的檢測能效都非常有限,因此一直以來circRNA被認為是RNA異常剪接的產物[7]。近年來隨著生物信息學的快速發展和高通量測序技術的不斷革新,目前已經在真核細胞中發現了大量內源circRNA,大部分circRNA結構穩定、豐度高且呈現出時空表達特異性,說明circRNA可能在調控基因表達過程中發揮著重要作用[1,8-10]。根據序列構成的差異可以將circRNA分為3類:外顯子circRNA (Exonic circRNA, ecRNA)[11]、內含子circRNA(Circular intronic RNA, ciRNA)[12]、外顯子-內含子circRNA(Exon-intron circRNA, ElciRNA)[13]。這些circRNA具有自身特有的產生方式,同時它們的生物學功能也有一定差異。與其他非編碼RNA一樣,circRNA的序列和結構決定了它的生物學功能,circRNA可能通過以下幾種方式發揮生物學功能:miRNA海綿[9,14],作為翻譯模板[15],調控基因轉錄[12-13],與RNA結合蛋白作用[1,9,16]。近年來研究發現,circRNA可以與疾病相關的miRNA相互作用,從而參與疾病發生和形成,另外circRNA在體液中表達豐度高且穩定性好,可以作為未來癌癥等疾病檢測的新型標志物。
本文綜述了circRNA產生機制、特征、生物學功能,并對近年來circRNA最新研究進展進行歸納總結,以期為真核生物基因表達調控研究、疾病檢測等提供基礎資料,并對circRNA在動物分子育種方面的研究進行展望。
1 環狀RNA的生成機制
眾所周知,DNA中的遺傳信息通過轉錄傳遞到RNA,再經過翻譯傳遞到蛋白質,從而實現遺傳信息的編碼,產生特定的生物學功能。通常情況下,真核生物基因在轉錄后通過RNA剪接去除內含子并連接外顯子形成成熟RNA分子,經典剪接過程是指原始轉錄本——核不均一RNA(Heterogeneous nuclear RNA, hnRNA)去除內含子后將外顯子按照基因組編碼鏈的順序連接[17]。而當一個或多個外顯子在剪接過程中被去除或外顯子不按照基因組編碼鏈的順序連接,這一過程稱為RNA可變剪接(Alternative splicing),可變剪接進一步豐富了遺傳信息的多樣性[18]。近年來,在真核生物中發現了另一種普遍存在的剪接方式,它可以將一個以上的外顯子或內含子的5′端與3′端共價連接,形成環狀結構的RNA分子,這一剪接方式被稱為反向剪接(Back-splicing)(圖1)[16,19]。反向剪接與前體mRNA剪接存在相互競爭作用,從而決定hnRNA的加工命運并影響circRNA產生效率[20]。
circRNA有兩種形成途徑:外顯子環化和內含子環化,可以產生3種circRNA,即ecRNA、ElciRNA和ciRNA[21]。目前普遍認為有兩種機制解釋外顯子環化:套索驅動環化(Lariat-driven circularization);內含子配對環化(Intron-pairing driven circularization)[1]。前者認為hnRNA在轉錄過程中RNA部分折疊拉近非相鄰的外顯子,發生外顯子跳躍(Exon skipping),形成的套索中間體進一步剪接產生外顯子circRNA(ecRNA);后者則認為側翼內含子通過反向互補序列配對形成環狀結構,然后剪切去除內含子并連接外顯子,最終形成ecRNA。通常情況下,許多由內含子形成的套索結構很快被脫分支酶降解[22],而Y.Zhang等[12]在海拉(Hela)細胞和人胚胎干細胞中發現一些內含子套索結構能躲避分支酶降解而在胞內聚集,這些內含子具有特定的核酸序列(即:5′剪接點含有7個富集GU的motif和3′端分支點含有11個富集C的motif),在剪接作用后不被脫分支酶降解從而加工形成ciRNA。最新研究發現,當兩個以上的外顯子在環化時,它們之間的內含子可能滯留其中,最終形成既有外顯子又有內含子的ElciRNA[13]。ciRNA由2′-5′磷脂鍵連接環化,而ecRNA和ElciRNA由3′-5′磷脂鍵連接環化[21]。
不是所有外顯子都能形成circRNA,研究表明序列長度或一些特定核酸序列對于circRNA形成具有重要作用。W.R.Jeck等[1]和X.O.Zhang等[11]發現參與環化外顯子的側翼內含子中具有互補重復序列(如ALU元件)有助于外顯子環化。另外,側翼內含子越大,外顯子越易環化[21],可能原因是越大的內含子包含更多的互補重復序列,更容易配對形成環狀結構。此外,一些蛋白質分子可以參與反向剪接過程,從而調節circRNA形成。R.Ashwal-Fluss等[20]發現,在人和果蠅中盲肌蛋白(Muscleblind, MBL)能夠促進自身轉錄本第二外顯子環化,進一步研究表明MBL能夠作為RNA結合蛋白連接側翼內含子以維持環狀結構,從而促進第二外顯子環化。S.J.Conn等[23]發現,在上皮細胞-間充質轉化(Epithelial-Mesenchymal Transition, EMT)過程中,RNA結合蛋白Quaking(QKI)能夠結合到側翼內含子中特定的motif從而促進外顯子環化。而A.Ivanov等[24]研究發現,RNA編輯酶ADARs(Adenosine deaminases acting on RNA)能夠結合側翼內含子互補的雙鏈區域,解除雙鏈的相互作用從而抑制circRNA形成。可見,circRNA的形成受到特定核酸序列的順式調節作用和一些蛋白質分子的反式調節作用,但circRNA形成的具體分子機制仍有待進一步研究。
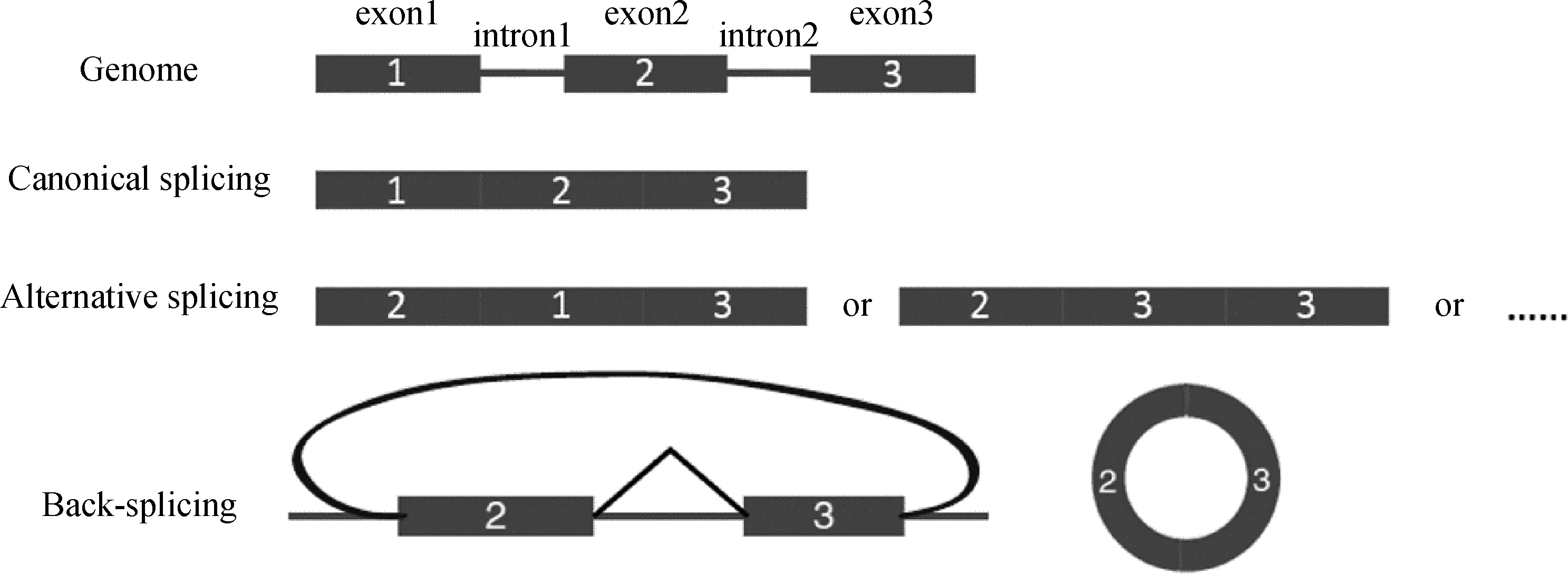
圖1 核不均一RNA分子剪切方式Fig.1 The splicing modes of hnRNA
2 環狀RNA的特征
與線性RNA分子相比,circRNA最典型的特征是形成了不具有5′端帽子和3′端poly(A)尾巴的共價閉合環狀結構,這使得circRNA比線性RNA分子更能夠抵抗核酸外切酶或核糖核酸酶的降解作用[25]。W.R.Jeck等[1]發現,在放線菌素D 處理時,circRNA的半衰期超過48 h,而與它們對應的線性轉錄本(HIPK3,KIAA0812,ASXL1,LPAR1)不超過20 h。J.H.Bahn等[26]在健康人體無細胞唾液中鑒定了超過400種circRNA,第一次發現circRNA也存在于體液中,佐證了circRNA具有較高的穩定性。近年來研究發現circRNA在某些組織中表達豐度較高,甚至超過相應的線性轉錄本10倍以上[1],當然circRNA與相應的線性RNA比例處于動態變化中,且表現出明顯的組織特異性。此外,通常情況下ecRNA主要位于細胞質中,且一些ecRNA含有miRNA作用元件,而ciRNA和ElciRNA定位于胞核中,可能參與基因表達調控(3種circRNA特征見表1)。外顯子來源的circRNA具有選擇性環化(Alternative circularization)作用(圖2),即5′端側翼內含子可以與鄰近3′端側翼內含子互補環化,也可以與間隔較遠的3′端側翼內含子互補環化,選擇性環化可產生不同組合的circRNA,與hnRNA的可變剪接類似,它可以進一步豐富circRNA的多樣性[11,27]。circRNA在進化上具有保守性,J.Salzman等[8]發現人類與小鼠有4%的直系同源基因能夠產生circRNA,J.Salzman等[8]和W.R.Jeck等[1]還發現在大腦中有44%的circRNA也存在于睪丸。S.Memczak等[9]發現含有編碼區(Coding sequence,CDS)的circRNA表現出較高的序列保守性,尤其是在第三位密碼子處,而來自基因間區和內含子的circRNA序列保守性相對較弱。circRNA這些特征提示它可能對基因表達具有重要的調控作用。
3 環狀RNA生物學功能
近年來通過高通量測序和生物信息學分析已經在真核生物中鑒定了大量的circRNA,這些circRNA呈現出時空表達特異性和物種間的保守性,說明circRNA可能在真核生物基因表達調控上具有重要作用。研究發現circRNA可能通過以下4種方式發揮其生物學功能(圖2):miRNA海綿[9,14]、作為翻譯模板[15,28];調控基因轉錄[12-13];與RNA結合蛋白作用[1,9,16]。
3.1 miRNA海綿
由外顯子構成的ecRNA具有穩定的環狀結構,且大多數ecRNA包含有miRNA結合位點,因此,ecRNA可以作為高效的競爭性內源RNA,有效吸附miRNA從而調控miRNA的靶基因。小腦變性相關蛋白1反義轉錄物(Antisense to the cerebellar degeneration-related protein 1 transcript,CDR1as)是一種ecRNA,CDR1as長度約為1.5 kb,定位于細胞質中,主要在人和小鼠的腦中表達,含有超過60個能夠與miR-7結合的保守性位點,從而作為miR-7分子海綿影響miR-7靶基因表達,因此,也被稱為miR-7 的circRNA 海綿(Circular RNA sponge for miR-7,ciRS-7)[9,14,29]。環狀RNA——CDR1as和miR-7共同高表達于中腦區域,在斑馬魚胚胎發育過程中,過表達CDR1as使腦體積減少,發育受到阻礙,而外源注射miR-7可以恢復正常,進一步說明CDR1as至少部分通過與miR-7結合而發揮作用[14,29]。此外,性別決定基因(Sex-determining region Y,Sry)對決定哺乳動物性別具重要作用,Sry基因在睪丸中可以產生環狀轉錄本,SrycircRNA至少有16個與miR-138結合的保守性位點,可以作為miR-138海綿發揮作用[14]。最近,國內學者Q.Zheng等[30]使用高通量測序從6個正常組織和7個癌組織中找到超過27 000個circRNA,并發現,來源于HIPK3基因2號外顯子的circRNA——circHIPK3,它表達豐度高,可以通過18個潛在結合位點吸附9個抑制細胞增殖的miRNA(miR-124,miR-152, miR-193a, miR-29a, miR-29b, miR-338, miR-379,miR-584,miR-654),從而影響細胞增殖。但目前發現大多數circRNA僅含有少量miRNA結合位點,CDR1as和SrycirRNA可能是作為分子海綿的特例[31-32]。另外,Y.Li等[33]發現,來源于MHCC-LM3肝癌細胞的exosome中比細胞具有更高豐度的circRNA,MHCC-LM3肝癌細胞中與circRNA相關的miRNA豐度可能影響circRNA進入exosome。Y.Li等[33]還在人血清exosome中鑒定了超過1 000種circRNA,癌癥患者和健康個體的血清exosome中circRNA的種類和豐度有明顯差異,這些差異表達的circRNA可能作為未來癌癥檢測和治療的標志分子。
表1 不同類型circRNA的特征
Table 1 The characteristics of different types of circRNA
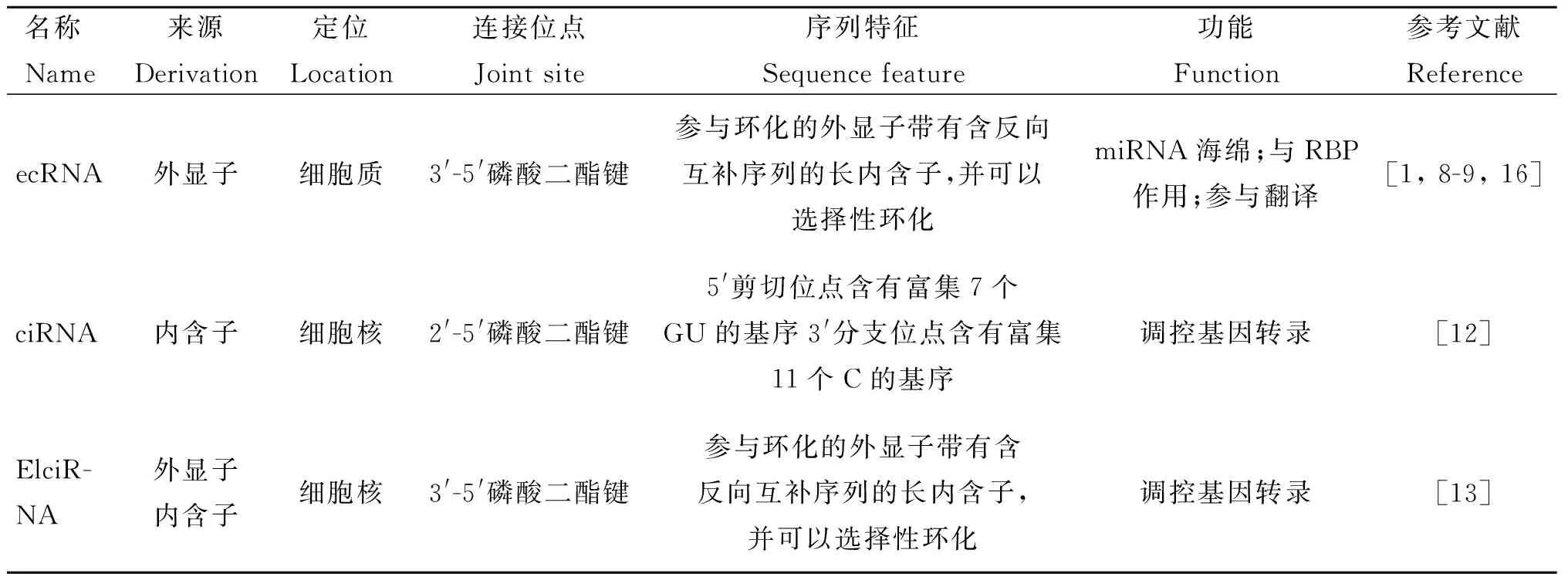
名稱Name來源Derivation定位Location連接位點Jointsite序列特征Sequencefeature功能Function參考文獻ReferenceecRNA外顯子細胞質3'-5'磷酸二酯鍵參與環化的外顯子帶有含反向互補序列的長內含子,并可以選擇性環化miRNA海綿;與RBP作用;參與翻譯[1,8-9,16]ciRNA內含子細胞核2'-5'磷酸二酯鍵5'剪切位點含有富集7個GU的基序3'分支位點含有富集11個C的基序調控基因轉錄[12]ElciR-NA外顯子內含子細胞核3'-5'磷酸二酯鍵參與環化的外顯子帶有含反向互補序列的長內含子,并可以選擇性環化調控基因轉錄[13]
ecRNA. 外顯子circRNA;ciRNA. 內含子circRNA;ElciRNA. 外顯子-內含子circRNA
ecRNA. Exon circRNA; ciRNA. Intron circRNA; ElciRNA. Exon-intron circRNA
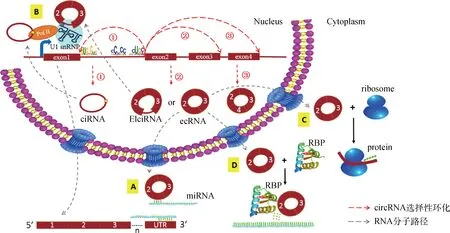
A.由外顯子形成的ecRNA可以在胞質中吸附miRNA,充當miRNA分子海綿;B. ciRNA或ElciRNA在胞核中與Pol II作用促進Parent gene轉錄;C.帶有內部核糖體進入位點IRES的circRNA可能在胞質中作為模板被翻譯成多肽或蛋白質;D. circRNA可以與RNA結合蛋白相互作用,作為分子腳手架為RNA結合蛋白質、RNA、DNA之間相互作用提供平臺。Pol II. RNA聚合酶Ⅱ;U1 snRNP. U1小核核糖核蛋白;RBP. RNA結合蛋白A. Exon-derived circRNA (ecRNA) can adsorb free miRNAs in the cytoplasm and acts as miRNA sponge; B. Intron-derived circRNA (ciRNA) or exon-intron circRNA (ElciRNA) can interact with the Pol II transcription complex at the promoters of parental genes to enhance gene expression in the nucleus; C. circRNA with internal ribosome entry site (IRES) as a template may be translated into a polypeptide or protein in the cytoplasm; D. As a molecular scaffolding, circRNA can interact with the RNA binding protein to provide a platform for interaction between RBP, RNA, DNA. Pol II. RNA polymerase II; U1 snRNP. U1 small nuclear ribonucleoproteins; RBP. RNA binding protein圖2 環狀RNA功能Fig.2 The biological function of circRNA
3.2 作為翻譯模板
通常情況下circRNA 是不可以被翻譯的,因此被定義為一類新的非編碼RNA[31]。而近年來研究發現一些circRNA包含有外顯子序列,可能被翻譯成蛋白質。最早報道的是δ肝炎病毒的單鏈環狀RNA基因組能夠在哺乳動物細胞中翻譯成病毒相關蛋白質,并在肝炎發生過程中發揮作用[34]。Y.Wang等[15]將內部核糖體進入位點(Internal ribozyme entry site,IRES)構建到綠色熒光蛋白(Green Fluorescent Protein ,GFP)的開放閱讀框(Open reading frame,ORF)前,然后經反向剪接形成circRNA,這種circRNA能夠翻譯成具有功能的GFP。此外,N.Abe等[35]發現,在大腸桿菌中,含有多個ORF的circRNA能被有效翻譯成蛋白質,這些circRNA沒有終止密碼子,且核苷酸數量為3的倍數。N.Abe等還發現在Hela細胞中circRNA也能夠被翻譯[36]。另有研究發現,一旦翻譯起始,circRNA的環狀結構使核糖體更易循環參與多肽延長過程[37]。由此可見,circRNA在某些特定條件下可以被翻譯成功能性的蛋白質。
3.3 調控基因轉錄
近期研究表明,circRNA能夠順式或反式(cis-/trans-)調控基因轉錄從而影響基因表達[38]。來源于內含子的circRNA (ciRNA/ElciRNA)主要定位于細胞核中,能夠與RNA聚合酶II相互作用促進自身編碼基因轉錄[12-13]。Y.Zhang等[12]首次在人細胞中發現了ciRNA,并找出了內含子環化依賴的motif,證實ci-ankrd52大量聚集于自身編碼基因的轉錄位點,通過與RNA聚合酶II作用,從而促進自身編碼基因轉錄。Z.Li等[13]用紫外交聯-免疫共沉淀(Cross-linking and immunoprecipitation , CLIP)RNA聚合酶II在Hela細胞中發現了111種circRNA,并鑒定了表達豐度TOP15的circRNA。后續研究發現ElciRNA(如:circEIF3J 和circPAIP2)能夠與U1互補配對,進而與U1小核核糖核蛋白(U1 small nuclear ribonucleoproteins, snRNP)相互作用以順式調控作用促進自身編碼基因轉錄。另外,有一部分ElciRNA在細胞核轉錄位點以外的其他區域富集,提示胞核中的ElciRNA可能發揮反式調控作用。
3.4 與RNA結合蛋白相互作用
一些線性非編碼RNA可以與RNA結合蛋白作用發揮生物學功能,因此提示circRNA也可能與RNA結合蛋白結合而發揮作用[39-40]。外顯子來源的circRNA可以穩定地與細胞內某些蛋白質分子特異性結合,作為腳手架(scaffolding)再通過互補序列與RNA或DNA結合,為RNA結合蛋白、RNA、DNA之間相互作用提供平臺,如ecRNA可以與AGO蛋白和RNA聚合酶II相互作用進而發揮miRNA海綿參與調控轉錄的作用[1,9,16]。另外,環化后的RNA分子具有與其線性轉錄本不同的三級結構,并產生新的蛋白結合位點,因此,circRNA能夠結合不同的蛋白分子,促進RNA結合蛋白、RNA、DNA之間相互作用從而發揮正常生理功能[1,9,16]。
4 circRNA的研究方法
4.1 分子生物學方法
CircRNA的環狀結構具有較高的穩定性,可以抵抗酶的消化作用,而線性RNA不具有此特征,因此可用于初步純化和鑒定circRNA[16]。用核酸外切酶RNase R、煙酸磷酸酶、5′端核酸外切酶等處理可以降解大部分線性RNA,而circRNA不被降解,然后用circRNA特定的背對背引物(Divergent primer)定量分析酶處理前后的樣品,可以鑒定或定量circRNA[16,41]。其次,circRNA不具有末端極性結構,在凝膠中比等長的線性RNA遷移速度慢,這種效應在強交聯凝膠中尤為明顯。而與來自同源基因的轉錄物相比,circRNA的核酸序列更少,在弱交聯凝膠中遷移速度更慢,因此可以通過Northern blot法鑒定circRNA[42]。另外,通過熒光原位雜交技術(Fluorescenceinsituhybridization, FISH)可以對circRNA進行亞細胞定位,利用siRNA或反義寡核苷酸(Antisense oligonucleotides,ASO)可以干擾circRNA表達以進行circRNA功能驗證[12-13]。
4.2 高通量測序法
傳統的分子生物學方法在circRNA功能驗證上具有重要作用,對于發現物種新的circRNA(尤其是豐度較低的)則不盡人意,而高通量測序技術的出現和生物信息學的發展則能很好地解決這一難題。circRNA是通過反向剪接產生的,早期RNA-seq技術的算法不能很好地分辨出反向剪接(Back-splicing)位點和相應的環狀結構。針對這一問題,研究人員在測序分析的策略和算法上做出了有效的改進,主要包括以下幾種:一是假定基因間存在不同形式的外顯子重排,構建可能形成circRNA的候選邊界序列組合,然后將其與測序數據進行比對[8];二是將測序數據通過不同的序列比對算法,直接與基因組序列進行匹配[1];三是通過設計多重剪接接頭序列,直接從cDNA序列中檢測circRNA分子的存在[43]。目前應用于circRNA分子研究算法有MapSplice[44]、CircSeq[1]、segemehl[43]等。另外,circRNA不具有poly(A)結構,傳統的oligo dT富集建庫法不能將其有效富集。使用ribo-zero kit去除rRNA,再利用RNase R去除線性RNA,這種建庫方法能夠有效富集circRNA[16,45]。
5 展望
隨著高通量測序技術和生物信息學快速發展,越來越多的circRNA已經被發現和鑒定,目前線蟲、小鼠、人等物種的circRNA研究信息收錄于circbase數據庫中,關于circRNA的研究方興未艾[45-46]。近年來研究表明circRNA與疾病發生密切相關,circRNA與疾病相關的miRNA相互作用從而調控某些疾病發生,例如:ciRS-7在人腦組織中豐富表達, ciRS-7含有多個串聯的miR-7結合位點,因此可以作為內源性的miRNA海綿吸附miR-7,在正常生理條件下ciRS-7-miR-7-miR-7的靶基因處于動態平衡,當這種穩態被打破時則可能誘發疾病[14,29]。另外,A.Bachmary-Heyda等[47]發現,在癌組織或癌細胞中,circRNA與其線性轉錄本的比值更低,即circRNA在癌細胞中豐度較正常相比更低,可能機制是:線性轉錄本在細胞中易達到合成與降解的平衡,而circRNA更加穩定,在正常增殖的細胞中circRNA不斷“累積”,而在癌細胞的無限增殖中circRNA被“稀釋”。同時,circRNA在體液(血液[33,48]、唾液[26]等)中表達豐度高且穩定性好,因此,這些特征表明circRNA可以作為未來癌癥等疾病檢測的新型標志物[49-50]。
目前有關circRNA的研究主要集中于疾病(尤其是癌癥),在農業動物上的研究還鮮有報道,T.Morten等[5]通過高通量測序研究豬妊娠期間(6個時間點)腦組織(5個分區)發育相關的circRNA,共鑒定4 634個circRNA,其中在妊娠中期皮質中circRNA表達豐度最高,且與其線性轉錄本相比呈現出明顯的差異表達,提示circRNA在豬胚胎腦發育方面具有重要調控作用。研究農業動物相關的circRNA,深度挖掘參與調控經濟性狀(脂肪沉積、肌肉發育、動物繁殖等)相關的circRNA,將成為未來動物遺傳育種研究的新方向。目前,circRNA命名尚未統一,有關circRNA的生物學功能有待進一步研究,circRNA參與疾病發生的詳細機制仍不清楚,因此,circRNA的研究還有很長的路要走。
[1] JECK W R, SORRENTINO J A, WANG K,et al. Circular RNAs are abundant, conserved, and associated with ALU repeats[J].RNA, 2013, 19(2): 141-157.
[2] SANGER H L, KLOTZ G, RIESNER D, et al. Viroids are single-stranded covalently closed circular RNA molecules existing as highly base-paired rod-like structures[J].ProcNatlAcadSciUSA, 1976, 73(11): 3852-3856.
[3] CAPEL B, SWAIN A, NICOLIS S, et al. Circular transcripts of the testis-determining gene Sry in adult mouse testis[J].Cell, 1993, 73(5): 1019-1030.
[4] ABDELMOHSEN K, PANDA A C, DE S, et al. Circular RNAs in monkey muscle: age-dependent changes[J].Aging(AlbanyNY), 2015, 7(11): 903-910.
[5] VEN? M T, HANSEN T B, VEN? S T,et al. Spatio-temporal regulation of circular RNA expression during porcine embryonic brain development[J].GenomeBiol, 2015, 16: 245.
[6] BURD C E, JECK W R, LIU Y,et al. Expression of linear and novel circular forms of an INK4/ARF-associated non-coding RNA correlates with atherosclerosis risk[J].PLoSGenet, 2010, 6(12):e1001233.
[7] COCQUERELLE C, MASCREZ B, HéTUIN D,et al. Mis-splicing yields circular RNA molecules[J].FASEBJ, 1993, 7(1): 155-160.
[8] SALZMAN J, CHEN R E, OLSEN M N, et al. Cell-type specific features of circular RNA expression[J].PLoSGenet, 2013, 9(9): e1003777.
[9] MEMCZAK S, JENS M, ELEFSINIOTI A, et al. Circular RNAs are a large class of animal RNAs with regulatory potency[J].Nature, 2013, 495(7441): 333-338.
[10] SALZMAN J, GAWAD C, WANG P L,et al. Circular RNAs are the predominant transcript isoform from hundreds of human genes in diverse cell types[J].PLoSOne, 2012, 7(2): e30733.
[11] ZHANG X O, WANG H B, ZHANG Y,et al. Complementary sequence-mediated exon circularization[J].Cell, 2014, 159(1): 134-147.
[12] ZHANG Y, ZHANG X O, CHEN T,et al. Circular intronic long noncoding RNAs[J].MolCell, 2013, 51(6): 792-806.
[13] LI Z, HUANG C, BAO C,et al. Exon-intron circular RNAs regulate transcription in the nucleus[J].NatStructMolBiol,2015, 22(3): 256-264.
[14] HANSEN T B, JENSEN T I, CLAUSEN B H,et al. Natural RNA circles function as efficient microRNA sponges[J].Nature, 2013, 495(7441): 384-388.
[15] WANG Y, WANG Z. Efficient backsplicing produces translatable circular mRNAs[J].RNA, 2015, 21(2): 172-179.
[16] JECK W R, SHARPLESS N E. Detecting and characterizing circular RNAs[J].NatBiotechnol, 2014, 32(5): 453-461.
[17] CRICK F. Split genes and RNA splicing[J].Science, 1979, 204(4390): 264-271.
[18] BLACK D L. Mechanisms of alternative pre-messenger RNA splicing[J].AnnuRevBiochem, 2003, 72: 291-336.
[19] VICENS Q, WESTHOF E. Biogenesis of circular RNAs[J].Cell, 2014, 159(1): 13-14.
[20] ASHWAL-FLUSS R, MEYER M, PAMUDURTI N R, et al. circRNA biogenesis competes with pre-mRNA splicing[J].MolCell, 2014, 56(1): 55-66.
[21] SHEN T, HAN M, WEI G,et al. An intriguing RNA species—perspectives of circularized RNA[J].ProteinCell, 2015, 6(12): 871-880.
[23] CONN S J, PILLMAN K A, TOUBIA J,et al. The RNA binding protein Quaking regulates formation of circRNAs[J].Cell, 2015, 160(6): 1125-1134.
[24] IVANOV A, MEMCZAK S, WYLER E,et al. Analysis of intron sequences reveals hallmarks of circular RNA biogenesis in animals[J].CellRep, 2015, 10(2): 170-177.
[25] SUZUKI H, TSUKAHARA T. A view of pre-mRNA splicing from RNase R resistant RNAs[J].IntJMolSci, 2014, 15(6): 9331-9342.
[26] BAHN J H, ZHANG Q, LI F,et al. The landscape of microRNA, Piwi-interacting RNA, and circular RNA in human saliva[J].ClinChem, 2015, 61(1): 221-230.
[27] CHEN L L, YANG L. Regulation of circRNA biogenesis[J].RNABiol, 2015, 12(4): 381-388.
[28] SURONO A, TAKESHIMA Y, WIBAWA T,et al. Circular dystrophin RNAs consisting of exons that were skipped by alternative splicing[J].HumMolGenet, 1999, 8(3): 493-500.
[29] HANSEN T B, KJEMS J, DAMGAARD C K. Circular RNA and miR-7 in cancer[J].CancerRes, 2013, 73(18): 5609-5612.
[30] ZHENG Q, BAO C, GUO W,et al. Circular RNA profiling reveals an abundant circHIPK3 that regulates cell growth by sponging multiple miRNAs[J].NatCommun, 2016, 7:11215.
[31] GUO J U, AGARWAL V, GUO H,et al. Expanded identification and characterization of mammalian circular RNAs[J].GenomeBiol, 2014, 15(7): 409.
[32] QU S, YANG X, LI X,et al. Circular RNA: a new star of noncoding RNAs[J].CancerLett, 2015, 365(2): 141-148.
[33] LI Y, ZHENG Q, BAO C,et al. Circular RNA is enriched and stable in exosomes: a promising biomarker for cancer diagnosis[J].CellRes, 2015, 25(8): 981-984.
[34] KOS A, DIJKEMA R, ARNBERG A C,et al. The hepatitis delta (δ) virus possesses a circular RNA[J].Nature, 1985, 323(6088): 558-560.
[35] ABE N, HIROSHIMA M, MARUYAMA H,et al. Rolling circle amplification in a prokaryotic translation system using small circular RNA[J].AngewChemIntEdEngl, 2013, 52(27): 7004-7008.
[36] ABE N, MATSUMOTO K, NISHIHARA M,et al. Rolling circle translation of circular RNA in living human cells[J].SciRep, 2015, 5: 16435.
[37] PERRIMAN R, ARES M Jr. Circular mRNA can direct translation of extremely long repeating-sequence proteinsinvivo[J].RNA, 1998, 4(9): 1047-1054.
[38] CHEN L L. The biogenesis and emerging roles of circular RNAs[J].NatRevMolCellBiol, 2016, 17(4):205-211.
[39] ROMEO T. Global regulation by the small RNA-binding protein CsrA and the non-coding RNA molecule CsrB[J].MolMicrobiol, 1998, 29(6): 1321-1330.
[40] YIN Q F, YANG L, ZHANG Y,et al. Long noncoding RNAs with snoRNA ends[J].MolCell, 2012, 48(2): 219-230.
[41] SUZUKI H, ZUO Y, WANG J,et al. Characterization of RNase R-digested cellular RNA source that consists of lariat and circular RNAs from pre-mRNA splicing[J].NucleicAcidsRes, 2006, 34(8): e63.
[42] TABAK H F, VAN DER HORST G, SMIT J,et al. Discrimination between RNA circles, interlocked RNA circles and lariats using two-dimensional polyacrylamide gel electrophoresis[J].NucleicAcidsRes, 1988, 16(14): 6597-6605.
[43] HOFFMANN S, OTTO C, DOOSE G,et al. A multi-split mapping algorithm for circular RNA, splicing, trans-splicing and fusion detection[J].GenomeBiol, 2014, 15(2): R34.
[44] WANG K, SINGH D, ZENG Z,et al. MapSplice: accurate mapping of RNA-seq reads for splice junction discovery[J].NucleicAcidsRes, 2010, 38(18): e178.
[45] EBBESEN K K, KJEMS J, HANSEN T B. Circular RNAs: Identification, biogenesis and function[J].BiochimBiophysActa, 2016, 1859(1):163-168.
[46] http://www.circbase.org/.
[47] BACHMAYR-HEYDA A, REINER A T, AUER K,et al. Correlation of circular RNA abundance with proliferation-exemplified with colorectal and ovarian cancer, idiopathic lung fibrosis, and normal human tissues[J].SciRep, 2015, 5:8057.
[48] MEMCZAK S, PAPAVASILEIOU P, PETERS O,et al. Identification and characterization of circular RNAs as a new class of putative biomarkers in human blood[J].PLoSOne, 2015, 10(10): e0141214.
[49] LI J, YANG J, ZHOU P,et al. Circular RNAs in cancer: novel insights into origins, properties, functions and implications[J].AmJCancerRes, 2015, 5(2): 472-480.
[50] LI P, CHEN S, CHEN H,et al. Using circular RNA as a novel type of biomarker in the screening of gastric cancer[J].ClinChimActa, 2015, 444: 132-136.
(編輯 郭云雁)
The Research Advance of Circular RNA
ZHANG Jin-wei, LONG Ke-ren, WANG Xun, LI Ming-zhou, MA Ji-deng*
(InstituteofAnimalGeneticsandBreeding,CollegeofAnimalScienceandTechnology,SichuanAgriculturalUniversity,Chengdu611130,China)
Circular RNAs (circRNAs) are a class of endogenous noncoding RNA, unlike linear RNAs, which form a covalently closed continuous loop by back-splicing. circRNA has no the 5′ end cap and 3′ end poly (A) tail structure, and exist steadily in many eukaryotic cells. Due to the limitation of traditional biological methods for circRNA detection in both species and abundance, circRNA is traditionally considered as the products of abnormal RNA splicing. With the development of bioinformatics and the innovation of high-throughput sequencing technology, the thousands of endogenous circRNA in mammalian cells were discovered, some of which are highly expressed in a tissue- and development stage-specific manner and display conservation across species, suggesting their potential regulating function on the gene expression. In combination with the characteristics, formation mechanism and biological function of circRNA, we summarize the research advance of circRNA in recent years, aiming to provide fundamental information for studying eukaryotic gene expression regulation and disease detection, and prospect circRNAs′ research in animal molecular breeding.
circRNA; characteristics; biogenesis; biological function
10.11843/j.issn.0366-6964.2016.11.001
2016-03-30
國家高技術研究發展計劃(863計劃)項目(2013AA102502);國家自然科學基金(31472081;31522055);四川省教育廳重點項目(16ZA0025;15ZA0008;15ZA0003);四川省青年科技創新研究團隊(2015TD0012)
張進威(1993-),男,四川儀隴人,碩士生,主要從事豬遺傳育種研究,E-mail:JinweiZhang50@163.com
*通信作者:馬繼登,助理研究員,主要從事豬遺傳育種研究,E-mail:jideng_ma@sina.com
S813.2
A
0366-6964(2016)11-2151-08

