合成生物學開關在代謝工程中的應用
龐慶霄 梁泉峰 祁慶生(山東大學生命科學學院 微生物技術國家重點實驗室,濟南 250100)
合成生物學開關在代謝工程中的應用
龐慶霄 梁泉峰 祁慶生
(山東大學生命科學學院 微生物技術國家重點實驗室,濟南 250100)
在代謝工程研究領域中合成生物學開關主要用于調控基因的表達。傳統的代謝工程改造主要通過敲除和過表達來改變特定基因的表達量。但基因敲除通常會導致菌體生長的下降。因此,我們需要適時的關閉和激活特定基因的表達。合成生物學開關就是解決這一問題的關鍵工具。目前,在代謝工程中常用的合成生物學開關有光控開關、溫度誘導開關、撥動開關和核糖開關。其中,撥動開關和核糖開關在動態調節基因表達上擁有更大優勢。介紹了代謝工程中常用的幾種合成生物學開關,以及它們在代謝工程的應用。
合成生物學;基因表達調控;代謝工程;合成生物學開關;動態調控
代謝工程的主要目標之一就是通過基因調控增加化合物產量。隨著DNA分子技術的進步,我們現在能夠在基因水平對代謝網絡進行操作。通過針對途徑中的關鍵酶的編碼基因,進行敲除、擴增和下調[1,2]。基因敲除方法主要優勢是增加底物向目標化合物的轉化率。然而基因敲除常常導致生長率的下降,從而致使產量的減少。權衡產量和產率使沖突最小化,這樣的設計和操作的優化是至關重要的[3]。在抵消基因敲除對產量的影響上[4],基因表達動態調節比靜態的方法在最大化產物濃度上更有利。對于基因敲除影響產量這一問題,動態調控的解決方法就是保持動態的高度表達直到獲得足夠的生物量,然后關掉這些基因。合成生物學開關就是實現動態基因表達控制系統的重要元件,它能感應相關條件的變化,在適當的時間控制基因表達的“開”或“關”。目前,在代謝工程中常用的合成生物學開關有光控開關、溫度誘導開關、撥動開關和核糖開關。
1 光控開關
光是一個高度靈活的外界刺激,它在不改變代謝條件的前提下,實現時間空間上控制基因的精確表達。因此,在功能基因組學、系統生物學和生物技術領域光介導的基因表達調控研究進展迅速。光誘導的生物反應主要通過使用光封閉分子,或者使用帶有光敏感結構域(發色團)的光感應器來實現光控開關的觸發。光封閉化合物和有發色團分子吸收一定波長的光,然后起始一個光觸發的反應。光誘導反應導致封閉基團不可逆釋放或者使光感受器處于可逆的激活態。因此這兩種機制都用于光依賴的基因表達控制[5]。
然而封閉化合物有一些限制,光信號到達特定細胞的過程和這種機制在大范圍不同的生物體的應用都有一定障礙,而且封閉化合物光分解后不可逆轉。因此,基因編碼的人工光受體蛋白的發展有助于規避這些系統固有的限制。相比較而言光敏感調控系統被用于代謝工程中進行重組開關的構建[6]。人工光受體蛋白在光照射下激活其激酶活性,從而磷酸化某些啟動子轉錄所需的關鍵轉錄因子,轉錄因子被激活起始轉錄(圖1)。Binder等[7]將光控開關用于谷氨酸棒狀桿菌中朱欒倍半萜的生產,獲得了目前為止朱欒倍半萜最高產量和滴定量。朱欒倍半萜對菌體生長有嚴重影響,而且Binder等使用的谷氨酸棒狀桿菌細胞膜對IPTG透性較差。他們將帶有封閉分子的IPTG加LAC操縱子的光控開關用于調節朱欒倍半萜合酶的表達,在生長期抑制朱欒倍半萜的合成,當菌體達到一定密度后,用光誘導朱欒倍半萜合酶基因表達。從而在不影響菌體生長情況下,獲得了高產量的朱欒倍半萜。
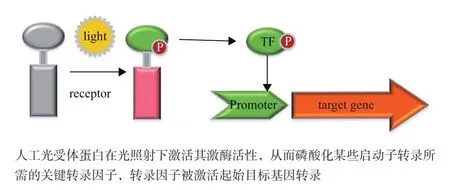
圖1 光介導的基因表達的控制
2 溫度誘導開關
溫度作為代謝過程的重要環境條件很早就已經應用在代謝調控中。在大腸桿菌中含有λ噬菌體的PL啟動子的質粒常用來表達外源基因。通過引入λ噬菌體的CI抑制子,PL啟動子下的基因表達可以被溫度控制[8]。這種溫度誘導需要使用一個溫度敏感的抑制子,在32℃時抑制子抑制基因的表達,在42℃時誘導基因表達。Gu等[9]發現了另一種溫度控制基因表達機制。這種機制通過溫度控制σE因子是否與LuxR啟動子(Plux)結合來控制基因表達。LuxR是群體感應的重要因子,將群體感應原理與溫度控制開關結合可以構建出“與”門邏輯通路。當溫度達到37℃并且Plux啟動子被群體信號分子AHL所誘導時,Plux啟動子下游的基因才能表達。溫度誘導在大規模應用上來說可能是最方便的方法,溫度可以通過加熱和冷卻來迅速切換。1990年,Shi等[10]將溫度誘導開關應用于聚-3-羥基丁酸(PHB)的生產,在34℃下培養菌體10 h促進菌體生長,然后在40℃誘導PHB合酶表達,5 h后溫度降至37℃緩解熱休克,他們使用這種方法大大提高了PHB的產量和產率。2016年,Zhang等[11]將溫度誘導開關用于α-酮丁酸的生產,在35℃保證細胞生長,然后切換到40℃誘導蘇氨酸脫氨酶表達,使α-酮丁酸的產量在發酵26 h后達40.8 g/L,達到了工業生產要求的產量。
3 撥動開關
撥動開關是一大類代謝調控開關,它們的共同特點是能夠隨環境(誘導劑或其他感應底物的有無)的改變來控制基因表達的開關。這里簡單介紹幾種代謝工程中使用的撥動開關。2000年,Gardner等[12]描述了一種構建在質粒上的撥動開關,它可以在兩個啟動子表達之間來回撥動來應答外部信號。開關由兩個抑制子和兩個保守的啟動子組成(圖2)。每個啟動子都會被另一個啟動子表達的抑制子所抑制[13]。這樣通過控制誘導劑的有無來控制開關在“開”和“關”之間切換,從而控制下游基因的表達。
隨著對微生物群體感應機制研究的深入,將群體感應機制與撥動開關結合形成了能夠感應菌體密度的動態調節元件。2004年,Kobayashi等[14]設計了一個由PL*啟動子、Ptrc啟動子、lacI基因和λcl基因組成的撥動開關。2008年,Anesiadis等[15]將群體感應應用到代謝工程中,在Kobayashi等的開關的基礎上設計出了感應群體密度的撥動開關。細胞間的交流是通過一群信號分子完成的,它們是自誘導物。高絲氨酸內酯(AHL)就是一個這樣的自誘導物,它的濃度跟環境中的細胞數量成一定比例。將AHL的表達基因設計到撥動開關中,就構建成了感應群體密度的撥動開關(圖3)。菌體密度決定AHl濃度,AHL濃度決定LacI表達,進而決定目的基因表達[16]。這實現了基因表達的動態調節,解決了因為基因敲除而影響細胞生長,進而影響產量的問題。

圖2 雙穩態撥動開關
在代謝工程生產目標產物時往往使用阻斷競爭途徑和過表達瓶頸酶等手段來增加產量,這種方法往往使中間體和酶積累較高水平,高水平的中間體和酶對菌體產生毒性,抑制細胞生長和降低產物產量。Liu 等在對大腸桿菌進行改造生產脂肪酸時發現改造后的菌株中乙酰輔酶A羧化酶積累很高。乙酰輔酶A羧化酶積累導致菌體生長的下降。Liu等[17]設計了一種可以感應乙酰輔酶A羧化酶的產物丙二酸單酰輔酶A濃度的撥動開關。該開關可以在丙二酸單酰輔酶A濃度低時提高乙酰輔酶A羧化酶的表達,在丙二酸單酰輔酶A濃度較高時降低乙酰輔酶A羧化酶的表達。這樣既可以增加產物積累,又能降低乙酰輔酶A羧化酶積累對細胞的毒性。
Tsuruno等[18]將撥動開關應用于生產3-羥基丙酸,獲得了比傳統敲除方法更高的產量和產率。本實驗室在對莽草酸生產的研究中,為了解決敲除莽草酸下游代謝基因莽草酸激酶而使菌株變成芳香族化合物營養缺陷型的問題,設計了一種能夠調節莽草酸激酶基因表達量的開關,增加了莽草酸的產量。這種方法與直接敲除莽草酸激酶相比避免了外源添加芳香族化合物[19],從而降低了成本。
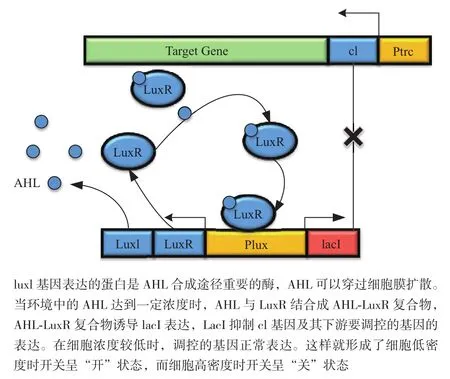
圖3 群體感應撥動開關
4 核糖開關
近些年來,一種新的不需要蛋白轉錄因子參與而直接由核酸感受胞內信號分子或胞外環境變化的調控模式得到越來越多關注并被應用于一些研究領域,這種調控模式被稱為核糖開關(riboswitch)。核糖開關結構首先由Winkler等[20]于2002年在大腸桿菌中發現并命名。迄今為止發現的核糖開關有20多種,包括Ado-cbl核糖開關、SAM核糖開關、TPP核糖開關、FMN核糖開關、賴氨酸核糖開關及glmS核糖開關等廣泛應用的核糖開關(表1)。因為核糖開關是由適體結構域和表達結構域的組合,一個結構域更改并不影響另一個區域功能的發揮。人們可以在不改變調控法則的情況下更改核糖開關配體結合的特異性。于是通過盡可能的綁定新的配體進而篩選出高能力的基因調控適體。核糖開關可實時感應配體的濃度,并在配體濃度低時表現出“開”的狀態,基因可以正常表達;在配體濃度高時表現出“關”的狀態,抑制基因表達。核糖開關通過感應底物濃度高低來實現對基因表達的動態調節。
核糖開關這種調節元件已經被用于代謝工程生產。Zhou和Zeng[21]將賴氨酸核糖開關應用于賴氨酸的生產。三羧酸循環(TCA)是氨基酸合成前體的重要來源,檸檬酸合酶是TCA循環的第一個酶(圖4)。有實驗證明檸檬酸合酶活性降低有助于賴氨酸產量增加。但是檸檬酸合酶是細菌能量代謝的重要酶,直接敲除將導致菌體生長嚴重下降。Wang等[22]用賴氨酸核糖開關替換掉檸檬酸合酶編碼基因gltA前端啟動子和起始密碼子之間的序列。核糖開關跟隨gltA一起轉錄,在生產菌株產生一定的賴氨酸時,賴氨酸與核糖開關結合,下調下游基因gltA的表達。這樣既避免了基因敲除導致的菌體生長下降又提高了賴氨酸的產量。本實驗室在對大腸桿菌生產賴氨酸的研究中,將雙重篩選基因tetA與核糖開關相結合組成核糖開關篩選系統,實現了賴氨酸生產菌株的高通量篩選,提高了賴氨酸的產量。
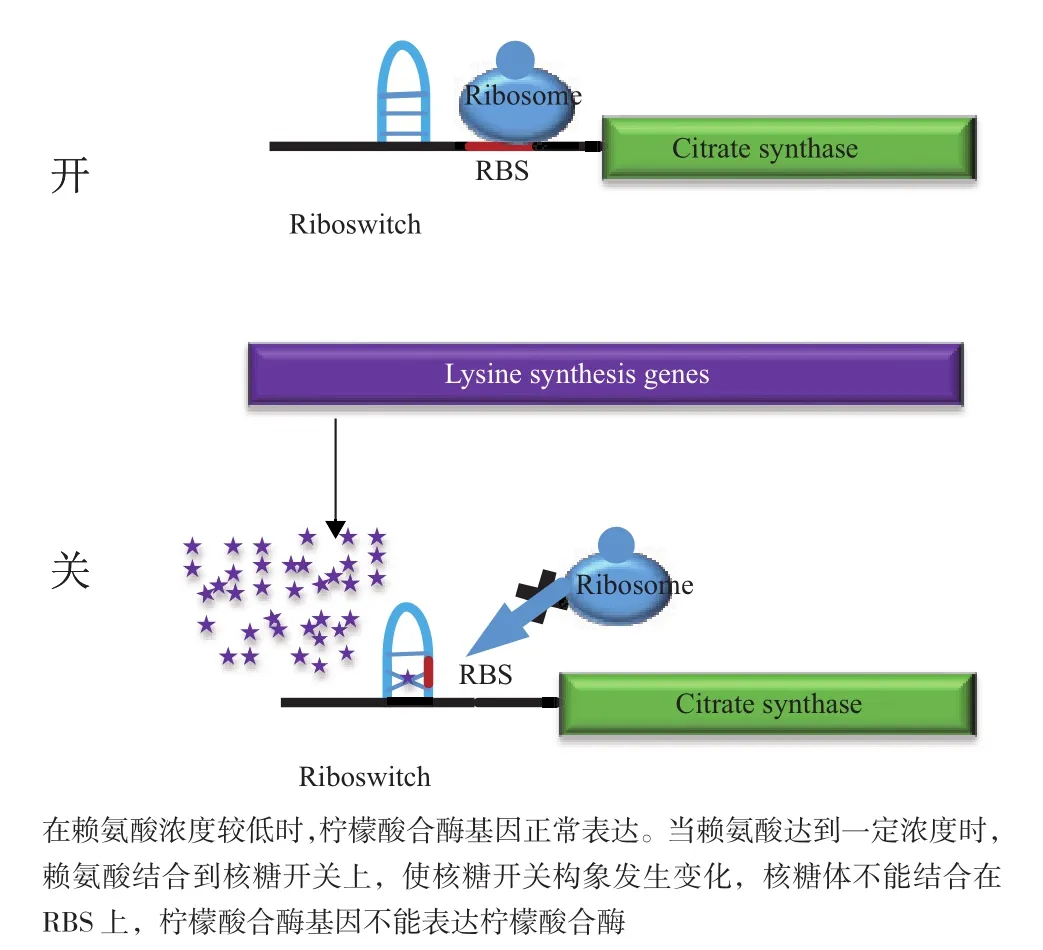
圖4 賴氨酸合成代謝途徑
5 展望
隨著微生物代謝工程產物不斷的研發并應用于生產中,對于代謝工程的各種工具的研究也不斷深入。由于基因敲除導致生產菌株生長率下降進而影響產物產量,研究的方向也開始轉移到了動態調控相關基因的表達。近年來合成生物學開關的研究已成為熱點,特別是代謝工程中開關的應用相關研究具有獨特的吸引力,逐步深入并取得了很大進展。預計合成生物學的開關將應用于代謝工程各種產品的生產,將極大推動相關領域的發展。

表1 常見的核糖開關
[1]Kim JY, Cha HJ. Down-regulation of acetate pathway through antisense strategy in Escherichia coli:improved foreign protein production[J]. Biotechnology and Bioengineering, 2003, 83(7):841-853.
[2]Herring CD, Glasner JD, Blattner F. Gene replacement without selection:regulated suppression of amber mutations in Escherichia coli[J]. Gene, 2003, 311:153-163.
[3]Schugerl K, Hubbuch J. Integrated bioprocesses[J]. Current Opinion in Microbiology, 2005, 8(3):294-300.
[4]Gadkar KG, Doyle Iii FJ, Edwards JS, et al. Estimating optimal profiles of genetic alterations using constraint-based models[J]. Biotechnology and Bioengineering, 2005, 89(2):243-251.
[5]Drepper T, Krauss U, Meyer zu Berstenhorst S, et al. Lights on and action! Controlling microbial gene expression by light[J]. Appl Microbiol Biotechnol, 2011, 90(1):23-40.
[6]Deiters A. Principles and applications of the photochemical control of cellular processes[J]. Chembiochem:a European Journal of Chemical Biology, 2010, 11(1):47-53.
[7]Binder D, Frohwitter J, Mahr R, et al. Light-controlled cell factoriesemploying photocaged isopropyl-β-d-thiogalactopyranoside for lightmediated optimization of lac promoter-based gene expression and(+)-valencene biosynthesis in Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2016, 82(20):6141-6149.
[8]Lowman HB, Bina M. Temperature-mediated regulation and downstream inducible selection for controlling gene expression from the bacteriophage lambda pL promoter[J]. Gene, 1990, 96(1):133-136.
[9] Gu D, Guo M, Yang M, et al. A σE-mediated temperature gauge controls a switch from LuxR-mediated virulence gene expression to thermal stress adaptation in Vibrio alginolyticus[J]. PLoS Pathogens, 2016, 12(6):e1005645.
[10] Shi H, Kyuwa K, Takasu M, et al. Temperature-induced expression of phb genes in Escherichia coli and the effect of temperature patterns on the production of poly-3-hydroxybutyrate[J]. Journal of Bioscience and Bioengineering, 2001, 91(1):21-26.
[11]Zhang C, Qi J, Li Y, et al. Production of α-ketobutyrate using engineered Escherichia coli via temperature shift[J]. Biotechnology and Bioengineering, 2016, 113(9):2054-2059.
[12]Gardner TS, Cantor CR, Collins JJ. Construction of a genetic toggle switch in Escherichia coli[J]. Nature, 2000, 403(6767):339-342.
[13]Judd EM, Laub MT, McAdams HH. Toggles and oscillators:new genetic circuit designs[J]. McAdams, BioEssays, 2000, 22(6):507-509.
[14]Kobayashi H, Kaern M, Araki M, et al. Programmable cells:interfacing natural and engineered gene networks[J]. Proc Natl Acad Sci U S A, 2004, 101(22):8414-8419.
[15]Anesiadis N, Cluett WR, Mahadevan R. Dynamic metabolic engineering for increasing bioprocess productivity[J]. Mahadevan, Metab Eng, 2008, 10(5):255-266.
[16]Anesiadis N, Kobayashi H, Cluett WR, Mahadevan R. Analysis and design of a genetic circuit for dynamic metabolic engineering[J]. ACS Synthetic Biology, 2013, 2(8):442-452.
[17]Liu D, Xiao X, Evans BS, Zhang F. Negative feedback regulation of fatty acid production based on a malonyl-CoA sensoractuator[J]. ACS Synthetic Biology, 2015, 4(2):132-140.
[18]Tsuruno K, Honjo H, Hanai T. Enhancement of 3-hydroxypropionic acid production from glycerol by using a metabolic toggle switch[J]. Microbial Cell Factories, 2015, 14:155.
[19]Gu P, Su T, Wang Q, et al. Tunable switch mediated shikimate biosynthesis in an engineered non-auxotrophic Escherichia coli[J]. Scientific Reports, 2016, 6:29745.
[20]Winkler W, Nahvi A, Breaker RR. Thiamine derivatives bind messenger RNAs directly to regulate bacterial gene expression[J]. Nature, 2002, 419(6910):952-956.
[21]Zhou LB, Zeng AP. Exploring lysine riboswitch for metabolic flux control and improvement of L-lysine synthesis in Corynebacterium glutamicum[J]. ACS Synthetic Biology, 2015, 4(6):729-734.
[22]Wang JM, Gao D, Yu X, et al. Evolution of a chimeric aspartate kinase for L-lysine production using a synthetic RNA device[J]. Appl Microbiol Biotechnol, 2015, 99(20):8527-8536.
[23]Li C, Lv D, Zhang L, et al. Approach to the unfolding and folding dynamics of add A-riboswitch upon adenine dissociation using a coarse-grained elastic network model[J]. The Journal of Chemical Physics, 2016, 145(1):014104.
[24] Howe JA, Xiao L, Fischmann TO, et al. Atomic resolution mechanistic studies of ribocil:A highly selective unnatural ligand mimic of the E. coli FMN riboswitch[J]. RNA Biol, 2016, 13(10):946-954.
[25]Esquiaqui JM, Sherman EM, Ye JD, Fanucci GE. Conformational flexibility and dynamics of the internal loop and helical regions of the kink-turn motif in the glycine riboswitch by site-directed spinlabeling[J]. Biochemistry, 2016, 55(31):4295-4305.
[26]Yu Y, Zhu X, Shen Y, et al. Enhancing the vitamin B12production and growth of Propionibacterium freudenreichii in tofu wastewater via a light-induced vitamin B12riboswitch[J]. Applied Microbiology and Biotechnology, 2015, 99(24):10481-10488.
[27] Wang J, Gao D, Yu X, et al. Evolution of a chimeric aspartate kinase for L-lysine production using a synthetic RNA device[J]. Applied Microbiology and Biotechnology, 2015, 99(20):8527-8536.
[28]Su Y, Hickey SF, Keyser SG, Hammond MC. In vitro and in vivo enzyme activity screening via RNA-based fluorescent biosensors for S-adenosyl-l-homocysteine(SAH)[J]. Journal of the AmericanChemical Society, 2016, 138(22):7040-7047.
[29]Lussier A, Bastet L, Chauvier A, Lafontaine DA. A kissing loop is important for btuB riboswitch ligand sensing and regulatory control[J]. J Biol Chem, 2015, 290(44):26739-26751.
[30]Suresh G, Srinivasan H, Nanda S, Priyakumar UD. Ligandinduced stabilization of a Duplex-like architecture is crucial for the switching mechanism of the SAM-III riboswitch[J]. Biochemistry, 2016, 55(24):3349-3360.
[31]Polaski JT, Holmstrom ED, Nesbitt DJ, Batey RT. Mechanistic insights into cofactor-dependent coupling of RNA folding and mRNA transcription/translation by a cobalamin riboswitch[J]. Cell Reports, 2016, 15(5):1100-1110.
[32] Trausch JJ, Marcano-Velazquez JG, Matyjasik MM, Batey RT. Metal ion-mediated nucleobase recognition by the ZTP riboswitch[J]. Chemistry & Biology, 2015, 22(7):829-837.
[33]Aytenfisu AH, Liberman JA, Wedekind JE, Mathews DH. Molecular mechanism for preQ1-II riboswitch function revealed by molecular dynamics[J]. RNA, 2015, 21(11):1898-1907.
[34]Ren A, Xue Y, Peselis A, et al. Structural and dynamic basis for low-affinity, high-selectivity binding of L-glutamine by the glutamine riboswitch[J]. Cell Reports, 2015, 13(9):1800-1813.
[35]Zhang J, Ferré-DAmaré AR. Trying on tRNA for size:RNase P and the T-box riboswitch as molecular rulers[J]. Biomolecules 2016, 6(2):E18.
(責任編輯 馬鑫)
Application of Switch for Synthetic Biology in Metabolic Engineering
PANG Qing-xiao LIANG Quan-feng QI Qing-sheng
(State Key Laboratory of Microbiology,College of Life Sciences,Shandong University,Ji’nan 250100)
Translational switches for synthetic biology are mainly used to regulate gene expression in the field of metabolic engineering research. Traditional metabolic engineering regulates the expression of specific genes by using knockout and overexpression. However,the knockout of genes usually leads to the decrease in growth,therefore,we need to close and activate the expression of specific gene at proper time,and translational switche for synthetic biology is the key tool to solve such issue. At present,light-controlled switch,temperatureinduced switch,toggle switch,and riboswitch are commonly used in metabolic engineering,of which the toggle switch and riboswitch present a great advantage in the dynamic regulation of gene expression. In this paper,several kinds of translational switches for synthetic biology and their applications in metabolic engineering are reviewed.
synthetic biology;gene expression regulation;metabolic engineering;switch for synthesis biology;dynamic regulation
10.13560/j.cnki.biotech.bull.1985.2017.01.006
2016-11-02
國家“973”項目子課題(2012CB725202),山東省科技發展計劃項目(2015GSF121042)
龐慶霄,男,碩士,研究方向:代謝工程及合成生物學;E-mail:lnupqx@163.com
梁泉峰,男,博士,副教授,研究方向:代謝工程及合成生物學;E-mail:liangquanfeng@sdu.edu.cn

