釀酒酵母人工雜合啟動子與天然啟動子活性比較
唐瑞琪熊亮白鳳武,趙心清(. 上海交通大學(xué)生命科學(xué)技術(shù)學(xué)院,上海 0040;. 大連理工大學(xué)生命科學(xué)與技術(shù)學(xué)院,大連 603)
釀酒酵母人工雜合啟動子與天然啟動子活性比較
唐瑞琪1熊亮2白鳳武1,2趙心清1
(1. 上海交通大學(xué)生命科學(xué)技術(shù)學(xué)院,上海 200240;2. 大連理工大學(xué)生命科學(xué)與技術(shù)學(xué)院,大連 116023)
系統(tǒng)地比較了人工啟動子PaTEF1與天然啟動子PTEF1和PTDH3在不同條件下的活性差別,結(jié)果表明,人工啟動子PaTEF1的活性并不是在任何條件下都高于天然啟動子的活性,而與宿主、培養(yǎng)基以及細(xì)胞生長階段有關(guān)。在3個宿主背景中,BY4741中PaTEF1的活性最高,而LX03中最低。在YPD100中,人工啟動子PaTEF1活性分別為天然啟動子PTEF1和PTDH3活性的1.4-4.6倍和0.9-2.0倍;而在YPE(5%和7%)中,PaTEF1活性與PTEF1和PTDH3活性之比在0.7-1.3以及0.8-1.3之間。在YPE中培養(yǎng)時,PaTEF1的活性為在YPD100中培養(yǎng)時的1.7-2.0倍,PTEF1和PTDH3的活性在YPE中為YPD100中培養(yǎng)時的2.7-7.1倍和1.3-3.4倍,啟動子在YPE中的活性較YPD100中更高,但人工啟動子的活性變化較天然啟動子更小。此外,在不同遺傳背景的菌株中,啟動子活性從對數(shù)期早期到對數(shù)期中期和從對數(shù)期中期到穩(wěn)定期的變化趨勢不同。
釀酒酵母;人工雜合啟動子;TEF1啟動子;宿主菌株;環(huán)境條件
釀酒酵母是目前最常用的生產(chǎn)生物燃料和生物基化學(xué)品的底盤細(xì)胞之一。作為重要的細(xì)胞工廠,對釀酒酵母細(xì)胞中代謝通路進(jìn)行精細(xì)調(diào)節(jié)對于提高生物產(chǎn)品的產(chǎn)量、產(chǎn)率和生產(chǎn)強度至關(guān)重要[1]。啟動子作為轉(zhuǎn)錄調(diào)控以及合成生物學(xué)的重要元件,被廣泛應(yīng)用于基因表達(dá)的研究和菌株構(gòu)建中。天然的啟動子主要可以分為組成型啟動子和誘導(dǎo)型啟動子兩類,前者能維持恒定的基因轉(zhuǎn)錄水平,而后者的表達(dá)則依賴于特殊誘導(dǎo)劑的添加[2]。研究者們鑒定了許多不同的啟動子,包括天然啟動子和啟動子的突變體[3,4]。研究表明,啟動子中的元件可以作為模塊進(jìn)行合成生物學(xué)改造來調(diào)節(jié)啟動子的表達(dá)強度,通過將不同的上游增強元件(UASCLB和UASCIT)與不同的天然啟動子核心(PGPD、PCYC、PTEF和PLEUM)串聯(lián),得到的雜合啟動子的活性是相應(yīng)的天然啟動子活性的1.63-10.02倍[5]。作為一種新的遺傳操作元件,人工啟動子的高強度和穩(wěn)定性對于精細(xì)調(diào)節(jié)細(xì)胞中關(guān)鍵途徑的表達(dá)具有重要意義。
目前發(fā)表的大部分研究結(jié)果傾向于創(chuàng)造更高的啟動子多樣性,然而,研究個別啟動子在不同條件下的啟動活性同樣重要。啟動子的活性會受到多種環(huán)境因素的影響,例如,釀酒酵母多個啟動子在不同碳源(葡萄糖、蔗糖、乳糖和乙醇)中的活性具有顯著差異[6]。當(dāng)細(xì)胞處在不同的生長時期時,啟動子的活性也略有不同。此外,啟動子在碳源轉(zhuǎn)換期(diauxic shift)的活性也存在明顯差異[6]。基因的最優(yōu)表達(dá)水平在不同宿主菌株中也不盡相同,并受到宿主遺傳背景的影響[7]。
葡萄糖是細(xì)胞工廠生產(chǎn)目標(biāo)產(chǎn)物時最常見的碳源,隨著發(fā)酵的進(jìn)行,當(dāng)葡萄糖耗盡之后,細(xì)胞轉(zhuǎn)換為利用培養(yǎng)基中其他碳源的模式。乙醇既是釀酒酵母乙醇發(fā)酵中的主要代謝產(chǎn)物,在發(fā)酵后期乙醇也可能被當(dāng)做支持菌株生長的碳源。在某些高附加值產(chǎn)品的生產(chǎn)過程中,乙醇也被用作生產(chǎn)碳源,例如,以乙醇作為碳源有利于青蒿素的生產(chǎn)[8]。高濃度乙醇也對宿主菌株產(chǎn)生明顯的抑制作用[9],因此,研究不同濃度乙醇存在條件下啟動子的活性具有重要參考價值。
雖然目前已經(jīng)有不少關(guān)于啟動子在不同碳源和不同細(xì)胞生長時期的研究,但是大部分研究只對單一宿主中或單一條件下的不用啟動子活性進(jìn)行了比較,沒有同時比較不同宿主、不同培養(yǎng)基以及不同細(xì)胞生長階段的啟動子活性的研究。此外,現(xiàn)有對人工啟動子和天然啟動子活性比較的研究還非常少。本研究選取了由3個串聯(lián)的上游增強元件UASCLB和TEF1啟動子核心組裝而成的人工啟動子P[10]aTEF1與天然啟動子進(jìn)行了比較,研究人工啟動子PaTEF1與天然啟動子PTEF1以及PTDH3在不同宿主及同一宿主不同細(xì)胞生長階段或者不同培養(yǎng)基條件下的活性差別,為進(jìn)一步應(yīng)用釀酒酵母人工啟動子進(jìn)行合成生物學(xué)研究以及人工啟動子的生物技術(shù)應(yīng)用和進(jìn)一步改造提供參考。
1 材料與方法
1.1 材料
1.1.1 菌株、質(zhì)粒和引物 本研究所用到的菌株、質(zhì)粒和引物,如表1-表3所示。
1.1.2 培養(yǎng)基 LB培養(yǎng)基:5 g/L酵母浸粉,10 g/L蛋白胨,10 g/L NaCl;YPD100培養(yǎng)基:10 g/L酵母浸粉,20 g/L蛋白胨,100 g/L葡萄糖;YPE培養(yǎng)基含10 g/L酵母浸粉和20 g/L蛋白胨,5% YPE和7% YPE分別加入5%(V/V)和7%(V/V)的無水乙醇;YPD活化培養(yǎng)基:10 g/L酵母浸粉,20 g/L蛋白胨,20 g/L葡萄糖。固體培養(yǎng)基添加瓊脂粉至20 g/L。大腸桿菌轉(zhuǎn)化子篩選時添加100 mg/L氨芐青霉素;釀酒酵母轉(zhuǎn)化子篩選和細(xì)胞培養(yǎng)時添加200 mg/L潮霉素。
1.2 方法
1.2.1 報告載體的構(gòu)建 首先以pKT127為模板,yEGFP-F/R為引物PCR擴(kuò)增得到帶多克隆位點Linker和ADH1終止子序列的yEGFP片段,經(jīng)Xma I/Kpn I雙酶切后連接到用相同酶處理的pRS41H載體中,得到載體pRS41H-yEGFP。以S288c基因組為模板,PTEF1-F/R和PTDH3-F/R為引物分別進(jìn)行PCR,擴(kuò)增得到PTEF1和PTDH3啟動子片段;以p416-UASCLB(3X)-PTEF為模板,PaTEF1-F/R為引物PCR擴(kuò)增得到PaTEF1啟動子片段。人工啟動子PaTEF1比野生型PTEF1多了3個重復(fù)的CLB2啟動子激活序列UASCLB,該序列位于基因CLB2起始密碼子上游-867至-627,長度為240 bp[5]。將上述啟動子片段通過相應(yīng)的酶切位點連接到pRS41H-yEGFP,得到相應(yīng)的報告載體 pRS41H-PTEF1-yEGFP、pRS41H-PTDH3-yEGFP和pRS41H-PaTEF1-yEGFP。
1.2.2 菌株的構(gòu)建和篩選 將測序驗證正確的啟動子報告載體及其空載(pRS41H-yEGFP)通過醋酸鋰轉(zhuǎn)化法[14]分別轉(zhuǎn)化到釀酒酵母BY4741、Ethanol Red和CEN.PK 113-5D LX03(下文簡稱LX03)菌株中。在含有200 mg/L潮霉素的抗性YPD固體培養(yǎng)基中篩選轉(zhuǎn)化子。挑取單菌落,在含有200 mg/L潮霉素的YPD培養(yǎng)基中擴(kuò)大培養(yǎng),含有啟動子的轉(zhuǎn)化子通過熒光顯微鏡檢測細(xì)胞GFP熒光。空載體的轉(zhuǎn)化子在含有200 mg/L潮霉素的YPD中培養(yǎng)后提取質(zhì)粒,轉(zhuǎn)化大腸桿菌并進(jìn)一步驗證質(zhì)粒的序列。

表1 本研究所用的菌株

表2 本研究所用的引物

表3 本研究所用的質(zhì)粒
1.2.3 釀酒酵母細(xì)胞培養(yǎng) 為了高通量檢測不同宿主以及不同培養(yǎng)基中的啟動子活性,本研究采用方形U底全擋板24孔板(常州英德生物科技有限公司,YD010124B),在30℃和200 r/min條件下,對上述12個釀酒酵母菌株進(jìn)行培養(yǎng)。培養(yǎng)體積為3 mL,并比較了各菌株在YPD100、5% YPE和7%YPE三種培養(yǎng)基條件下的生長和啟動子活性。取在-80℃甘油管中保存的菌在5 mL YPD活化培養(yǎng)基中培養(yǎng)過夜后,將活化后的菌液按初始OD600為0.2轉(zhuǎn)接至上述培養(yǎng)基中進(jìn)行培養(yǎng),并取樣檢測細(xì)胞生長及啟動子活性。每個菌株在培養(yǎng)和檢測時設(shè)置兩個平行。1.2.4 細(xì)胞生長的測定 每隔12 h(YPD100)或24 h(YPE)取0.1 mL不同培養(yǎng)基中培養(yǎng)的菌液,稀釋到合適濃度后,用酶標(biāo)儀(Thermo-Fisher ScientificTM,MultiskanTMGO,MA,USA),在600 nm波長下檢測細(xì)胞濃度。
1.2.5 GFP熒光檢測 取24孔板中培養(yǎng)的菌液0.1 mL,在4℃下3 000 ×g離心3 min收集細(xì)胞,用1 mL預(yù)冷的ddH2O洗滌細(xì)胞,離心后棄上清并用預(yù)冷的ddH2O重懸細(xì)胞至適合流式細(xì)胞儀檢測的濃度。通過流式細(xì)胞儀(BD FACSAria II?,NJ,USA)檢測單個細(xì)胞中yEGFP的熒光強度,進(jìn)而對啟動子強度進(jìn)行分析。yEGFP熒光通過藍(lán)光(488 nm)激發(fā),用530/30規(guī)格的濾光片檢測。每份樣品(細(xì)胞懸液)共收集50 000個細(xì)胞進(jìn)行檢測和統(tǒng)計。通過前向散射光檢測器(FSC-A)和側(cè)向散射光檢測器(SSC-A)對細(xì)胞大小進(jìn)行檢測。GFP熒光與細(xì)胞的大小有關(guān),本研究根據(jù)Peng等[6]描述的方法進(jìn)行校正。
2 結(jié)果
2.1 報告載體和菌株的構(gòu)建
根據(jù)1.2.1所描述的方法,構(gòu)建得到不含啟動子的pRS41H-yEGFP載體和含有3種不同啟動子的報 告 載 體:pRS41H-PaTEF1-yEGFP、pRS41H-PTEF1-yEGFP和pRS41H-PTDH3-yEGFP(圖1)。將上述4個載體分別轉(zhuǎn)化釀酒酵母BY4741、Ethanol Red和LX03菌株,得到表1所示的12個報告載體過表達(dá)菌株,并用于后續(xù)研究。
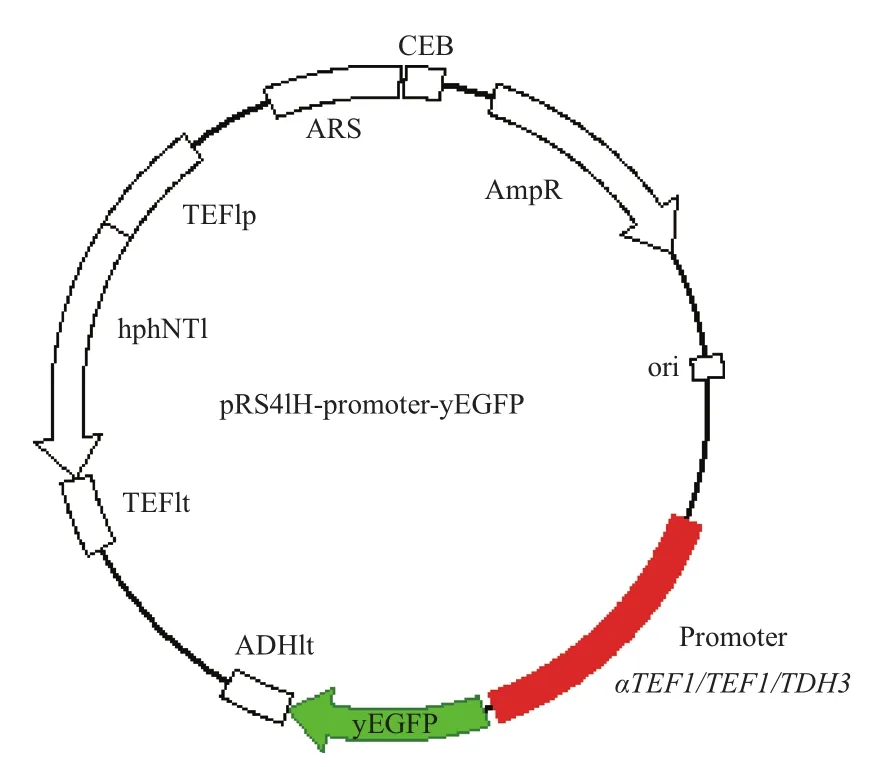
圖1 報告載體示意圖
2.2 細(xì)胞生長的比較
釀酒酵母BY4741、Ethanol Red和LX03菌株在YPD100、5% YPE和7% YPE條件下的生長情況如圖2所示。YPD100中培養(yǎng)的各個菌株無明顯延滯期,在36 h之后進(jìn)入穩(wěn)定期,BY4741背景的菌株生長速率顯著低于以另外兩個菌為宿主的菌株,而以Ethanol Red為宿主的菌株生長速率略高于以LX03為宿主的菌株。同時,5% YPE和7% YPE中BY4741背景的菌株生長速率也要慢于以Ethanol Red或LX03為宿主的菌株,但LX03背景的菌株生長速率略快于以Ethanol Red為宿主的菌株。
2.3 各菌株在不同條件下的啟動子強度
菌株在不同培養(yǎng)基中生長情況不同,因此經(jīng)過預(yù)實驗確定各個菌株在3種培養(yǎng)基中培養(yǎng)時的生長時期,并按照1.2.5描述的方法檢測細(xì)胞熒光。
釀酒酵母BY4741、Ethanol Red和LX03菌株在YPD100、5% YPE和7% YPE條件下的GFP熒光強度如圖3所示。含有空載體的菌株檢測到的熒光強度幾乎可以忽略不計,因此細(xì)胞中的熒光強度直接反映相應(yīng)的啟動子強度。在YPD100中3種遺傳背景的菌株的人工啟動子PaTEF1活性均高于天然啟動子PTEF1活性,前者是后者的1.4-4.6倍;而BY4741、Ethanol Red和LX03菌株的PaTEF1活性分別是PTDH3活性的0.93、2.03和1.14倍。在5%和7% YPE中,LX03背景的菌株中PaTEF1活性較PTEF1更高,較PTDH3更低;而Ethanol Red背景的菌株中PaTEF1的活性較PTEF1更低,較PTDH3更高,BY4741背景的菌株中PaTEF1的活性較PTDH3更高,與PTEF1相比則是有高有低;3個菌株在YPE培養(yǎng)基中的PaTEF1活性與PTEF1和PTDH3活性之比分別在0.7-1.3和0.8-1.3之間。在不同遺傳背景的菌株中,啟動子在YPE中的活性均較YPD中更高,在YPE中培養(yǎng)時細(xì)胞的PaTEF1、PTEF1和PTDH3啟動子活性分別為在YPD100中培養(yǎng)時的1.7-2.0、2.7-7.1和1.3-3.4倍。在3種培養(yǎng)基中,PaTEF1和PTDH3的活性在BY4741背景的菌株中最高,在LX03背景的菌株中最低;而PTEF1的活性則在Ethanol Red背景的菌株中最高,在LX03背景的菌株中最低。
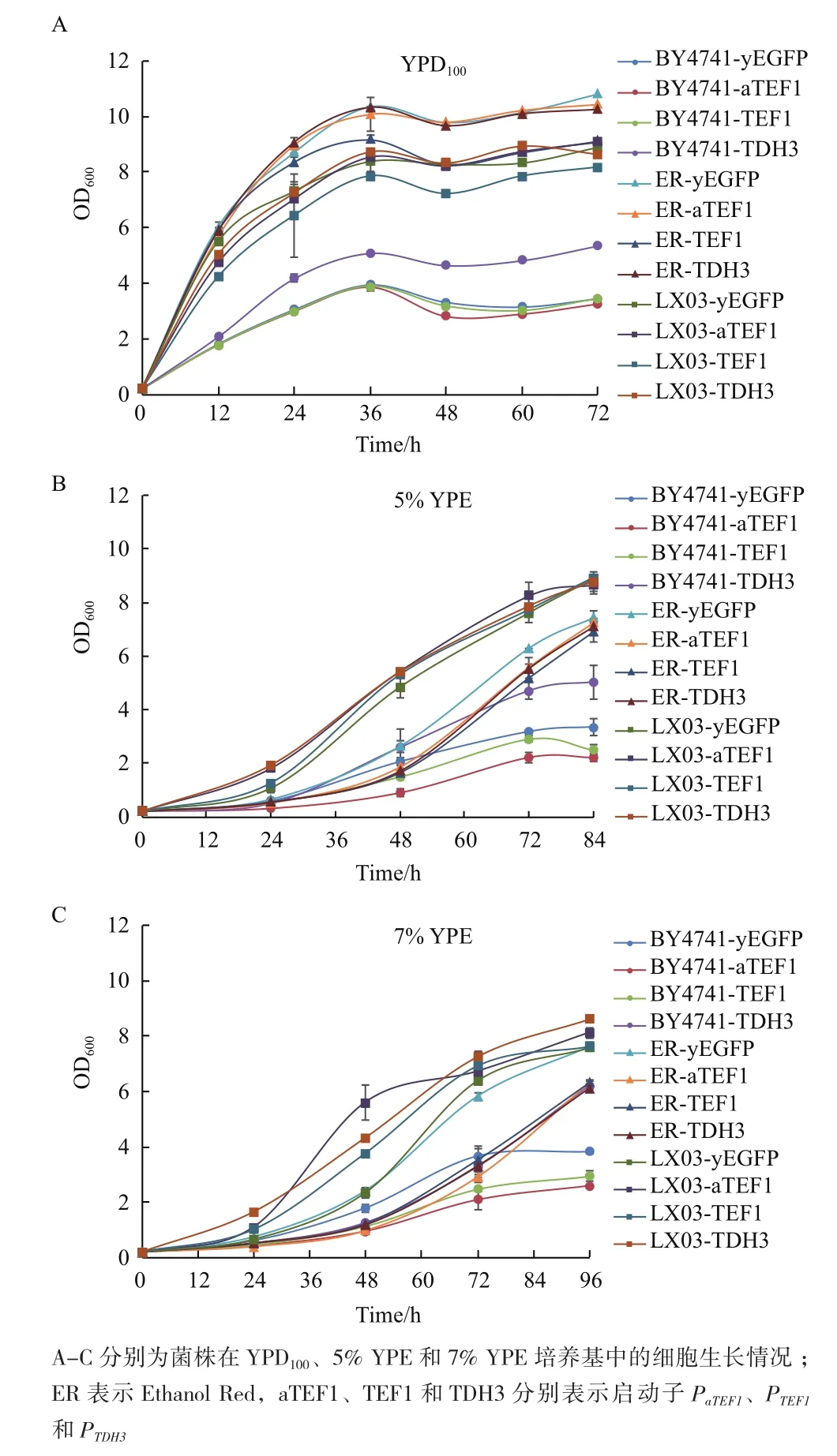
圖2 菌株在不同培養(yǎng)基條件下的生長情況比較
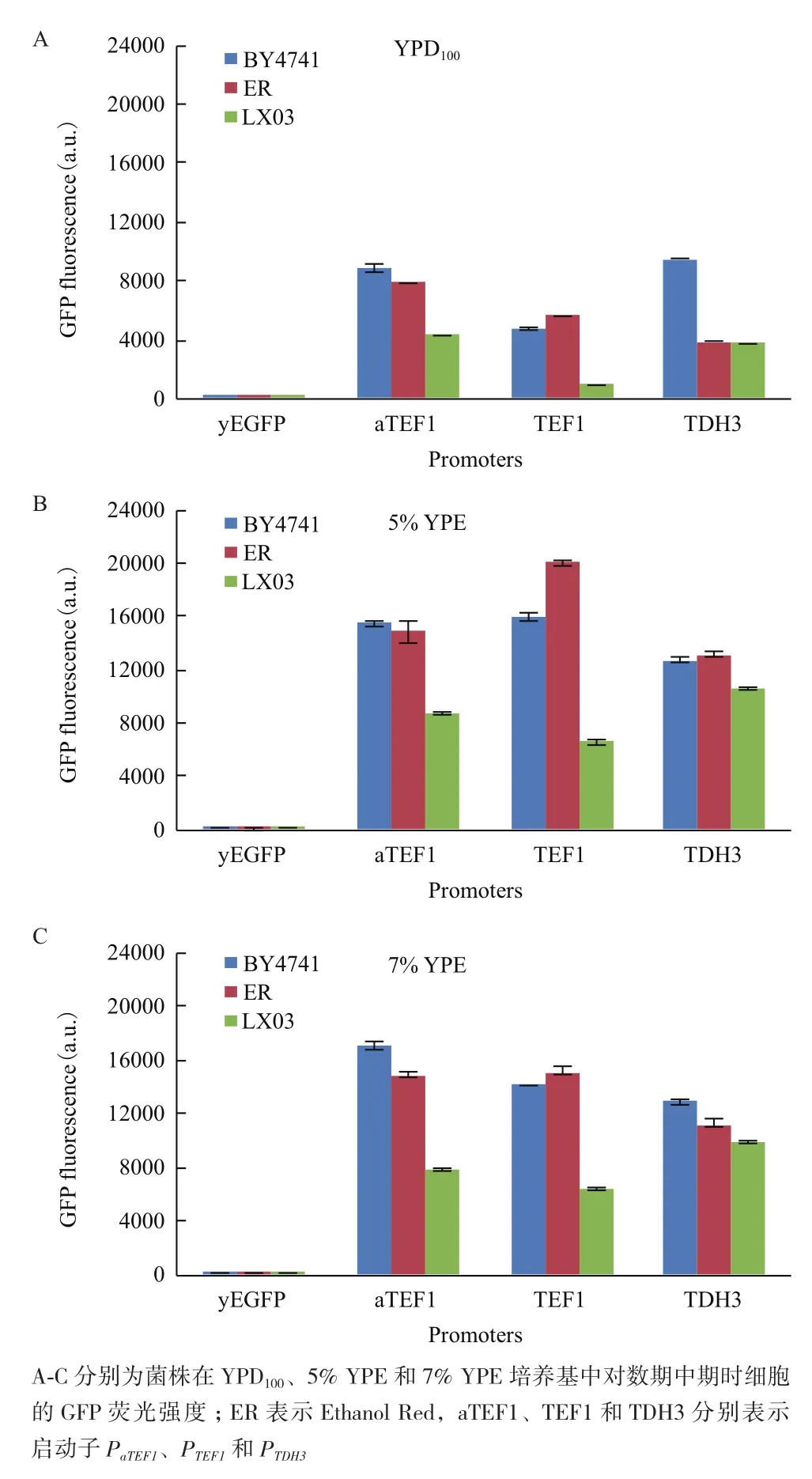
圖3 菌株在不同培養(yǎng)基條件下的GFP熒光比較
此外,對3個菌株在YPD100培養(yǎng)基中,對數(shù)期早期、對數(shù)期中期和穩(wěn)定期的細(xì)胞熒光強度進(jìn)行了比較,相應(yīng)的取樣時間點分別為8、24和48 h(圖4,表4)。啟動子活性從對數(shù)期早期到對數(shù)期中期的變化較對數(shù)期中期到穩(wěn)定期的變化大。對比對數(shù)期中期和對數(shù)期早期,PTDH3啟動子在3種背景的菌株中啟動子活性變化小,而PaTEF1啟動子在BY4741背景的菌株中對數(shù)期中期的活性較對數(shù)期早期的活性減弱(前者是后者的0.73倍),PTEF1啟動子在LX03背景的菌株中活性增強(前者是后者的1.43倍)。
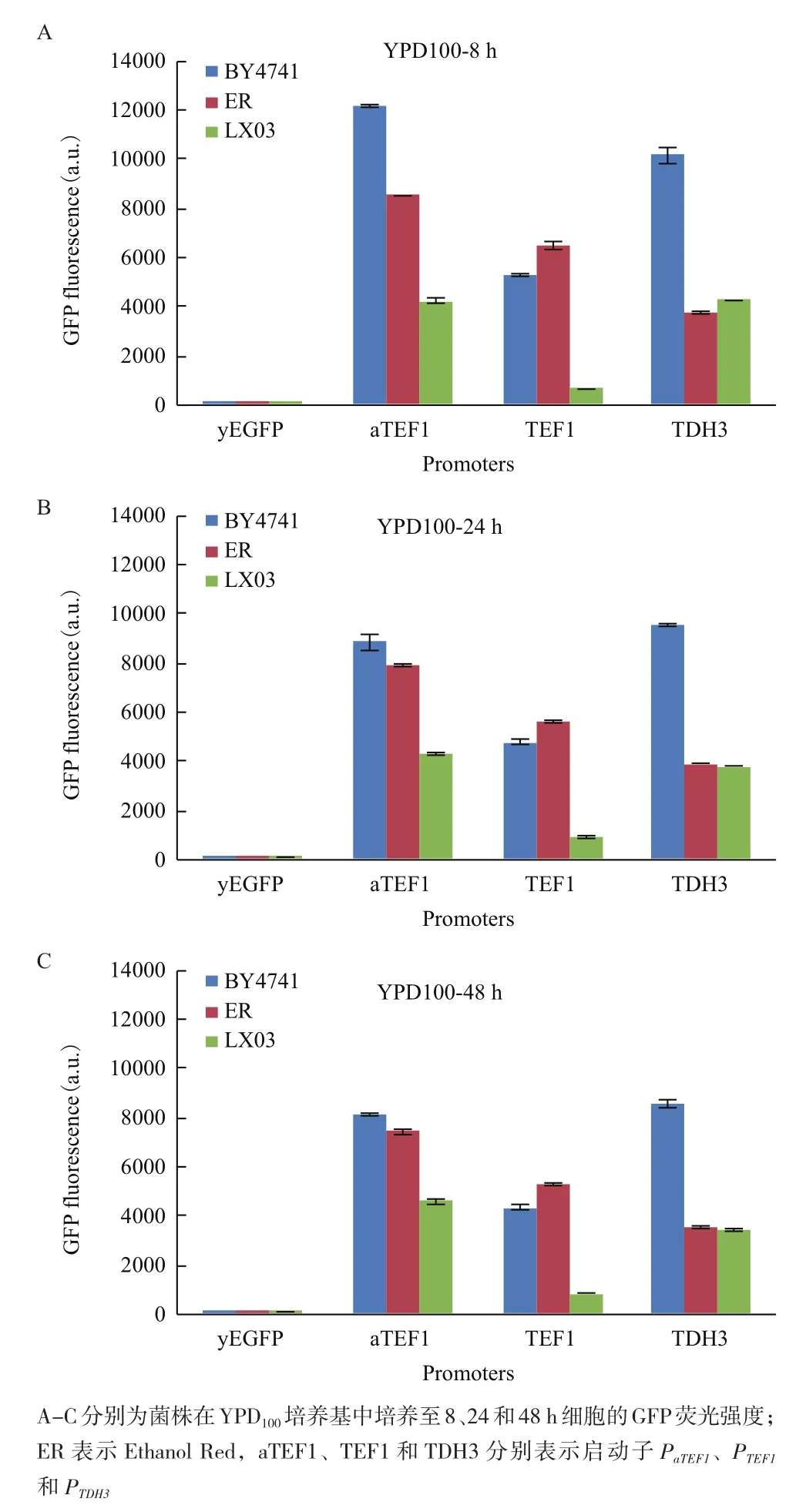
圖4 菌株在YPD100培養(yǎng)基中不同細(xì)胞生長階段的GFP熒光比較
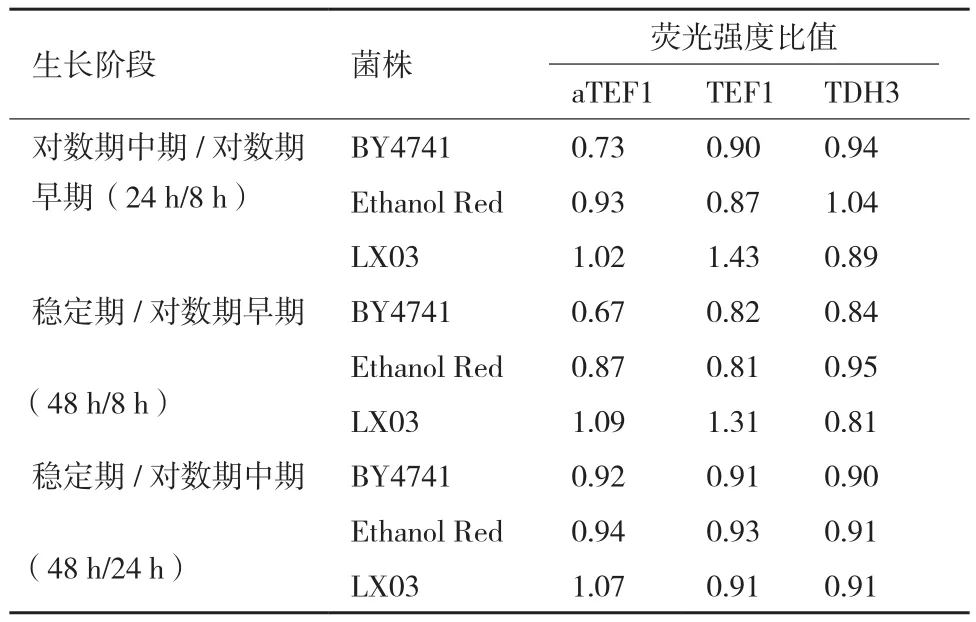
表4 菌株在YPD100培養(yǎng)基中不同生長階段的GFP熒光強度比較
3 討論
釀酒酵母是普遍使用的底盤細(xì)胞,對其細(xì)胞中關(guān)鍵代謝途徑的基因表達(dá)強度進(jìn)行精細(xì)調(diào)節(jié)對于提高生產(chǎn)菌株的性能至關(guān)重要。細(xì)胞內(nèi)代謝的調(diào)控水平包括轉(zhuǎn)錄水平、翻譯水平和翻譯后修飾水平。蛋白質(zhì)的翻譯后修飾調(diào)控較為復(fù)雜,不便于進(jìn)行遺傳操作。相較于翻譯水平,轉(zhuǎn)錄水平的調(diào)控對基因表達(dá)水平的提升倍數(shù)更多,可達(dá)到102數(shù)量級,而翻譯水平調(diào)控對基因表達(dá)水平的提升卻很低,例如密碼子優(yōu)化使釀酒酵母雙加氧酶基因表達(dá)水平提高2.9倍左右[15]。因此,通過采用不同強度的啟動子來調(diào)控轉(zhuǎn)錄水平進(jìn)而改變代謝流是一個常用的方法[16]。例如,通過在RIM15基因的5'-UTR區(qū)域插入PCK1基因的啟動子可以抑制RIM15基因在早期葡萄糖豐富時的表達(dá)而激活其在后期乙醇發(fā)酵時的表達(dá),進(jìn)而提高乙醇發(fā)酵性能[17]。此外,在其他代謝途徑中,研究者們也通過選擇不同強度的啟動子來調(diào)控不同酶的表達(dá)水平。如我國學(xué)者[18]對釀酒酵母中萜類化合物合成途徑中多達(dá)12個基因的啟動子活性進(jìn)行了表征,并采用具有不同活性的啟動子對代謝途徑進(jìn)行了重新設(shè)計,進(jìn)而獲得了角鯊烯、油酸和麥角固醇產(chǎn)量顯著提高的釀酒酵母菌株。在過去20多年里,研究者們鑒定了許多不同的啟動子,包括天然的啟動子及其突變體。Partow等[19]對多個組成型啟動子以及誘導(dǎo)型啟動子PGAL1和PGAL10在葡萄糖和半乳糖中的活性進(jìn)行了表征;Alper等[5]通過對野生型TEF1啟動子進(jìn)行突變得到了活性跨度較大的TEF1啟動子突變體;Peng等[6]則同時鑒定了19個啟動子在不同碳源中的活性。在現(xiàn)有啟動子的基礎(chǔ)上,研究者們又創(chuàng)造性地將啟動子中的調(diào)控元件進(jìn)行了組裝,得到了具有更強活性的人工啟動子[4]。
與挖掘啟動子的多樣性相比,研究個別啟動子在不同條件下,尤其是工業(yè)發(fā)酵環(huán)境條件下的啟動活性和穩(wěn)定性對于提升細(xì)胞工廠的性能同樣重要。基因的最優(yōu)表達(dá)水平在不同宿主中也不盡相同,并受到宿主遺傳背景的影響。因此本研究選擇了不同宿主進(jìn)行啟動子活性分析,包括BY4741、Ethanol Red和LX03,其中BY4741為常用的實驗室菌株,Ethanol Red為工業(yè)菌株,LX03則是本課題組之前研究工作中構(gòu)建的木糖代謝菌株[10]。Partow等[19]研究發(fā)現(xiàn),當(dāng)把8 h時的PTEF1啟動子活性定義為100%時,24 h和48 h時PTEF1啟動子活性分別為156%和136%。在本研究中,僅LX03背景菌株在葡萄糖條件下的啟動強度變化與Partow等[19]的研究結(jié)果一致,而BY4741和Ethanol Red背景菌株中PTEF1在24 h和48 h相對于8 h的啟動子活性均出現(xiàn)了下降(表4)。Partow等[19]所用的菌株為CEN.PK 113-5D,與LX03的出發(fā)菌株相同。由此說明,在相同遺傳背景的菌株中,啟動子活性隨著細(xì)胞生長階段的變化趨勢相同。PaTEF1在YPD100中的活性高于PTEF1的活性,而在YPE中則表現(xiàn)為在LX03背景的菌株中PaTEF1的活性高于PTEF1的,在Ethanol Red背景的菌株中PaTEF1的活性較PTEF1更低,而在BY4741中有高有低。在3種培養(yǎng)基中,PaTEF1的活性在BY4741背景的菌株中最高,在LX03背景的菌株中最低。而PTEF1的活性則在Ethanol Red背景的菌株中最高,在LX03背景的菌株中最低。表明啟動子活性會隨宿主遺傳背景的不同而變化。同一啟動子的活性在不同宿主中不盡相同,這可能與不同宿主的代謝調(diào)控網(wǎng)絡(luò)不同有關(guān)。此外,人工啟動子的活性并非總是高于天然啟動子活性,而是隨著釀酒酵母遺傳背景和培養(yǎng)基條件的不同發(fā)生變化。
研究表明,啟動子的活性也會受到包括碳源和生長時期等多種因素的影響[19,20],因此本研究檢測了3種培養(yǎng)基條件下對數(shù)期早期、對數(shù)期中期和穩(wěn)定期的啟動子活性。葡萄糖是細(xì)胞工廠生產(chǎn)目標(biāo)產(chǎn)物時最常用的碳源,當(dāng)葡萄糖耗盡之后,細(xì)胞轉(zhuǎn)換為利用培養(yǎng)基中其他碳源的模式。乙醇既是釀酒酵母乙醇發(fā)酵中的主要代謝產(chǎn)物,在發(fā)酵后期也作為細(xì)胞生長的碳源。乙醇也被用作某些高附加值產(chǎn)品,如角鯊烯、游離脂肪酸等[18]生產(chǎn)過程的碳源,研究表明以乙醇作為碳源有利于青蒿素的生產(chǎn)[8]。此外,高濃度乙醇對宿主菌株產(chǎn)生明顯的抑制作用[21]。因此研究乙醇為碳源條件下的啟動自強度也具有重要的意義。Peng等[6]研究了以2%乙醇為碳源時啟動子活性,發(fā)現(xiàn)PTEF啟動子活性較PPGK1和PTDH3等啟動子的活性更強。Yamauchi等[22]發(fā)現(xiàn)BTN2啟動子可在高濃度(>9%,V/V)乙醇條件下強表達(dá),但在溫和乙醇脅迫條件下PBTN2活性則相對較低,采用該啟動子也能夠在高濃度乙醇條件下啟動CUR1、GIC2以及YUR1等異源基因的表達(dá)。本研究對不同宿主中野生型啟動子和人工啟動子在3種培養(yǎng)基YPD100、5% YPE和7% YPE的活性進(jìn)行了研究和比較。其中,培養(yǎng)基YPE中5%和7%的乙醇既可作為碳源又是一種細(xì)胞脅迫因子,相較于2%的乙醇濃度也更接近生產(chǎn)發(fā)酵過程中的乙醇濃度。有趣的是,啟動子PaTEF1、PTEF1和PTDH3在YPE中的活性均高于在YPD100中的活性,前者是后者的1.3-7.1倍,提示乙醇對上述啟動子有一定的激活作用。YPE中啟動子活性較YPD中更強,可能是菌株響應(yīng)乙醇脅迫的一種表現(xiàn)。通過菌株在不同培養(yǎng)基條件下的生長情況(圖2)可知,在YPE條件下培養(yǎng)時,菌株的延滯期延長,即乙醇對菌株的生長產(chǎn)生了抑制,這可能使得細(xì)胞對蛋白質(zhì)的需求更大,增加了啟動子活性。另外,轉(zhuǎn)錄因子與啟動子的結(jié)合對啟動子的活性影響較大[23,24],乙醇作為一種脅迫的同時也是一種不同于葡萄糖的碳源,可能影響轉(zhuǎn)錄因子的結(jié)合,從而影響啟動子活性。
隨著合成生物學(xué)的發(fā)展和啟動子研究的深入[4],人工雜合啟動子成為一種新的元件構(gòu)建方法。人工雜合啟動子由上游激活序列和核心啟動子組合而成,Alper等通過將不同的上游增強元件(UASCLB和UASCIT)與不同的天然啟動子核心(PGPD、PCYC、PTEF和PLEUM)串聯(lián)得到的雜合啟動子的活性是相應(yīng)的天然啟動子活性的1.63-10.02倍。盡管人工啟動子比野生型啟動子活性更強,但前人的研究只考察了釀酒酵母BY4741菌株在YSC-URA(20 g/L glucose)培養(yǎng)基中培養(yǎng)48 h時的人工啟動子活性,未對人工啟動子在不同宿主、不同碳源以及不同生長時期的活性進(jìn)行進(jìn)一步研究。本研究選取的人工啟動子是Alper等[5]報道的將3個串聯(lián)的240-bp UASCLB與PTEF1核心組裝而成的啟動子UASCLB(3X)- PTEF1,該啟動子在葡萄糖條件下的活性是野生型PTEF1的1.63倍,并研究了人工啟動子及其天然啟動子PTEF1以及常用的強啟動子PTDH3在不同宿主、不同培養(yǎng)基條件和細(xì)胞不同生長階段的活性。值得一提的是,在YPD100和YPE培養(yǎng)基中,人工啟動子PaTEF1的活性變化倍數(shù)為1.7-2.0,而天然啟動子PTEF1和PTDH3的變化倍數(shù)分別為2.7-7.1和1.3-3.4,這可能從側(cè)面反映出人工啟動子的活性在不同培養(yǎng)基中較天然啟動子更穩(wěn)定。
4 結(jié)論
本研究系統(tǒng)全面地分析和比較了人工啟動子和天然啟動子在不同條件下和不同宿主中的性能。宿主的遺傳背景和培養(yǎng)基條件對啟動子活性的影響較大,同時,啟動子活性可能隨著細(xì)胞生長階段的不同而不同,另外,在不同遺傳背景的菌株中,啟動子活性從對數(shù)期早期到對數(shù)期中期和從對數(shù)期中期到穩(wěn)定期的變化趨勢不同。這些結(jié)果表明啟動子的選用需要考慮宿主遺傳背景和不同的培養(yǎng)基條件以及基因的表達(dá)在不同細(xì)胞生長階段的需求。人工啟動子在不同培養(yǎng)基中的活性比天然啟動子更加穩(wěn)定,因此在需要基因穩(wěn)定表達(dá)的情況下,人工啟動子可能是一種更好的選擇。
致謝:感謝美國德克薩斯大學(xué)奧斯汀分校Hal S. Alper教授惠贈質(zhì)粒p416-UASCLB(3X)-PTEF。
[1]Kav??ek M, Stra?ar M, Curk T, et al. Yeast as a cell factory:current state and perspectives[J]. Microbial Cell Factories, 2015, 14(1):1.
[2]Mnaimneh S, Davierwala AP, Haynes J, et al. Exploration of essential gene functions via titratable promoter alleles[J]. Cell, 2004, 118(1):31-44.
[3]Weinhandl K, Winkler M, Glieder A, et al. Carbon source dependent promoters in yeasts[J]. Microbial Cell Factories, 2014, 13(1):1.
[4]Blazeck J, Alper HS. Promoter engineering:recent advances in controlling transcription at the most fundamental level[J]. Biotechnology Journal, 2013, 8(1):46-58.
[5]Blazeck J, Garg R, Reed B, et al. Controlling promoter strength and regulation in Saccharomyces cerevisiae using synthetic hybrid promoters[J]. Biotechnology Bioengineering, 2012, 109:2884-2895.
[6]Peng B, Williams TC, Henry M, et al. Controlling heterologous gene expression in yeast cell factories on different carbon substrates and across the diauxic shift:a comparison of yeast promoter activities[J]. Microbial Cell Factories, 2015, 14:91.
[7]Alper H, Fischer C, Nevoigt E, et al. Tuning genetic control through promoter engineering[J]. Proceedings of the National Academy of Sciences of the United States of America, 2005, 102(36):12678-12683.
[8]Westfall PJ, Pitera DJ, Lenihan JR, et al. Production of amorphadiene in yeast, and its conversion to dihydroartemisinic acid, precursor to the antimalarial agent artemisinin[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109:E111-E118.
[9]Zhao XQ, Bai FW. Mechanisms of yeast stress tolerance and its manipulation for efficient fuel ethanol production[J]. Journal of Biotechnology, 2009, 144(1):23-30.
[10]Blazeck J, Liu L, Redden H, et al. Tuning gene expression in Yarrowia lipolytica by a hybrid promoter approach[J]. Applied and Environmental Microbiology, 2011, 77(22):7905-7914.
[11]熊亮, 程誠, 李凱, 等. 菊芋秸稈高濃度物料分步糖化及乙醇發(fā)酵[J]. 應(yīng)用與環(huán)境生物學(xué)報, 2016, 22(3):382-387.
[12]Sheff MA, Thorn KS. Optimized cassettes for fluorescent protein tagging in Saccharomyces cerevisiae[J]. Yeast, 2004, 21:661-670.
[13]Taxis C, Knop M. System of centromeric, episomal, and integrative vectors based on drug resistance markers for Saccharomyces cerevisiae[J]. Biotechniques, 2006, 40(1):73-78.
[14]Gietz RD, Schiestl RH. High-efficiency yeast transformation using the LiAc/SS carrier DNA/PEG method[J]. Nature Protocols, 2007, 2(1):31-34.
[15]Leavitt JM, Alper HS. Advances and current limitations in transcript-level control of gene expression[J]. Current Opinion in Biotechnology, 2015, 34:98-104.
[16]Blazeck J, Alper H. Systems metabolic engineering:Genome-scale models and beyond[J]. Biotechnology Journal, 2010, 5(7):647-659.
[17]Watanabe D, Kaneko A, Sugimoto Y, et al. Promoter engineering of the Saccharomyces cerevisiae RIM15 gene for improvement of alcoholic fermentation rates under stress conditions[J]. Journal of Bioscience and Bioengineering, 2016, pii:S1389-1723(16)30202-X. doi:10. 1016/j. jbiosc. 2016. 08. 004.
[18]Rasool A, Zhang G, Li Z, et al. Engineering of the terpenoid pathway in Saccharomyces cerevisiae co-overproduces squalene and the non-terpenoid compound oleic acid[J]. Chemical Engineering Science, 2016, 152:457-467.
[19]Partow S, Siewers V, Bj?rn S, et al. Characterization of different promoters for designing a new expression vector in Saccharomyces cerevisiae[J]. Yeast, 2010, 27(11):955-964.
[20]Vickers CE, Bydder SF, Zhou Y, et al. Dual gene expression cassette vectors with antibiotic selection markers for engineering in Saccharomyces cerevisiae[J]. Microbial Cell Factories, 2013, 12:96.
[21]Bai FW, Anderson WA, Moo-Young M. Ethanol fermentation technologies from sugar and starch feedstocks[J]. Biotechnology Advances, 2008, 26(1):89-105.
[22]Yamauchi Y, Izawa S. Prioritized expression of BTN2 of Saccharomyces cerevisiae under pronounced translation repression induced by severe ethanol stress[J]. Frontiers in Microbiology, 2016, 7:1319.
[23]Redden H, Alper HS. The development and characterization of synthetic minimal yeast promoters[J]. Nature Communications, 2015, 6:7810.
[24]Ganapathi M, Palumbo MJ, Ansari SA, et al. Extensive role of the general regulatory factors, Abf1 and Rap1, in determining genomewide chromatin structure in budding yeast[J]. Nucleic Acids Research, 2011, 39(6):2032-2044.
(責(zé)任編輯 馬鑫)
Activity Comparison of the Artificial Hybrid Promoter with Its Native Promoter in Saccharomyces cerevisiae
TANG Rui-qi1XIONG Liang2BAI Feng-wu1,2ZHAO Xin-qing1
(1. School of Life Science and Biotechnology,Shanghai Jiao Tong University,Shanghai 200240;2. School of Life Science and Biotechnology,Dalian University of Technology,Dalian 116023)
The responses of promoter strengths of artificial TEF1 promoter(PaTEF1)and native promoter PTEF1as well as PTDH3were comprehensively compared. The strength of PaTEF1was not always higher than PTEF1,but varied along with the genetic background of host,medium,and cell growth phase. Among the three investigated hosts,PaTEF1showed the highest strength in BY4741,while the lowest in LX03. The strengths of PaTEF1were 1.4-4.6 and 0.9-2.0 times of those of native promoters(PTEF1and PTDH3)in YPD100medium,respectively. The activities of PaTEF1in YPE(5% and 7%)were 0.7-1.3 and 0.8-1.3 times of PTEF1and PTDH3. The activity of PaTEF1in YPE was 1.7-2.0 times of that in YPD100,whereas 2.7-7.1 and 1.3-3.4 times for PTEF1and PTDH3,respectively. The activities of the promoters in YEP were higher than those in YPD100,however,the activity variation of artificial promoters was less than that of native promoter. In addition,the variation trends of promoter strengths from early-log to mid-log phase and from mid-log to stationary phase varied in different host strains.
Saccharomyces cerevisiae;artificial hybrid promoter;TEF1 promoter;host strain;environmental condition
10.13560/j.cnki.biotech.bull.1985.2017.01.013
2016-10-20
國家自然科學(xué)基金項目(31461143029,5151101168)
唐瑞琪,女,博士研究生,研究方向:釀酒酵母代謝工程改造;E-mail:rq_tang@sjtu.edu.cn
趙心清,女,博士,教授,研究方向:釀酒酵母及絲狀真菌代謝工程改造和生物能源生產(chǎn);E-mail:xqzhao@sjtu.edu.cn

