基于宏基因組學技術分析不同貯藏條件下 鯰魚片中的菌相變化
朱迎春,王洋,樊曉盼,馬儷珍,王凱麗
?
基于宏基因組學技術分析不同貯藏條件下 鯰魚片中的菌相變化
朱迎春1,王洋2,樊曉盼3,馬儷珍3,王凱麗1
(1山西農業大學食品科學與工程學院,山西太谷 030801;2天津農學院水產學院/天津市水產生態及養殖重點實驗室,天津 300384;3天津農學院食品科學與生物工程學院/天津市農副產品深加工技術工程中心,天津 300384)
【目的】分析鯰魚肉片貯藏過程中微生物多樣性和菌群動態變化,探究天然保鮮劑對鯰魚肉片菌相及菌落組成的影響。【方法】試驗分8組,即新鮮鯰魚片(CK0);空氣包裝(air-package,AP)鯰魚片低溫貯藏(4±1)℃第4、7天(AP4和AP7)樣品;氣調包裝(modified atmosphere package,MAP,60% CO2/40% N2)鯰魚片冰溫貯藏(-0.7±0.02℃)第10、30天(MAP10和MAP30)樣品;添加5%天然保鮮液(由殼聚糖、蜂膠、溶菌酶、Nisin和茶多酚復配而成)MAP鯰魚片冰溫貯藏第10、30和40天(MAPP10、MAPP30和MAPP40)樣品。采用宏基因組學技術,測定并分析不同貯藏方式鯰魚片全部微生物的16S rDNA序列,比較各組菌群的物種組成和豐度信息,通過Alpha多樣性分析和主成分分析,考察包裝方式、貯藏溫度和天然保鮮液對微生物的抑制作用。【結果】8組樣品中454焦磷酸測序共鑒定出25個門、433個屬的細菌。MAP10、MAPP10、MAPP30中的最優勢菌屬和CK0一致,均為;而AP4和AP7中的最優勢菌分別為(70.49%)和(59.01%)。從微生物角度來看,MAP+冰溫貯藏優于AP+冷藏。隨著貯藏時間的延長,氣調包裝中的豐度由15.78%(MAP10)急劇上升為82.85%(MAP30)。添加天然保鮮液的MAPP10和MAPP30中幾乎檢測不到。【結論】氣調包裝協同冰溫貯藏顯著降低了鯰魚片中細菌菌群的豐度和多樣性,有利于延長鯰魚肉的保質期。天然保鮮液中的茶多酚、Nisin和殼聚糖等能顯著抑制MAP+冰溫貯藏的鯰魚片中的主要腐敗菌屬的生長繁殖。
鯰魚片;宏基因組學;菌相變化;天然保鮮液
0 引言
【研究意義】革胡子鯰魚()肉質細嫩,無肌間刺,蛋白質、脂肪含量高,深受人們喜愛。但宰殺后的鯰魚肉極易因多種微生物的生長繁殖而腐敗變質。對于食品中微生物的群落組成及變化,常用的傳統培養分離方法存在費時、費力、片面的缺陷,無法真正解析微生物的組成、豐度及其變化情況,而省去培養過程的宏基因組學(metagenomics)技術直接從樣品中提取基因組DNA后進行測序分析,借助高通量測序方法,結合生物信息學工具,能夠發現大量過去無法分離培養的微生物[1-2]。因此,利用宏基因組學技術研究和分析冰溫貯藏過程中鯰魚肉菌群構成及其演替規律并研究其控制措施,具有重要的理論意義和潛在的實用價值。【前人研究進展】目前在食品領域中已將宏基因組學技術用于食品中微生物群落動態的追蹤[3-5],分析了干酪[6]、韓國泡菜[7-9]等發酵食品中微生物群落結構和動態變化規律,揭示了發酵食品釀造過程中微生物的演替變化及其與代謝產物之間的聯系。在肉制品加工方面,BENSON等[10]研究了不同處理的冷藏豬肉香腸的微生物變化。發現香腸經過乳酸/雙醋酸酯處理對微生物動力學有顯著影響,導致微生物多樣性發生改變,而微生物變化與化學變化緊密相關。在肉制品貯藏方面,XIAO等[11]利用宏基因組學技術,鑒定出真空包裝水晶肴肉含有169個屬的細菌,是貯藏0、7和30 d時的優勢菌群;及是貯藏15、22和30 d時的優勢菌群;所有樣品在貯藏期內微生物多樣性呈增加趨勢且有不同的群落結構,15 d后群落結構開始發生變化,30 d后群落結構有顯著性差異。Zhao等[12]考查了真空包裝冷卻豬肉21 d貯藏過程中的菌相變化規律,發現新鮮豬肉(0 d)的微生物多樣性高,貯藏0—7 d,豐度急劇下降,而和豐度逐漸上升并在第7天達到峰值,之后急劇下降至檢測不出,豐度值持續上升最終成為優勢菌。【本研究切入點】通過前期試驗已經獲知鯰魚片進行氣調包裝(modified atmosphere package,MAP,60% CO2/40% N2)后冰溫(-0.7℃)貯藏,比常規的空氣包裝(air-package,AP)低溫貯藏(4℃)更有利于維持鯰魚片的食用品質及理化特性[13]。但是雖然大多數微生物在此貯藏條件下生長受到抑制,仍有一些嗜冷性微生物可以生長繁殖,從而對鯰魚肉的品質及人類健康安全造成威脅,有必要進一步開展相關研究。【擬解決的關鍵問題】利用宏基因組學技術對鯰魚肉片貯藏過程中微生物種群構成和演替變化進行監測,并將筆者實驗室已研究出的天然保鮮劑[14]作用于鯰魚片,考察其對鯰魚肉片菌相變化規律及菌落組成的影響,了解不同貯藏條件下鯰魚片的優勢腐敗菌群變化,以針對性地靶向抑制優勢腐敗菌,提高鯰魚片的微生物安全性。
1 材料與方法
試驗于2014年11月在天津農學院食品加工車間進行。
1.1 試驗材料及樣品采集
10條革胡子鯰魚(),平均每條重(1 250±25)g,體長(30±2)cm,30 min內鮮活運至天津農學院食品加工車間進行宰殺。首先放入(5—7)℃冰水中10 min使其致暈,立即去頭、去皮、去內臟,去骨剖片后,用無菌冰水洗滌,瀝干水分,切成4 cm×3 cm×1 cm魚片,每片重為(20±2)g,將得到的160片鯰魚片充分混合,隨機分4組:(1)20片魚片用于0 d微生物群落結構的分析,標記為CK0;(2)40片魚片空氣包裝(AP)之后冷藏庫((4±1)℃)貯藏,在貯藏的4 d(貨架期終點)和7 d(腐敗階段)取樣分析,分別用AP4和AP7表示;(3)40片魚片直接進行氣調包裝(MAP,60% CO2/40% N2)后置于國家農產品保鮮工程技術研究中心冰溫庫((-0.7±0.02)℃)中貯藏,在10 d(新鮮階段)和30 d(腐敗階段)取樣,分別標記為MAP10、MAP30;(4)60片魚片加入肉重5%的保鮮液(由0.5%殼聚糖、0.1%蜂膠、0.075%Nisin、0.075%溶菌酶、0.5%茶多酚配制而成),攪拌均勻,再以同樣方式進行氣調包裝(MAP),置于冰溫庫((-0.7±0.02)℃)中貯藏,分別在10 d(新鮮階段)、30 d(次新鮮階段)和40 d(貨架期終點)取樣分析,分別標記為MAPP10、MAPP30和MAPP40。所取樣品裝入無菌自封袋中,于-80℃超低溫冰箱保存。將全部8個樣品(CK0、AP4、AP7、MAPP10、MAPP30、MAPP40、MAP10、MAP30)以冷凍運輸方式運送到上海派森諾生物科技有限公司,進行宏基因組測序和生物信息學分析。
1.2 試驗方法
1.2.1 宏基因組DNA的提取 按照1.1所取的樣品,參照OMEGA Soil DNA Kit試劑盒說明,分別抽提不同鯰魚肉樣品的細菌基因組DNA。所得DNA采用紫外分光光度計檢測濃度和純度,并采用0.8%瓊脂糖凝膠電泳檢測DNA完整性。
1.2.2 16S rDNA PCR擴增 以1.2.1獲得的基因組DNA為模板,擴增 16S V1—V3區全長,引物(F:5′- 454adapter-mid-AGAGTTTGATCCTGGCTCAG-3′;R:5′-454adapter-TTACCGCGG CTGCTGGCAC-3′)。
PCR反應體系為:8.75 μL滅菌超純水;5.00 μL 5×Q5 Buffer,5×GC Enhancer,5.00 μL dNTP(2.5 mmol·L-1);2.00 μL模板(2 ng·μL-1);1.00 μL引物F(10 μmol·L-1);1.00 μL引物R(10 μmol·L-1);0.25 μL Q5 DNA polymeras。PCR循環梯度反應參數:98℃預變性4 min;98℃變性30 s,55℃退火45 s,72℃延伸1 min,27個循環;72℃延伸7 min,冷卻至4℃。PCR產物用1.5%瓊脂糖凝膠電泳檢測。
1.2.3 焦磷酸測序 使用AMPure Beads對PCR產物進行純化,將純化后的PCR產物使用PicoGreen dsDNS Assay Kit在酶標儀上對文庫進行定量,然后將PCR純化產物稀釋,等量混合。混合后的樣本進行454焦磷酸測序。454測序按照標準的454/Roche GS-FLX Titanium方案進行,利用GS DNA Library Preparation kit制備成單鏈模板DNA(single-stranded DNA)文庫,并利用GS emPCR kit 固定在磁珠上,在一個油包水的體系里進行擴增。
1.2.4 序列分析
1.2.4.1 原始雙端序列的過濾和連接 采用Illumina MiSeq平臺進行測序。去除準確率低于99%的以及模糊堿基N;然后利用軟件Flash(http://www. genomics. jhu.edu/software/FLASH/index. shtml)對通過質量過濾的序列進行連接,要求read1和read2的overlap≥10 bp,且不容許堿基錯配。最后,根據index信息提取每個樣品的有效序列(要求Index完全匹配)。
1.2.4.2 優質序列的獲取及統計 為了保證分析結果的準確性,運用Qiime軟件[15](version 1.7.0,http://qiime.org/)進行序列過濾,運用mothur[16](version 1.31.2,http://www.mothur.org/)軟件中uchime的方法去除嵌合體序列,得到用于后續分析的優質序列。
1.2.4.3 生物信息學分析 本試驗在Qiime中調用uclust的方法對優質序列按相似度0.97進行OUT(Operational Taxomomic Units,操作分類單元)聚類分析,選取每個類最長的序列為代表序列。在Qiime中采用blast的方法對序列數據庫進行比對,比對后得到每個OTU的分類學信息,并設置置信區間為95%,默認為cut off=0.03,即序列相似度在3%以下就認為“未分類”。使用OUT表,利用Qiime軟件繪制稀釋曲線圖。
群落結構多樣性分析選用Chao-the Chao1 estimator(http://www.mothur.org/wiki/Chao),Ace(abundance based coverage estimator)-the ACE estimator(http: //www.mothur.org/wiki/Ace),Shannon-the Shannon index(http://www.mothur.org/wiki/Shannon),Simpson- the Simpson index(http://www.mothur.org/wiki/ Simpson)等參數進行評估,根據OTU列表中的各樣品物種豐度情況,應用軟件mothur中的summary.single命令,計算4種常用的生物多樣性指數。物種豐度差異分析根據門和屬兩個層次上序列數的統計信息,將各物種豐度歸一到同數量級,應用軟件mothur中的metastats(http://metastats. cbcb.umd.edu/)命令,進行兩組之間的差異顯著性分析,多組之間采用SPSS進行方差分析和LSD多重比較,sig值小于0.05的物種認為在組間存在顯著性差異。此外,將所有細菌按分類學統計并利用軟件Qiime進行主成分分析(PCA),觀察不同樣本間微生物群落結構多樣性的分類學差異。
2 結果
2.1 宏基因組DNA的提取
表1為鯰魚微生物基因組DNA的純度檢測結果。基因組DNA在OD260處具有完整的波峰,證明所提取的微生物基因組DNA純度較好。基因組 DNA的平均濃度為87.38 ng·μL-1,所提取基因組DNA的總量為4.40 μg,符合瓊脂糖凝膠電泳的基本要求。
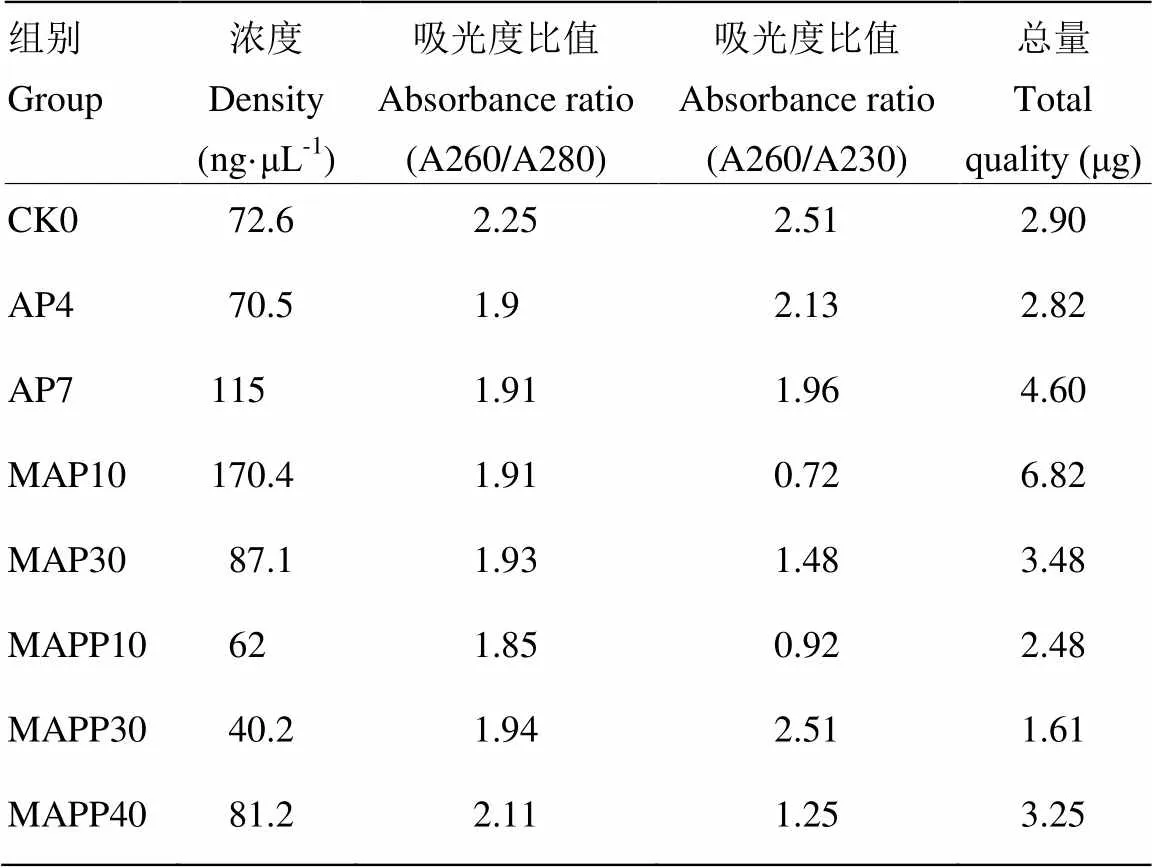
表1 鯰魚片細菌宏基因組質量檢測結果
圖1為細菌16S rDNA V1—V3區片段瓊脂糖凝膠電泳圖譜,可知,以樣品DNA為模板可以擴增出細菌的16S V1—V3區片段,大小500 bp左右且條帶清晰,大小正確,濃度適當,可進行后續試驗。
2.2 優質序列的獲取與統計
本試驗采用454 FLX+平臺進行測序,將測序得到的原始序列進行篩選,丟棄長度短于200 bp、含有模糊堿基、引物堿基含2個以上錯配、單堿基重復超過6個的序列,得到質量較高、符合要求的有效序列。但是高通量測序建庫過程中PCR擴增會產生嵌合體序列(chimera sequence),測序過程中會產生點突變和同聚物等測序錯誤,隨著測序長度的增加,序列末端的質量會降低。為了保證分析結果的準確性,需要對有效序列進行過濾和去除嵌合體處理,得到最終用于后續分析的優質序列。表2為8個樣品中有效序列和優質序列的統計結果,共獲得有效序列40 834個,優質序列為29 646個,優質序列的平均長度為458 bp。
M:DL2000 marker,1:CK0;2:AP4;3:AP7;4:MAP10;5:MAP30;6:MAPP10;7:MAPP30;8:MAPP40
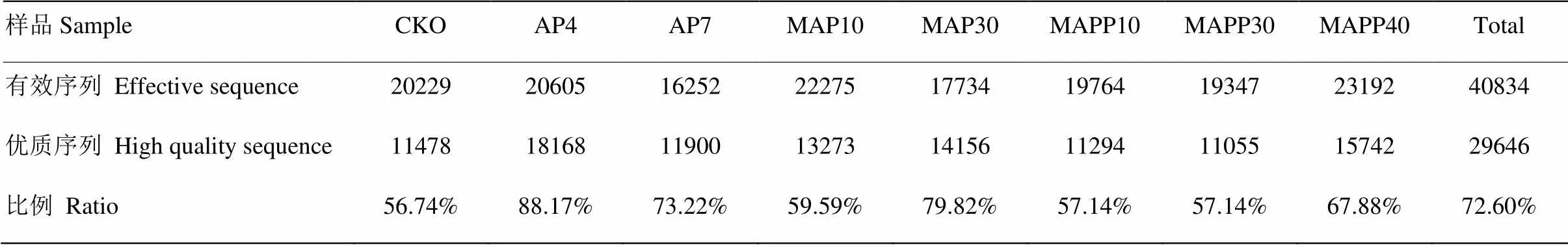
表2 樣品序列數統計表
2.3 MAP鯰魚片冰溫貯藏過程中細菌群落結構及動態變化
2.3.1 鯰魚片基于門水平的微生物群落結構分析 圖2是基于門繪制的微生物群落結構變化圖,由圖2可知,所有的樣品細菌群落可以分為25個門,其中放線菌門(Actinobacteria)、擬桿菌門(Bacteroidetes)、藍藻門(Cyanobacteria)、厚壁菌門(Firmicutes)、變形菌門(Proteobacteria)的微生物占了極大的比例。新鮮鯰魚片(CK0)中Proteobacteria、Actinobacteria和Firmicutes是鯰魚肉樣品的主要微生物,分別占到29.70%、29.35%和22.25%,其次為Bacteroidetes和Cyanobacteria,所占比例為8.97%和7.89%,5種微生物的豐度總和達到98.06%,其他20種不同門的微生物只占很小比例。隨著包裝方式和貯藏溫度的不同,基于門的微生物群落結構發生了變化。
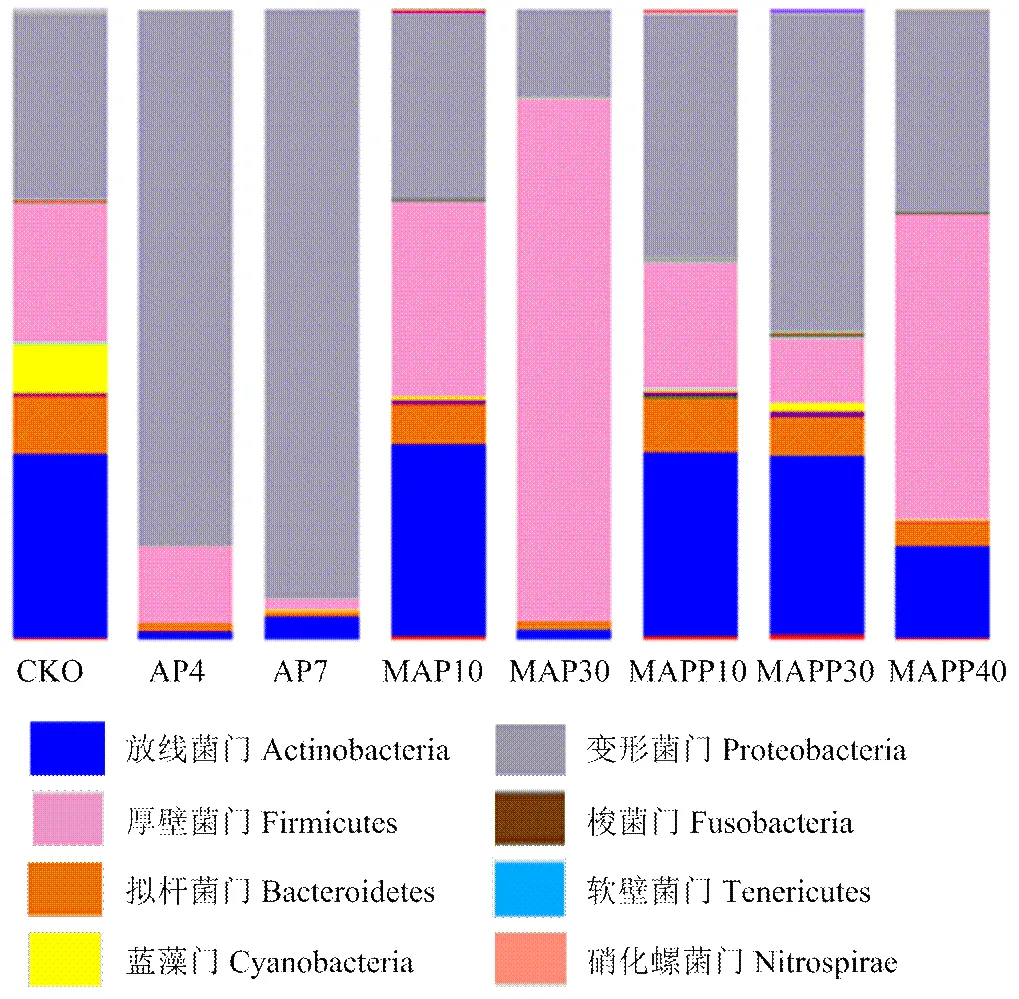
圖2 基于門的細菌群落結構變化
AP鯰魚片4℃貯藏期間Proteobacteria豐度顯著增加,新鮮魚片豐度為29.70%,貯藏4 d上升為82.82%,7 d達到93.58%,成為優勢腐敗菌;Actinobacteria豐度下降,從29.35%下降到1.31%(4 d),之后略有上升但比例仍然很低(3.69%,7 d);Firmicutes豐度逐漸下降,從22.25%下降到12.15%(4 d)至1.84%(7 d)。MAP鯰魚片冰溫貯藏4 d與CK0相比,群落結構未發生顯著變化,貯藏至第30天時,Proteobacteria和Actinobacteria的豐度分別下降到13.96%和1.59%;Firmicutes的豐度大幅上升為83.01%。添加保鮮液的MAPP鯰魚片Proteobacteria的豐度在31.81%— 50.03%呈現波動變化,貯藏第10天、30天和40天時Actinobacteria豐度下降速率小于其他處理組,最終達到14.78%(40 d)。Firmiciutes在貯藏期間的豐度分別為20.25%(10 d)、10.45%(30 d)和48.50%(40 d),呈先下降后上升的趨勢。
擬桿菌門(Bacteroidetes)和藍藻門(Cyanobacteria)在鯰魚片所有樣品中所占比例較小且變化規律相同,均隨著貯藏時間的延長呈下降趨勢,新鮮鯰魚片Bacteroidetes豐度值為8.97%,至樣品貯藏終點,達到0.84%—3.53%。Cyanobacteria豐度值則從7.89%下降至0.03%—0.09%。
由此可知,在鯰魚片貯藏過程中,Proteobacteria、Actinobacteria和Firmicutes是優勢菌群。冰溫貯藏及加入天然保鮮液可以抑制Proteobacteria、Cyanobacteria和Bacteroidetes的生長,但冰溫貯藏結合保鮮液作用對Actinobacteria的作用不明顯,而對Firmicutes無顯著抑制作用,這可能是由于很多Firmicutes會產生芽孢,可以抵抗脫水和極端環境,具有較強抗性的緣故。
2.3.2 基于屬水平的微生物群落結構分析 圖3—圖5是基于屬水平繪制的微生物群落結構變化圖,它可以更清楚地表示鯰魚肉在貯藏過程中菌群結構的動態變化。本試驗共鑒定出433個屬的細菌,表明鯰魚片菌落形態極為豐富。新鮮鯰魚片共含有170個屬的細菌,其中豐度大于1%的菌屬共有16個,豐度總和為96.90%,放線菌()豐度達到22.26%,占優勢地位。其他依次為芽孢桿菌屬()(15.98%),蒼白桿菌屬()(6.10%)和不動桿菌屬()(4.01%),其余的菌屬雖然豐度不高但數量眾多。
AP鯰魚片在4℃貯藏4 d 時菌相和新鮮魚片(CK0)相比有了很大的變化,氣單胞菌屬()豐度最高,達到70.49%,成為優勢菌群;乳酸球菌菌屬()所占比例為11.93%,假單胞菌屬()為9.95%。貯存至7 d,氣單胞菌屬()比例下降為5.54%,假單胞菌屬()成為優勢菌群,豐度達到59.01%。因此,普通的空氣包裝食品導致其腐敗的主要優勢菌是氣單胞菌屬和假單胞菌屬,為了更好地保持AP包裝鯰魚肉片的品質,應當設法控制它們的生長繁殖。
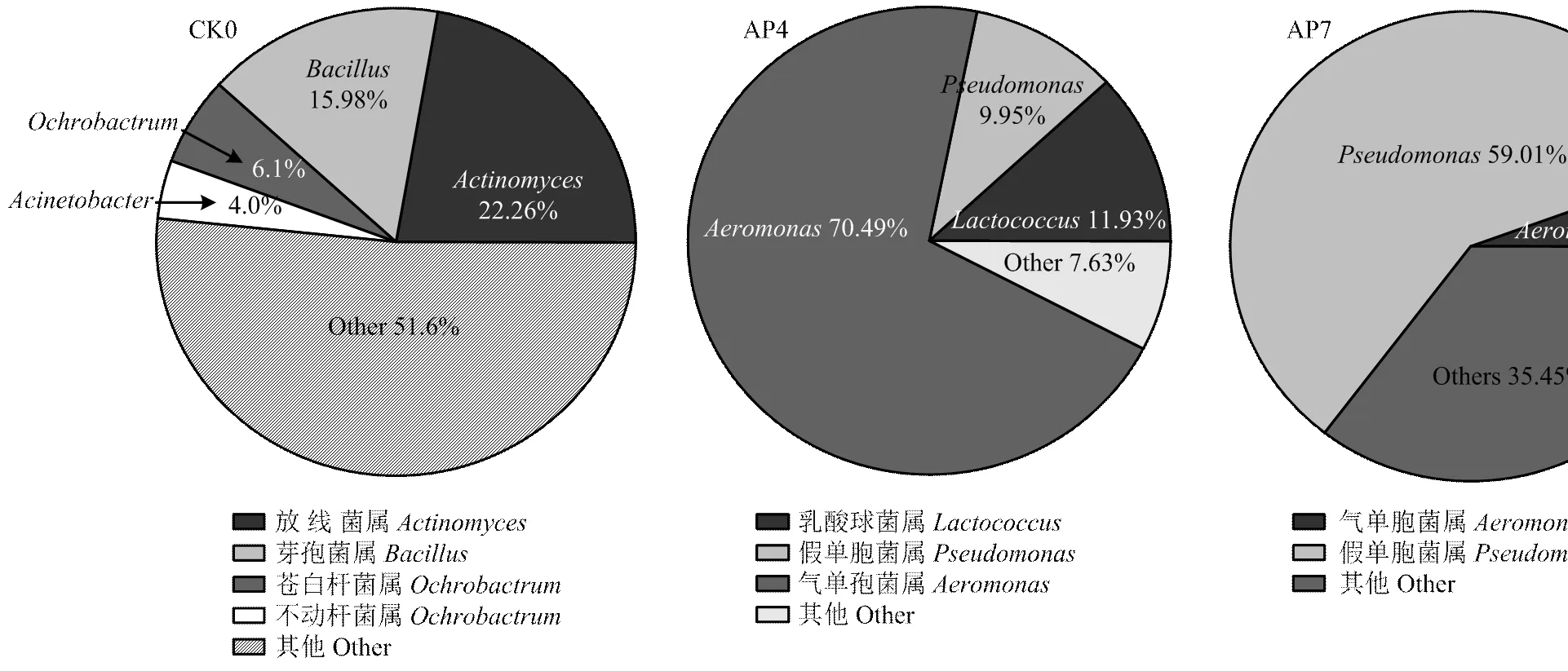
圖3 基于屬水平的鯰魚片微生物群落結構(CK0組及AP組)
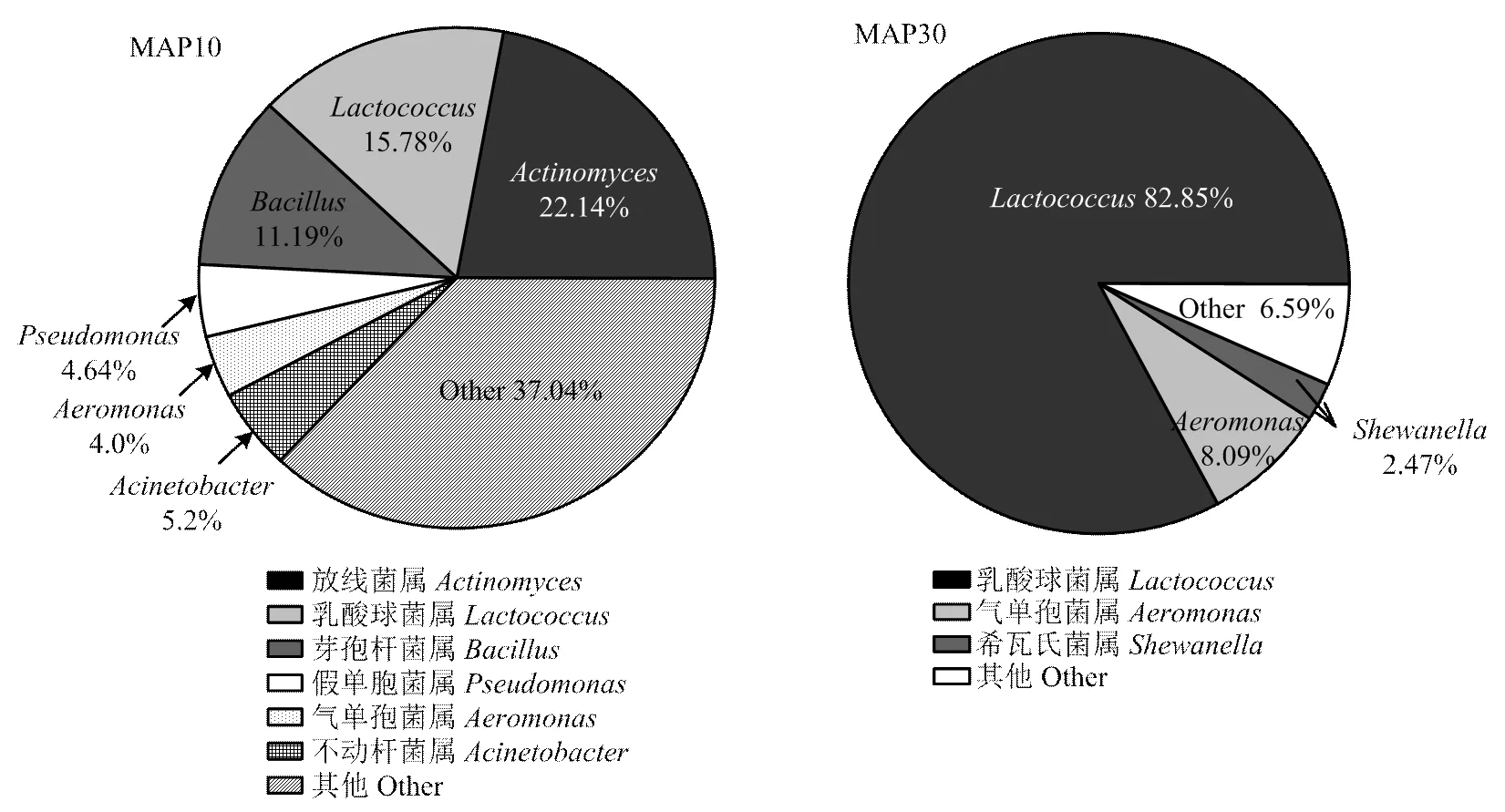
圖4 基于屬水平的鯰魚片微生物群落結構(MAP組)
MAP鯰魚片冰溫貯藏10 d時,微生物群落結構與新鮮鯰魚片(CK0)比較并沒有顯著差異。至貯藏終點(30 d)時,豐度急劇上升為82.85%,其次為占8.09%,占2.47%,這3種菌屬占總微生物的比例為94%,菌落組成結構逐漸變得單一。成為MAP鯰魚片的優勢腐敗菌。
天然保鮮液作用的MAPP組鯰魚片在冰溫貯藏過程中所占比例逐漸下降,由21.73% (10 d)降低到20.13%(30 d)和12.29%(40 d)。10 d時的葡萄球菌屬()(3.72%)、腸球菌屬()(8.24%)和蒼白桿菌屬()(5.20%)至貯藏第30、40天時逐漸下降到較小的比例:(1.51%,0.57%)、(2.18%,0.41%)和(3.26%,1.89%)。而奈瑟菌屬()的豐度由0.07%(10 d)上升到22.76%(30 d);在貯藏前期豐度較低,為 2.03%(10 d),而貯藏40 d達到46.44%;不動桿菌屬()的比例由10.79%(10 d)上升為17.98%(40 d)。所以,和成為MAPP鯰魚片貯藏后期的優勢致腐菌屬。
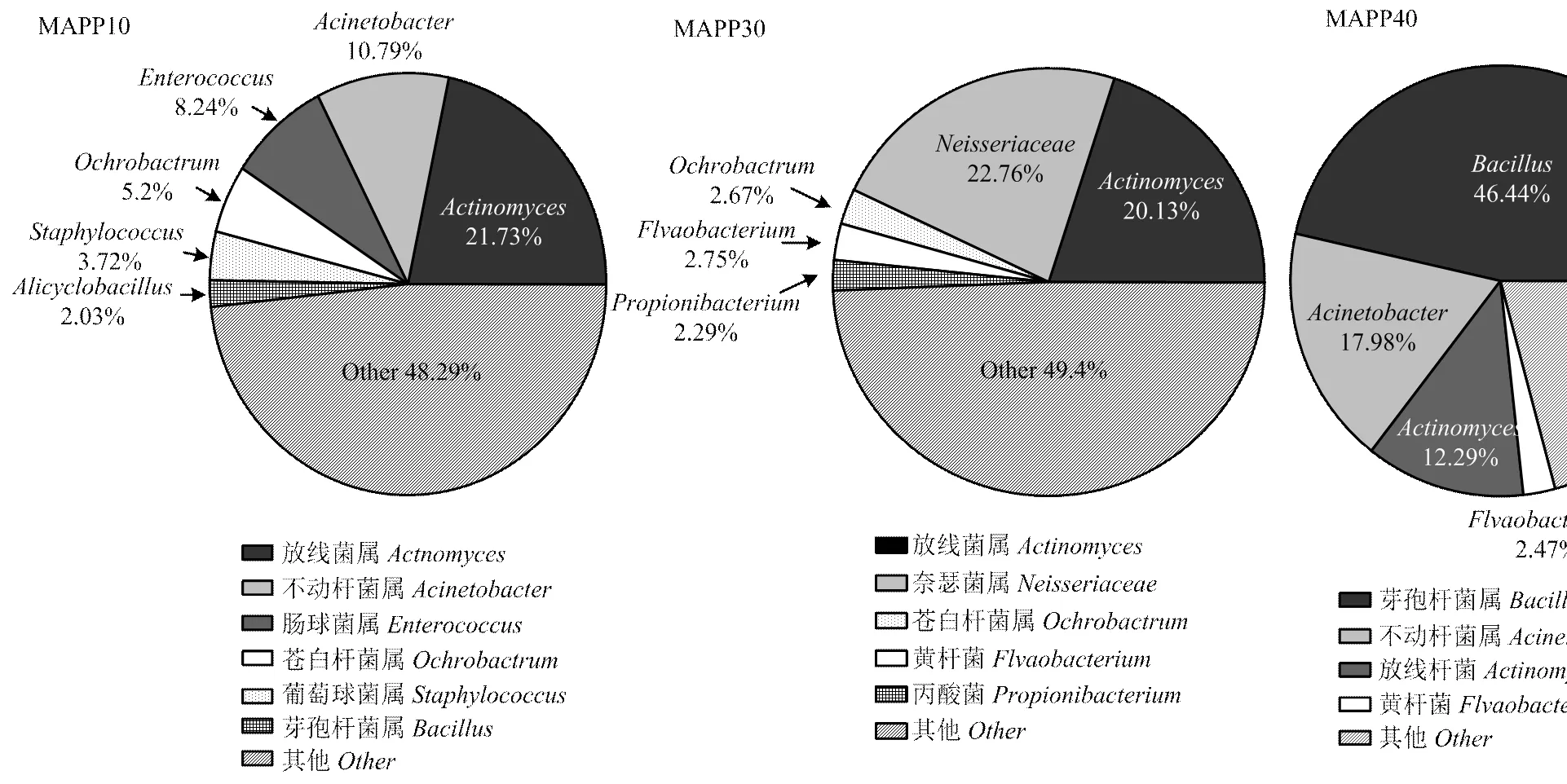
圖5 基于屬水平的鯰魚片微生物群落結構(MAPP組)
2.4 鯰魚片貯藏過程中細菌群落的Alpha多樣性分析
表3為鯰魚片微生物Alpha多樣性指數表。Chao1、ACE、Chao/ACE為群落豐度指數(community richness),指數越大,群落豐度越高;Shannon和Simpson代表群落多樣性(Community diversity),Shannon值越大,群落多樣性越高,Simpson指數值越大,則群落多樣性越低。
由細菌群落的Alpha多樣性分析表明,隨著貯藏時間的延長,細菌群落組成發生較大的變化,新鮮鯰魚片(CK0)Chao1指數、Ace指數和Shannon指數均較高,Simpson指數較低,這表明新鮮鯰魚肉細菌群落豐富,具有較高的多樣性。隨著貯藏時間的延長,AP鯰魚片冷藏4 d細菌的豐度和多樣性逐漸降低,說明低溫對某些嗜溫性細菌具有抑制性,導致其生長受阻。貯藏7 d,進入鯰魚片腐敗階段,蛋白質和脂肪分解產生一些低分子堿性化合物,肉的pH升高,為某些適于分解蛋白質和脂肪的微生物創造了生長和繁殖的機會,細菌多樣性提高,群落豐度隨之提高。
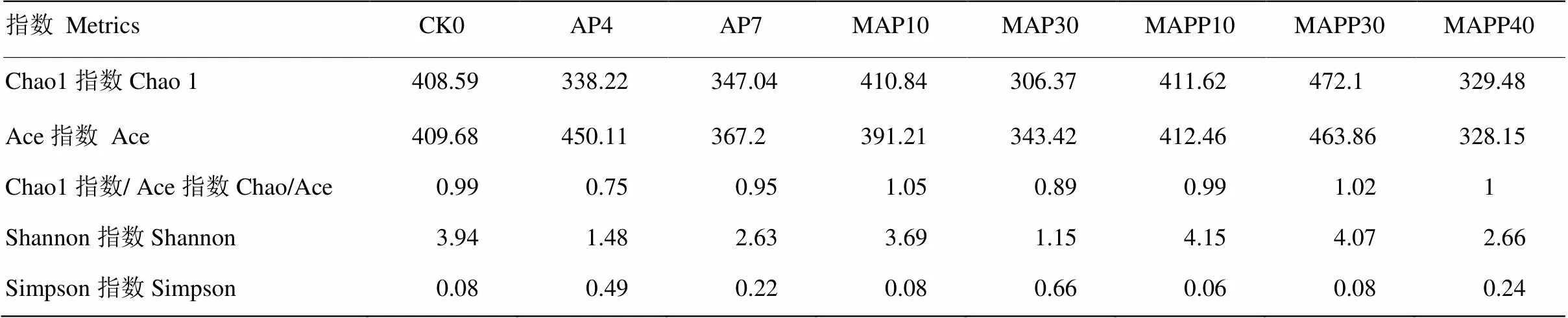
表3 生物多樣性指數表
對冰溫貯藏MAP鯰魚片而言,在貯藏前期,細菌多樣性和豐度(Chao 1、Ace,Chao/Ace和Shannon、Simpson指數)與新鮮鯰魚片相比無較大變化。在貯藏終點,多樣性和豐度仍明顯低于AP冷藏組相同階段(即AP7)。經保鮮液作用的MAPP組細菌多樣性和豐度在貯藏第10天與CK0組無明顯差異,貯藏30 d高于MAP組,這可能是添加殼聚糖、蜂膠、Nisin等保鮮液后改變了微生態的內環境所致,但隨著貯藏時間的延長,細菌多樣性和豐度逐漸降低,也說明冰溫+氣調+保鮮液三重作用對細菌群落的抑制作用。
2.5 鯰魚片貯藏過程中細菌群落的主成分分析
由圖6可知,CK0、MAP10、MAPP10、MAPP30和MAPP40樣品的微生物組成較為接近,可認為是一個群體;AP7則和這個群體的組成有一定的差別;而MAP30、AP4和這個群體的差別很大。由此可知,冰溫貯藏+氣調包裝可以很好地維持鯰魚肉的品質,使鯰魚肉的微生物菌群結構在整個貯藏過程中與CK0(即新鮮鯰魚片)相比并沒有發生顯著的變化,這種保鮮作用在添加保鮮劑的MAPP樣品組貯藏30、40 d的時候依然有效。而MAP30樣品根據感官判定已經達到腐敗階段,其中腐敗菌大量繁殖,致病菌活力增強,微生物組成發生了很大的變化。和氣調包裝冰溫貯藏的條件不同,空氣包裝的鯰魚片在低溫條件下微生物組成與前者有所差異,部分需氧或兼性厭氧菌如大量繁殖(AP4),進而成為腐敗的優勢菌群(AP7),樣品的細菌群落結構有了顯著性的差異。圖6說明冰溫貯藏的樣品在貨架期內其細菌群落結構與新鮮樣品差異性小,而空氣包裝樣品在貯藏期間均有不同的細菌群落分布,與新鮮樣品的差異性大,而添加天然保鮮液的樣品在貯藏期內(40 d之內)能很好的保持微生物組成不發生顯著變化。

圖6 鯰魚片細菌群落貯藏過程中主成分分析
3 討論
新鮮鯰魚片初始微生物數量不高,但豐度較大,各種微生物相互制衡。無論采用哪種方式包裝和貯藏,隨著貯藏時間的延長,部分微生物生長繁殖,成為優勢腐敗菌,而其他微生物受到抑制,因此微生物豐度急速下降。和成為鯰魚片空氣包裝低溫貯藏的優勢腐敗菌。是水產品中的常見致腐菌,它與其他腐敗菌協同作用,水解蛋白質、淀粉和脂肪等,產生的尸胺是腐敗時不良氣味的直接來源[17]。KROVACEK[18]和MANO[19]等研究表明,能夠在-2—10℃的低溫下生存和繁殖,在冷藏的牛肉、烤牛肉和豬肉中都發現過該菌。李苗云等[20]也發現是冷卻豬肉貯藏過程中主要的優勢腐敗菌之一。是冷鏈流通中高蛋白水產品腐敗的主要原因[21-22]。DAINTY[23]研究表明,肉品的腐敗在很大程度上取決于肉品初期的數量和該菌在菌系中所占的比例。本試驗中,AP鯰魚片貯藏4 d以后,豐度大大超過成為冷藏條件下有氧包裝的優勢腐敗菌,這是因為屬好氧菌,屬兼性厭氧菌,所以在有氧包裝條件下,逐漸成為優勢菌并使魚肉發黏、變色,產生NH3和H2S等散發出刺鼻的臭味,使魚肉在感官上難以接受,這與許多研究報道一致[24-26]。
包裝方式和貯藏溫度的不同帶來微生物群落結構的差異,與AP鯰魚片相比,MAP鯰魚片菌相構成不及前者豐富,體現了氣調包裝對細菌菌群的抑制作用。氣調包裝中高濃度CO2通過改變微生物細胞內酶結構或影響細胞膜功能等方式來抑制細菌的生長代謝[27],特別是對等需氧菌的抑制。貯藏終點,豐度降低為0.14%。成為MAP鯰魚片冰溫貯藏后期的優勢腐敗菌,豐度值高達82.85%。勵建榮等[28]研究發現冷藏((3±0.5)℃)的氣調包裝(75% CO2/25% N2及50% CO2/50% N2)魚丸中為優勢腐敗菌。STAMATIS[29]也發現是氣調包裝(75% CO2/25% N2及50% CO2/50% N2)鯖魚在3℃或6℃下微生物最多的種群之一。
本研究中并未成為MAPP鯰魚片的優勢菌屬,貯藏過程中豐度在0.12%—1.42%,MAP鯰魚片中豐度較高的在MAPP鯰魚片中豐度值始終小于0.10%,豐度也逐漸降低,至貯藏40 d為0.41%,試驗說明天然保鮮液抑制其生長,改變了鯰魚片菌群結構。這是因為本試驗所用的復合保鮮液由溶菌酶、殼聚糖、蜂膠和Nisin等按比例配合而成。Nisin和溶菌酶等對革蘭氏陽性菌作用強;蜂膠對革蘭氏陰性菌作用強;茶多酚既有抑菌作用又有抗氧化作用;殼聚糖則對腸桿菌、沙門氏菌及葡萄球菌等多種微生物都有較強的抑制作用[30],研究報道殼聚糖還可以有效抑制三甲胺生成和脂肪氧化,降低生物胺生成量,使尸胺被完全抑制,組胺質量分數小于50 mg·kg-1[31]。抑菌劑不同,抑菌譜亦不同。試驗說明5種天然抑菌劑按照一定比例復配起來可以拓寬抑菌譜,起到明顯的抑菌作用。
MAPP組鯰魚片貯藏后期、奈瑟菌屬()和的豐度較高。為革蘭氏陽性菌,其芽孢位于菌體內,具有厚而含水量低的多層結構和高含量吡啶二羧酸,所以對熱、干燥、輻射、化學消毒劑和其他理化因素有較強的抵抗力,對保鮮液有比較強的抵抗性,保鮮液并不能將其很好抑制,當保鮮液發揮抑菌效能,將其他細菌抑制之后,它的豐度相應提高。專性需氧,本不是MAP貯藏的優勢菌,但隨著包裝袋內CO2的逐步消耗及外界O2通過包裝袋的滲透改變了鯰魚片貯藏的內環境,為奈瑟菌屬()的生長繁殖創造了條件。此外,也成為MAPP組鯰魚片貯藏后期的優勢菌群之一。孫麗霞[32]對氣調包裝結合茶多酚、Nisin和殼聚糖涂膜保鮮后的大黃魚貨架期終點(20 d)的菌落結構進行分析,也得出為主要致腐菌的結論。
本試驗發現,在新鮮(CK0)及氣調包裝鯰魚片(MAP10、MAPP10、MAPP30、MAPP40)中一直存在并占較大比例,說明也是貯藏過程中的主要污染微生物之一,應該引起足夠重視。無芽孢、無運動性、呈分枝狀或棍棒狀,厭氧或兼性厭氧菌,二氧化碳能促進其生長。在其他文獻中,極少報道,也未見在水產品中的生物學特性的研究報道,而本試驗中成為鯰魚片冰溫貯藏的優勢腐敗菌之一,因此有必要在以后的研究中對其做進一步的深入研究。據報道,是厭氧包裝的優勢腐敗菌,但是本試驗中,的豐度較低,與傳統微生物培養的結果一致,可能與原料魚的品種、飼養環境等有關。
本試驗采用以454 FLX+平臺高通量測序為基礎的宏基因組學技術全面研究了鯰魚片貯藏過程中微生物的物種組成及豐度信息,這為充分認識不可培養微生物并從完整的群落水平上研究微生物活動提供了可能,對于解析貯藏過程中微生物的動態變化以及貯藏過程的質量安全監測具有重要的意義和應用價值。
然而,任何一種微生物的研究手段都有其自身的優勢和局限性。高通量測序技術涵蓋的信息量巨大,某些未知序列因沒有足夠的參照標準,使得海量信息的歸類整合相對模糊。同時高通量測序技術是以微生物DNA作為檢測基礎,無法判別菌體的存活狀態[33]。傳統純培養鑒定方法盡管只能進行一般的表性特征描述,但其具有成本低、分離出的菌體純度高等特點,在水產品貯藏保鮮研究中仍占有重要地位,可以作為高通量測序技術的有益補充。
4 結論
采用宏基因組學方法分析了鯰魚片貯藏過程中細菌群落的組成及其豐度變化,考察了天然保鮮劑對貯藏過程中菌相變化規律及菌落結構的影響,共鑒定出25個門、433個屬的細菌。基于門水平,Proteobacteria、Actinobacteria和Firmicutes是優勢菌群。基于屬水平,新鮮鯰魚肉初始菌群主要是和,空氣包裝鯰魚片低溫貯藏過程中優勢菌為和,氣調包裝鯰魚片在冰溫貯藏過程中逐漸成為優勢菌群。添加天然保鮮液后,豐度降低。茶多酚、Nisin和殼聚糖等天然保鮮液能顯著抑制鯰魚片中等菌屬的生長繁殖,降低了鯰魚片細菌菌群的豐度和多樣性。
References
[1] HANDELSMAN J. Metagenomics application of genomics to uncultured in microorganisms., 2004, 68(4): 669-685.
[2] HANDELSMAN J, RONDON M R, BRADY S F, CLARDY J, GOODMAN R M. Molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products., 1998, 5(10): 245-249.
[3] KERGOURLAY G, TAMINIAU B, DAUBE G, VERGES M C. Metagenomic insights into the dynamics of microbial communities in food., 2015, 213: 31-39.
[4] WOLFE B E, DUTTON R J. Fermented foods as experimentally tractable microbial ecosystems., 2015, 161(1): 49-55.
[5] LOPEZ-VELASCO G, WELBAUM G E, BOYER R R, MANE S P, PONDER M A. Changes in spinach phylloepiphytic bacteria communities following minimal processing and refrigerated storage described using pyrosequencing of 16S rRNA amplicons., 2011, 110(110): 1203-1214.
[6] ALDRETE-TAPIA A, ESCOBAR-RAMIREZ M C, TAMPLIN M, HERNANDEZ-ITURRIAGA M. High-throughput sequencing of microbial communities in Poro cheese, an artisanal Mexican cheese., 2014, 44(6): 136-141.
[7] JUNG J Y, LEE S H, KIM J M, PARK M S, BAS J W, HAHN Y, MADSEN E L, JEON C OMetagenomic analysis of kimchi, a traditional Korean fermented food., 2011, 77(7): 2264-2274.
[8] JUNG J Y, LEE S H, LEE H J, SEO H Y PARK W S.Effects ofstarter cultures on microbial communities andmetabolites during kimchi fermentation., 2012, 153(3): 378-387.
[9] PARK E J, CHUN J, CHA C J, PARK W S, CHE O J.Bacterial community analysis during fermentation of ten representative kinds of kimchi with barcoded pyrosequencing., 2012, 30(1): 197-204.
[10] BENSON A K, DAVID J R, GILBRETH S E, SMITH G, NIETFELDT J, LEGGE R, KIM J, SINHA R,DUNCAN C E, MA J, SINGH I. Microbial successions are associated with changes in chemical profiles of a model refrigerated fresh pork sausage during an 80-day shelf life study., 2014, 80(17): 5178-5194.
[11] XIAO X, DONG Y, ZHU Y, CUI H. Bacterial diversity analysis of Zhenjiang yao meat during refrigerated and vacuum-packed storage by 454 pyrosequencing., 2013, 66(4): 398-405.
[12] ZHAO F, ZHOU G, YE K,WANG S, XU X, LI C. Microbial changes in vacuum-packed chilled pork during storage., 2014, 100(100): 145-149.
[13] ZHU Y C,MA L Z, XIONG Y L. Super-chilling (-0.7℃) with high-CO2packaging inhibits biochemical changes of microbial origin in catfish () muscle during storage, 2016, 206: 182-190.
[14] 馬儷珍. 冷卻豬肉生物和物理綜合保鮮技術及保鮮機理的研究[D]. 北京: 中國農業大學, 2003.
MA L Z. Studies on the biological and physical integrated preservation techniques and preservation effect of chilled pork [D]. Beijing: China Agricultural University, 2003. (in chinese)
[15] CAPORASO J G, KUCZYNSKI J, STOMBAUGH J, BITTINGER K, BUSHMAN F D. QIIME allows analysis of high-throughput community sequencing data., 2010, 7(5): 335-336.
[16] SCHLOSS P D,WESTCOTT S L, RYABIN T, HALL J R, HARTMANN M. Introducing mother: Open-source,platform-independent,community-supported software for describing and comparing microbial communities., 2009, 75(23): 7537-7541.
[17] DUAN J, CHERIAN Q, ZHAO Y. Quality enhancement in fresh and frozen lingcod () fillets by employment of fish oil incorporated chitosan coatings., 2010, 119(2): 524-532.
[18] KROVACEK K, FARIS A, BALODA S B, PETERZ M, LINDBERG T. Prevalence and characterization ofsppisolated from foods in Uppsala, Sweden., 1992, 9(1): 29-36.
[19] MANO S B, ORDONEZ J A, FERNANDO G D. Growth/survival of natural flora andon refrigerated uncooked pork and turkey packaged in modified atmospheres., 2000, 17(6): 657-669.
[20] 李苗云, 周光宏, 徐幸蓮.應用PCR-DGGE研究冷卻豬肉貯藏過程中的優勢菌.西北農林科技大學學報(自然科學版), 2008, 36(9): 185-189.
LI M Y, ZHOU G H, XU X L. Study on the dominant bacteria during the storage period on chilled pork using PCR-DGGE., 2008, 36(9): 185-189. (in Chinese)
[21] OLSSON C, AHRNE S, PETTERSSON B, MOLIN G. The bacterial flora of fresh and chill-stored pork: Analysis by cloning and sequencing of l6S rRNA genes., 2003, 83(3): 245-252.
[22] GILL C O, DUSSAULT F, HOLLEY R A, HOUDE A, JONES T. Evaluation of the hygienic performances of the processes for cleaning,dressing and cooling pig carcasses at eight packing plants., 2000, 58(1/2): 65-72.
[23] DAINTY H, MACKEY B M. The relationship between the phenotypic properties of bacteria from chilled-stored meat and spoilage processes., 1992, 73(supplement s21): 103s-114s.
[24] LABADIE J. Consequences of packaging on bacterial growth: Meat is an ecological niche., 1999, 52(3): 299-305.
[25] ERCOLINI D, RUSSO F, BLAIOTTA G, PEPE O, MAURIELLO G.Simultaneous detection of, andfrom meat by use of a multiplex PCR assay targeting the carA gene., 2007, 73(7): 2354-2359.
[26] ARNAUT-ROLLIER I, ZUTTER L D, HOOF J V. Identities of thespp. in flora from chilled chicken., 1999, 48(2): 87-96.
[27] JYDEGARRD-AXELSEN A M, H?IBY P E, HOLMSTROM K, ROSSELL N, KN?CHEL S. CO2and anaerobiosis-induced changes inphysiology and gene expression of different Listeriamonocytogenes strains., 2004, 70(7): 4111-4117.
[28] 勵建榮, 劉永吉, 朱軍莉, 張旭光, 傅俏, 趙羽, 周小敏, 李鈺金. 真空、空氣和氣調包裝對冷藏魚糜制品品質的影響. 水產學報, 2011, 35(3): 446-455.
LI J R, LIU Y J, ZHU J L , ZHANG X G, FU Q, ZHAO Y, ZHOUX M, LI Y J. Influence of vacuum packaging, air packaging and modified atmosphere Packaging on the quality of refrigerated surimi-based product., 2011, 35(3): 446-455. (in Chinese)
[29] STAMATIS N N, ARKOUDEL S J. Quality assessment of Scomber colias japonicus under modified atmosphere and vacuum packaging., 2007, 18(4): 292-300.
[30] CHANTARASATAPOM P, TEPKASIKUL P, KINGCHA Y, YOKSAN R, PICHYANGKURA R. Water-based oligochitosan and nanowhisker chitosan as potential food preservatives for shelf-life extension of minced pork., 2014, 159(1): 463-470.
[31] 李苗苗, 王江峰, 徐大倫, 張后程, 楊文鴿. 4種保鮮處理對冰溫貯藏金槍魚片生物胺的影響. 中國食品學報, 2015, 15(2): 111-119.
LI M M, WANG J F, XU D L, ZHANG H C,YANG W G. Effects of 4 packaging methods on biogenic amines formation in yellowfin Tuna () fillets under controlled freezing-point temperature., 2015, 15(2): 111-119. (in Chinese)
[32] 孫麗霞. 氣調包裝結合生物保鮮劑對冷藏大黃魚品質及菌相的研究[D]. 杭州: 浙江工商大學, 2014.
Sun L X. The Effect of Modified atmosphere packaging with biological antistaling agent on storage quality of large yellow croaker and bacteria phase [D]. Hangzhou: Zhejiang Gongshang University, 2014. (in Chinese)
[33] 曹榮, 劉淇, 趙玲, 孟輝輝. 基于高通量測序的牡蠣冷藏過程中微生物群落分析. 農業工程學報, 2016, 32(20): 275-280.
Cao R, Liu Q, Zhao L, Meng H H. Microbial flora analysis of oyster during refrigerated storage by high throughput sequencing technology., 2016, 32(20): 275-280. (in Chinese)
(責任編輯趙伶俐)
Metagenomic Analysis of Bacterial Phases of Catfish Fillets under Different Storage Conditions
ZHU YingChun1, Wang Yang2, Fan XiaoPan3, MA LiZhen3, Wang KaiLi1
(1College of Food Science and Engineering, Shanxi Agriculture University, Taigu 030801, Shanxi;2The Department of Aquaculture Science of Tianjin Agriculture University/Tianjin Key Laboratory of Aqua-Ecology and Aquaculture, Tianjin 300384;3The Department of Food Science and Biological Engineering of Tianjin Agriculture University/Tianjin Engineering and Technology Research Center of Agricultural Products Processing, Tianjin 300384)
【Objective】The objective of this experiment is to analyze the dynamic changes of bacterial phase of catfish fillet stored under different storage conditions, evaluate the effect of natural preservative on the special spoilage bacteria. 【Method】There were 8 groups in this experiment including fresh catfish () fillets (CK0), AP (air-package) catfish fillets stored at (4±1)℃ for 4 and 7 d (AP4, AP7),MAP (modified atmosphere package, 60%CO2/40%N2) catfish fillets stored at controlled freezing-point temperature (0.7±0.02)℃ for 10 and 30 d(MAP10,MAP30), MAP catfish fillets added with 5% natural preservative stored at controlled freezing-point temperature (0.7±0.02)℃ for 10, 30 and 40 d (MAPP10, MAPP30 and MAPP40). The natural preservative consists of 0.5% chitosan, 0.1% propolis, 0.075% lysozyme, 0.075% Nisin and 0.5% tea polyphenol. Microbial16S rDNA of different samples were sequenced in metagenomics analysis, bacterial composition and abundance of these groups were compared. Alpha diversity and principal component analysis were carried out to investigate the effect of the natural preservative, package and storage temperature on bacterial phase changing.【Result】Totally 25 phylum, 433 genuses of bacteria were identified in 8 sample groups. The same as CK0, the principal genus in MAP10, MAPP10, MAPP30 was, while it was(70.49%) and(59.01%) in AP4 and AP7, respectively. From the respect of microbiology, MAP + controlled freezing-point temperature was better than AP + low temperature storage. During the storage, the abundance ofincreased sharply from 15.78% (MAP10) to 82.85% (MAP30), while in the MAPP group (MAPP10 and MAPP30) which supplemented with natural preservativesrarely could be detected. 【Conclusion】MAP + controlled freezing-point storage significantly lowered the bacterial abundance and diversity, therefore is useful to prolong the shelf life of catfish fillets. The components of tea polyphenol, Nisin and chitosan in the natural preservative used in this study markedly inhibited the growth and the multiplication ofwhich is the special spoilage bacteria in the MAP fillets stored at controlled freezing point. This is important for improving the security of catfish meat against microbial contamination through precise target inhibition.
catfish fillet; metagenomics; bacteria phase changes; natural preservative
2016-08-01;接受日期:2017-01-05
天津市水產產業技術體系創新團隊項目、山西農業大學博士科研啟動基金(2015ZZ06)
朱迎春,Tel:13835487476;E-mail:yingchun0417@163.com。 通信作者馬儷珍,Tel:18622200780;E-mail:Malizhen-6329@163.com

