烏蘇里擬鲿微衛星文庫的構建及分析
朱傳坤潘正軍王 輝吳 楠常國亮丁懷宇周鳳建,
(1. 淮陰師范學院生命科學學院, 江蘇省特色水產繁育工程實驗室, 淮安 223300; 2. 淮陰師范學院江蘇省區域現代農業與環境保護協同創新中心, 淮安 223300; 3. 江蘇省淮安市水產技術指導站, 淮安 223001)
烏蘇里擬鲿微衛星文庫的構建及分析
朱傳坤1,2潘正軍1,2王 輝1,2吳 楠1,2常國亮1,2丁懷宇1,2周鳳建2,3
(1. 淮陰師范學院生命科學學院, 江蘇省特色水產繁育工程實驗室, 淮安 223300; 2. 淮陰師范學院江蘇省區域現代農業與環境保護協同創新中心, 淮安 223300; 3. 江蘇省淮安市水產技術指導站, 淮安 223001)
烏蘇里擬鲿(Pseudobagras ussuriensis)又名烏蘇里(Leiocassis ussuriensis), 隸屬于鯰形目, 鲿科, 擬鲿屬,主要分布于我國東北地區, 但在我國其他各大水系也有分布[1]。烏蘇里擬鲿肉質細嫩、味道鮮美、風味獨特、適口性好、市場價值高, 深受廣大消費者和養殖戶青睞。在20世紀80年代, 烏蘇里擬鲿的天然捕撈量曾僅次于同科的黃顙魚(Pelteobagrus fulvidsco), 然而由于捕撈強度的持續增長、環境污染及生境破壞等因素的影響,其野生資源量銳減, 目前在很多自然分布區已很難捕獲野生個體[2]。
目前已有較多針對烏蘇里擬鲿的研究報道, 其中大多數研究集中于其人工繁殖[3,4]、人工養殖[5]、營養及生理[6,7]等方面, 關于其遺傳學的相關研究則鮮有報道, 僅有少量利用同工酶、RAPD、線粒體DNA及SRAP等標記對其進行群體遺傳多樣性分析的研究報道[8—11]。微衛星標記是由2—6堿基為重復單元的串聯重復序列, 根據串聯重復連續與否, 可分為完美型、非完美型及復合型微衛星三類[12]。微衛星標記不僅在真核生物基因組中分布廣泛, 而且具有多態性高、可重復性強及共顯性遺傳等優點[12], 在水產動物遺傳學和基因組學研究中被廣泛應用[13]。然而, 目前尚無利用微衛星標記對烏蘇里擬鲿進行種質資源和遺傳結構分析的相關報道, 原因可能主要是由于該物種微衛星標記的缺乏。因此, 本研究采用磁珠富集法構建了烏蘇里擬鲿基因組微衛星富集文庫,并對文庫的微衛星富集效率及特征進行了評估和分析,同時, 從中開發了一批多態性微衛星標記。本研究結果將為今后烏蘇里擬鲿中基于微衛星標記的種質資源保護、遺傳圖譜構建及分子標記輔助育種等研究工作的開展奠定基礎。
1 材料與方法
1.1 樣本采集及DNA抽提
本研究所用32尾烏蘇里擬鲿樣本均于2015年5月采自江蘇省淮安市水產技術指導站, 每尾個體剪取尾鰭組織并保存于無水乙醇中, 基因組DNA抽提采用傳統的苯酚-氯仿法進行[14]。
1.2 基因組酶切及片段篩選
取50 ng/μL的基因組DNA 10 μL, 加入5 U限制性內切酶Mse I (NEB), 于37℃水浴中消化3h, 酶切產物用1.0%的瓊脂糖凝膠進行電泳, 并利用DNA純化試劑盒(康為世紀)回收純化250—800 bp的DNA片段。之后, 將等體積的上、下游接頭引物Mse I-A: 5′-GACGATGAG TCCTGAG-3′和Mse I-B: 5′-TACTCAGGACTCAT-3′混合, 并于95℃變性5min, 緩慢冷卻至室溫后, 即制備成終濃度50 μmol/L的連接接頭。利用T4 DNA連接酶(Promega)將所回收的DNA片段和新制備的連接接頭于16℃生化培養箱中過夜連接。連接產物稀釋10倍后利用引物Mse I-N: 5′-GATGAGTCCTGAGTAAN-3′進行預擴增, PCR反應體系總體積為25 μL, 包含: MseI-N引物(10 μmol/L) 0.8 μL, 10×Taq Buffer (不含Mg2+) 2.5 μL, MgCl2(25 mmol/L) 1.6 μL, dNTPs (10 mmol/L) 0.4 μL, 模板DNA 1 μL, Taq DNA聚合酶(Promega) 0.2 μL (5 U/μL),滅菌超純水18.5 μL。PCR反應條件為: 94℃預變性3min, 94℃變性1min, 53℃退火1min, 72℃延伸1min, 經N (N=14、17、20、23、27、30)個循環, 確定最佳循環次數為17; 72℃延伸5min, 通過1.0%瓊脂糖凝膠電泳后, 再次進行切膠和純化, 從而獲得大量250—800 bp的DNA片段。
1.3 生物素探針雜交
取1.2中PCR預擴增純化產物29 μL與1 μL生物素標記的(GT)13探針(5′-bio-GTGTGTGTGTGTGTGTGTGT GTGTGT, 100 μmol/L)及70 μL雜交緩沖液(6×SSC+0.1% SDS)配制成總體積100 μL的雜交反應體系, 于95℃變性5min, 65℃退火1h, 再緩慢冷卻至室溫, 從而使預擴增產物中含微衛星的片段和(GT)13探針充分雜交。
1.4 磁珠富集
取150 μL鏈霉親和素包被的磁珠(Promega), 用 300 μL TEN100溶液(10 mmol/L Tris-HCl, 1 mmol/L EDTA, 100 mmol/L NaCl, pH 7.5)在室溫下洗滌3次, 每次5min,清洗后用300 μL的TEN100溶液懸浮磁珠。將1.3中獲得的雜交產物和預處理的磁珠在室溫下混勻, 放置30min進行磁珠雜交反應。磁珠雜交完成后利用磁力架固定磁珠, 移去雜交混合液。用400 μL TEN1000緩沖液(10 mmol/L Tris-HCl, 1 mmol/L EDTA, 1 mol/L NaCl, pH 7.5)在室溫下對磁珠進行非嚴謹洗脫, 共洗脫3次, 每次5min。之后, 用洗滌液400 μL (0.2×SSC+0.1% SDS)于室溫下對磁珠進行3次嚴謹洗脫, 每次5min。最后, 加入50 μL TE緩沖液(pH=8.0)于沸水中處理磁珠5min, 并迅速固定磁珠吸取上清液, 之后重復進行一次洗脫, 最終獲得兩次洗脫的單鏈目的DNA片段。
1.5 目的片段的擴增和回收
分別以兩次洗脫獲得的單鏈目的DNA片段為模板, MseI-N為引物, 對目的片段進行擴增以獲得雙鏈目的DNA片段, PCR擴增體系和程序同1.2。將兩次洗脫片段的預擴增產物通過1.0%的瓊脂糖凝膠電泳后, 切取250—800 bp的片段并利用DNA純化試劑盒進行純化。
1.6 目的片段的克隆及陽性克隆篩選
將1.5中獲得的純化PCR產物連接入pMD18-T載體中(TaKaRa), 并轉化入DH5α感受態大腸桿菌菌株中, 于含氨芐青霉素的固體LB培養基上進行過夜培養, 從而獲得烏蘇里擬鲿的微衛星富集文庫。
挑取單克隆于150 μL含氨芐青霉素的液體LB培養基中, 并于37℃搖床中以200 r/min速度振蕩培養24h。克隆培養結束后, 利用M13通用引物(-47與-48)對每個克隆進行PCR擴增和1.0%瓊脂糖凝膠電泳檢測, 挑選擴增片段大小在250—800 bp的克隆送測序公司測序。
1.7 微衛星引物設計及多態性篩選
克隆測序結果獲得后, 利用SSRhunter軟件中的默認參數對每條序列進行微衛星搜尋, 并輔以人工查找和校正。對含有微衛星序列的片段, 采用Primer Premier 5.0軟件進行引物設計, 并送上海生工合成。合成好的引物在32尾烏蘇里擬鲿樣本中進行PCR擴增效果檢測及多態性篩選, PCR反應體系總體積為12.5 μL, 包含10×Taq Buffer (含Mg2+)1.3 μL, dNTP 0.4 μL(2.5 mmol/L), 上、下游混合引物0.4 μL(2.5 μmol/L), 模版DNA 1 μL (50 ng /μL), Taq DNA聚合酶(5 U/μL) 0.1 μL, 滅菌超純水9.3 μL。PCR產物利用10%聚丙烯酰胺凝膠進行電泳分型, 以PBR322/MspI(天根生物)作為標準DNA, 凝膠通過溴化乙錠(EB)染色及凝膠成像系統拍照后進行多態性判斷和分析。
2 結果
2.1 微衛星富集文庫克隆陽性率檢測
利用菌落計數器估算, 所構建微衛星富集文庫中共獲得克隆約3000個, 隨機挑選其中233個進行了陽性檢測。通過檢測發現, 其中有224個為陽性單克隆, 7個為陽性雙克隆, 2個為假陽性克隆, 因此, 本文庫的陽性率達到99.1%。
2.2 文庫微衛星富集效率
將隨機挑選的208個陽性單克隆進行了測序, 其中16個由于抽提質粒失敗而未獲得測序結果, 最終共獲得序列192條。通過微衛星序列查找后, 發現在192條序列中, 含有微衛星的序列共有162條, 占序列總數的84.4%,即該文庫的微衛星富集陽性率為84.4%。
2.3 微衛星序列分析
本研究獲得的162條含微衛星序列中, 有91條(56.2%)為完美型微衛星序列, 39條(24.1%)為非完美型, 32條(19.8%)為復合型序列。這些序列中共有微衛星位點271個, 平均每條序列含微衛星1.68個, 大部分序列(64.8%)含有一個微衛星位點, 也有少量序列微衛星位點數較多(6.2%), 其中最多的含有10個微衛星位點(圖1A)。重復單元以GT/TG和CA/AC類型為主(75.0%), 但同時也發現了其他兩堿基及三、四堿基重復類型(圖1B), 甚至還發現了重復單元為12—40堿基的6條小衛星DNA序列。微衛星的重復次數介于5—81, 其中重復次數大于10次的有171個(62.9%), 重復次數大于30的有33個(12.2%)(圖 1C),
2.4 微衛星引物設計與多態性篩選
在162條含微衛星的序列中, 有15條側翼序列太短,無法設計引物。在147條含有足夠側翼長度的序列中, 隨機挑選了20條進行引物設計, 并將這20對引物在32尾烏蘇里擬鲿個體中進行了PCR及電泳檢測。結果表明, 20對引物中有17對能擴增出穩定條帶, 其中有8對呈現出良好的多態性(表 1、圖 2), 多態性比例為40.0%。
3 討論
3.1 微衛星文庫富集效率
微衛星標記具有分布廣泛、穩定性好、多態性高、可重復性強、共顯性遺傳等諸多優點, 在群體遺傳結構分析、遺傳背景分析、種質資源評估、基因鑒定、遺傳連鎖圖譜構建、數量性狀位點定位及分子標記輔助育種等方面具有廣泛應用。因此, 微衛星標記的開發是其應用研究的基礎環節, 目前主要可通過4種途徑獲得微衛星標記。(1)磁珠富集法。該方法是目前微衛星標記開發的主流方法, 通過構建微衛星磁珠富集文庫, 進行微衛星標記篩選。其微衛星標記開發效率高, 目的性強, 因此被廣大研究者廣泛采用, 已在鳙(Aristichthys nobilis)[15]、胭脂魚(Myxocyprinus asiaticus)[16]、蘭州鲇(Silurus lanzhouensis)[17]等多種魚類中成功應用。(2)數據庫篩選法。在公共數據庫中下載目的物種已發表的EST或DNA序列,從中進行微衛星標記的篩選。雖然該方法具有成本低、耗時短及工作量較小的優點, 但對于公共數據庫中序列資源貧乏的物種來說, 該方法并不適用。(3)跨種擴增法。通過近緣物種已發表的微衛星標記的跨種擴增來篩選目的物種的微衛星標記。該方法具有和公共數據庫篩選法相同的優點, 但該方法的應用前提是近緣物種中已有微衛星標記的報道, 若親緣關系太遠, 則該方法的效率較低。(4)高通量測序法。該方法主要是通過基因組和轉錄組的高通量測序, 從測序序列中篩選微衛星標記, 該方法成本相對較高。由于目前公共數據庫中烏蘇里擬鲿的核苷酸序列極少, 同時, 擬鲿屬的近緣物種中也鮮有微衛星的相關報道, 因此, 采用主流的磁珠富集法構建微衛星文庫, 是在烏蘇里擬鲿中開發微衛星標記經濟、有效的手段。
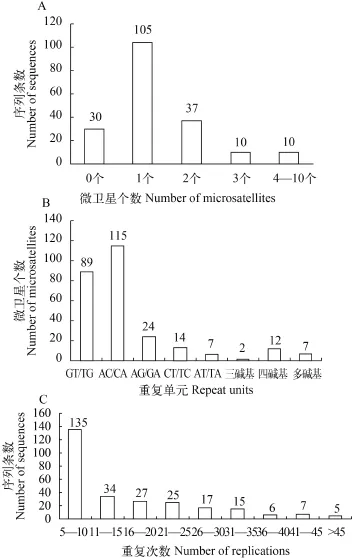
圖 1 烏蘇里擬鲿微衛星文庫中微衛星位點特征分析Fig. 1 Characterization of microsatellites in the enriched library of Pseudobagrus ussuriensisA. 每條序列中含微衛星數目的比例分布; B. 各重復類型微衛星的比例分布; C. 微衛星重復次數頻率分布A. Proportion of microsatellite numbers in each sequence; B. Proportional distribution of microsatellites with different repeat motifs; C. Frequencies of microsatellites with different core motif
已有研究表明, 在真核生物基因組中, (CA)n或(GT)n重復類型的微衛星含量最為豐富[18], 因此, 為獲得更高的微衛星富集效率, 本研究選用(GT)n重復探針類型, 同時為了防止探針長度過長影響雜交效率, 采用長度適中的(GT)13作為雜交探針進行富集文庫構建。本研究構建的微衛星富集文庫的微衛星富集效率達到了84.4%,與合浦珠母貝(Pinctada fucata)(83.2%)[19]類似, 低于銀鯽(Carassius auratus gibelio)(95.3%)[20], 但高于大多數已報道水產動物中的富集效率, 如胭脂魚(40.0%)[16]、蘭州鲇(63.0%)[17]、鱖(Siniperca chuatsi)(66.7%)[21]、刀鱭(Coilia ectenes)(69.4%)[22]等。影響陽性率的因素有很多, 如實驗所用試劑、操作者的熟練程度及洗脫溫度和時間等差異都可能影響最終的陽性率; 另外, 探針的類型也是影響陽性率的重要因素, 若所用探針的微衛星重復類型在目的物種基因組中的豐度太低或重復次數不合理, 都會影響雜交效率, 進而影響微衛星富集效率。因此, 為提高富集效率, 有學者提倡采用多種探針進行微衛星富集文庫的構建[23], 該方法已在多種水產中得到應用[17,24,25]。本研究雖然以GT重復類型作為探針, 但獲得的微衛星中除GT/CA重復類型外, 也發現了其他重復類型, 如CT/AG及三、四堿基重復類型, 類似結果在其他水產動物中均有報道[16, 22, 26]。
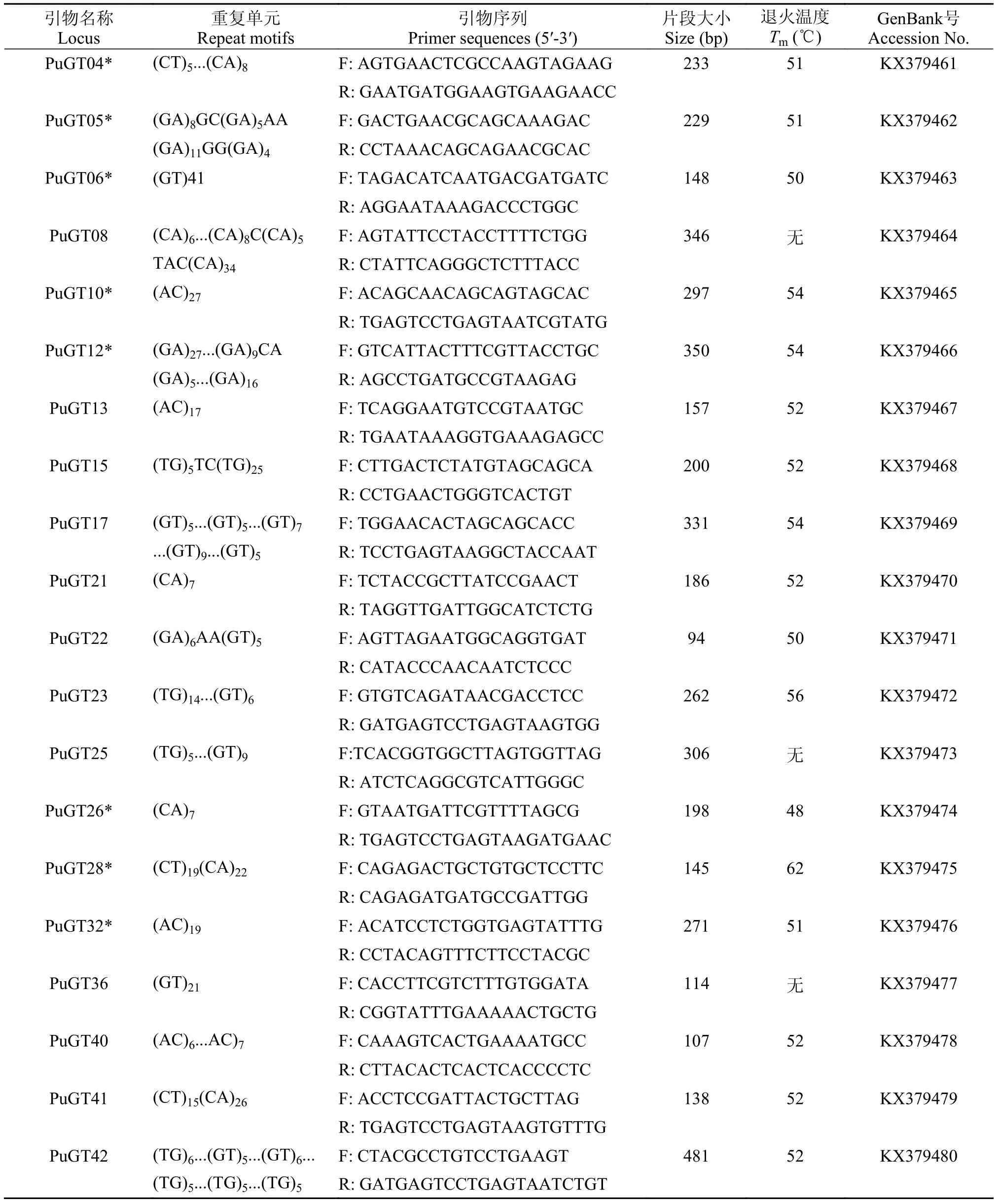
表 1 20個烏蘇里擬鲿微衛星位點的引物信息Tab. 1 Information for 20 microsatellite loci of Pseudobagras ussuriensis
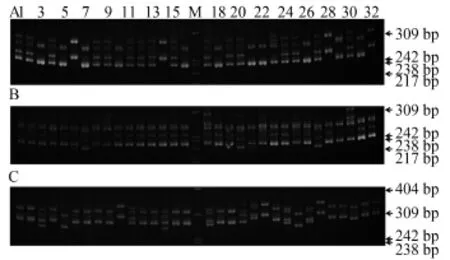
圖 2 微衛星文庫中3個多態性標記在32尾烏蘇里擬鲿中的電泳檢測圖譜Fig. 2 Electrophoretic results of 3 polymorphic microsatellites in 32 individuals of Pseudobagrus ussuriensisA. PuGT04電泳圖; B. PuGT05電泳圖; C. PuGT32電泳圖A. Electrophoretogram of PuGT04; B. Electrophoretogram of
3.2 微衛星組成特點
本研究獲得的162條含微衛星序列中, 完美型、非完美型和復合型所占比例分別為56.2%、24.1%和19.8%,可見完美型微衛星為主要類型, 該結果也與蘭州鲇[17]、黑斑原鰷(Glyptosternum maculatum)[26]、中國鱟(Tachypleus tridentatus)[27]及合浦珠母貝[19]等水產動物具有相似的微衛星組成特點。由于微衛星本身的結構特點, 其復制滑動及點突變頻率均較高[28,29], 完美型微衛星可能主要由核心重復區的復制滑動產生, 而非完美型則可能是由核心重復區的點突變導致, 相對于點突變, 復制滑動的概率可能更大些, 因此完美型微衛星的比例在已報道物種中總是最高的。
一般來說, 重復次數低于5 的微衛星很少會檢測出多態性[30], 因此, 微衛星位點的核心重復次數至少為5次,重復次數越多, 該位點的多態性也就越高[12], 但在真核生物中微衛星核心重復大多小于30次, 如本研究中重復次數小于30次的微衛星所占比例為87.8%。因此, 微衛星核心重復次數可反映相應位點的多態性信息, 然而, 重復次數過多會導致測序結果側翼序列過短, 造成引物設計困難, 本研究中15條無法設計引物的序列多數屬于此類情況。在隨機合成的20對引物中, 有17對可擴增出穩定清晰的目的條帶, 其中8對在32尾烏蘇里擬鲿中表現為多態性, 引物擴增成功率高于刀鱭[22]、蘭州鲇[17]及太湖新銀魚(Neosalanx taihuensis)[24]等物種, 多態性比例與蘭州鲇[17]相當, 高于刀鱭[22]、寬口光唇魚(Acrossocheilus monticola)[25]等物種, 低于太湖新銀魚[24]等。影響擴增成功率的主要原因可能在于引物設計的成功與否, 而引物多態性比例則和物種本身的多樣性及用于多態性檢測的樣本的遺傳背景相關。
4 結論
本研究首次構建了烏蘇里擬鲿微衛星富集文庫, 該文庫不僅克隆陽性率高, 而且微衛星陽性率也處于較高水平, 因此, 利用該文庫可以達到高效開發微衛星標記的目的。本研究結果將有助于推動烏蘇里擬鲿的遺傳結構分析、遺傳連鎖圖譜構建、數量性狀位點定位及分子標記輔助育種等研究工作的進程。
[1]Chu X L, Zheng B S, Dai D Y, et al. Fauna Sinica [M]. Beijing: Science Press. 1999, 60 [褚新洛, 鄭葆珊, 戴定遠, 等. 中國動物志. 北京: 科學出版社. 1999, 60]
[2]Liu Q, Zuo Z Y, Wang Z T, et al. Molecular and morphologic identification of wild Pseudobagrus ussuriensis in Qinhe River and morphologic comparison with wild and cultured populations in the Heilongjiang River Basin [J]. Journal of Hydroecology, 2016, 37(2): 76—82 [劉青, 左中原, 王志濤, 等. 山西沁河烏蘇里擬鲿形態、分子鑒定及與黑龍江群體比較. 水生態學雜志, 2016, 37(2): 76—82]
[3]Wang M H, Cai Y X, Chen X H, et al. Growth comparison of offsprings in Ussuri bullhead Pseudobagrus ussuriensis and darkbarbled catfish Pelteobagrus vachelli and their reciprocal hybrids [J]. Fisheries Science, 2013, 32(1): 50—54 [王明華, 蔡永祥, 陳校輝, 等. 烏蘇里擬鲿、 瓦氏黃顙魚雜交與自交子代生長性能比較. 水產科學, 2013, 32(1): 50—54]
[4]Cui K K, Miao J C, Li H M, et al. Study on artificial breeding technology of Pseudobagras ussuriensis [J]. Hebei Fisheries, 2012, 219: 23—25 [崔寬寬, 苗建春, 李賀密, 等. 烏蘇里的人工繁育技術研究. 河北漁業, 2012, 219: 23—25]
[5]Wu Y D. Technology on nurturing of fingerlings and food fish production of Pseudobagras ussuriensis using net cages [J]. Scientific Fish Farming, 2016, (5): 37—39 [吳宜東. 烏蘇里擬鲿魚種培育和網箱養殖技術探討. 科學養魚, 2016, (5): 37—39]
[6]Wang Y Y, Zhou Q C, Bu X Y, et al. Effects of replacing fish meal with soybean meal on growth performance, body composition and apparent digestibility coefficients of Pseudobagrus ussuriensis [J]. Acta Hydrobiologica Sinica, 2016, 40(4): 681—689 [王裕玉, 周歧存,卜憲勇,等. 飼料中不同含量豆粕對烏蘇里擬鲿生長、體成分及表觀消化率的影響. 水生生物學報, 2016, 40(4): 681—689]
[7]Yang G, Wang X H, Hu Y H, et al. Adaptability of Pseudobagras ussuriensis to Salinity and Alkalinity [J]. Journal of Tianjin Agricultural University, 2012, 19(3): 32—35 [楊廣, 王心華, 胡玉花, 等. 烏蘇里擬鲿對鹽度、堿度的適應性. 天津農學院學報, 2012, 19(3): 32—35]
[8]Pan W Z, Yin H B, Sun Z W, et al. Isozyme of Pseudobagrus ussuriensis in Heilongjiang Water System [J]. Jour-nal of Northeast Forestry University, 2006, 34(6): 66—69 [潘偉志, 尹洪濱, 孫中武, 等. 烏蘇里擬鲿(Pseudobagrus ussuriensis)同工酶分析. 東北林業大學學報, 2006, 34(6): 66—69]
[9]Chen Y M, Chen X H, Pan Y, et al. RAPD analysis of genetic variation between Pelteobagrus vachelli (♀) and Pseudobagrus ussuriensis (♂) and their hybrids [J]. Journal of Shanghai Ocean University, 2010, 19(1): 12—18 [陳友明, 陳校輝, 潘瑩, 等, 江黃顙(♀)和烏蘇里擬鲿(♂)及其雜交子代遺傳變異的 RAPD分析. 上海海洋大學學報, 2010, 19(1): 12—18]
[10]Wu J, Zhang Y, Huo T B, et al. Genetic structure of Pseudobagrus ussuriensis from Heilongjiang and Songhua River based on mtDNA sequences [J]. Freshwater Fisheries, 2013, 43(2): 16—20 [吳靜, 張研, 霍堂斌,等. 利用線粒體序列分析黑龍江和松花江流域烏蘇里擬鲿種群遺傳結構. 淡水漁業, 2013, 43(2): 16—20]
[11]Xu H F, Huang H Z, Fan W S, et al. Genetic diversity analyses of wild and cultured Pseudobagrus ussuriensis populations [J]. Marine Sciences, 2011, 35(3): 17—22 [徐汗福, 黃鶴忠, 范皖蘇, 等. 烏蘇里擬鲿野生群體和人工養殖群體遺傳多樣性的比較研究. 海洋科學, 2011, 35(3): 17—22]
[12]Weber J L. Informativeness of human (dC-dA)n.(dG-dT)n polymorphisms [J]. Genomics, 1990, 7(4): 524—530
[13]Sun X W, Zhang X F, Zhao Y Y, et al. Development and application of microsatellite markers in aquatic species [J]. Journal of Fishery Sciences of China, 2008, 15(4): 689—703 [孫效文, 張曉鋒, 趙瑩瑩, 等. 水產生物微衛星標記技術研究進展及其應用. 中國水產科學, 2008, 15(4): 689—703]
[14]Sambrook J, Russell D W. Molecular Cloning: A Laboratory Manual, 3rd ed [M]. UK: Cold Spring Harbor Laboratory Press. 2001, 463—464
[15]Cheng L, Liu L, Yu X, et al. Sixteen polymorphic microsatellites in bighead carp (Aristichthys nobilis) and cross-amplification in silver carp (Hypophthalmichthys molitrix) [J]. Molecular Ecology Resources, 2008, 8(3): 656—658
[16]Yang Z, Shi F, Que Y F, et al. Construction of microsatellite library enriched and its application prospect in Chinese sucker (Myxocyprinus asiaticus) [J]. Journal of Hydroecology, 2009, 2(2): 107—112 [楊鐘, 史方, 闕延福, 等. 胭脂魚微衛星富集文庫的構建及其應用前景展望. 水生態學雜志, 2009, 2(2): 107—112]
[17]Wei D W, Lian Z Q, Wu X D, et al. Construction and identification of (CAG)n and (GATA)n microsatellites from the Silurus lanzhouensis genome [J]. Freshwater Fisheries, 2014, 44(5): 15—20 [魏大為, 連總強, 吳旭東,等. 蘭州鲇(CAG)n、(GATA)n微衛星文庫構建與鑒定.淡水漁業, 2014, 44(5): 15—20]
[18]Brenner S, Elgar G, Sandford R, et al. Characterization of the pufferfish (Fugu) genome as a compact model vertebrate genome [J]. Nature, 1993, 366(6452): 265—268
[19]Qu N N, Gong S Y, Huang G J, et al. Isolation and screening of microsatellite markers from the Chinese pearl oyster Pinctada fucata based on FIASCO [J]. Journal of Tropical Oceanography, 2010, 29(3): 47—54 [曲妮妮, 龔世園, 黃桂菊, 等. 基于FIASCO 技術的合浦珠母貝微衛星標記分離與篩選研究. 熱帶海洋學報, 2010, 29(3): 47—54]
[20]Ji C H, Sun X W. Rapid isolation of microsatellites from genome of silver crucian carp by magnetic beads [J]. Journal of Dalian Fisheries University, 2007, 22(6): 460—464 [姬長虹, 孫效文. 用磁珠富集法快速制備銀鯽微衛星標記. 大連水產學院學報, 2007, 22(6): 460—464]
[21]Kuang G Q, Liu Z, Lu S Q, et al. Isolation and characterization of microsatellite loci from Siniperca chuatsi [J]. Journal of Fishery Sciences of China, 2007, 14(4): 608—614 [匡剛橋, 劉臻, 魯雙慶, 等. FIASCO法篩選鱖魚微衛星標記. 中國水產科學, 2007, 14(4): 608—614]
[22]Deng P P, Shi Y H, Zhang G Y, et al. Isolation of Microsatellite in C. ectenes by Magnetic Beads [J]. Biotechnology Bulletin, 2014, 6: 145—149 [鄧平平, 施永海, 張根玉, 等. 磁珠富集法分離刀鱭微衛星標記. 生物技術通報, 2014, 6: 145—149]
[23]Edwards K J, Barker J H, Daly A, et al. Microsatellite libraries enriched for several microsatellite sequences in plants [J]. BioTechniques, 1996, 20(5): 758—760
[24]Li C Y, Liu H Y, Xiong F. Isolation and Sequence Characterization of Microsatellite loci in Neosalanx taihuensis [J]. Journal of Hydroecology, 2015, 36(2): 55—60 [李存耀, 劉紅艷, 熊飛. 太湖新銀魚微衛星位點的分離與序列特征分析. 水生態學雜志, 2015, 36(2): 55—60]
[25]Liu R C, Zhang F T, Dan S G, et al. Isolation and characterization analysis of microsatellite loci in Acrossocheilus monticola [J]. Sichuan Journal of Zoology, 2013, 32(2): 161—166 [劉瑞成, 張富鐵, 但勝國, 等. 寬口光唇魚微衛星位點的篩選與特征分析. 四川動物, 2013, 32(2): 161—166]
[26]Guo B Y, Xie C X, Qi P Z, et al. Construction and identification of DNA libraries enriched for microsatellite repeat sequences of Glyptosternum maculatum [J]. Acta Hydrobiologica Sinica, 2011, 35(6): 908—912 [郭寶英, 謝從新, 祁鵬志, 等. 黑斑原鮡微衛星DNA 富集文庫構建與鑒定. 水生生物學報, 2011, 35(6): 908—912]
[27]Ning Y F, Li Z B, Dai G, et al. Construction and identification of enriched microsatellite DNA libraries from Tachypleus tridentatus [J]. Marine Sciences, 2015, 39(4): 15—20 [寧曄鳳, 黎中寶, 戴剛, 等. 中國鱟基因組微衛星富集文庫的構建及分析. 海洋科學, 2015, 39(4): 15—20]
[28]Weber J L, Wong C. Mutation of human short tandem repeats [J]. Human Molecular Genetics, 1993, 2(8): 1123—1128
[29]Schlotterer C, Tautz D. Slippage synthesis of simple sequence DNA [J]. Nucleic Acids Research, 1992, 20(2): 211—215
[30]Valdes A M, Slatkin M, Freimer N B. Allele frequencies at microsatellite loci: the stepwise mutation model revisited [J]. Genetics, 1993, 133(3): 737—749
CONSTRUCTION AND ANALYSIS OF AN ENRICHED MICROSATELLITE LIBRARY IN PSEUDOBAGRAS USSURIENSIS
ZHU Chuan-Kun1,2, PAN Zheng-Jun1,2, WANG Hui1,2, WU Nan1,2, CHANG Guo-Liang1,2, DING Huai-Yu1,2and ZHOU Feng-Jian2,3
(1. Jiangsu Engineering Laboratory for Breeding of Special Aquatic Organisms, School of Life Science, Huaiyin Normal University, Huai’an 223300, China; 2. Jiangsu Collaborative Innovation Center of Regional Modern Agriculture & Environmental Protection, Huaiyin Normal University, Huai’an 223300, China; 3. Huai’an Fisheries Technical Guidance Station, Huai’an 223001, China)
烏蘇里擬鲿; 磁珠富集文庫; 微衛星
Pseudobagras ussuriensis; Magnetic beads-enriched library; Microsatellite
Q781
A
1000-3207(2017)02-0473-06
10.7541/2017.59
2016-06-15;
2016-10-24
江蘇省區域現代農業與環境保護協同創新中心項目(HSXT307); 淮陰師范學院博士科研啟動項目(31ZCK00); 國家自然科學基金項目(31602146)資助 [Supported by Jiangsu Collaborative Innovation Center of Regional Modern Agriculture & Environmental Protection (HSXT307), the Start-up Funds of Scientific Research for Dr. C. Zhu from Huaiyin Normal University (31ZCK00) and the National Natural Science Foundation of China (31602146)]
朱傳坤(1984—), 男, 山東臨沂人; 講師; 主要從事魚類基因組學與分子標記輔助育種研究。E-mail: zhuchuankun@hytc.edu.cn

