團頭魴MSTN 基因cDNA 結構、表達及過表達對胚胎發育的影響
杜尚可 沈睿杰 陳 杰 蔣霞云 鄒曙明
(上海海洋大學農業部淡水水產種質資源重點實驗室, 上海 201306)
團頭魴MSTN 基因cDNA 結構、表達及過表達對胚胎發育的影響
杜尚可 沈睿杰 陳 杰 蔣霞云 鄒曙明
(上海海洋大學農業部淡水水產種質資源重點實驗室, 上海 201306)
研究通過cDNA末端快速擴增法(RACE)克隆得到團頭魴生長抑制素(MSTN)基因的cDNA全長并分析了MSTN基因在團頭魴胚胎、成魚組織中表達以及MSTN基因在胚胎中過表達情況。結果表明團頭魴MSTN基因的cDNA全長為2187 bp, ORF(開放閱讀框)大小為1128 bp, 編碼376個氨基酸。組織逆轉錄PCR (RT-PCR)結果顯示, MSTN基因在肌肉、腦和精巢組織中大量表達, 肝臟、脾臟和卵巢組織中的少量表達, 腸、腮、心、眼和腎組織中的微量表達。胚胎逆轉錄PCR (RT-PCR)結果顯示, 在0—44 hpf胚胎發育階段, MSTN基因表達量較低; 而在48—52 hpf胚胎發育階段, MSTN基因表達量逐漸升高。整胚原位雜交(WISH)結果顯示, 胚胎發育的16 hpf時期MSTN基因主要在脊索中表達, 胚胎發育的28 hpf和55 hpf時期MSTN基因在腦中表達。MSTN基因過表達結果顯示, 胚胎在體節發生期出現前-后軸拉長, 背-腹軸變短; 脊索發生扭曲, 強烈抑制體節發育而導致不分化等現象。研究為后續團頭魴MSTN基因的功能研究及團頭魴分子育種提供相關參考依據。
MSTN基因; 團頭魴; 原位雜交; 過表達
肌肉生長抑制素(Myostatin, MSTN), 又稱GDF8 (Growth differentiation factor 8, 生長分化因子 8),屬于TGF-β (Transforming growth factor β, 轉化生長因子β)家族的成員[1]。該基因抑制骨骼肌的生長、發育。1997年, MSTN首次在小鼠中被發現, 之后鳥類[2]和牛、豬等哺乳動物中[3,4]也克隆得到了該基因。截止目前為止, 魚類中斑馬魚[5]、刀鱭[6]、牙鲆[7]、草魚[8]等十多個物種的MSTN基因已被克隆[9—12]。但關于團頭魴MSTN基因功能、表達的研究卻鮮有報道。
蛋白酶酶切MSTN前體蛋白, 形成N端前肽和C端活性成熟肽, 酶切之后的N端前肽與C端活性成熟肽仍以非共價結合方式存在[13]。有研究表明, MSTN前肽不僅對MSTN成熟肽二聚體的準確形成有重要的作用, 還能夠通過與成熟肽結合, 抑制MSTN基因與其受體結合, 阻斷MSTN基因功能[14]。MSTN成熟肽二聚體與受體結合激活細胞內Smad2/ 3蛋白磷酸化, 并與Smad4蛋白形成復合體, 再與靶基因的DNA調控序列結合, 進而促進或抑制相關基因的轉錄表達[15]。利用轉基因[16]、Morpholino[17]或RNAi[18]等技術, 已證實MSTN的突變或轉錄表達降低能促進斑馬魚肌肉的生長和發育。
團頭魴(Megalobrama amblycephala)草食性, 從20世紀60年代開始, 團頭魴就作為我國重要的淡水養殖種類[19], 2014年的總產量達7.8×108kg[20]。本研究克隆得到團頭魴MSTN基因全長cDNA的序列,并采用RT-PCR研究分析MSTN mRNA在組織、胚胎中的表達情況; 采用整胚原位雜交(WISH)方法研究MSTN mRNA在胚胎中的空間分布、表達情況; MSTN mRNA過表達對團頭魴胚胎發育的影響通過胚胎顯微注射的方法來研究。本研究可為團頭魴MSTN基因的功能研究及團頭魴分子育種提供相關參考依據。
1 材料與方法
1.1 實驗用魚、胚胎
本研究所需的團頭魴雌雄親魚為上海海洋大學濱海基地團頭魴遺傳育種中心培育的“浦江1號”。所用胚胎為開展人工授精所得, 胚胎發育過程中, 每隔2—3h換水1次。受精卵置于室溫下人工培養、孵化, 每隔一段時間取成活胚胎50個, 在顯微鏡下觀察發育時相, 然后將其放置于有RNA store保存液中以備后續提取總RNA。提取組織所需團頭魴成魚為2齡魚, 體重約500 g。
1.2 不同發育時期胚胎和成魚不同組織總RNA的提取
活體剖殺團頭魴成魚提取組織, 迅速置于液氮中快速冷凍。使用Trizol (Invitrogen)法提取經高速組織研磨儀研磨過的胚胎和成魚組織中總RNA。使用瓊脂糖凝膠電泳檢測提取的總RNA的質量, 使用紫外分光光度計(Bio-Rad, 美國)檢測提取的總RNA的濃度, 胚胎及各組織總RNA置于–80℃冰箱中低溫保存。
1.3 MSTN基因全長cDNA的獲得
根據斑馬魚(Danio rerio)MSTN基因序列設計一對特異性引物(mstn-F和mstn-R, 表 1)。以團頭魴肌肉組織總RNA反轉錄獲得的cDNA為模板, PCR擴增MSTN基因小片段。3′-和5′-RACE擴增使用SMART RACE cDNA amplification kit (Clontech公司), 按試劑盒說明設計RACE和巢式引物(3′-mstn、3′-mstn nest、5′-mstn和 5′-mstn nest, 表 1), PCR擴增3′-和5′-RACE目的片段[21], 所用引物均由上海生工生物技術公司合成。
巢式擴增產物經過割膠回收(使用凝膠回收膠試劑盒割膠回收目的片段PCR產物)、連接[將上述PCR產物與pMD19-T (TaKaRa) 載體連接構建重組質粒]、轉化(將重組質粒轉化到感受態細胞DH5α中)、克隆(含氨芐青霉素的LB平板培養)、測序(使用瓊脂糖凝膠電泳檢測插入特定片段的重組子, 所獲得的陽性克隆由上海生工生物技術有限公司進行測序)[8]。測序結果用BioEdit7.0 軟件中的Clustal W程序比對, 去掉重疊、序列兩端引物部分, 拼接得到團頭魴MSTN基因cDNA全長序列。
1.4 序列比較及進化樹分析
采用BLAST (http://www.ncbi.nlm.nih.gov/ BLAST)、Clustal W軟件進行核苷酸序列驗證、翻譯及氨基酸序列相似性分析。尋找正確的開放閱讀框使用ORF (Open reading frame) finder程序(http:// www.ncbi.nlm.nih.gov/projects/gorf)。應用Compute pI-Mw程序預測氨基酸序列等電點、Sigal P 3.0 server預測氨基酸序列相對分子質量和信號肽[21]。用Expasy在線軟件進行蛋白翻譯。用SMART (http://smart.embl-heidelberg.de/)預測結構域。運用Clustal W[22]比對序列, 用MEGA 5.05軟件構建NJ (Neighbor- joining)系統進化樹[23]。
1.5 逆轉錄PCR (RT-PCR)分析
根據已克隆出的團頭魴MSTN和魚類18s基因序列信息, 設計團頭魴MSTN RT-PCR引物(MSTN RT-F和MSTN RT-R, 表 1)和18sRT-PCR引物(18sRTF和18s RT-R, 表 1)。以18s基因為內參, 對團頭魴不同時期胚胎和成魚不同組織進行RT-PCR分析MSTN基因mRNA在團頭魴組織、胚胎中的表達。
1.6 整胚原位雜交(WISH)
根據已克隆出的團頭魴MSTN基因序列, 跨越ORF和3′-UTR區域設計一對引物, 表 1 中引物(mstn-in situ F/R)。擴增出大小為877 bp的目的片段, 將目的片段與pGM-T (TaKaRa) 載體連接。使用特定限制性內切酶(SacⅡ)37℃體外進行線性化酶切, 回收的酶切產物體外經RNA聚合酶(Promega)轉錄獲得WISH所需RNA探針。
WISH所需胚胎, 使用3%的胰蛋白酶浸泡去除卵膜并將其置于4%的PFA中, 4℃保存24h。固定好的胚胎使用含有0.1% Tween-20的磷酸緩沖液(PBS)進行沖洗, 再轉移到100%的甲醇中, 于–20℃放置1d以上, 4℃長期保存。WISH所使用的RNA核糖核酸探針采用地高辛標記, 胚胎與探針在60℃雜交超過16h, 與PBST-SB抗(抗地高辛抗體)與AP (堿性磷酸酶)偶聯, 通過羅氏BM染料產生紫色不溶沉淀。顯色完成后的胚胎在尼康SMZ1500顯微鏡下進行拍照。
1.7 團頭魴胚胎顯微注射
依據克隆出的團頭魴MSTN基因全長序列, 設計過表達引物(表 1, mstn cds-F和mstn cds-R), 以團頭魴肌肉組織cDNA為模板進行PCR擴增。使用TIAN quick Maxi Purification Kit回收PCR擴增產物, 用限制性內切酶BamHⅠ和XhoⅠ (Promega)體外進行酶切, 將酶切產物轉化入pCS2載體中(本實驗室保存中)。經凝膠電泳檢測合適的重組pCS2-MSTN質粒送由生工生物技術公司測序、檢測。使用限制性內切酶NotⅠ對檢測合適的重組pCS2-MSTN質粒進行酶切, 酶切產物采用mMessage mMachine kit (Ambion, TX, USA)體外轉錄獲得5′端加帽mRNA[24]。
顯微注射裝置由PV830 Pneumatic PicoPump顯微注射器和SMZ-445體式顯微鏡(Nikon)組成, 裝置由氮氣氣壓驅動, 過轉錄表達MSTN mRNA濃度為500 pg/nL, 每次注射團頭魴1—2細胞期胚胎卵黃(靠近細胞部分)的量為1 nL。注射好的胚胎置于胚胎培養液中室溫下孵化, 顯微鏡下觀察胚胎的發育情況并拍照[24]。
2 結果
2.1 團頭魴MSTN基因cDNA序列分析
本研究克隆獲得團頭魴MSTN基因全長cDNA序列(KY072939)。其cDNA序列全長2187 bp, 包括88 bp的5′ UTR (非編碼區), 971 bp的3′ UTR (非編碼區)。ORF大小為1128 bp, 可編碼375個氨基酸,推測的MSTN前體蛋白等電點為5.02, 相對分子質量為92.34 kD。團頭魴MSTN基因編碼蛋白包含有兩大TGF-β蛋白結構域, 也有RXXR蛋白酶水解位點RIRR; 還有9個保守的半胱氨酸殘基(圖 1)。
2.2 同源性及分子進化分析
利用CLUSTAL W比對團頭魴、斑馬魚、草魚和人類氨基酸序列分析(圖 1), 團頭魴氨基酸序列與草魚和斑馬魚MSTN1氨基酸序列的相似度分別為99%和97%, 而與草魚和斑馬魚MSTN2氨基酸序列的相似度分別為68%和70%, 與人類氨基酸序列的相似度為68%。氨基酸序列分析結果表明, 克隆得到的團頭魴MSTN與草魚、斑馬魚MSTN 1具有較高的相似度。Neighbor-joining系統進化樹(圖 2)結果表明, 克隆得到的團頭魴MSTN與草魚、斑馬魚MSTN 1和鯉MSTN 1a, 1b型緊密聚為一支, 由此可以得知克得到的團頭魴MSTN基因為1型MSTN基因。
2.3 團頭魴MSTN mRNA在不同時期胚胎和成魚不同組織中的表達分析
以18s基因為內參, 用表 1中引物(MSTN RTF/R和18s F/R)對團頭魴成魚腦、腸、肝胰臟、鰓、心臟、眼睛、腎臟、肌肉、脾臟、精巢和卵巢等11個組織進行RT-PCR分析。結果顯示MSTN mRNA在所有11個組織中均有表達, 但組織間表達量存在差異。肌肉、腦和精巢中的大量表達, 肝胰臟、脾臟和卵巢少量表達, 而在其他組織中微量表達(圖 3A)。
以18s基因為內參, 用表 1中引物(MSTN RTF/R和18s F/R)對團頭魴0—52 hpf不同時期胚胎進行RT-PCR分析。結果顯示團頭魴MSTN mRNA在胚胎發育的各個時期均有表達, 0—44 hpf微量表達, 4—52 hpf表達量逐漸升高(圖 3B)。
2.4 WISH (整胚原位雜交)結果分析
團頭魴胚胎WISH (整胚原位雜交)結果(圖 4)表明: 在16 hpf時, MSTN mRNA在16 hpf在脊索中表達(圖 4A); 在28 hpf時, MSTN mRNA在眼睛和整個腦部(前腦、間腦、中腦、中腦-后腦邊界、后腦)表達(圖 5B); 而在55 hpf時, MSTN mRNA僅在眼睛、前腦和間腦中表達(圖 4C)。對照組均無信號表達(圖 4D, E, F)。
2.5 MSTN mRNA過表達結果分析

圖 1 團頭魴與人類、斑馬魚、草魚MSTN氨基酸序列比對及結構分析Fig. 1 Alignment of the deduced amino acid sequences of grass carp duplicated MSTNs with homologs from zebrafish, grass carp and humansRXXR蛋白酶水解位點黑色下劃線表示, 黑色邊框為TGF-β前肽結構域, 波浪邊框為TGF-β或類TGF-β結構域保守的半胱氨酸殘基用黑色箭頭表示RXXR hydrolysis sites underlined black, black border for the TGF-β propeptide domain, a wave border for the TGF-β or TGF-β type domain conserved cysteine, residues with a black arrow
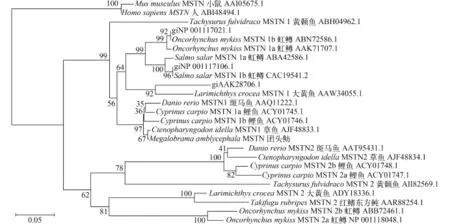
圖 2 團頭魴MSTN基因氨基酸序列構建的NJ系統進化樹Fig. 2 Neighbor-Joining phylogenetic tree of blunt snout bream MSTN putative peptides in vertebrates團頭魴MSTN用下劃線標出, 數值代表置信的百分比The blunt snout bream MSTN is indicated by underline; Numbers of branches are percentage of times that the two clades branched as sisters
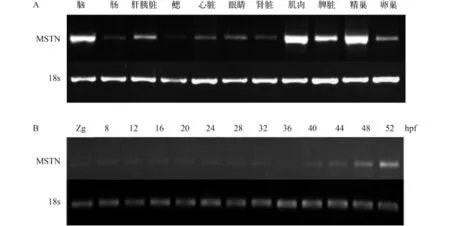
圖 3 團頭魴MSTN在成體不同組織(A)和胚胎不同時期(B)的表達情況Fig. 3 The MSTN mRNA level in adult tissues (A) and during embryogenesis (B) in Megalobrama amblycephala
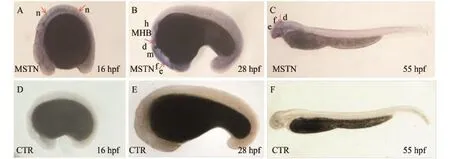
圖 4 團頭魴MSTN mRNA在胚胎時期的整胚原位雜交結果Fig. 4 Whole-mount embryo in situ hybridization analysis of MSTN mRNA during different embryonic stages in blunt snout breamA、B和C. MSTN反義探針; D、E和F. MSTH正義探針; n. 脊索; e. 眼睛; f. 前腦; d. 間腦; MHB. 中腦、后腦邊界; h. 后腦The MSTN antisense probe (A, B and C), the sense probe (D, E and F); n. notochord; e. eyes; f. forebrain; d. midbrain; MHB. midhindbrain; h. hindbrain
團頭魴MSTN mRNA過表達結果(圖 5), 16 hpf后團頭魴胚胎發育出現明顯異常, 在體節發生期顯示為前-后軸伸長, 背-腹軸縮短, 包被層萎縮, 體型呈紡錘形(圖 5A–H); 脊索和肌節的生長發育受到強烈抑制, 并造成脊索的輕微扭曲(圖 5H)。顯微注射結果表明, MSTN mRNA過表達對團頭魴胚胎背-腹軸和體節的形成過程造成重大的影響。在12 hpf,對照組、注射組的存活率分別為91, 6%和20, 7%;在16 hpf, 對照組、注射組的存活率分別為 75.4%和8.6%; 17 hpf時注射組胚胎全部死亡。
3 討論
本研究克隆獲得團頭魴MSTN基因全長cDNA序列, 序列全長2187 bp; ORF編碼375個氨基酸, 屬于TGF-β超家族, 有的RIRR蛋白水解位點和9個保守的半胱氨酸殘基。或許是由于魚類第三輪全基因組復制的緣故, 一些魚類研究表明存在兩個MSTN基因[25,26]。序列比對結果顯示, 團頭魴MSTN基因氨基酸序列與斑馬魚、草魚MSTN基因氨基酸序列相似性較高, 而與人類MSTN基因氨基酸序列相似性較低, 表明MSTN基因在長期的進化中高度保守。團頭魴MSTN與斑馬魚MSTN 1的相似性高達97%, 與斑馬魚MSTN 2的相似率僅為66%, 表明研究克隆得到的團頭魴MSTN應為1型基因。
研究小鼠發現, MSTN基因只在某一特定組織(骨骼肌)中大量表達, 少數組織(脂肪、心肌、乳腺)中少量表達[1], 而魚類中MSTN基因卻廣泛表達[27]。團頭魴組織RT-PCR結果顯示, 所有檢測組織中MSTN mRNA均有表達, 但組織間MSTN mRNA表達量存在明顯差異; 據此推斷, 團頭魴MSTN基因除調節肌肉生長和發育外, 可能還存在其他的功能[17,28]。團頭魴不同發育時期的胚胎RT-PCR結果顯示, MSTN mRNA在0—44 hpf的表達量較低, 48—52 hpf表達量逐漸升高。前期檢測到的MSTN mRNA或許是少量保留的母源性轉錄本, 后期檢測到的MSTN mRNA可能由于團頭魴胚胎發育36h后體節基本發育完成, 激活相關肌肉特異性基因的轉錄表達[29], 因而導致MSTN mRNA轉錄表達量逐漸升高。Vianello等[30]報道了斑馬魚一細胞期就檢測到MSTN mRNA, 8 hpf降低; 而在16 hpf, MSTN mRNA表達水平顯著增加, 由此可以驗證前期檢測到的MSTN mRNA是由少量保留的母源性轉錄本而來, 后期是由于胚胎發育過程中肌肉細胞分化而引起的MSTN mRNA表達量的上升。
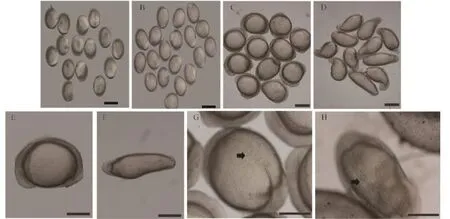
圖 5 MSTN過表達對團頭魴胚胎發育的影響Fig. 5 Overexpression of MSTN causes obvious embryonic abnormalities in Megalobrama amblycephala at 12 hpf and 16 hpfA為發育至12h對照組胚胎, B為發育至12h過表達胚胎; C-H均為發育至16h胚胎; C、E和G為正常對照組; D、F和H為MSTN過表達組; 黑箭頭指示體節位置; 短線長度均為600 μmA are the wildtype embryos that developed at 12 h; B are MSTN mRNA-injected embryos that developmented at 12 h; C-H are all embryos that developmented at 16h; C, E and G are the wildtype embryos; D, F and H are MSTN mRNA-injected embryos. Dark arrows show the somites. Scale bar=600 μm
WISH結果顯示, 胚胎發育早期MSTN mRNA廣泛表達, 在脊髓中表達最明顯; 這與草魚MSTN mRNA的早期12 hpf胚胎原位雜交結果是一致的。團頭魴MSTN mRNA在中期28 hpf和晚期55 hpf胚胎中主要集中在腦中表達; 與草魚MSTN mRNA中期胚胎中在腦中和尾節中; 晚期胚胎中在腦和脊索中表達結果不同[31,32]; 而斑馬魚MSTN mRNA的表達與團頭魴、草魚MSTN mRNA完全相反, 斑馬魚MSTN mRNA在早期表達信號微弱, 在中、后期表達強烈[33];表明在不同種魚之間MSTN mRNA的表達存在差異。團頭魴MSTN mRNA過表達造成表型發生變化, 主要體現在胚胎前-后軸伸長, 背-腹軸縮短, 使整個胚胎成紡錘形, 這與BMPs蛋白受到抑制后引起的胚胎形態變化非常相似[34,35], 這或許是由于MSTN基因的調控作用通過Smad蛋白發揮的同時,促進抑制性Samd蛋白的表達, 抑制了BMPs信號轉遞的結果[36]。本研究開展了團頭魴MSTN基因結構、功能、表達以及過表達研究, 可為下階段MSTN基因的morpholino基因敲降或crispr/cas9敲除研究MSTN功能性缺失對胚胎發育的影響打下良好基礎。
[1]McPherron A C, Lawler A M, Lee S J. Regulation of skeletal muscle mass in mice by a new TGF-beta superfamily member [J]. Nature, 1997, 387(6628): 83—90
[2]Yang W, Wang H X. cDNA cloning, expression and purification of protein of GDF-8 in chicken [J]. Chinese Journal of Biotechnology, 2001, 17(4): 460—462 [楊威,王海霞. 雞生長分化因子GDF-8 cDNA的克隆表達及蛋白質純化. 生物工程學報, 2001, 17(4): 460—462]
[3]Li S H, Xiong Y Z, Zheng R. The polymorphism and SNPs research of myostatin gene in pig (porcine) [J]. Journal of Genetica and Genomics, 2002, 29(4): 326—331 [李邵華, 熊遠著, 鄭嶸. 豬MSTN基因多態性及其SNPs的研究. 遺傳學報, 2002, 29(4): 326—331]
[4]Grober L, Martin L J R, Poncelet D, et al. A deletion in the bovine myostatin gene causes the double-muscled phenotype in cattle [J]. Nature Genetics, 1997, 17(1): 71—74
[5]Amali A A, Lin C J, Chela Y. Up-regulation of musclespecific transcription factors during embryonic somhogenesis of zebrafish (Danio rerio) by knock-down of myostafin-1 [J]. Developmental Dynamics, 2003, 229: 847—856
[6]Du F K, Nie Z J, Xu G C, et al. Cloning and tissue expression of the MSTN gene in Coilia nasus [J]. Journal of Fishery Sciences of China, 2014, 4: 684—692 [杜富寬,聶志娟, 徐鋼春, 等. 刀鱭MSTN基因的克隆及其組織表達. 中國水產科學, 2014, 4: 684—692]
[7]Xu J Y, Cheng S L. Cloning and expression analysis of the myostatin (MSTN) gene in Paralichthys olivaceus [J]. Journal of Fisheries of China, 2008, 32(4): 497—506 [徐建勇, 陳松林. 牙鲆肌肉生長抑制素(MSTN)基因克隆.水產學報, 2008, 32(4): 497—506]
[8]Pu J W, Sun C F, Jiang X Y, et al. Two cDNAs cloning, expression and overexpression in embryo of myostatin from grass carp (Ctenopharyngodon idellus) [J]. Biotechnology Bulletin, 2011, 8: 153—160 [濮劍威, 孫成飛, 蔣霞云, 等. 草魚兩個肌肉生長抑制素cDNA克隆、表達及過量表達對胚胎發育的影響. 生物技術通報, 2011, 8: 153—160]
[9]Maccatrozzo L, Bargelloni L, Radaelli G, et al. Characterization of the myostatin gene in the gilthead seabream (Sparus aurata): sequence, genomic structure, and expression pattern [J]. Marine Biotechnology, 2001, 3(3): 224—230
[10]Rodgers B D, Weber G M, Sullivan C V, et al. Isolation and characterization of moystatin complementary deoxyribonucleic acid clones from two commercially important fish: Oreochromis mossambicus and Morone chrysops [J]. Endocrinology, 2001, 142: 1412—1418
[11]Rescan P Y, Jutel I, Ralliere C. Two myostatin genes are differentially expressed in myotomal muscles of the trout (Oncorhynchus mykiss) [J]. The Journal of Experimental Biology, 2001, 204(20): 3523—3529
[12]Ostbye T K, Galloway T F, Nielsen C, et al. The two myostatin genes of Atlantic salmon (Salmo salar) are expressed in a variety of tissues [J]. European Journal of Biochemistry, 2001, 268(20): 5249—5257
[13]Lee S J, McPherron A C. Regulation of myostatin activity and muscle growth [J]. Proceedings of the National Academy of Sciences of the United States of America, 2001, 98(16): 9306—9311
[14]Hill J J, Davies M V, Pearson A A, et al. The myostatin propeptide and the follistatin-related gene are inhibitory binding proteins of myostatin in normal serum [J]. The Journal of Biological Chemistry, 2002, 277(43): 40735—40741
[15]Zhu X, Topuzis S, Liang L, et al. Myostatin signaling through Smad2, Smad3 and Smad4 is regulated by the inhibitory Smad7 by a negative feedback mechanism [J]. Cytokine, 2004, 26(6): 263—272
[16]Xu C, Wu G, Zohar Y, et al. Analysis of myostatin gene structure, expression and function in zerbafish [J]. Journal of Experimental Biology, 2003, 206(22): 4067—4079
[17]Amali A A, Lin C J F, Chen Y H, et al. Upregulation of muscle-specific transcription factors during embryonic somitogenesis of zebrafish (Danio rerio) by knock-down of myostatin-1 [J]. Developmental Dynamics, 2004, 229(4): 847—856
[18]Acosta J, Carpio Y, Borroto L, et al. Myostatin gene silenced by RNAi show a zebrafish giant phenotype [J]. The Journal of Biology, 2005, 119(4): 324—331
[19]Li S F, Zou S M, Cai W Q, et al. Production of interploid triploids by 4n × 2n blunt snout bream (Megalobrama amblycephala) and their first performance data [J]. Aquaculture Research, 2006, 37(4): 374—349
[20]FBMA. China Fisheries Year Book [C]. Beijing: China Agriculture Press. 2015 [中國漁業年鑒. 北京: 中國農業出版社. 2015]
[21]Zhou C X. Molecular cloning and functional characterization of IGF-1R and duplicated IGFBP-5 in grass carp (Ctenopharyngodon idellus) and SNP analysis of different grass carp’s muscle-growth control genes [D]. Thesis for Master of Science. Shanghai Ocean University, Shanghai, 2014 [周春雪. 草魚胰島素樣生長因子1受體和結合蛋白5的克隆與功能研究及不同草魚群體肌肉生長調控基因SNP分析. 碩士學位論文, 上海海洋大學,上海. 2014]
[22]Larkin M A, Blackshields G, Brown N P, et al. Clustal W and Clustal X version 2.0 [J]. Bioinformatics, 2007, 23(21): 2947—2948
[23]Tamura K, Peterson D, Peterson N, et al. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]. Molecular Biology and Evolution, 2011, 28(10): 2731—2739
[24]Sheng R J. Structural and functional studies of hypoxia inducible factors in Megalobrama amblycephala [D]. Thesis for Master of Science. Shanghai Ocean University, Shanghai. 2011 [沈睿杰. 團頭魴缺氧誘導因子的結構和功能研究. 碩士學位論文, 上海海洋大學, 上海. 2011]
[25]Ostbye T K, Galloway T F, Nislsen C, et al. The two myostatin genes of Atlantic salmon (Salmo salar) are expressed in a variety of tissues [J]. European Journal of Biochemistry, 2001, 268(20): 5249—5357
[26]Robert S B, Goetz F W. Myostatin protein and RNA tran-script levels in adult and developing brook trout [J]. Molecular and Cellular Endocrinology, 2003, 210(1-2): 9—20
[27]Rodgers B D, Weber G M, Sullivan C V, et al. Isolation and characterization of myostatin complementary deoxyribonucleic acid clones from two commercially important fish: Oreochromis mossambicus and Morone chrysops [J]. Endocrinology, 2001, 142(4): 1412—1418
[28]Garikipati D K, Gahr S A, Roalson E H, et al. Characterization of rainbow trout myostatin-2 genes (rtMSTN-2a and -2b): genomic organization, differential expression, and pseudogenization [J]. Endocrinology, 2007, 148(5): 2106—2115
[29]Zhang H W. Developmental Biology (second edition) [M]. Beijing: Higher Education Press. 2006, 178—179 [張紅衛. 發育生物學(第二版). 北京: 高等教育出版社. 2006, 178—179]
[30]Vianello S, Brazzoduro L, Dalla Valle L, et al. Myostatin expression during development and chronic stress in zebrafish (Danio rerio) [J]. The Journal of Endocrinology, 2003, 176(1): 47—59
[31]Zheng G D, Sun C F, Pu J W, et al. Two myostatin genes exhibit divergent and conserved functions in grass carp (Ctenopharyngodon idellus) [J]. General & Comparative Endocrinology, 2016, 214: 68—76
[32]Pu J W. cDNA cloning, expression in embryo of myostain in grass carp (Ctenopharyngodon idellus) [D]. Thesis for Master of Science. Shanghai Ocean University, Shanghai. 2011 [濮劍威. 草魚肌肉生長抑制素克隆及功能研究. 碩士學位論文, 上海海洋大學, 上海. 2011]
[33]Xu C, Wu G, Zohar Y, et al. Analysis of myostatin gene structure, expression and function in zebrafish [J]. Journal of Experimental Biology, 2003, 206(22): 4067—4079
[34]Gan S, Huang Z, Liu N, et al. MicroRNA-140-5p impairs zebrafish embryonic bone development via targeting BMP-2 [J]. FEBS Letters, 2016, 590(10): 1438—1446
[35]Xue Y, Zheng X, Huang L, et al. Organizer-derived Bmp2 is required for the formation of a correct BMP activity gradient during embryonic development [J]. Nature Communication, 2014, 5(4): 3766
[36]Ishisaki A, Yamato K, Hashimoto S, et al. Differential inhibition of Smad6 and Smad7 on bone morphogenetic protein- and activin-mediated growth arrest and apoptosis in B cells [J]. Journal of Biological Chemistry, 1999, 274(19): 13637—13642
MOLECULAR STRUCTURE AND EXPRESSION OF MYOSTATIN IN MEGALOBRAMA AMBLYCEPHALA AND ITS OVEREXPRESSION EFFECTS IN EMBRYO
DU Shang-Ke, SHEN Rui-Jie, CHEN Jie, JIANG Xia-Yun and ZOU Shu-Ming
(Key Laboratory of Genetic Resources for Freshwater Aquaculture and Fisheries, Shanghai Ocean University, Shanghai 201306, China)
The current study cloned a 2187 bp full-length cDNA of myostatin from blunt snout bream (Megalobrama amblycephala) by rapid amplification of cDNA ends (RACE). Its open reading frame is 1128 bp encoding 376 amino acids. RT-PCR analysis demonstrated that MSTN are extensively expressed in tissues of blunt snout bream with high level in the muscle, brain and testis is the highest, modest level in the liver, spleen, and ovary, and low level in intestine, gills, heart, eye, and kidney. During embryos development, mRNA level of MSTN is low from 0 to 44 hpf, whereas its expression increases gradually from 48 hpf to 52 hpf. The whole mount in situ hybridization demonstrated that MSTN mRNA was transcribed at different tissues of blunt snout bream’s embryos. MSTN mRNA were detected at the Notochord at 16 hpf. In 28 hpf and 55 hpf embryos, the MSNT mRNA level was very high in brain. Overexpression MSTN mRNA in embryos caused elongated anterior-posterior axis, shorter dorsal-ventral axis, slightly distorted notochord, and strongly inhibited somites. This study provides knowledge for subsequent blunt snout bream MSTN gene function research and molecular breeding of blunt snout bream.
Myostatin; Megalobrama amblycephala; In situ hybridization; Overexpression
Q344+.1
A
1000-3207(2017)03-0573-08
10.7541/2017.74
2016-07-11;
2016-11-25
國家科技支撐計劃(2012BAD26B00); 國家自然科學基金(31272633和31201760)資助 [Supported by the Key Projects in the National Science & Technology Pillar Program (2012BAD26B00); the National Natural Science Foundation of China (31272633, 31201760)]
杜尚可(1988—), 男, 河南新鄉人; 碩士; 研究方向為魚類遺傳育種。E-mail: dushangke1988@163.com
鄒曙明, 教授, 博導; 研究方向為魚類遺傳育種。E-mail: smzou@shou.edu.cn

