甘薯TD—SSR—PCR反應體系的優化
南文卓 鄭丹煒 朱國鵬 張小貝
摘 要 為探索甘薯最佳TD-SSR-PCR體系,利用L16(43)正交設計研究2×PCR Mix、引物、模板DNA等主要影響因素的適宜濃度,并在此基礎上對擴增程序和聚丙烯酰胺凝膠電泳上樣量進行優化。結果表明,優化后的TD-SSR-PCR反應體系包括:10 μL 2×PCR Mix、100 ng模板DNA、0.4 μmol/L引物,1 μL甘油,總體積20 μL;優化后的擴增程序為:94 ℃預變性4 min,94 ℃變性45 s、Tm+5 ℃~Tm-5 ℃(每循環退火溫度下降0.5 ℃,Tm選用一對引物中的較小Tm值)退火30 s(退火時間因擴增片段大小而異)、72 ℃延伸1 min共20個循環,94 ℃變性45 s、Tm-5 ℃退火30 s、72 ℃延伸1 min共15個循環,最后72 ℃延伸7 min,4 ℃保存;聚丙烯酰胺電泳上樣量以1.5~2 μL為宜。在此條件下,利用引物Z37對10份甘薯材料進行PCR擴增,得到的條帶清晰、多態性高,表明此條件適用于甘薯的TD-SSR-PCR反應體系。
關鍵詞 甘薯;TD-SSR-PCR;正交設計;2×PCR Mix;最佳上樣量
中圖分類號 Q945 文獻標識碼 A
Optimization of TD-SSR-PCR System on Sweet Potato
NAN Wenzhuo,ZHEGN Danwei,ZHU Guopeng*,ZHANG Xiaobei
College of Horticulture and Landscape Architecture,Hainan University,Haikou, Hainan 570228, China
Abstract To explore the best TD-SSR-PCR system on sweet potato, an L16(43)orthogonal design was used to study the suitable concentration of the main influence factors such as 2×PCR Mix, primers and template DNA. And on this basis, the amplification process and the best sample amount of polyacrylamide gel electrophoresis were optimized. The results showed that the optimized 20 μL TD-SSR-PCR reaction system included: 10 μL 2×PCR Mix, 100 ng template DNA, 0.4 μmol/L primers, 1 μL glycerol, total volume is 20 μL; The optimized PCR amplification system started at initial denaturation for 4 min at 94 ℃, followed by 20 cycles of denaturation for 45 s at 94 ℃, anneal for 30 s(Annealing time varied depending on the DNA fragment size)at Tm+5~Tm-5 ℃(annealing temperature decreased 0.5 ℃ per cycle, chose the smaller Tm of a pair of primers as the using one), extension for 1 min at 72℃, then 15 cycles of denaturation for 45 s at 94 ℃, anneal for 30 s at Tm-5 ℃, extension for 1 min at 72 ℃, the amplification was completed after extension for 7 min at 72 ℃, finally stored at 4 ℃. The best sample amount of polyacrylamide gel electrophoresis was 1.5-2 μL. Under this condition, clear banding pattern with high polymorphic was obtained by using primer Z37 to amplify 10 sweet potato materials. It indicated that this condition was suitable for TD-SSR-PCR of sweet potato.
Key words sweet potato; TD-SSR-PCR; orthogonal design; 2×PCR Mix; the best sample amount
doi 10.3969/j.issn.1000-2561.2017.01.016
甘薯[Ipomoea batatas(L)Lam.]作為主要糧食作物,不論在發達國家還是發展中國家都具有重要意義[1]。甘薯為同源六倍體作物,染色體數量多,遺傳背景復雜[2],且繁殖方式為無性繁殖,嚴重制約甘薯育種和生產的發展。
SSR(Simple sequence repeat)即簡單序列重復,通常又稱為微衛星,是一類主要由1~6個堿基組成的基序串聯重復而成的DNA序列。每個SSR位點兩側一般是相對保守的單拷貝序列,根據這個特點可設計一對特異引物來擴增不同重復次數的SSR序列。SSR標記為共顯性標記,多態性高,并可區分種質資源的純合和雜合狀況。另外SSR在實驗中對DNA模板的用量和質量要求相對較低,結果重復性好[3],相比于AFLP、RAPD、ISSR等分子標記技術具有更高的可靠性[4]。
對甘薯材料進行PCR反應體系優化方面的研究已有報道。宋吉軒[5]和張世安[6]建立了甘薯ISSR-PCR反應體系,雷劍[7]、蒲志剛[8]分別優化了甘薯RAPD-PCR和AFLP-PCR反應體系,而關于甘薯SSR-PCR反應體系的優化則未見報道。多數已報道的其他物種[9-11]的SSR-PCR反應體系優化都是根據單個影響因素進行不同梯度間的組合從而選擇最適合的反應體系,關于現今廣為應用的商品化PCR Mix體系優化研究則相對較少[12-13]。不同研究對象從SSR擴增到最后的電泳,每個步驟都會影響到最終效果。本研究擬利用正交試驗設計對甘薯SSR-PCR反應體系(2×PCR Mix、引物和模板3個因素4個水平)進行優化,并在此基礎上對SSR-PCR反應程序、聚丙烯酰胺凝膠電泳上樣量繼續優化,以期建立一套適用于甘薯TD-SSR-PCR的反應體系。
1 材料與方法
1.1 材料
10份甘薯材料取自海南大學海甸校區試驗基地。
2×PCR Mix為東盛生物產品,其組分包括100 mmol/L KCL、20 mmol/L Tris-HCL、3 mmol/L MgCL2、400 umol Dntps、0.1 U/μL TaqDNA聚合酶、溴酚藍、其他增強劑與穩定劑。
試驗引物ZY5序列Fwd:TCGTCACTTTCTCTC
TCCTG;Rev:GCTCTCCTCCATCTCTTCTG,重復序列為(AG)10。
結論驗證引物Z37序列Fwd:GGCGACTGTAA
TGTGGTGAA;Rev:CGGGAGGTATCTTGGATTGA,重復序列為(CTGCTC)3。引物均由北京三博遠志生物技術有限責任公司合成。
Marker為東盛生物公司的DS 2 000。
1.2 方法
1.2.1 DNA的提取與檢測 采用CTAB法提取基因組DNA,用NanoDrop檢測DNA的濃度和質量,并用瓊脂糖跑膠。將樣品稀釋到100 ng/μL后,置于4 ℃冰箱中保存備用。
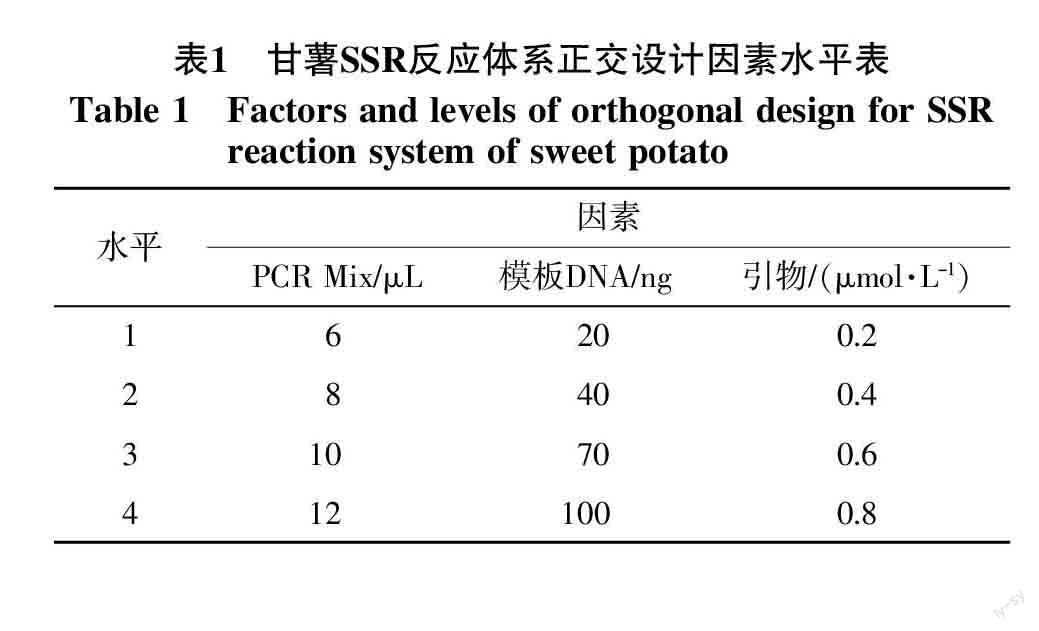
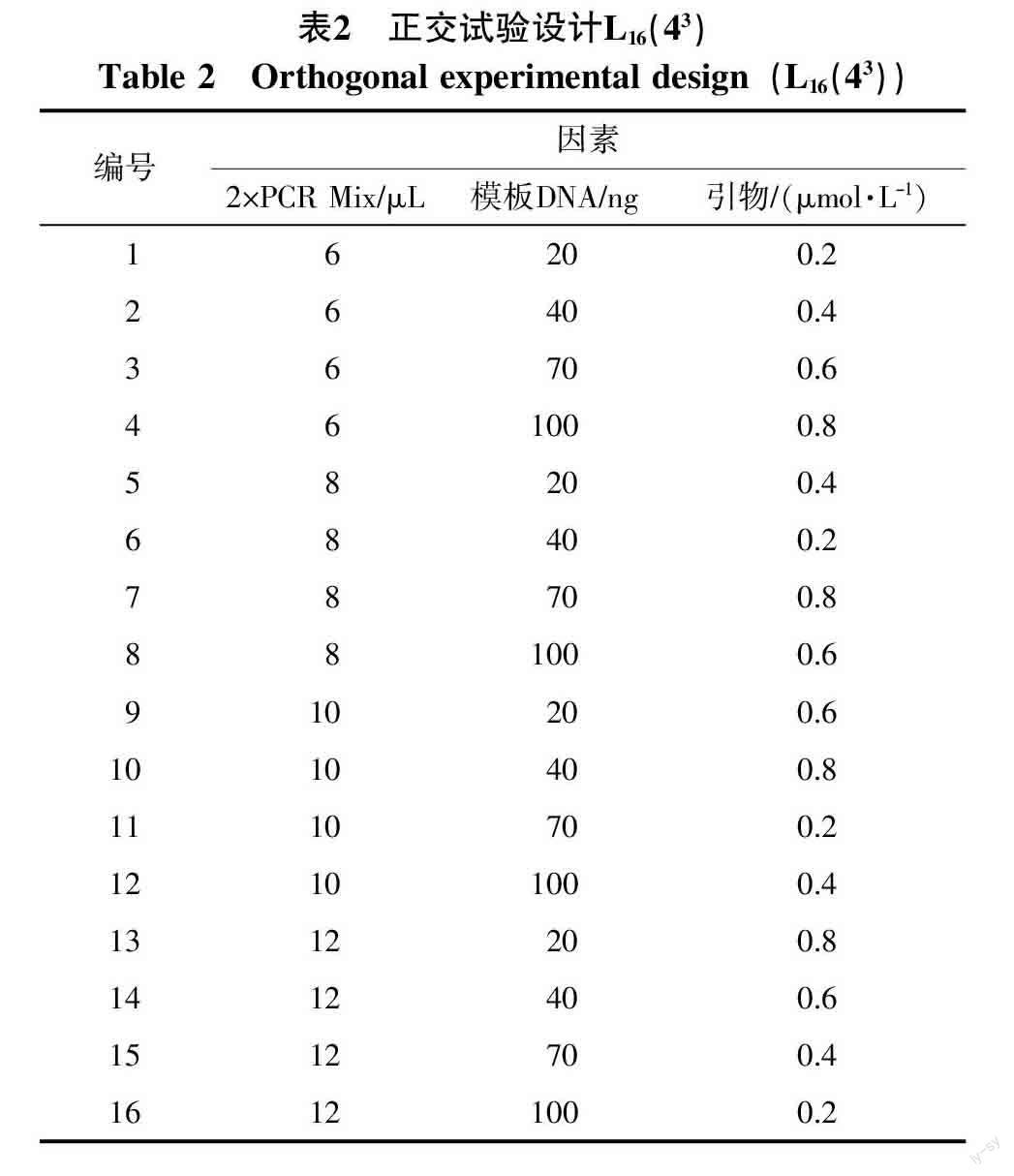
1.2.2 SSR-PCR擴增體系優化 對編號16的甘薯材料設計3因素、4個水平的L16(43)正交表(表1、2),選擇最佳SSR擴增體系。SSR擴增在Heal Force T960 PCR儀上進行。
1.2.3 甘薯SSR-PCR方法的選擇 對優化后的體系分別進行梯度PCR(G-PCR)和降落PCR(TD-PCR)。G-PCR反應程序:94 ℃預變性4 min;94 ℃變性45 s,55 ℃退火30 s(55 ℃為預試驗中確定的最佳退火溫度),72 ℃延伸1 min,共32個循環;然后,72 ℃延伸7 min,4 ℃保存。TD-PCR反應程序:94 ℃預變性4 min,94 ℃變性45 s、Tm+5 ℃~Tm-5 ℃退火30 s(每循環退火溫度降低0.5 ℃,Tm選用一對引物中的較小Tm值)、72 ℃延伸1 min共20個循環,94 ℃變性45 s、Tm-5 ℃退火30 s、72 ℃延伸1 min共12個循環,最后72 ℃延伸7 min,4 ℃保存。
擴增產物在6%非變性聚丙烯酰胺凝膠上檢測,每孔點樣4 μL。所用電泳儀為北京六一電泳儀廠的DYCZ-30C型電泳儀,1.0 mm 40齒梳子,100 V恒壓電泳2 h,銀染顯色,照相保存。
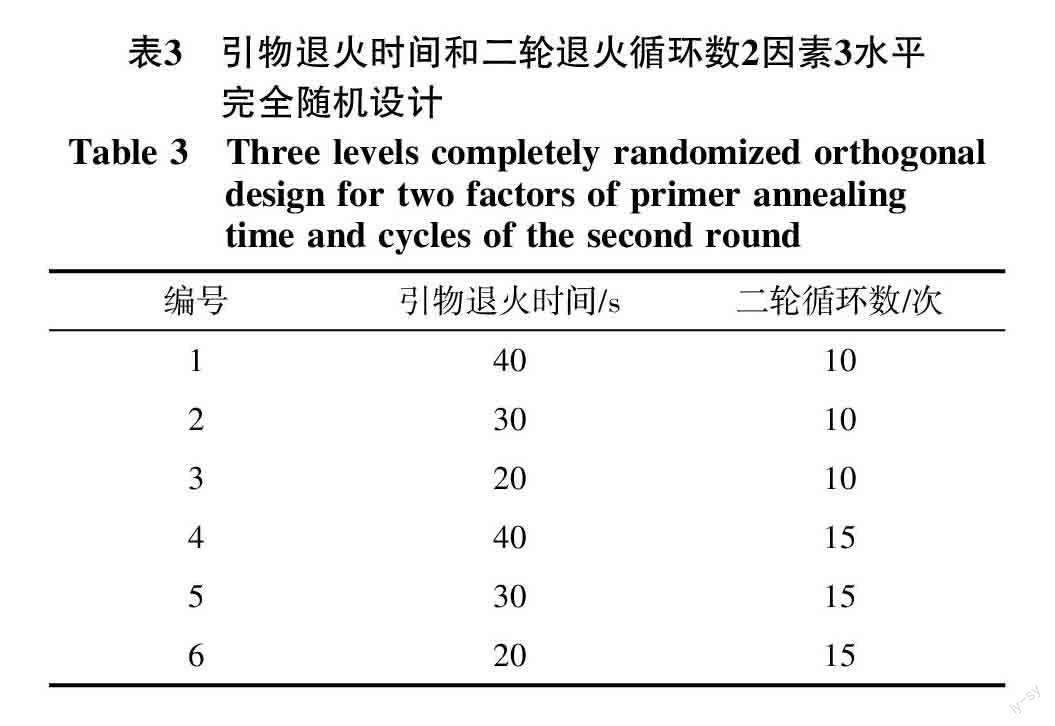
1.2.4 TD-SSR-PCR反應程序的優化 以一對上下游引物的較低Tm±5 ℃值為范圍對10份甘薯材料進行TD-PCR。對退火時間和二輪循環次數(一輪循環退火溫度漸降,二輪循環退火溫度不變)2個因素設置3個水平完全隨機設計,選擇最優TD-SSR-PCR組合。
1.2.5 聚丙烯酰胺凝膠電泳上樣量的優化 利用優化后的TD-SSR-PCR最佳反應體系對上樣量進行優化,共設置5個梯度,分別為1、1.5、2、2.5、3 μL,選擇最佳上樣量。
2 結果與分析
2.1 正交設計SSR-PCR擴增結果分析
正交設計SSR-PCR反應體系擴增見圖1。從圖1可看出,16個組合都可以擴增出條帶,其中第1、2、5、6、9、13組合擴增條帶較為清晰但敏感性差,且每組相同PCR Mix梯度的條帶隨著DNA含量的增加逐漸變強,說明模板DNA為PCR擴增體系主要影響因素。其余組合敏感性較好,尤其第12組合擴增條帶最為清晰,為本研究最優組合,即20 μL PCR體系包含:10 μL PCR Mix、100 ng模板DNA、0.4 μmol/L引物,5%甘油,其余加ddH2O補齊。
2.2 G-PCR與TD-PCR結果比較
G-PCR與TD-PCR的擴增結果見圖2。由圖2可看出,TD-PCR較G-PCR特異性更高,但部分條帶顏色較淺。考慮到試驗結果可靠性,TD-PCR更適合SSR的擴增,條帶深淺則進一步優化。
2.3 不同TD-SSR-PCR程序擴增結果分析
不同TD-SSR-PCR程序對SSR擴增的影響見圖3。由圖3可看出,在一定循環數和退火時間范圍內,循環數與PCR擴增條帶強度正相關,循環數越多,條帶越強;特異性擴增則與退火時間成負相關,退火時間越短,特異性越強。圖3中的組合1,由于其退火時間較組合2、3長,非特異性擴增較多,組合2和3雖然特異性較強,但產物量較少,條帶較淺。相較而言,組合4~6擴增產物量較多,特異性與非特異性擴增也可以區分,其中組合5、6由于退火時間的縮短,有效抑制了非特異性條帶的擴增,使條帶更加清晰易讀,為較適宜TD-SSR-PCR組合。
2.4 聚丙烯酰胺凝膠不同上樣量的電泳結果分析
不同上樣量對聚丙烯酰胺凝膠電泳的影響見圖4。由圖4可知,上樣量的多少幾乎不影響特異性和非特異性條帶的區分,但是在上樣量為1.5~2 μL時,泳道背景淺且原本堆積成片的較小片段也可以被分離成條,使條帶更加清晰易讀。因此,聚丙烯酰胺電泳建議上樣量為1.5~2 μL。
2.5 TD-SSR-PCR優化體系的驗證
引物Z37的TD-SSR擴增結果見圖5。由圖5可看出,根據優化結果利用引物Z37進行TD-SSR-PCR擴增時,退火30 s和20 s都可擴增出條帶,但退火30 s時較大片段更加清晰。與引物ZY5更適合的20 s之所以造成差異可能是擴增片段大小不一,擴增片段大則所需退火時間長,反之則短。
3 討論
在優化PCR反應條件時需考慮到各因素對反應的影響。梁玉琴等[12]在優化柿樹反應體系中發現影響PCR反應最顯著的因素是Mix,而本研究中模板DNA含量為最主要影響因素,可能與柿樹為喬木而甘薯為草本植物有關。李玉恒等[14]對大鼠RNA的擴增結果表明,TD-PCR省略了G-PCR摸索最適退火溫度的過程,且其靈敏度和特異性均高于G-PCR;張瑞強等[15]發現相同的反應體系,TD-PCR更有益于提高PCR特異性,這些觀點在本研究中也得到驗證。
本研究證明TD-PCR是甘薯PCR擴增較好的選擇,但其起始退火溫度的升高在提高PCR擴增特異性的同時,也提高了引物結合難度,降低了擴增效率。一般普通PCR的擴增30個循環即可,但本研究同樣循環數的TD-PCR產物卻較少,電泳條帶較淺。故使用TD-PCR時應根據試驗需要適當增加循環數,得到足夠產物量。
本研究預試驗發現,PCR的優化有時很難區分設計的各組合的優劣,對此建議:①優化PCR反應體系,可先利用稍低的退火溫度,溫度太高可能某些特異性的條帶也消失,條帶結果無二,難以區分;溫度稍低雖然有非特異性條帶產生,但容易區分組合優劣。②優化PCR退火時間,要根據體系中的酶延伸速度和擴增片段大小適當選擇,然后根據結果決定是否繼續優化。③優化聚丙烯酰胺凝膠電泳最佳上樣量,參考本研究設計梯度,擇優選用。本研究中,3 μL上樣量也可讀帶,但泳道背景深,若增加樣本數量,則整個電泳圖會顯得雜亂。反之,1.5~2 μL時泳道背景清亮,一目了然,且長度接近的較小片段也可很好地區分。究其原因,進行SSR-PCR的擴增最重要的是引物,特異性高的引物不用繁瑣的優化也可得到漂亮且可靠的條帶,特異性低的引物則需要一步步的優化以達到理想的效果。
4 結論
利用不同分子標記對不同物種進行擴增時使用的反應體系各有差異,對所研究物種建立穩定的PCR反應體系是深入分子研究的基礎。本研究建立了適用于甘薯材料的20 μL TD-SSR-PCR反應體系:10 μL 2×PCR Mix、100 ng模板DNA、0.4 μmol/L引物,1 μL甘油,其余加ddH2O補齊;優化后的反應程序為:94 ℃預變性4 min,94 ℃變性45 s、Tm+5 ℃~Tm-5 ℃(每循環退火溫度下降0.5 ℃,Tm選用一對引物中的較小Tm值)退火30 s(使用DNA聚合酶聚合速度約1 kbp/min,退火時間因DNA片段大小而異)、72 ℃延伸1 min共20個循環,94 ℃變性45 s、Tm-5 ℃退火30 s、72 ℃延伸1 min共15個循環,最后72 ℃延伸7 min,4 ℃保存;聚丙烯酰胺電泳上樣量以1.5~2 μL為宜。
參考文獻
[1] 馬劍鳳, 程金花, 汪 潔, 等. 國內外甘薯產業發展概況[J]. 江蘇農業科學, 2012(12): 1-5.
[2]唐 茜, 何鳳發, 王季春, 等. 甘薯SRAP遺傳圖譜構建及淀粉含量QTL初步定位[J]. 西南大學學報(自然科學版), 2010, 32(6): 40-45.
[3] 方宣鈞, 無為人, 唐紀良. 作物DNA標記輔助育種[M]. 北京: 科學出版社, 2000.
[4] Schlotterer C. Opinion: The evolution of molecular markers-just a matter of fashion[J]. Nature Reviews Genetics, 2004, 5(1): 63-69.
[5] 宋吉軒, 雷尊國, 黃 團, 等. 甘薯ISSR-PCR反應體系的優化[J]. 貴州農業科學, 2009(11): 25-26, 29.
[6] 張安世, 張利民, 劉 瑩. 均勻設計優化甘薯ISSR-PCR反應體系[J]. 焦作師范高等專科學校學報, 2015(1): 66-68.
[7] 雷 劍, 楊新筍. 甘薯RAPD-PCR反應條件的優化[J]. 湖北農業科學, 2009(10): 2 343-2 346.
[8] 蒲志剛, 唐 靜, 鐘昌松, 等. 甘薯AFLP分子標記體系建立[J]. 分子植物育種, 2006(S1): 40-44.
[9] 謝文剛, 張新全, 彭 燕, 等. 鴨茅SSR-PCR反應體系優化及引物篩選[J]. 分子植物育種, 2008(2): 381-386.
[10] 李亞利, 陳書霞, 孟煥文, 等. 利用正交設計優化黃瓜的SSR-PCR反應體系[J]. 西北農業學報, 2008(3): 280-284.
[11] 范小寧, 林 萍, 張盛周. 油茶SSR-PCR反應體系的優化研究[J]. 安徽農業科學, 2011(23): 14 098-14 102.
[12]梁玉琴, 李芳東, 傅建敏, 等. 正交設計優化柿屬植物SSR-PCR反應體系[J]. 經濟林研究, 2011(4): 17-22.
[13] 李 季, 黃天帶, 華玉偉, 等. 多重PCR技術在橡膠樹轉基因分子鑒定中的應用[J]. 熱帶作物學報, 2014, 35(5): 882-889.
[14] 李玉恒, 趙明慧, 趙紫陽, 等. 利用梯度PCR和降落PCR擴增CIRP基因的比較[J]. 黑龍江八一農墾大學學報, 2011(5): 46-49, 75.
[15] 張瑞強, 繁 萍, 張紅見, 等. 利用降落PCR擴增KMT-1基因[J]. 青海大學學報(自然科學版), 2010(2): 1-3.

