一種加權K—均值基因聚類算法
姚登舉+詹曉娟+張曉晶

摘要:針對微陣列表達數據集中基因-基因之間存在復雜相關關系的問題,基于隨機森林變量重要性分數,提出了一種新的加權K-均值基因聚類算法。首先,以微陣列表達數據中的樣本為對象、基因為特征,訓練隨機森林分類器,計算每個基因的變量重要性分數;然后,以基因為對象、樣本為特征、基因的變量重要性分數為權重進行K-均值聚類。在Leukemia、Breast、DLBCL等3個微陣列表數據集上進行了實驗,結果表明:所提出的加權K-均值聚類算法與原始的K-均值聚類算法相比,類間距離與總距離的比值平均高出177個百分點,具有更好的同質性和差異性。
關鍵詞:微陣列表達數據;聚類分析;隨機森林;K-均值
DOI:1015938/jjhust201702021
中圖分類號: TP391
文獻標志碼: A
文章編號: 1007-2683(2017)02-0112-05
Abstract:In view of the complex correlation between gene and gene in the microarray data set, a weighted K mean gene clustering algorithm based on random forest variable importance score was proposed First, the proposed algorithm begins with training random forest classifier on the microarray data, using the samples as objects and the genes as features, variable importance scores were calculated for each gene; then, a weighted Kmeans clustering were performed with genes as objects, samples as features, and variable importance score as weighted value Experiments were carried out on Leukemia, Breast and DLBCL three datasets The experimental results show that the proposed weighted K mean clustering algorithm has an average of 177 percentage points higher than the original K mean clustering algorithm with respective to the ratio of the distance between the class and the total distance and has better homogeneity and difference
Keywords:microarray expression data; clustering analysis; random forest; Kmeans
0引言
聚類是將物理或抽象對象的集合分組為由類似的對象組成的多個集合的過程,其中屬于同一個集合的對象之間彼此相似,屬于不同集合的對象之間彼此相異[1]。聚類是機器學習和數據挖據中的重要研究內容,被廣泛應用于經濟、管理、地質勘探、圖像識別、生物醫學、生物信息學等領域中[2-6]。隨著高通量測序技術(Highthroughput Sequencing)的迅速發展,各物種的基因表達數據(Gene expression data)出現了爆炸式增長,同時大量的基因表達數據能夠在公共數據庫(如由美國NCBI管理和維護的GEO數據庫、由美國斯坦福大學管理和維護的SMD數據庫、由歐洲EBI管理和維護的ArraryExpress數據庫和由日本多所大學合作提供的CGED數據庫等)中得到[7-11]。在基因表達數據分析任務中,基因聚類分析有著非常廣泛的應用。當前,基因聚類分析方法主要有三類:基于基因的聚類(Genebased clustering)、基于樣本的聚類(Samplebased clustering)和兩路聚類(Biclustering)[12,13]。基于基因的聚類將基因看成聚類的對象,將樣本看成描述基因的特征,表達模式類似的基因(即共表達的基因,Coexpression gene)通常被劃分為同一類,一般具有相同的功能,因此可以根據聚類中已知基因的功能推斷某些未知基因的功能;基于樣本的聚類則以基因為特征,以樣本為對象,通過樣本聚類,可以發現樣本的顯性結構(Phenotype structure),自動對病理特征或實驗條件進行分類;兩路聚類是指同時對基因和樣本進行的聚類,目的是找出在某些條件下參與調控的基因聚類以及與某些基因相關聯的條件,從而更精確、更細致地探索基因和樣本間的相互關系。
基因聚類的主要對象是基因表達微陣列數據。原始的基因表達微陣列數據中存在著大量的冗余基因、噪聲基因和不相關基因,并且研究表明,對于某類疾病的發生發展,通常是多個基因共同作用的結果,亦即基因表達微陣列中多個基因之間存在著復雜的相互作用,所以一般的基于統計的度量標準,如皮爾森相關系數、信息熵等,難以準確地表達基因的相對重要性[14]。隨機森林作為一種流行的機器學習算法,由于在訓練決策樹的過程中,既考慮了單個變量對于目標變量的影響,又考慮了多個變量之間的相互作用,其變量重要性分數被廣泛應用于評價數據集中特征變量的相對重要性,尤其是應用在生物醫學與生物信息學研究中[15-17]。當前,基于隨機森林和K-均值聚類相結合的方法已經被應用在網絡入侵檢測[18]等研究中,然而在基因聚類任務中,基于隨機森林變量重要性分數對基因進行加權聚類研究較少,仍然是一個值得探索的領域。本文主要針對基于基因的聚類分析任務,將隨機森林的變量重要性分數引入到K-均值聚類的過程中,提出了一種基于隨機森林變量重要性分數的加權K-均值聚類算法,能夠提高基因聚類結果的質量。
1算法設計
12隨機森林
隨機森林(Random Forest,RF)[19]是一個由一組決策樹分類器{h(X,θk),k=1,2,,K}組成的集成分類器,其中θk是服從獨立同分布的隨機向量,K表示隨機森林中決策樹的個數,在給定自變量X下,每個決策樹分類器通過投票來決定最優的分類結果[5]。隨機森林是許多決策樹集成在一起的分類器,如果把決策樹看成分類任務中的一個專家,隨機森林就是許多專家在一起對某種任務進行分類。生成隨機森林的步驟如下[20]:
1)從原始訓練數據集中,應用Bootstrap方法有放回地隨機抽取K個新的自助樣本集,并由此構建K棵分類回歸樹,每次未被抽到的樣本組成了K個袋外數據(outofbag, OOB)。
2)設有n個特征,則在每一棵樹的每個節點處隨機抽取mtry個特征(mtry<=n),通過計算每個特征蘊含的信息量,在mtry個特征中選擇一個最具有分類能力的特征進行節點分裂。
3)每棵樹最大限度生長,不做任何剪裁。
4)將生成的多棵樹組成隨機森林,用隨機森林對新的數據進行分類,分類結果按樹分類器的投票多少而定。
由于決策樹算法在節點分裂過程中考慮了特征之間的相互影響,隨機森林算法能夠有效地揭示多個特征之間的相互作用,對于單個特征具有小的邊際效應但多個特征的組合對目標變量有較大影響的數據集合,表現出優異的分類和預測性能,而且隨機森林算法不需要先驗假設[7]。目前,RF已經被廣泛應用于各種分類、預測、變量重要性研究、特征選擇以及異常點檢測問題中,尤其在生物醫學和生物信息學領域,隨機森林由于能識別多個特征變量之間的相互作用而受到青睞[21-22]。
隨機森林算法的一個重要產物是變量重要性分數,它可以很好地反映訓練數據集中分類變量對于目標變量的影響程度,目前,隨機森林變量重要性分數已經被廣泛應用于各種數據挖掘任務中。隨機森林提供了4種變量重要性分數供選擇,本文采用基于置換的變量重要性分數[16]。基于置換的變量重要性分數定義為在袋外數據(OOB)上當變量發生輕微擾動前后分類模型的分類正確率的平均減少量,它采用了直覺排列策略,既考慮到每一個變量單獨的影響,又考慮了多個變量之間的相關作用。
給定訓練樣本集合D,集合中的特征標記為Xj,j=1,2,,N,Xj的基于置換的變量重要性分數表示為IMj,則IMj的計算過程如下[20]:
1)對訓練集D進行Bootstrap隨機重采樣B次,得到B個樣本子集Db,b=1,2,,B;
2)設置b=1;
3)在樣本集合Db上訓練決策樹Tb,袋外數據標記為Loobb;
4)在袋外數據Loobb上,應用決策樹分類器Tb對測試數據進行分類,正確分類的樣本個數標記為Roobb;
5)對特征Xj,j=1,2,,N,隨機地擾動Loobb中每一個樣本直到它與目標變量的原始關系被打斷,擾動后的數據集標記為Loobbj;
6)在擾動后的數據集Loobbj上,應用決策樹分類器Tb對數據進行分類,正確分類的樣本個數標記為Roobbj;如果特征Xj與目標變量相關,那么分類器的分類性能將明顯降低;
7)對于b=2,,B,重復第3)-6)步;
8)按照下式計算特征Xj的變量重要性分數:
IMj=1B∑Bi=1Roobb-Roobbj;
9)輸出所有特征的重要性分數:
IM={IM1,IM2,,IMN}。
13基于隨機森林變量重要性分數的加權K-均值基因聚類算法
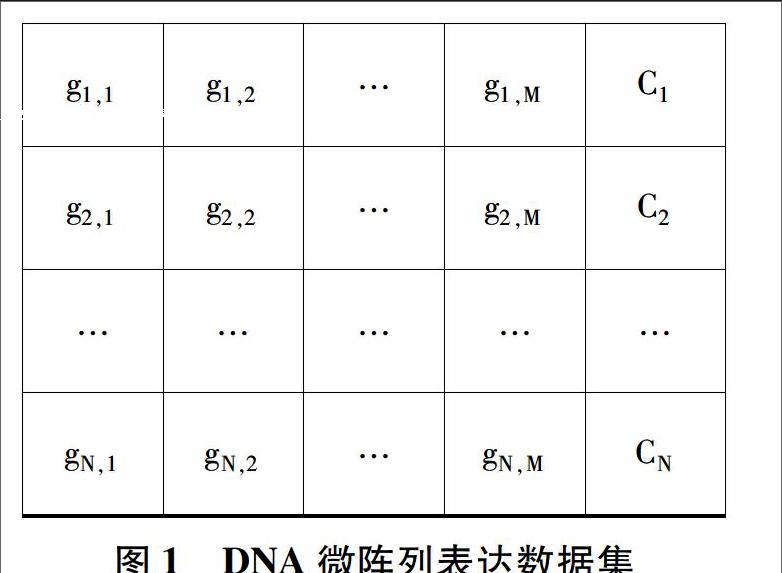
基因數據通常以DNA微陣列表達數據形式存儲。一般而言,微陣列表達數據集是一個N×(M+1) 的矩陣,矩陣中的每一行表示一個樣本,除最后列以外的每一列表示該樣本的一個基因,每一個元素gi,j是一個數值,表示第i個樣本第j個基因的基因表達水平,最后一列表示第i個樣本的類標簽,如圖1所示。
g1,1g1,2…g1,MC1
g2,1g2,2…g2,MC2
……………
gN,1gN,2…gN,MCN
2實驗與結果分析
21數據集
為了驗證本文提出的算法的有效性,在Leukemia(白血病)、Breast(乳腺癌)、DLBCL(彌漫性大B細胞淋巴瘤)等3個微陣列表數據集上進行了實驗。這些數據集的基本信息如表1所示。
原始的微陣列表達數據集中通常包含大量的噪聲基因,為了降低計算時間和存儲空間需求,在執行基因聚類分析之前首先采用四分位距方法和單因素方差分析法來過濾掉明顯不相關的基因和噪聲基因,所有表達水平低于總體IQR 1/5的基因在這一步被過濾掉。基因過濾后的數據信息如表2所示。
23實驗結果及分析
在3個實驗數據集上對原始的K-均值算法和本文提出的基于隨機森林變量重要性分數的加權K-均值算法進行了實驗,指定聚類數目k=100,最大迭代次數T=60。采用類內離散度和J和類間加權距離和D指標來衡量算法的性能,10次實驗結果的平均值如表3所示。
從表3可以看出,本文提出的基于隨機森林變量重要性分數的加權K-均值聚類算法的J值明顯低于原始的K-均值算法,說明類內基因表達模式高度相關;所提出的算法的R值比原始的K-均值算法平均高177個百分點,表明基于隨機森林變量重要性分數的加權K-均值聚類算法得到的聚類劃分中,類間差異比原始的K-均值聚類算法顯著。
3結語
提出了一種基于隨機森林變量重要性分數的加權K-均值基因聚類算法,相對于原始的K-均值聚類算法,該算法能夠有效地提高類內相似度和類間差異度,即提高聚類結果的質量。如何針對基因表達數據的特點,選擇或設計合適的聚類準則函數,利用本文提出的算法探索生物醫學信息,有待于進一步研究。
參 考 文 獻:
[1]周志華.機器學習[M].北京:清華大學出版社,2016:211-213
[2]劉帥,林克正,孫旭東,等.基于聚類的SIFT人臉檢測算法[J].哈爾濱理工大學學報,2014,19(1):31-35
[3]吳娛,鐘誠,尹夢曉.基因表達數據的分層近鄰傳播聚類算法[J].計算機工程與設計,2016,37(11):2961-2966
[4]陳偉,程詠梅,張紹武,潘泉.鄰域種子的啟發式454序列聚類方法[J].軟件學報,2014,25(5):929-938
[5]黃偉華,馬中,戴新發,徐明迪,高毅,劉利民.一種特征加權模糊聚類的負載均衡算法[J].西安電子科技大學學報(自然科學報),2017,44(2):138-143
[6]余曉東,雷英杰,岳韶華,王睿.基于粒子群優化的直覺模糊核聚類算法研究[J].通信學報,2015,36(5):1-7
[7]李霞,雷健波,李亦學,李勁松.生物信息學[M].北京:人民衛生出版社,2015:286-287
[8]李雨童,姚登舉,李哲,侯金利.基于R的醫學大數據挖掘系統研究[J].哈爾濱理工大學學報,2016,21(2):38-43
[9]高敬陽,齊飛,管瑞.基于高通量測序技術的基因組結構變異檢測算法[J].生物信息學,2014,12(1):5-9
[10]李晟,程福東,孫嘯.高通量DNA測序技術與疾病診斷及預防[J].生物醫學工程與臨床,2016,20(2):210-215
[11]吳林寰,陸震鳴,龔勁松,史勁松,許正宏.高通量測序技術在食品微生物研究中的應用[J].生物工程學報,2016,32(9):1164-1174
[12]岳峰,孫亮,王寬全,王永吉,左旺孟.基因表達數據的聚類分析研究進展[J].自動化學報,2008,34(2):113-120
[13]張國印,程慧杰,劉詠梅,姚愛紅.一種新算法在基因表達譜聚類中的應用[J].計算機工程與應用,2009,45(36):216-218
[14]王愛國.微陣列基因表達數據的特征分析方法研究[D].安徽:合肥工業大學,2015:1-5
[15]ALI Anaissi, PAUL J KENNEDY, Madhu Goyal1 Daniel R Catchpoole A Balanced Iterative Random Forest for Gene Selection from Microarray Data[J] BMC Bioinformatics, 2013, 14: 261P
[16]QI, Y Random Forest for Bioinformatics [J]. Ensemble Machine Learning, 2012: 307-323
[17]孫磊,許馳,胡學龍.一種基于隨機森林的長非編碼RNA預測方法[J].揚州大學學報:自然科學版,2016,19(4):50-53
[18]ELBASIONY R M, SALLAM E A, ELTOBELY T E, et al A Hybrid Network Intrusion Detection Framework Based on Random Forests and Weighted kmeans [J]. Ain Shams Engineering Journal, 2013, 4(4):753-762
[19]BREINMAN L Random Forests [J]. Machine Learning, 2001, 45: 5–32
[20]姚登舉,楊靜,詹曉娟.基于隨機森林的特征選擇算法[J].吉林大學學報工學版,2014,44(1):137-141
[21]VERIKAS A,GELZINIS A,BACAUSKIENE M Mining Data with Random Forests: A Survey and Results of New Tests [J]. Pattern Recognition, 2011, 44: 330–349
[22]劉勘,袁蘊英,劉萍.基于隨機森林分類的微博機器用戶識別研究[J].北京大學學報(自然科學版),2015,51(2):289-300
(編輯:王萍)

