陜西省黃鼠的遺傳學特征分析
安翠紅,陳寶寶,范鎖平,孫養信,呂 文,魯 亮
?
陜西省黃鼠的遺傳學特征分析
安翠紅,陳寶寶,范鎖平,孫養信,呂 文,魯 亮
目的 明確分布于陜西省的黃鼠的遺傳學特征。方法 測定COI、Cyt-b基因序列,與內蒙古科右中旗、正鑲白旗達烏爾黃鼠、寧夏海原縣阿拉善黃鼠同類基因序列進行比對,分析遺傳距離,構建系統發育NJ樹。結果 陜西省定邊縣黃鼠COI基因序列與寧夏阿拉善黃鼠的遺傳距離≤0.5%,與內蒙達烏爾黃鼠遺傳距離在7.9%~9.3%之間,Cyt-b基因序列與寧夏阿拉善黃鼠的遺傳距離為≤2.2%,與內蒙達烏爾黃鼠遺傳距離在8.9%~11.2%之間。基于COI和Cyt-b基因的NJ系統樹可見所有樣本序列形成兩個高支持度的單一分支,陜西定邊縣黃鼠與寧夏阿拉善黃鼠聚為一類,兩者與內蒙達烏爾黃鼠形成兩個獨立分支, 基于COI基因的 NJ系統樹顯示陜西各地樣本聚為一類。結論 陜西省的黃鼠應為阿拉善黃鼠。
黃鼠;DNA條形碼;COI基因;Cyt-b基因;阿拉善黃鼠;陜西;達烏爾黃鼠
阿拉善黃鼠(Spermophilusalashanicus)曾是一個有爭議的物種。20世紀中葉國內外學者多以其形態特征與達烏爾黃鼠(Spermophilusdauricus)相近,而將其歸為達烏爾黃鼠的亞種[1-2]。對于這兩種黃鼠的分類地位的討論經歷了很長時間。近年來,利用細胞生物學手段,國內外學者利用染色體核型來區分這兩種黃鼠,這兩種黃鼠的分類地位逐步明確[3-4]。
在陜西省的陜北、關中地區分布的黃鼠,多年來一直被認為是達烏爾黃鼠[5-6]。中國學者趙天飆、秦長育等多將分布于內蒙古南部和陜甘寧青及蒙古國西南部等地的阿拉善黃鼠視為達烏爾黃鼠的亞種[1-2]。上述區域中,涵蓋了陜西北部地區的長爪沙鼠鼠疫疫源地和寧夏甘肅的阿拉善黃鼠鼠疫疫源地。小型獸類宿主動物的準確分類是鼠疫動物流行病學調查的重要內容。但這些地區的黃鼠到底是哪種,不僅是單純分類學的問題,更是和公共衛生相關的分類學問題。
DNA條形碼技術能夠準確地評估物種和遺傳多樣性[8],并對生物多樣性進行有效地監測[7-9]。
陜西省鼠疫疫區1987-2014年發生3起鼠間鼠疫,在疫情處理及監測過程中,經常會遇到一些形態學上很難鑒定的宿主動物。2012年課題組開展了DNA條形碼技術在陜西鼠疫疫區應用研究[10],現將結果報告如下。
1 材 料
陜西省定邊縣、大荔縣、府谷縣、靖邊縣、吳起縣、寧夏海原縣、內蒙古科右中旗、正鑲白旗現場采集黃鼠鼠體和臟器標本。DB01-15采自定邊縣,DL01-10,13采自大荔縣,WQ01-05采自吳起縣,FG02-04,10采自府谷縣,JB18,19采自靖邊縣。NX01-7采自寧夏海原縣,NM49、50、52、53采自內蒙科右中旗,ZB01-17采自內蒙正鑲白旗。
2 方 法
2.1 DNA模板的制備 現場采集的樣本用無水乙醇保存帶回,使用Qiagen公司生產QIAamp DNA Mini kit試劑盒操作手冊制備DNA模板,測定其DNA含量。
2.2 PCR反應 線粒體細胞色素C氧化酶I亞基(COI)基因選用雞尾酒引物[11],其中LepF1_t1、VF1_t1、VF1d_t、VF1i_t1的比例為1∶1∶1∶3。LepRI_t1、 VR1d_t1、 VR1_t1、VR1i_t1的比例也是1∶1∶1∶3,見表1。在25 μL反應體系中, 2×Taq Mastermix 12.5 μL, 上下游引物各1 μL, 模板2 μL。擴增條件: 94 ℃預變性5 min, 94 ℃變性30 s, 55 ℃退火30 s, 72 ℃延伸1 min, 共35個循環。Cyt-b基因引物序列為L14724 5′-CGAAGCTTGATATGAAAAACCATCGTTG-3′,H15915 5′-CGGAATTCCATTTTTGGTTTACAAGAC-3′,PCR擴增方法:50 μL PCR反應體中,2×Taq Mastermix25 μL,上下游引物各2 μL,模板4 μL。擴增條件為:94 ℃預變性3 min,94 ℃變性30 s,51 ℃退火30 s,72 ℃延伸1 min,共30個循環。PCR產物1.5%的瓊脂糖凝膠電泳分析,若電泳圖中出現目標條帶即送樣進行雙向測序。
表1 擴增COI基因的PCR引物序列
Tab.1 Sequence of primers for amplifyingCOIgene

引物類型Typeofprimer引物名稱Name引物序列(5'-3')Sequenceofprimers(5'-3')雞尾酒引物(上游)VF1LFt1Cocktailprimer(upstream)VF1LFt1LepF1_t1TGTAAAACGACGGCCAGTATTCAACCAAT-CATAAAGATATTGGVF1_t1TGTAAAACGACGGCCAGTTCTCAACCAAC-CACAAAGACATTGGVF1d_tTGTAAAACGACGGCCAGTTCTCAACCAAC-CACAARGAYATYGGVF1i_t1TGTAAAACGACGGCCAGTTCTCAACCAAC-CAIAAIGAIATIGG雞尾酒引物(下游)VR1LRt1Cocktailprimer(downstream)VR1LRt1LepRI_t1CAGGAAACAGCTATGACTAAACTTCTG-GATGTCCAAAAAATCAVR1d_t1CAGGAAACAGCTATGACTAGACTTCT-GGGTGGCCRAARAAYCAVR1_t1CAGGAAACAGCTATGACTAGACTTCT-GGGTGGCCAAAGAATCAVR1i_t1CAGGAAACAGCTATGACTAGACTTCT-GGGTGICCIAAIAAICA
2.3 DNA序列分析:用Chromos軟件觀察評價測序峰圖質量, 如果峰圖質量差,不能準確判斷堿基, 則重新進行擴增和測序。將測定序列在NCBI上運行BLAST程序進行序列同源性比較。運用Mega 6軟件比對各基因序列的堿基組成和變異,刪除序列兩端不能完全對齊的堿基。基于Kimura-2-parameter(K2P)[12]模型計算各物種的遺傳距離;采用鄰接法構建全部COI基因序列的NJ系統樹,對NJ樹進行內部分支檢驗與1,000次Bootstrap檢驗分析, 來確定各支系的置信度[13]。
3 結 果
3.1 DNA條形碼技術檢測結果與分析 對采自陜西省定邊縣、寧夏海原縣、內蒙古科右中旗、正鑲白旗的28份黃鼠樣本進行DNA條形碼技術檢測。其中NM49、NM50、NM52、NM53采自內蒙科右中旗,其余內蒙樣品采自正鑲白旗。28份樣本全部獲得COI基因、Cyt-b基因序列。
3.2 基因序列分析 陜西定邊黃鼠Cyt-b基因序列與寧夏阿拉善黃鼠的遺傳距離為≤2.2%,與內蒙達烏爾黃鼠遺傳距離在8.9%~11.2%之間,陜西各地黃鼠COI基因序列與寧夏阿拉善黃鼠的遺傳距離≤2%,與內蒙達烏爾黃鼠遺傳距離在8%~10%之間。
3.3 構建系統發育樹 應用Mega 6軟件構建NJ系統樹,見圖1、圖2。圖1COI、Cyt-b基因NJ系統樹顯示內蒙黃鼠與陜西定邊、寧夏海原黃鼠分成兩個獨立分支,陜西定邊黃鼠與寧夏海原黃鼠聚為一類。圖2COI基因NJ系統樹顯示陜西各地黃鼠聚為一類。

圖1 陜西定邊與寧夏、內蒙古黃鼠COI、Cyt-b基因NJ系統樹Fig.1 Neighbor-joining phylogenetic tree of COI and Cyt-b of the Citellus samples from Dingbian County in Shaanxi,Nigxia and Inner Mongolia
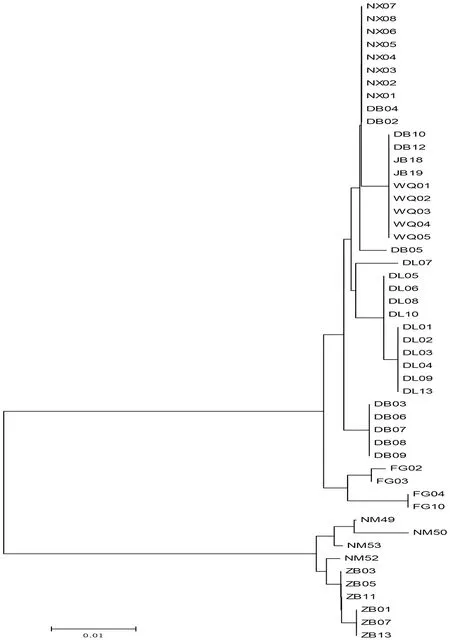
圖2 陜西各地黃鼠COI基因NJ系統樹Fig.2 Neighbor-joining phylogenetic tree of the Citellus samples from different places of Shaanxi Province
4 討 論
線粒體(Mitochondria)是真核細胞中小分子量的復制單位, 由于其具有拷貝數多、分子量小、編碼效率高、母系遺傳和進化速度較核基因組快等特點[14], 近年來越來越多的被作為分子標記, 廣泛應用到動物進化遺傳學、分子生態學、遺傳多樣性、物種及品系鑒定等方面[15]。DNA 條形碼是利用短的標準 DNA 序列來鑒定物種的一種行之有效的方法,因其片段小且易于獲得而被廣泛應用于多個物種的種類鑒定工作中。目前在動物分類中最常用的基因序列是線粒體細胞色素C氧化酶Ⅰ亞基(COⅠ)序列和Cyt-b基因序列。
據Hebert等對GenBank中同一個屬的物種COI序列數據進行研究,結果表明種內的COI序列差異程度小于2%[9]。馬英等對青海省海東地區小型獸類110只樣本的COI基因進行序列分析, 發現種內遺傳距離≤3%, 種間遺傳距離5%~10%[16], 安翠紅等對陜西鼠疫疫區139只宿主動物COI基因進行序列分析,發現種內遺傳距離≤2%,種間遺傳距離8.9%~15.1%[10]。
Meyer 的研究表明, 對于初步進行某些物種分子系統發育研究是可以首先采用線粒體細胞色素b 基因, 不僅它是13 個蛋白質編碼基因中了解的最為清楚的基因, 也被認為是最可信的分子標記之一[17], 同時用保守引物擴增線粒體Cyt-b基因較其他線粒體基因要容易的多, 因此研究較為廣泛[18]。已有研究表明線粒體Cyt-b基因在研究親緣關系較近的物種分類階元系統關系和種系地理學方面非常有用[19]。
本研究發現,陜西省定邊縣黃鼠COI基因序列與寧夏海原縣阿拉善黃鼠的遺傳距離≤0.5%,與內蒙達烏爾黃鼠遺傳距離在7.9%~9.3%之間。Cyt-b基因序列與寧夏阿拉善黃鼠的遺傳距離為≤2.2%,與內蒙達烏爾黃鼠遺傳距離在8.9%~11.2%之間。NJ系統樹顯示陜西黃鼠與寧夏阿拉善黃鼠聚為一類,兩者與內蒙達烏爾黃鼠形成兩個獨立分支。遺傳距離與NJ系統樹說明內蒙科右中旗、正鑲白旗黃鼠和寧夏海原阿拉善黃鼠、陜西黃鼠為不同種,陜西黃鼠與寧夏海原阿拉善黃鼠為同種。
綜上,依據COI、Cyt-b基因序列比對結果,表明陜西省的黃鼠為阿拉善黃鼠。
[1] Zhao TB,Li XM,Duan QH,et al, Fuzzy cluster analysis of four kinds of ground squirrel skeletal morphology[J]. J Inner Neimengolia Prevent Med, 1994,19(2):67-68. (in Chinese)
趙天飆, 李新民, 段全紅等.四種黃鼠骨骼形態的模糊聚類分析[J]. 內蒙古地方病防治研究. 1994,19(2):67-68.
[2] Qin CY,Li KC.Glires of Ningxia Autonomous[M].Yinchuan:People’s Publishing House of Ningxia.2003: 99-103. (in Chinese)
秦長育,李克昌.寧夏嚙齒動物與防制[M].銀川:寧夏人民出版社,2003:99-103.
[3] Tsvirka MV, Chelomina GN, Korabiev VP. Genetic evidence of hybridization between pale tailedSpermophiluspallidicaudaSatunin, 1903 andS.alaschanicusB Chner, 1888 ground squirrels in Mongolia[J]. Russian J Genetics, 2006, 42(4): 421-428. DOI: 10.1134/S1022795406040090
[4] Alcaide M, Rico C, Ruiz S, et al. Disentangling vector-borne transmission networks: a universal DNA barcoding method to identify vertebrate hosts from arthropod bloodmeals[J]. PLoS One, 2009, 4: e7092. DOI:10.1371/journal.pone.0007092
[5] Wang TZ,Xu WX. Fauna of Shaanxi Glires[M].Xi’an:Publishing House of Shaanxi Normal University. 1992:82-85. (in Chinese)
王廷正,許文賢.陜西嚙齒動物志[M].西安:陜西師范大學出版社,1992:82-85.
[6] Liu JS.Study on the important medical animals in Shaanxi province and its prevention and control[M].Xi’an,China.Xi’an: Publishing House of Northwest University. 2001:3-20. (in Chinese)
劉建書.陜西省重要醫學動物及防制研究[M].西安:西北大學出版社,2001:3-20.
[7] Hebert PD, Stoeckle MY, Zemlak TS, et al. Identification of birds through DNA barcodes[J]. PLoS Biol, 2004, 2(10): 312.DOI:10.1371/journal.pbio.0020312
[8] Hebert PDN, Ratnasingham S, deWaard JR. Barcoding animal life: cytochrome c oxidase subunit 1 divergences among closely related species[J]. Proc Royal Soci London, Series B: Biologic Sci, 2003, 270(Suppl): 96-99.DOI:10.1098/rsbl.2003.0025
[9] Smith MA, Fisher BL, Hebert PD. DNA barcoding for effective biodiversity assessment of a hyperdiverse arthro-pod group: the ants of Madagascar[J]. Philosophic Transact Royal Soc B: Biologic Sci, 2005, 360: 1825-1834. DOI:10.1098/rstb.2005.1714
[10] An CH,Sun YX,Chen BB,et al,Identification on host animals for plague by DNA barcoding technology in Shaanxi province[J]. J Epidem,2014, 35(9):1042-1045. (in Chinese)
安翠紅, 孫養信,陳寶寶,等.DNA條形碼技術在陜西省鼠疫疫區宿主動物鑒定中的應用[J]. 中華流行病學雜志, 2014, 35(9):1042-1045.
[11] Ivanovo NV, Zemlak TS, Hanner RH, et al. Universal primer cocktails for fish DNA barcoding[J]. Mol Ecol Notes, 2007, 7: 544-548. DOI: 10.1111/j.1471-8286.2007.01748.x
[12] Kimura M. A simple method for estimating evolutionary rates of base substitutions through comparative studies of nucleotide sequences[J]. J Mol Evolut, 1980, 16: 111-120. DOI: 10.1007/BF01731581
[13] Saitou N, Nei M. The neighbor-joining method:a new method for reconstructing phylogenetic trees[J]. Mol Biol Evol, 1987, 4(4): 406-425.
[14] Gray MW. Origin and evolution of mitochondrial DNA[J]. Ann Rev Cell Biol, 1989, 5: 25-50. DOI:10.1146/annurev.cb.05.110189.000325
[15] Xiao WH, Zhang YP. Genetics and evolution of mitocchondrial DNA in fish[J]. Acta Hydrobiologica Sinica, 2000, 24(4): 384-391. (in Chinese)
肖武漢, 張亞平. 魚類線粒體DNA的遺傳與進化[J]. 水生生物學報, 2000, 24(4): 384-391.
[16] Ma Y, Li HL, Lu L. Application for identification of small mammals by DNA barcoding in Haidong area, Qinghai ProvinceChina[J].Biodiversity Sci, 2012, 20(2): 193-198. (in Chinese)
馬英, 李海龍, 魯亮. DNA條形碼技術在青海海東地區小型獸類鑒定中的應用[J].生物多樣性, 2012, 20(2):193-198.
[17] Meyer A. Shortcoming of the cytochrome b gene as a molecular marker[J]. Trends Ecol Evolut, 1994, 9: 278-289. DOI: 10.1016/0169-5347(94)90028-0
[18] Katriina LI, Erie BT. Molecular resolution of the systematics of a problematic group of fishes (Teleostei: Osmeridae) and evidence for morphological homoplasy[J]. Mol Phylogenetics Evolut, 2009, 50(1): 163-175. DOI:10.1016/j.ympev.2008.10.021
[19] Song CB, Near TJ, Page LM. Phylogenetic relations among percid fishes as inferred from mitochondrial cytochrome b DNA sequence data[J]. Mol Phylogenetics Evolut, 1998, 10(3): 343-353. DOI:10.1006/mpev.1998.0542
Genetic characteristics ofSpermophilusin Shaanxi Province, China
AN Cui-hong, CHEN Bao-bao, FAN Suo-ping, SUN Yang-xin, LV Wen, LU Liang
(ShaanxiCenterforDiseaseControlandPrevention,Xi’an710054,China)
We studied the genetic characteristics ofSpermophilusin Shaanxi Province, China. TheCOI,Cyt-bgene were sequenced and the results were compared with those ofdauricusfrom Inner Mongolia Keyouzhong Banner and Zhengxiangbai Banner, andS.alaschanicusfrom Haiyuan County of Ningxia. And genetic distance was analyzed and Neighbor-Joining tree was built. Results showed that the genetic distance ofCOIgene sequences betweenSpermophilusfrom Dingbian County in Shaanxi andS.alaschanicusin Ningxia was ≤0.5%, and the genetic distance was ranged from 7.9% to 9.3% withCitellusdauricusfrom Inner Mongolia. The genetic distance ofCyt-bgene betweenSpermophilusfrom Dingbian County in Shaanxi andS.alaschanicusin Ningxia was ≤2.2%, and ranged from 8.9% to 11.2% withCitellusdauricusfrom Inner Mongolia. The Neighbor-Joining tree ofCOI,Cyt-bgene showed two major clusters. One of them were clustered bySpermophilusfrom Dingbian County in Shaanxi andS.alaschanicusin Ningxia, and another one wasCitellusdauricusfrom Inner Mongolia. The Neighbor-Joining tree ofCOIgene showed that all samples from Shaanxi Province clustered in a group. In conclusion, theSpermophilusin Shaanxi Province wereS.alaschanicus.
Spermophilus; DNA barcoding;COIgene;Cyt-bgene; Shaanxi Province;S.alaschanicus;S.dauricus
Sun Yang-Xin, Email: sxpco@126.com
10.3969/j.issn.1002-2694.2017.06.013
孫養信,Email: sxpco@126.com
陜西省疾病預防控制中心,西安 710054
R394
A
1002-2694(2017)06-0538-04
2017-02-21 編輯:李友松
陜西省科學技術研究發展計劃項目(No.2012K16-12-03)資助
Supported by a grant from the Science and Technology Research and Development Program of Shaanxi Province,(No. 2012K16-12-03)

