紅掌2個SOC1基因的克隆、序列與表達分析
馬廣瑩,朱開元,史小華,鄒清成,劉慧春,詹 菁,田丹青*
(1.浙江省農(nóng)業(yè)科學(xué)院花卉研究開發(fā)中心,杭州311202;2.浙江省杭州蕭山技師學(xué)院,杭州311202)
紅掌2個SOC1基因的克隆、序列與表達分析
馬廣瑩1,朱開元1,史小華1,鄒清成1,劉慧春1,詹 菁2,田丹青1*
(1.浙江省農(nóng)業(yè)科學(xué)院花卉研究開發(fā)中心,杭州311202;2.浙江省杭州蕭山技師學(xué)院,杭州311202)
為了明確SOC1轉(zhuǎn)錄因子在紅掌中的成員及作用機制,采用5'/3'末端快速擴增技術(shù)獲得這2個SOC1基因的完整編碼區(qū),通過生物信息學(xué)軟件分析這2個基因的相關(guān)信息,并結(jié)合熒光定量反轉(zhuǎn)錄-聚合酶鏈?zhǔn)椒磻?yīng)驗證這2個基因的表達模式。結(jié)果顯示:獲得的2個基因核苷酸長度分別為651 bp和642 bp,命名為AaSOC1-1和AaSOC1-2;2個轉(zhuǎn)錄因子在二級結(jié)構(gòu)上均有螺旋、折疊和轉(zhuǎn)角,未發(fā)現(xiàn)其他結(jié)構(gòu)。系統(tǒng)進化分析顯示,二者與單子葉植物同類蛋白距離較近,與紅掌的植物學(xué)分類地位一致。表達分析結(jié)果顯示,2個基因均在營養(yǎng)器官和生殖器官中表達,但表達水平略有差異。研究認為,從紅掌中克隆的2個SOC1轉(zhuǎn)錄因子可能定位于線粒體而非細胞核中,二者在序列組成和表達模式上既有保守性也存在一定的差異性,預(yù)示它們在功能上可能也存在一定的區(qū)別。
紅掌;SOC1基因;生物信息學(xué);表達分析
Summary SUPPRESSOR OF OVEREXPRESSION OF CO1(SOC1)is a flowering integration factor that widely existed in flowering plants,which is necessary in multiple flowering induction pathways,including vernalization,photoperiod,gibberellin, and otherendogenousorexogenousenvironmentalsignals.Therefore,research oftheeffectof SOC1 on plantgrowth and development can reveal the plant flowering mechanism and direct molecular breeding.Anthurium is a kind of high?grade potted flower in the worldwide.However,molecular biology research on this plant had just started.As a flowering integration factor,SOC1 in Anthurium may be a key to deeply understand the flower conversion process.
In this study,rapid amplification ofcDNA ends(RACE)technology was used to obtain the fulllength oftwo SOC1 genes of Anthurium.Severalbiologicalsoftwares were used to analyze the biologicalinformation,including NCBIBlast,NCBIConserved Domain Search,ProtParam,TMHMM Server version 2.0,SignalP 4.1 Server,iPSORT,PSIPRED,DNAman,and MEGA 6.0. Gene expression levels of the two SOC1 genes were detected by real?time reverse transcription?polymerase chain reaction(RT?PCR).
The main results were as follows:AaSOC1-1 and AaSOC1-2 were obtained from two independent SOC1 full codingsequence(CDS)regions,which shared 75.6%similarity at the nucleotide level.Their encoded proteins were composed of 216 and 213 amino acid residues,respectively.Both ofthe putative proteins had secondary structures such as helix,sheet,and turn, but no other special structures were found.Based on the online software analysis results,it was concluded that AaSOC1-1 and AaSOC1-2 were located in the mitochondria but not in the nucleus.Phylogenetic tree analysis showed that the two SOC1 transcription factors from Anthurium were tightly clustered with each other,and also with other monocotyledons.Fluorescence quantitative RT?PCR analysis revealed that both genes from Anthurium were ubiquitously expressed in the vegetative and reproductive organs,but different in the expression patterns.Particularly,AaSOC1-1 was mainly expressed in the bract and tepal,whereas AaSOC1-2 was mainly expressed in the stem and pedicel.
In sum,this study reported the nucleotide and deduced amino acid composition of the two SOC1 transcription factors. Preliminary analysis of their characteristics,structure,and potential function were performed through bioinformatics software, combined with multiple sequence alignment and phylogenetic tree construction.Based on the results,the two genes found in Anthurium were identified asmembersof SOC1 gene family.Gene expression experimentsconfirmed the similaritiesand differences of their expression patterns,which were relatively conservative compared with other species.This study lays the foundation for understanding the functional properties of these two transcription factors,which also requires in?depth research to verify the above speculation and views.
植物需要一個精準(zhǔn)的調(diào)控系統(tǒng)來完成由營養(yǎng)生長到生殖生長的轉(zhuǎn)變。植物在進化過程中已經(jīng)形成了這種精準(zhǔn)的調(diào)控體系,其中以MADS盒(MADS?box)為主的轉(zhuǎn)錄因子調(diào)控網(wǎng)絡(luò)發(fā)揮了十分重要的作用。近30年來,植物學(xué)家和分子生物學(xué)家在對擬南芥(Arabidopsis thaliana)、水稻(Oryza sativa)、矮牽牛(Petunia hybrida)、金魚草(Antirrhinum majus)等植物的研究中,逐漸總結(jié)出植物開花所涉及的4種途徑:光周期途徑、自主控制途徑、低溫春化途徑和赤霉素途徑[1-3]。植物在受到外界環(huán)境或內(nèi)源物質(zhì)等成花信號的刺激后,會通過一系列的信號轉(zhuǎn)導(dǎo),匯總到已經(jīng)發(fā)現(xiàn)的FT(FLOWERING LO?CUS T)、LFY(LEAFY)、SOC1(SUPPRESSOR OF OVEREXPRESSION OF CO1)這3種成花整合因子中,它們隨后再將成花信號傳遞給花器官決定基因,從而誘導(dǎo)成花,實現(xiàn)由營養(yǎng)生長向生殖生長的轉(zhuǎn)變[4]。
SOC1是MADS盒轉(zhuǎn)錄因子家族中的一員,其氨基酸組成較為保守,具有典型的M、I、K、C區(qū)段[5]。該轉(zhuǎn)錄因子最早在篩選過表達CO基因的突變體時在擬南芥中被發(fā)現(xiàn)[6]。SOC1功能缺失會嚴(yán)重抑制35S::CO植株的早花現(xiàn)象,說明SOC1作為CO基因的下游調(diào)控者,參與成花誘導(dǎo),并且先于花原基出現(xiàn),在部分營養(yǎng)器官如莖頂端分生組織、葉片、莖段,甚至根部中均有表達[7]。在擬南芥以外的其他物種中也存在類似的表達模式[8-10]。然而,研究發(fā)現(xiàn),它也在花器官形成初期表達,并參與花器官的決定[11-12]。所以,SOC1從營養(yǎng)生長階段到生殖發(fā)育階段,都發(fā)揮著重要的調(diào)控作用。在擬南芥中,SOC1的表達會對B功能基因造成壓制[13],但在花分生組織出現(xiàn)后,SOC1的表達會受到短時的抑制,以利于其他花器官決定基因的表達,然而這種短時的抑制會很快消失,所以在擬南芥的雌雄蕊中,也可以檢測到該基因的表達[14-15]。在調(diào)控方式上,SOC1的表達受到自身及其他轉(zhuǎn)錄因子的調(diào)控。有研究證明,SOC1本身可以與AG和AP1轉(zhuǎn)錄因子形成蛋白復(fù)合體,反過來結(jié)合于自身順式元件上,抑制自身的表達[16-17]。除了AG和AP1這2個同源異型轉(zhuǎn)錄因子,SVP和FLC作為MADS家族一員,也可以蛋白二聚體的形式調(diào)控SOC1的表達,表現(xiàn)為與SOC1的啟動子區(qū)域結(jié)合來抑制它的活性[18]。擬南芥SOC1雖然是一個轉(zhuǎn)錄因子,但是其亞細胞定位在細胞質(zhì)中,必須依靠MADS轉(zhuǎn)錄因子AGL24的協(xié)助才能順利進入細胞核,發(fā)揮其調(diào)控功能。可見,AGL24在SOC1的調(diào)控網(wǎng)絡(luò)中具有十分重要的作用[19]。
鑒于SOC1的重要調(diào)控功能及其在花器官發(fā)育過程中扮演的重要角色,近年來,有越來越多的同源基因被發(fā)掘研究,如牡丹(Paeonia suffruticosa)、矮牽牛、石斛蘭(Dendrobium)、草莓(Fragaria ananassa)、棉花(Gossypium hirsutum)等,涉及草本、木本、單子葉、雙子葉植物,已為深入研究SOC1轉(zhuǎn)錄因子的進化、調(diào)控、功能等工作做了積極探索[10-11,20-22]。本課題組已經(jīng)對紅掌中其他MADS基因的研究取得了一定的進展[23]。在此基礎(chǔ)上開展SOC1基因的研究,既能豐富紅掌MADS盒轉(zhuǎn)錄因子家族信息,也是解析該家族調(diào)控紅掌器官發(fā)生、成花轉(zhuǎn)換的必要環(huán)節(jié),對深入開展紅掌分子育種工作意義重大。
1 材料與方法
1.1 實驗材料
紅掌品種“阿拉巴馬”為第一作者所在單位的組培品種,取樣時間為初花期。LA taq酶、pMD-18T載體、5'/3'末端快速擴增(rapid amplification ofcDNA ends,RACE)試劑盒購自TaKaRa(大連)公司,RNA提取試劑(Plant RNA Reagent)購自美國Invitrogen公司,反轉(zhuǎn)錄cDNA第1鏈合成使用天根公司(北京)反轉(zhuǎn)錄試劑盒(FastquantRT Kit)。熒光定量分析采用TaKaRa(大連)公司熒光染料混合液(SYBR Fast qRT?PCR Mix),使用儀器為7500 Fast型熒光定量PCR儀(AB公司,美國)。其他耗材、生化試劑均為國產(chǎn)。
1.2 RNA提取及反轉(zhuǎn)錄
用于基因克隆的總RNA提取自紅掌幼嫩的花梗、苞片、花序復(fù)合體。總RNA分為2份,1份用于合成5'RACE模板,1份用于合成3'RACE模板,操作步驟嚴(yán)格按照試劑盒說明書進行。模板合成后,保存?zhèn)溆谩1磉_分析用總RNA提取自紅掌各幼嫩器官,包括根(根尖)、莖、葉(未展開)、花梗(托葉包裹中)、苞片(托葉包裹中)、花被片(顯微可見)、雄蕊(顯微可見)、雌蕊(顯微可見)、果實(未成熟),然后經(jīng)由DNase處理,按照試劑盒說明書反轉(zhuǎn)錄合成cDNA,備用。
1.3 基因克隆及表達分析
在前期轉(zhuǎn)錄組測序基礎(chǔ)上,已經(jīng)獲得了2個SOC1基因的表達序列標(biāo)簽(expressed sequence tag,EST),分別長294 bp和280 bp。以此序列為核心,設(shè)計特異引物(P1-5'和P1-3'擴增AaSOC1-1;P2-5'和P2-3'擴增AaSOC1-2),結(jié)合試劑盒通用引物UPM,采用LA taq酶并嚴(yán)格按照其擴增體系進行擴增,獲得2個基因的兩端序列,然后通過拼接、驗證,得到2個基因的完整編碼區(qū)。所用引物序列如下。P1-5':ACATGATCCCAAACTTTCCCCCAGCAG,P1-3':TCTACGAGTTCTCCAGCAACAGCAT;P2-5':CAGCCTCTCCTTTTCCTTCAGCAGCGT;P2-3':TC ACGCTGCTGAAGGAAAAGGAGAGGC。通用引物為UPM:CTAATACGACTCACTATAGGGCAAGCAG TGGTAT CAACGCAGAGT。
在獲得2個基因的完整編碼區(qū)后,以2個基因C-端不保守區(qū)段為模板,設(shè)計特異引物(GSP1-F:AGCAGATTACATCGCTGAAGG,GSP1-R:TTCCCG CTGTCATTGTCATAG,產(chǎn)物長127 bp;GSP2-F:GG AAAAGGAGAGGCTGCTGTT,GSP2-R:ATCAA CG GTGTCCAGAGCATC,產(chǎn)物長97 bp),用于熒光定量反轉(zhuǎn)錄-聚合酶鏈?zhǔn)椒磻?yīng)(reverse transcription?polymerase chain reaction,RT-PCR)。內(nèi)參基因采用紅掌UBQ基因,引物序列為UBQ-F:GGATT CTAGCT GCCAGGTTTG,UBQ-R:CGGGCATAATC GCAACTTG。反應(yīng)體系(20μL)嚴(yán)格按照試劑盒說明書進行,反應(yīng)程序為:95℃30 s;95℃5 s,58℃10 s,40個循環(huán)。每個樣本設(shè)置3個生物學(xué)重復(fù)。表達水平采用2-△△CT法計算。
1.4 序列生物信息學(xué)分析
對獲得的基因序列進行生物信息學(xué)分析,其中編碼區(qū)分析采用NCBI在線網(wǎng)站ORF Finder(http:// www.ncbi.nlm.nih.gov/gorf/gorf.html),編碼蛋白質(zhì)的保守區(qū)段分析采用NCBIConserved Domain Search軟件(http://www.ncbi.nlm.nih.gov/Structure/cdd/wrpsb.cgi),編碼區(qū)蛋白質(zhì)特征分析采用在線軟件ProtParam(http: //web.expasy.org/protparam/),TMHMM Server v2.0 (http://www.cbs.dtu.dk/services/TMHMM/),SignalP 4.1 Server(http://www.cbs.dtu.dk/services/SignalP/),iPSORT(http://ipsort.hgc.jp/),PSIPRED(http://bioinf. cs.ucl.ac.uk/psipred/)。多序列比對采用軟件DNAman;系統(tǒng)進化樹構(gòu)建采用Clustal X+MEGA 6.0:氨基酸全序列參與分析,選擇鄰位連接法(neighbor?joining method),用Bootstrap法檢驗1 000次。
2 結(jié)果與分析
2.1 紅掌2個SOC1基因的克隆
通過RACE技術(shù),在已知2個SOC1片段的基礎(chǔ)上,分別獲得了每個基因的上、下游序列,通過拼接以及全長PCR驗證,確認了2個基因的核苷酸組成,分別命名為AaSOC1-1和AaSOC1-2。AaSOC1-1編碼區(qū)長651 bp,AaSOC1-2編碼區(qū)長642 bp(圖1)。從軟件分析結(jié)果得出,2個基因序列的一致性只有75.6%,基本排除是同一基因不同剪接體的可能。

圖1 AaSOC1-1和AaSOC1-2基因編碼區(qū)核苷酸序列比對Fig.1 Necleotide sequence alignmentofgene coding region of AaSOC1-1 and AaSOC1-2
2.2 AaSOC1-1和AaSOC1-2的生物信息學(xué)分析
在獲得2個SOC1基因核苷酸序列的基礎(chǔ)上,對其編碼的氨基酸以及相關(guān)特征屬性進行分析。結(jié)果顯示,AaSOC1-1編碼216個氨基酸殘基,其中谷氨酸(Glu)共23個,占比10.6%,比例最高,其次為亮氨酸(Leu),共計20個,占比9.3%,最少的為色氨酸(Trp),共1個,占比0.5%;所有負電荷氨基酸(Asp+Glu)共計29個,正電荷氨基酸(Arg+Lys+His)共計38個。AaSOC1-1編碼蛋白的相對分子質(zhì)量為24 896.2,理論等電點pI=9.16,理論化學(xué)分子式為C1064H1736N322O340S13,預(yù)測的親水性平均系數(shù)(GRAVY)為-0.929:說明該結(jié)構(gòu)親水性能較好,屬于親水性蛋白質(zhì)。通過TMHMM 2.0在線軟件分析發(fā)現(xiàn),該編碼蛋白不具有跨膜螺旋結(jié)構(gòu),可認為是非跨膜蛋白。SignalP 4.1 Server和iPSORT軟件分析顯示,該蛋白不含有核定位信號,而含有線粒體定位信號,預(yù)測該基因編碼產(chǎn)物可能定位于線粒體,通過其他途徑(例如與互作蛋白結(jié)合)轉(zhuǎn)移到細胞核發(fā)揮作用。該蛋白二級結(jié)構(gòu)含有常見的螺旋(粉紅色)和折疊(黃色)結(jié)構(gòu)以及卷曲(無陰影)結(jié)構(gòu)(圖2A);預(yù)測的三級結(jié)構(gòu)如圖3A所示。
AaSOC1-2編碼213個氨基酸,其中亮氨酸(Leu)含量最高,共計29個,占比13.6%,其次為谷氨酸(Glu),共計27個,占比12.7%,含量最少的為色氨酸(Trp),共1個,占比0.5%;在該組成中,帶負電荷氨基酸(Asp+Glu)共計35個,帶正電荷氨基酸(Arg+Lys+His)共計41個。預(yù)測的相對分子質(zhì)量為24 546.0,理論等電點pI=6.68,理論化學(xué)分子式為C1064H1754N314O334S8,預(yù)測的親水性平均系數(shù)(GRAVY)為-0.642:說明AaSOC1-2編碼蛋白也屬于親水性蛋白。通過TMHMM 2.0在線軟件分析發(fā)現(xiàn),該蛋白不具有跨膜螺旋結(jié)構(gòu),也可認為是非跨膜蛋白。SignalP 4.1 Server軟件分析顯示,該蛋白不含有信號肽。SignalP 4.1 Server和iPSORT軟件分析顯示,該蛋白不含有核定位信號,而含有線粒體定位信號,預(yù)測該基因編碼產(chǎn)物定位于線粒體,也是通過其他途徑轉(zhuǎn)移到細胞核參與轉(zhuǎn)錄調(diào)控。從圖2B可看出,該蛋白的二級結(jié)構(gòu)同樣含有常見的螺旋、折疊和卷曲結(jié)構(gòu)。預(yù)測的蛋白質(zhì)三級結(jié)構(gòu)如圖3B所示。
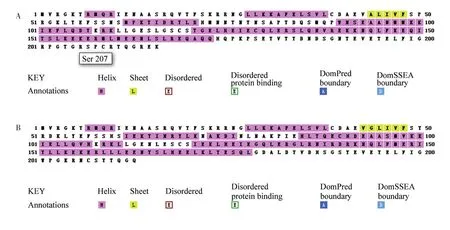
圖2 AaSOC1-1(A)和AaSOC1-2(B)預(yù)測氨基酸的二級結(jié)構(gòu)組成Fig.2 Secondary structure composition ofdeduced amino acid from AaSOC1-1(A)and AaSOC1-2(B)
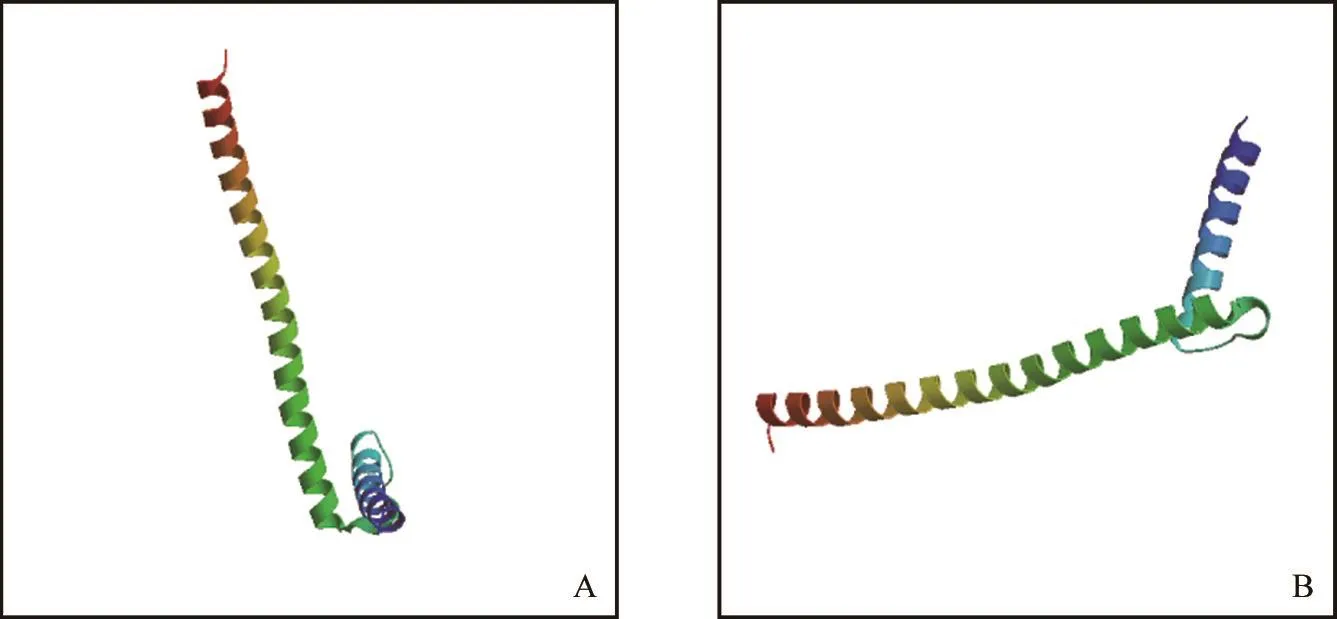
圖3 AaSOC1-1(A)和AaSOC1-2(B)蛋白質(zhì)三級結(jié)構(gòu)預(yù)測Fig.3 Deduced protein tertiary structure of AaSOC1-1(A)and AaSOC1-2(B)
運用DNAman軟件對這2個轉(zhuǎn)錄因子及不同物種的同源蛋白進行比對,結(jié)果如圖4所示。從中可見:紅掌和其他物種的SOC1轉(zhuǎn)錄因子普遍具有氨基酸組成保守性,尤其在MADS區(qū),無論是氨基酸數(shù)目還是組成,都保持了高度的保守性;在K區(qū)各序列之間保守區(qū)段較少,主要存在于零星的氨基酸殘基中;在相對不保守的C端,各序列均含有一個較為保守的DVETELFIGLP區(qū)段,該區(qū)段被認為是SOC1家族共有的識別特征,本研究報道的2個SOC1序列均含有此保守區(qū)段,可以認定這2個轉(zhuǎn)錄因子確為紅掌的SOC1同源成員。
為了更好地說明紅掌中2個SOC1轉(zhuǎn)錄因子與其他物種同源基因的進化關(guān)系,采用MEGA 6.0軟件構(gòu)建了不同物種SOC1轉(zhuǎn)錄因子的系統(tǒng)進化樹,分析了總共25個不同來源的SOC1同源轉(zhuǎn)錄因子。結(jié)果(圖5)顯示:單、雙子葉植物區(qū)分明顯,來源于棕櫚、石斛、玉米、小麥、紅掌的序列都歸于單子葉植物組,其余都歸于雙子葉植物組,序列的聚類信息與植物所屬類別完全一致;來源于紅掌的2個SOC1,雖然聚于同一分支,然而置信分值只有86,間接說明二者在氨基酸組成、功能等方面存在較顯著的差異。
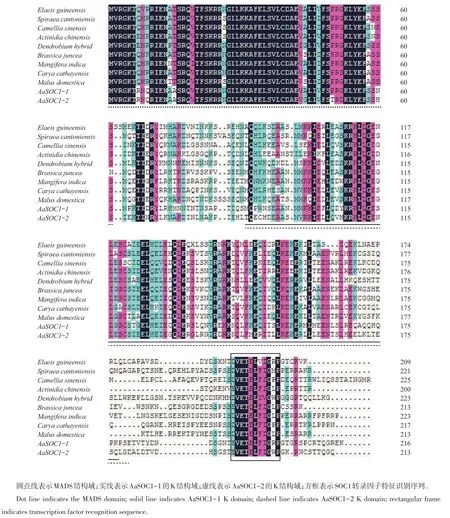
圖4 紅掌SOC1與其他物種同源蛋白多序列比對Fig.4 Multiple sequence alignmentofhomologous SOC1 proteins from Anthurium and otherspecies
2.3 AaSOC1-1和AaSOC1-2的表達分析
為明確紅掌中2個SOC1基因的表達情況,對根、莖、葉等9個有代表性的部位進行了熒光定量RT?PCR分析。結(jié)果(圖6)表明:AaSOC1-1的轉(zhuǎn)錄本在所檢測的各部位都有表達,其中在根部表達水平極低,在苞片中表達水平最高,在剛剛形成的果實中也存在低水平表達;AaSOC1-2的最低表達同樣在根部,其次是幼嫩的果實,而最高表達出現(xiàn)在花梗處;2個基因的表達在花器官中由外向內(nèi)逐漸遞減,表達主要集中于營養(yǎng)器官向生殖器官過渡的部位。

圖5 不同來源SOC1轉(zhuǎn)錄因子的系統(tǒng)進化樹分析Fig.5 Phylogenetic tree analysis of SOC1 transcription factors from differentsources
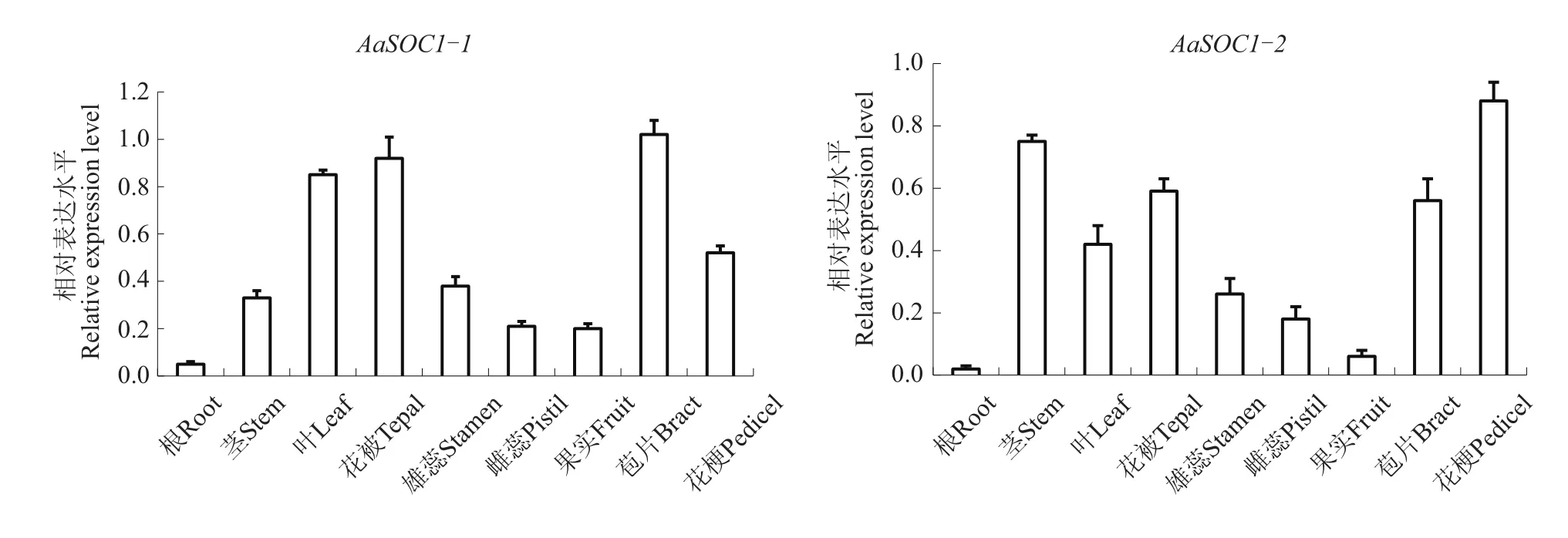
圖6 AaSOC1-1和AaSOC1-2在紅掌不同部位的表達分析Fig.6 Expression analysis of AaSOC1-1 and AaSOC1-2 in differenttissues of Anthurium
3 討論
在目前報道的有關(guān)SOC1的研究中,多數(shù)植株中只發(fā)現(xiàn)了1種轉(zhuǎn)錄本,也有在1種植物中發(fā)現(xiàn)多種轉(zhuǎn)錄本的情況[24]。在本研究中,已經(jīng)發(fā)現(xiàn)的2個SOC1基因序列的相似度并不高,預(yù)測的氨基酸序列差異也比較大,但通過多序列比對、系統(tǒng)進化分析和氨基酸組成分析,都可以判定它們屬于SOC1轉(zhuǎn)錄因子家族成員;因此,可以認定紅掌中至少存在2個或2個以上的SOC1同源基因。
生物信息學(xué)分析顯示,紅掌中的這2個SOC1轉(zhuǎn)錄因子都不含有核定位信號,這就意味著它們發(fā)揮調(diào)控功能需要借助外力進入細胞核,進而調(diào)節(jié)下游基因的表達。在擬南芥以及草莓等物種上,SOC1定位于細胞質(zhì)并與其他蛋白質(zhì)互作進而發(fā)揮轉(zhuǎn)錄因子的調(diào)控功能已經(jīng)被多次報道[11,19]。據(jù)此推斷,本研究的2個轉(zhuǎn)錄因子也有可能是通過類似的途徑參與紅掌的生長發(fā)育調(diào)控。
從表達分析結(jié)果看,源于紅掌的這2個SOC1基因在不同的組織部位皆有表達,包括營養(yǎng)器官和生殖器官。這與前人在其他物種上報道的同源基因的表達模式類似[25-26]。特別之處在于,紅掌的生殖器官著生在一根長長的花梗上,包括苞片、肉穗花序、花器官。本研究結(jié)果顯示,紅掌的2個SOC1基因在花梗中都有較高的表達水平,尤其是AaSOC1-2,這說明2個SOC1基因在成花轉(zhuǎn)換中發(fā)揮著重要作用。在營養(yǎng)器官和生殖器官中的普遍表達,暗示紅掌中2個SOC1基因除了扮演開花整合因子的角色,也很有可能在花器官分生組織決定方面起到某種作用,這一推論前人在其他物種的相關(guān)研究中也多有報道[20,27]。
本研究雖未涉及2個SOC1轉(zhuǎn)錄因子的功能驗證,但是從已報道的不同物種SOC1轉(zhuǎn)錄因子的研究結(jié)果來看,促進植物開花幾乎是絕大多數(shù)SOC1的共有功能[28-30]。此外,在擬南芥中的SOC1轉(zhuǎn)錄因子還可以通過調(diào)控一些氣孔開放相關(guān)基因的表達,進而間接控制氣孔的開放[29]。對早竹SOC1的研究發(fā)現(xiàn),它除了具有促進開花的功能外,還可以應(yīng)答赤霉素信號,隨赤霉素含量的增加而提高表達水平,且過表達該基因還能影響葉片的形態(tài)發(fā)育[30]。這些發(fā)現(xiàn)都豐富了SOC1的功能研究。我們對紅掌中2個SOC1基因的功能研究也正在進行,二者是否存在功能特異性將在后續(xù)報道中呈現(xiàn)。
4 結(jié)論
本研究采用經(jīng)典RACE克隆法,以2條已獲得的獨立EST序列為模板,從紅掌生殖器官cDNA中獲得了2個SOC1類基因,通過序列比較和生物信息學(xué)軟件分析,認定這2個基因均屬于SOC1基因家族。這2個基因在長度、核苷酸相似度和預(yù)測蛋白質(zhì)相似度方面的差異都可佐證二者不是由同一個RNA剪接而來。
將紅掌的2個SOC1與其他物種的同源蛋白比較表明,它們均含有保守的SOC1模體(motif),系統(tǒng)進化分析結(jié)果與紅掌所處的植物學(xué)分類地位一致。本研究還分析了2個SOC1基因在紅掌不同組織部位的表達水平,發(fā)現(xiàn)它們在營養(yǎng)和生殖器官中均有表達,且在花器官中的表達水平由外向內(nèi)遞減,但它們的轉(zhuǎn)錄本表達豐度不一致。至于二者在組成、表達上的異同是否會導(dǎo)致其功能差異,尚有待進一步研究。
[1]SIMPSONGG,DEANC.Arabidopsis,the Rosetta stone offlowering time?Science,2002,296(5566):285-289.
[2]SUNG S,AMASINO R M.Vernalization and epigenetics:How plants rememberwinter.Current Opinion in Plant Biology,2004,7 (1):4-10.
[3]B?URLE I,DEAN C.The timing of developmental transitions in plants.Cell,2006,125(4):655-664.
[4]PARCY F.Flowering:A time forintegration.International Journal of Development Biology,2005,49(5/6):585-593.
[5]LEE H,SUH S S,PARK E,etal.The AGAMOUS?LIKE 20 MADS domain protein integrates floralinductive pathways in Arabidopsis. Genes and Development,2000,14(18):2366-2376.
[6]ONOUCHIH,IGE?O M I,PéRILLEUX C,et al.Mutagenesis of plants overexpressing CONSTANS demonstrates novelinteractions among Arabidopsis flowering?time genes.The Plant Cell,2000,12 (6):885-900.
[7]SAMACH A,ONOUCHI H,GOLD S E,et al.Distinct roles of CONSTANS target genes in reproductive development of Arabidopsis.Science,2000,288(5471):1613-1616.
[8]NAKAMURA T,SONG I J,FUKUDA T,et al.Characterization of TrcMADS1 gene of Trillium camtschatcense(Trilliaceae)reveals functional evolution of the SOC1/TM3?like gene family.Journal of Plant Research,2005,118(3):229-234.
[9]ZHONG XF,DAIX,XUJH,etal.Cloning and expression analysis of GmGAL1,SOC1 homolog gene in soybean.Molecular Biology Reports,2012,39(6):6967-6974.
[10]WANG S L,BERUTO M,XUE J Q,et al.Molecular cloning and potential function prediction of homologous SOC1 genes in tree peony.Plant Cell Reports,2015,34(8):1459-1471.
[11]LEIHJ,YUANHZ,LIUY,etal.Identification and characterization of FaSOC 1,a homolog of SUPPRESSOR OF OVEREXPRESSION OF CONSTANS1 from strawberry.Gene,2013,531(2):158-167.
[12]SREEKANTAN L,THOMAS M R.VvFT and VvMADS8,the grapevine homologues ofthe floralintegrators FT and SOC1,have unique expression patterns in grapevine and hasten flowering in Arabidopsis.Functional Plant Biology,2006,33(12):1129-1139.
[13]GREGIS V,SESSAA,DORCA?FORNELL C,etal.The Arabidopsis floralmeristem identity genes AP1,AGL24 and SVP directly repress class B and C floral homeotic genes.The Plant Journal,2009,60 (4):626-637.
[14]BORNER R,KAMPMANN G,CHANDLER J,et al.A MADS domain gene involved in the transition to flowering in Arabidopsis. The Plant Journal,2000,24(5):591-599.
[15]LIU C,ZHOU J,BRACHA?DRORI K,et al.Specification ofArabidopsis floral meristem identity by repression of flowering time genes.Development,2007,134(10):1901-1910.
[16]LIU C,CHEN H,ER H L,et al.Direct interaction of AGL24 and SOC1 integrates flowering signals in Arabidopsis.Development, 2008,135(8):1481-1491.
[17]IMMINK R G H,POSéD,FERRARIO S,etal.Characterization of SOC1's central role in flowering by the identification of its upstream and downstream regulators.Plant Physiology,2012,160 (1):433-449.
[18]LI D,LIU C,SHEN L S,et al.A repressor complex governs the integration of flowering signals in Arabidopsis.Developmental Cell,2008,15(1):110-120.
[19]LEE J,OH M,PARK H,et al.SOC1 translocated to the nucleus by interaction with AGL24 directly regulates LEAFY.The Plant Journal,2008,55(5):832-843.
[20]MA G Y,NING G G,ZHANG W,et al.Overexpression of Petunia SOC1?like gene FBP21 in tobacco promotes flowering without decreasing flower or fruit quantity.Plant Molecular Biology Reporter,2011,29(3):573-581.
[21]DING L H,WANG Y W,YU H.Overexpression of DOSOC1,an ortholog of Arabidopsis SOC1,promotes flowering in the orchid Dendrobium Chao Parya Smile.Plant and Cell Physiology,2013, 54(4):595-608.
[22]ZHANG X H,WEIJH,FAN S L,etal.Functionalcharacterization of GhSOC1 and GhMADS42 homologs from upland cotton (Gossypium hirsutum L.).Plant Science,2016,242:178-186.
[23]馬廣瑩,劉慧春,鄒清成,等.紅掌花發(fā)育B類基因AaPI的克隆及表達分析.分子植物育種,2014,12(5):950-955. MA G Y,LIU H C,ZOU Q C,etal.Cloning and expression analysis of B class gene AaPI from Anthurium andraeanum.Molecular Plant Breeding,2014,12(5):950-955.(in Chinese with English abstract)
[24]VOOGD C,WANG T C,VARKONYI?GASIC E.Functional and expression analyses ofkiwifruit SOC1?like genes suggestthatthey may nothave a role in the transition to flowering butmay affect the duration of dormancy.Journal of Experimental Botany,2015,66 (15):4699-4710.
[25]LIU C,ZHU C,ZENG H M.Key KdSOC1 gene expression profiles during plantlet morphogenesis under hormone,photoperiod,and droughttreatments.Genetics and Molecular Research,2016,15(1): gmr.15017579.
[26]NA X F,JIAN B,YAO WW,etal.Cloning and functionalanalysis ofthe flowering gene GmSOC 1?like,a putative SUPPRESSOR OF OVEREXPRESSION CO1/AGAMOUS?LIKE 20(SOC1/AGL 20) ortholog in soybean.PlantCellReports,2013,32(8):1219-1229.
[27]RUOKOLAINENS,YANPN,ALBERTVA,etal.Over?expression ofthe Gerbera hybrida At?SOC1?like1 gene Gh?SOC1 leadsto floral organ identity deterioration.Annals of Botany,2011,107(9):1491-1499.
[28]ZHAO S Z,LUO Y Z,ZHANG Z L,et al.ZmSOC1,a MADS?box transcription factor from Zea mays,promotes flowering in Arabidopsis.International Journal of Molecular Sciences,2014,15 (11):19987-20003.
[29]KIMURA Y,AOKI S,ANDO E,et al.A flowering integrator, SOC1,affects stomatal opening in Arabidopsis thaliana.Plant and Cell Physiology,2015,56(4):640-649.
[30]LIU S N,MA T F,MA L Y,et al.Ectopic expression of PvSOC1, a homolog of SOC1,from Phyllostachys violascens,promotes flowering in Arabidopsis and rice.Acta Physiologiae Plantarum, 2016,38(7):166.
Cloning,sequence and expression analysis of two SOC1 genes from Anthurium.Journal of Zhejiang University(Agric.&Life Sci.),2017,43(3):289-297
MA Guangying1,ZHU Kaiyuan1,SHI Xiaohua1,ZOU Qingcheng1,LIU Huichun1,ZHAN Jing2,TIAN Danqing1*
(1.Flower Research and Development Center,Zhejiang Academy of Agricultural Sciences,Hangzhou 311202,China;2.Hangzhou Xiaoshan Technician College of Zhejiang Province,Hangzhou 311202,China)
Anthurium;SOC1 gene;bioinformatics;expression analysis
):馬廣瑩(http://orcid.org/0000-0002-4081-7870),E?mail:mgyflower@163.com
2016-10-11;接受日期(Accepted):2017-04-28
S 682.14;Q 943.2
A
10.3785/j.issn.1008-9209.2016.10.111
國家自然科學(xué)基金(31200527);浙江省農(nóng)業(yè)新品種選育重大專項(2016C02056-13)。
田丹青(http://orcid.org/0000-0002-8646-4206),Tel:+86-571-82704525,E?mail:tdq0123@163.com
(

