草莓輕型黃邊病毒外殼蛋白抗原表位預測、克隆、表達及其免疫原性分析
楊菊梅,馬建忠,*,王永剛,鄧子兵,魏 艷,潘 博,李印武
草莓輕型黃邊病毒外殼蛋白抗原表位預測、克隆、表達及其免疫原性分析
楊菊梅1,馬建忠1,*,王永剛1,鄧子兵1,魏 艷1,潘 博2,李印武2
(1.蘭州理工大學生命科學與工程學院,甘肅 蘭州 730050;2.甘肅啟明星節能科技有限公司,甘肅 白銀 730913)
應用IEDB、DNAStar、DNAMAN和SnapGene等生物信息學工具對草莓輕型黃邊病毒外殼蛋白的氨基酸殘基序列進行分析。選擇一段抗原性較強的肽段(位于外殼蛋白27~38氨基酸殘基處),依據大腸桿菌(Escherichia coli)的密碼子偏好性化學合成了該肽段的DNA編碼序列(AE)。AE片段克隆至E. coli表達載體pET32a(+)的EcoRⅠ和XhoⅠ位點,獲得重組質粒pET32a(+)-AE。含AE片段的開放閱讀框長561 bp,編碼了一個187氨基酸殘基的重組融合蛋白,理論分子質量為20.29 kD。重組質粒pET32a(+)-AE分別在E. coli BL21(DE3)和E. coli Rosetta中進行了誘導表達和條件優化。重組融合蛋白在E. coli Rosetta中的最佳表達條件為1.5 mmol/L異丙基-β-D-硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG)、35 ℃誘導表達2 h,產率為13.22 mg/L;在E. coli BL21(DE3)中的最佳表達條件是為0.5 mmol/L IPTG、30 ℃誘導表達2 h,產率為9.55 mg/L。重組融合蛋白經十二烷基硫酸鈉-聚丙烯酰胺凝膠電泳分離、質譜鑒定表明,其含有所預測的草莓輕型黃邊病毒外殼蛋白抗原表位肽段AE。AE重組融合蛋白經Ni2+親和色譜純化、EK酶切、分子篩除去融合蛋白標簽后,免疫產蛋雞。從免疫雞所產雞蛋中提取雞卵黃免疫球蛋白(immunoglobulin of yolk,Igy),Dot-Blot檢測抗體結合活性。結果表明,所提取的Igy可特異性識別AE肽段和SMyEV外殼蛋白。這一結果表明所預測的AE肽段具有較好的免疫原性。
草莓輕型黃邊病毒;外殼蛋白;抗原表位預測;原核表達;質譜鑒定;免疫原性
目前已經報道的可侵染草莓的病毒有30余種[1],且類型多樣,對草莓的農業生產危害很大。草莓輕型黃邊病毒(strawberry mild yellow edge virus,SMyEV)隸屬甲型線形病毒科(Alphaflexiviridae)馬鈴薯X病毒屬(Potexvirus)[2]。SMyEV侵染草莓植株后,植株長勢衰退、葉緣褪綠、小葉凹陷[3]。SMyEV單獨侵染草莓可致其產量下降20.8%,和其他病毒混合侵染可致其產量下降80%[4]。SMyEV的基因組包含1 條單鏈RNA,長5 966 bp,含有6 個不同的開放閱讀框(open reading frame,ORF)。其中,ORF5編碼其外殼蛋白,含有242 個氨基酸殘基、理論分子質量為26 kD[5]。病毒的外殼蛋白決定其免疫學特異性[6]。據此,通過表達、純化SMyEV外殼蛋白或其抗原表位、制備其抗體,可建立SMyEV的免疫學檢測方法。
大腸桿菌(Escherichia coli)表達系統簡單、成熟,是目前使用最為廣泛的蛋白質表達系統[7]。E. coli具有培養條件簡單、生長繁殖快、外源基因表達量高等優點。隨著分子生物學研究的深入,E. coli表達系統具備了可用于不同來源基因、不同蛋白質修飾的表達菌株[7]。E. coli BL21是E. coli B菌株的蛋白酶缺陷株,通常用來表達異源蛋白。DE3是一段插入E. coli基因組中并穩定遺傳的DNA片段,主要由乳糖操縱子的啟動子、操縱基因、T7噬菌體RNA聚合酶編碼基因等部分組成。在乳糖或異丙基-β-D-硫代半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG)誘導下可表達T7噬菌體RNA聚合酶,后者專一識別pET32a(+)載體上的T7噬菌體啟動子后的讀碼框并對其大量轉錄[8]。E. coli Rosetta宿主菌是E. coli BL21衍生菌,能明顯增強帶有E. coli稀有密碼子的真核蛋白的表達。該菌株通過一個相容性氯霉素抗性質粒補充密碼子AUA、AGG、AGA、CUA、CCC和GGA的tRNAs[9]。
抗原數據庫的建立、抗原表位共性的深入研究,產生了一系列抗原表位預測的方法或算法[10]。這些方法的準確性雖有待于進一步的提高,但毫無疑問,基于這些算法預測高抗原活性表位,進一步利用基因工程手段實現抗原表位的克隆、表達和純化,可大大簡化傳統抗原的篩選流程、縮短抗原的制備工作。
抗體(免疫球蛋白,immunoglobulin,Ig)是機體受到外來抗原物質刺激后,機體免疫系統細胞產生的可以特異性地識別和結合外來抗原并使之失活的一類球蛋白。雞(或者說禽類)經特定抗原免疫后,可產生相應的特異性抗體,并不斷儲存于卵黃中,即雞卵黃抗體(immunoglobulin of yolk,Igy)。Igy分離自雞蛋卵黃,不需要處死動物,其熱穩定性和抗酸性均優于IgG,在免疫學的診斷技術和免疫治療方面的應用日益廣泛[11]。由于鳥類和哺乳動物之間較遠的進化距離,哺乳動物的蛋白質更易免疫雞使其產生抗體[12]。本研究應用生物信息學分析軟件對SMyEV外殼蛋白的親水性、可及性、可塑性、抗原性和二級結構等進行了綜合預測。通過比較潛在抗原表位區段的平均抗原指數并選擇活性強、特異性高的抗原表位肽段,設計、合成了其DNA編碼片段,進一步實現了其原核表達、分離純化和質譜鑒定。以此策略制備免疫用抗原快速、成本低,并可避免外源基因在E. coli中表達時的密碼子偏好性等問題。
1 材料與方法
1.1 材料、菌種和載體
感染SMyEV的草莓(Fragaria nipponica,栽培名為紅顏)植株為蘭州理工大學馬建忠教授實驗室所保存;受疫雞(Gallus gallus)品種為京紅一號,購自正大養雞場。
E. coli DH5α、E. coli BL21(DE3)、E. coli Rosetta、表達載體pET32a(+)為蘭州理工大學馬建忠教授實驗室所保存。
1.2 試劑
SanPrep柱式質粒DNA小量抽提試劑盒、SanPrep柱式膠回收試劑盒、IPTG、β-巰基乙醇、一步法植物活性蛋白質提取試劑盒 生工生物工程(上海)股份有限公司;限制性內切酶EcoRⅠ、XhoⅠ、T4 DNA連接酶、DNA Marker、蛋白質Marker 寶生物工程(大連)有限公司;腸激酶(enterokinase,EK)、Ni-Agarose Resin 華瑞德生物科技有限公司;Sephadex G-50 北京索萊寶科技有限公司;胸苷激酶1檢測試劑盒 華瑞同康生物技術(深圳)有限公司。
1.3 方法
1.3.1 蛋白質性質分析及抗原表位優勢肽段AE的確定
檢索NCBI(http://www.ncbi.nlm.nih.gov)數據庫,提取SMyEV外殼蛋白的基因序列(AJ577358.1)和蛋白質序列(AKN20471.1)。應用IEDB、DNAStar、DNAMAN和SnapGene等軟件進行蛋白質性質分析和抗原表位預測。基于Garnier-Robson和Chou-Fasman兩種算法預測SMyEV外殼蛋白抗原表位[13-14];Hopp-Woods算法預測親水性[15];Karplus-Schulz算法預測柔韌性[16];Emini算法預測表面可及性[17];Jameson-Wolf算法預測抗原性指數[18]。參考吳玉章等[19]的算法計算潛在抗原表位區段的平均抗原指數。以Protein BLAST檢索NCBI數據庫(http:// blast.ncbi.nlm.nih.gov/Blast.cgi)分析優勢抗原肽段的特異性。利用GeneDesign(http://54.235.254.95/gd)以及E. coli的密碼子偏好性反轉譯獲得優勢肽段的核苷酸編碼序列[17]。之后以在線軟件Rare Coden Caltor(http://www. doembi.ucla.edu/sumchan/caltor.htmL)分析核苷酸編碼序列中的稀有密碼子[18]。
本研究中所預測的優勢抗原表位肽段AE的DNA編碼序列如下:
F片段:5’-aattcCTGCCGGGTCGTACCCCGAACCC GAACGCTAACGTTc-3’
R片段:5’-tcgagAACGTTAGCGTTCGGGTTCGGGG TACGACCCGGCAGg-3’
上述片段退火后,可形成編碼AE肽段11 個氨基酸殘基的密碼子。其5’端含有限制性內切酶EcoRⅠ的黏性末端(AATT),3’端含有限制性內切酶XhoⅠ的黏性末端(TCGA)。核苷酸片段F和R由生工生物工程有限公司(上海)合成。
1.3.2 抗原優勢表位肽段AE的克隆
將AE的編碼序列F片段和R片段的濃度調至1 pmoL/μL。F片段和R片段的退火反應10 μL:F片段5 μL,R片段5 μL;退火條件:95 ℃、3 min,69 ℃、10 min。參考SanPrep柱式質粒DNA小量抽提試劑盒提取質粒DNA pET32a(+),用限制性內切酶EcoRⅠ和XhoⅠ將載體pET32a(+)進行雙酶切。酶切反應體系60 μL:pET32a(+)DNA 3 μg,10×H Buffer 6 μL,EcoRⅠ 2 μL (10 U/μL),XhoⅠ 2 μL(10 U/μL),ddH2O補充至60 μL,37 ℃保溫2 h。酶切產物經0.8%瓊脂糖凝膠電泳分離,SanPrep柱式凝膠回收試劑盒回收載體片段。回收后的載體pET32a(+)與AE混合后16 ℃連接過夜[20]。連接產物轉化E. coli DH5α感受態細胞,轉化菌涂布于含100 μg/mL氨芐青霉素的LB平板上,37 ℃培養過夜[20]。次日挑取單菌落接種于4 mL含100 μg/mL氨芐青霉素的LB液體培養基中,37 ℃培養過夜,菌液送蘇州金唯智生物科技有限公司測序。
1.3.3 表達條件的優化
為了目的蛋白能夠高效表達,分別選用E. coli BL21(DE3)和E. coli Rosetta進行表達條件(誘導劑IPTG濃度、誘導時間和誘導溫度)的優化。重組表達載體轉化E. coli BL21(DE3)和E. coli Rosetta感受態細胞,轉化菌涂布于含50 μg/mL氨芐青霉素的LB平板上,37 ℃倒置培養過夜,挑單菌落于4 mL含50 μg/mL氨芐青霉素的LB液體培養基中37 ℃培養過夜,過夜培養物按1%的接種量接入20 mL含50 μg/mL氨芐青霉素LB液體培養基中,37 ℃培養至OD600nm為0.847時加入0、0.5、1.0、1.5、2.0 mmol/L IPTG 37 ℃誘導表達4 h,取1 mL樣品12 000 r/min離心2 min收集菌體,加入100 μL水和100 μL 2×Loading Buffer煮沸5 min,15%的十二烷基硫酸鈉-聚丙烯酰胺凝膠電泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)進行電泳[18],BandScan掃描計算誘導表達蛋白帶的相對含量。以相同的方法,在最佳IPTG濃度條件下以20、25、30、35、40 ℃培養E. coli,確定誘導表達蛋白帶相對含量最高的培養溫度。類似地,在最佳IPTG濃度和最佳誘導溫度條件下培養E. coli 0、1、2、4、6、8 h,確定誘導表達蛋白帶達最高相對含量時的最短培養時間。
在E. coli BL21(DE3)和E. coli Rosetta菌株的最佳表達條件下,誘導表達重組融合蛋白,SDS-PAGE分離,BandScan掃描目的蛋白的含量,并用考馬斯亮藍法[21]測定總蛋白質量濃度,分別計算目的蛋白在E. coli BL21(DE3)和E. coli Rosetta中的產率。
1.3.4 重組融合蛋白的鑒定
為了鑒定含AE肽段的重組融合蛋白是否正確,在E. coli Rosetta菌株中誘導表達的可溶性蛋白進行SDSPAGE分離,參照蛋白質低分子質量標志,從凝膠上切下與重組融合蛋白理論分子質量相近的誘導表達蛋白條帶,送生工生物工程(上海)有限公司進行串聯質譜分析,以確定誘導表達蛋白的氨基酸殘基序列。
1.3.5 重組融合蛋白的純化及免疫原性分析
原核表達、質譜鑒定氨基酸序列正確的重組融合蛋白,經過Ni2+親和色譜純化[20]、EK酶切,酶切體系為200 μL(融合蛋白150 μL(2 mg)、EK 16 μL(0.032 μg)、10×Buffer 20 μL,ddH2O補至200 μL),23 ℃條件下反應16 h。用SephadexG-50分子篩層析回收AE小片段[20]。1 μg/μL的AE抗原500 μL(500 μg)與完全弗氏佐劑500 μL混勻,肌肉注射免疫雞(京紅一號)。2 周之后,同樣劑量注射第2次。間隔2 周,用2 倍劑量的AE抗原免疫第3次。水稀釋-冰乙醇沉淀法[22]提取免疫后雞蛋中的雞卵黃免疫球蛋白(Igy)。根據一步法植物活性蛋白質提取試劑盒從感染SMyEV草莓植株葉片中提取總可溶性蛋白,作為檢測樣品。以pET32a(+)空載體在E. coli Rosetta中的表達產物(10 ng)為陰性對照,pET32a-AE在E. coli Rosetta中的表達產物(10 ng)陽性對照,通過Dot-Blot分析抗體活性。Dot-Blot檢測時草莓病株總可溶性蛋白為200 ng。Igy稀釋200 倍,質量濃度為5 μg/mL。
2 結果與分析
2.1 抗原表位預測
2.1.1 潛在抗原表位預測
抗原表位的親水性、可及性和可塑性較高,一般不會分布在α-螺旋、β-折疊區段,α-螺旋和β-折疊因其內部鍵能較高,形態固定,常處于蛋白質分子內部,難以與抗體嵌合,而β-轉角和無規則卷曲多處于蛋白質分子的表面,結構松散,有利于與抗體嵌合,成為抗原表位的可能性大[18]。根據以上原則,分別分析SMyEV外殼蛋白的親水性、可及性、可塑性、抗原性和二級結構。在預測二級結構時,為提高其準確性,首先通過IEDB工具在線預測SMyEV外殼蛋白二級結構中的α-螺旋和β-折疊區段,又用DNAStar軟件中提供的Garnier-Robson、Chou-Fasman兩種算法來預測其二級結構,綜合分析后得到了SMyEV外殼蛋白的潛在抗原表位(表1)。

表1 SMYEV外殼蛋白抗原表位綜合預測結果Table 1 Comprehensive prediction of antigen epitopes of the SMYEV coat protein
2.1.2 優勢抗原表位肽段AE的確定
目前的多個抗原表位預測方法即可預測具表位活性的氨基酸殘基區段,也可預測每個區段活性的強弱。據此,按吳氏平均抗原指數算法[19],對表1的抗原表位活性區段的強弱進行了比較(表2)。其中,27~38氨基酸殘基區段(LPGRTPNPNANVE,即AE片段)的平均抗原指數最高。為避免多肽免疫產生的抗體與天然蛋白之間的親和力低的風險,抗原表位的氨基酸殘基數應當在8~20 個之間[15]。AE片段的氨基酸殘基數為12 個,符合上述要求。此外,為避免所預測的抗原表位區域隱藏在蛋白質分子的內部,應優先選擇位于蛋白質分子N、C兩端的肽段。因為在完整的蛋白質分子中,N、C兩端通常暴露在蛋白質分子表面。AE肽段則剛好位于SMyEV外殼蛋白的N端,因此,該肽段被確定為優勢抗原表位。

表2 SMYEV外殼蛋白B細胞表位優勢區段的平均抗原指數Table 2 Average antigenic index of the B cell epitope dominant regions in the SMYEV coat protein
2.2 優勢抗原表位編碼片段AE的克隆
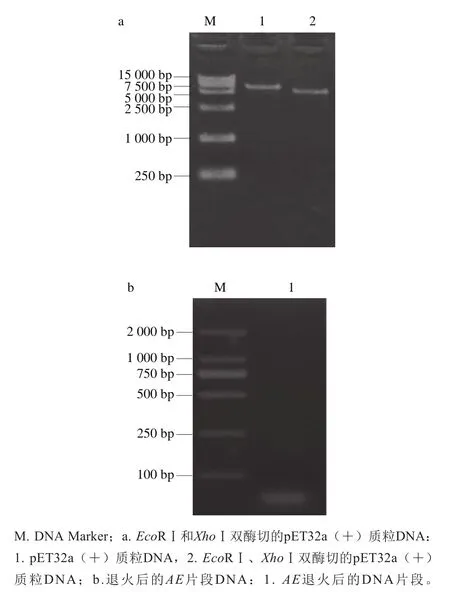
圖1 酶切載體DNA和退火后的AE片段DNA的瓊脂糖凝膠電泳Fig. 1 Separation of the enzymatically-digested vector DNA and the annealed AE fragment by agarose gel electrophoresis
載體pET32a(+)經EcoRⅠ、XhoⅠ雙酶切,瓊脂糖凝膠電泳分離后回收大片段(圖1a)。F片段與R片段退火后形成的雙鏈DNA片段AE的大小為42 bp,2%瓊脂糖凝膠電泳進行分離,出現一條均一的條帶,大小與AE的理論預期值基本一致(圖1b)。將AE與雙酶切的載體pET32a(+)連接后,挑取重組質粒進行核苷酸序列分析以確定AE片段的正確插入。含AE片段的重組克隆pET32a(+)-AE的誘導表達區的ORF長561 bp,編碼了一個187氨基酸殘基的重組融合蛋白(圖5a),理論分子質量為20.29 kD。
2.3 重組融合蛋白誘導表達條件的優化
外源基因在E. coli表達時影響因素較多,密碼子偏好性、稀有密碼子、發夾結構等都可影響到外源基因的表達。本研究的AE片段雖然是依據E. coli的密碼子偏好性設計,而且排除了稀有密碼子的存在,但其表達效率仍依賴于實驗檢驗。據此,選擇E. coli BL21(DE3)和E. coli Rosetta來表達pET32a(+)-AE,以此了解不同E. coli菌株對同一外源基因的表達是否效果不同。
2.3.1 誘導劑IPTG最適濃度的確定
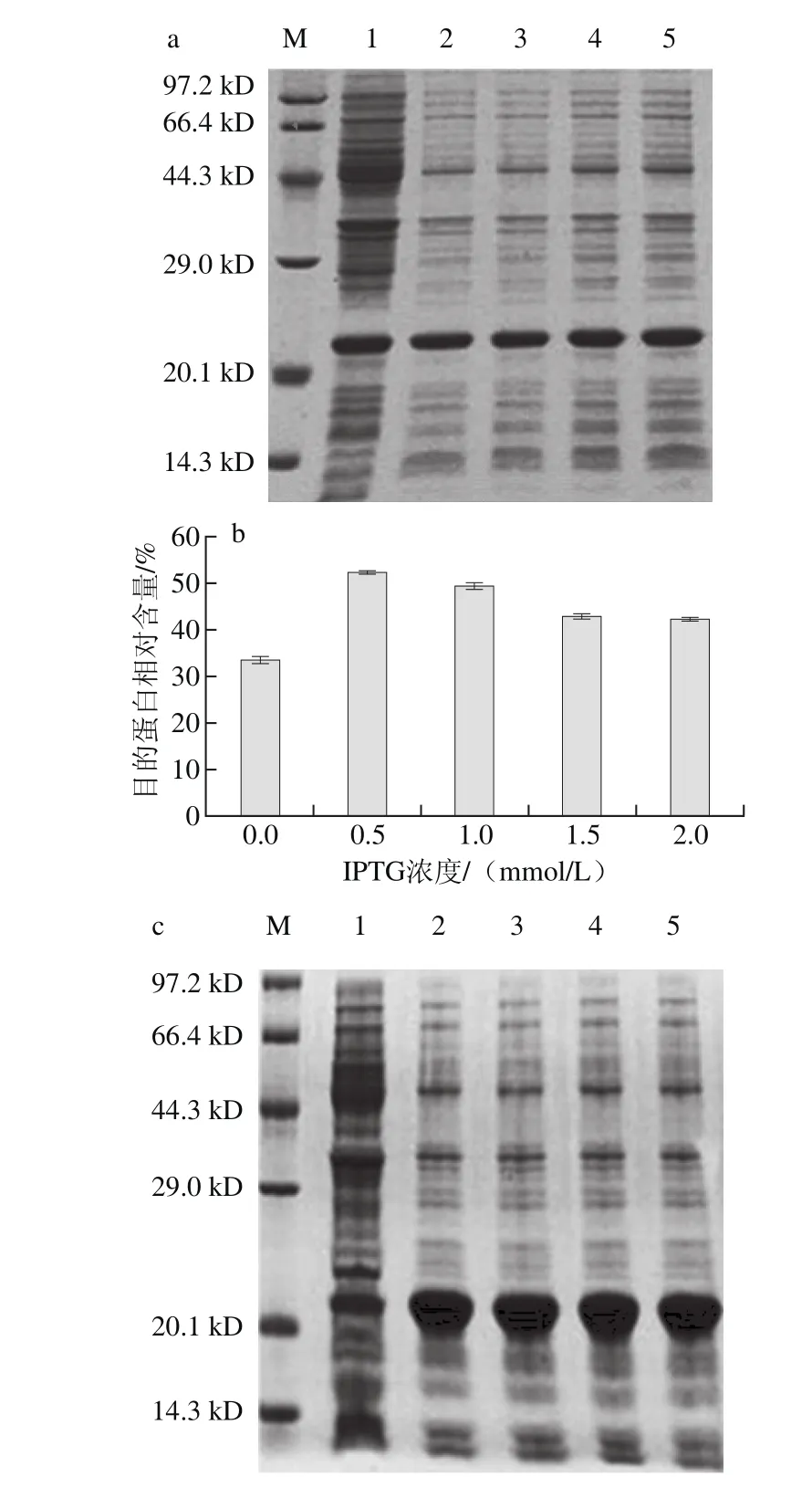
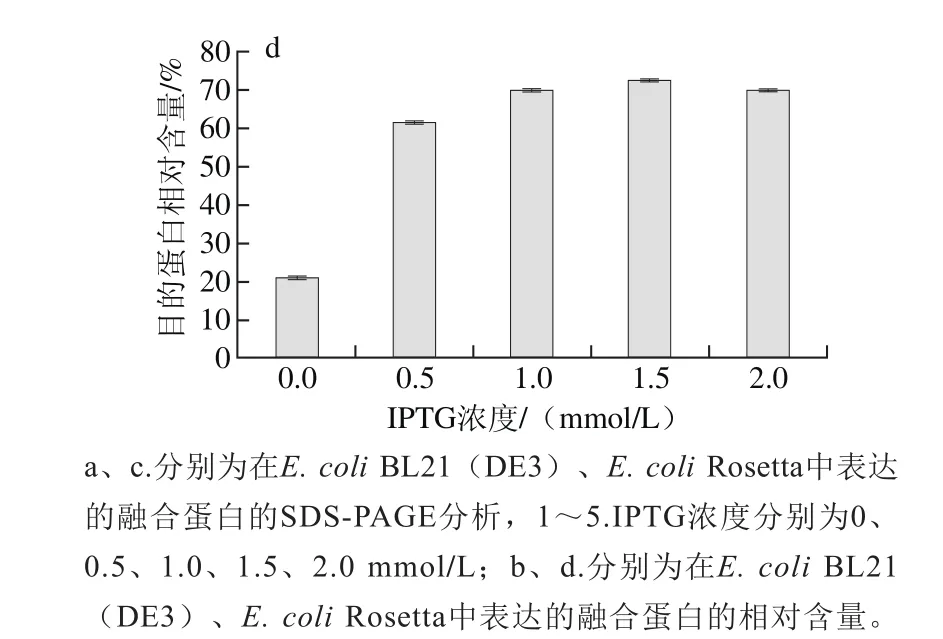
圖2 誘導劑IPTG的濃度對重組融合蛋白表達的影響Fig. 2 Effects of IPTG concentration on expression of the recombinant fusion protein
IPTG作為誘導劑,能夠解除阻遏物對lac操縱子的抑制,使啟動子啟動下游基因的轉錄。在低IPTG濃度下,基因表達隨IPTG濃度升高而增強。但IPTG有一定的毒性,濃度過高又會抑制細菌的生長。所以,確定IPTG的最佳誘導濃度是重組融合蛋白高效表達的基礎。圖2結果表明,當宿主菌株為E. coli BL21(DE3),IPTG濃度在0~2.0 mmol/L時,重組融合蛋白的相對含量在0.5 mmol/L IPTG時最高,占可溶性總蛋白的51.1%。當宿主菌株為E. coli Rosetta時,重組融合蛋白的相對含量在1.5 mmol/L IPTG時最高,占到可溶性總蛋白的72.4%。在E. coli BL21(DE3)菌株中,在誘導重組融合蛋白表達最高的IPTG濃度(0.5 mmol/L)之后,隨IPTG濃度增加,重組融合蛋白的表達量較E. coli Rosetta明顯下降(圖2)。在E. coli Rosetta菌株中,IPTG誘導表達的融合蛋白含量都高于60%;而在E. coli BL21(DE3)菌株中,目的蛋白的最高含量也只有51.1%。據此可以確定,E. coli Rosetta菌株比E. coli BL21(DE3)菌株更適合表達本實驗中的重組融合蛋白。
2.3.2 最佳誘導溫度的確定
培養溫度通過影響細菌體內新陳代謝的速率進而影響到細菌的生長,也就是說,溫度會影響到細菌體內基因的表達、蛋白的積累。E. coli的最適生長溫度為37 ℃,但是不同外源蛋白的最佳誘導溫度不一定是其最佳生長溫度。培養溫度高,細菌生長快,蛋白表達量大,但多以包涵體的形式存在;培養溫度低,菌體生長緩慢,蛋白表達量通常也降低。不同培養溫度對重組融合蛋白表達量的影響結果表明,在E. coli BL21(DE3)中,誘導溫度為30 ℃時,重組融合蛋白的相對含量最高,為48.8%;在E. coli Rosetta中,誘導溫度為35 ℃時,重組融合蛋白的相對含量最高,為64.1%。不同溫度條件下,重組融合蛋白在E. coli Rosetta中的相對含量均高于E. coli BL21(DE3)菌株(圖3)。

圖3 誘導溫度對重組融合蛋白表達的影響Fig. 3 The effect of the induced temperatures on expression of the recombinant fusion protein
2.3.3 最佳誘導時間的確定
誘導時間通過影響菌體的數量及外源蛋白的積累量而影響外源蛋白的表達,誘導時間過短菌體數量太少,外源蛋白表達積累量低。誘導時間過長,菌體進入衰退期。而且,若外源蛋白不穩定,則有可能隨培養時間的延長而降解。不同誘導時間對重組融合蛋白表達量的影響結果表明,在E. coli BL21(DE3)中,誘導時間為2 h時,重組融合蛋白的相對含量最高,為47.0%;在E. coli Rosetta中,誘導時間為2 h時,重組融合蛋白的相對含量最高,為69.3%。不同誘導時間下,重組融合蛋白在E. coli Rosetta中的相對含量均高于E. coli BL21(DE3)菌株(圖4)。隨著誘導時間的延長,目的蛋白的相對含量有所下降,這可能是其他蛋白的表達量相對增加或是目的蛋白出現的降解速率較快所致。
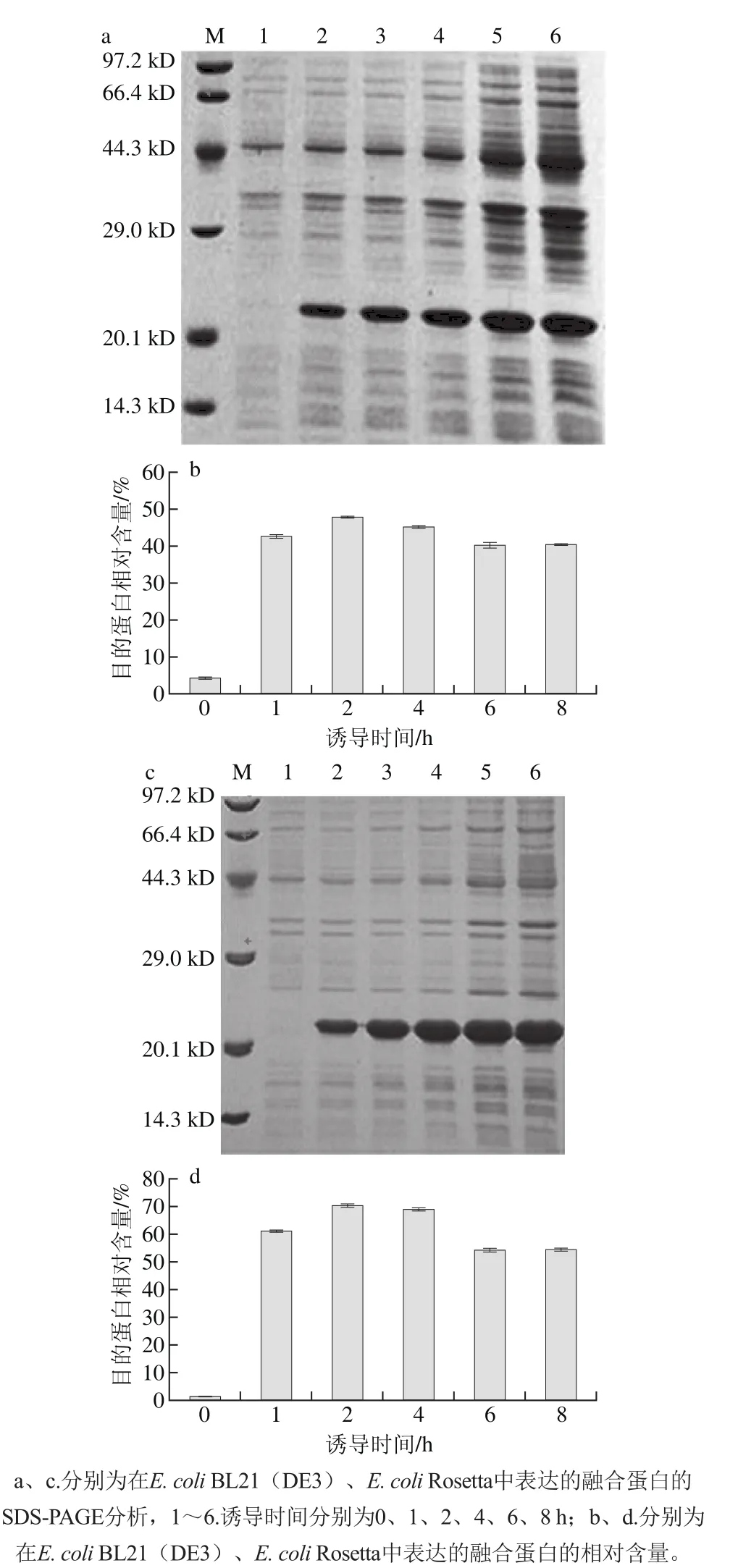
圖4 重組融合蛋白表達的時間進程Fig. 4 Time-courses of fusion protein expression
通過上述實驗,分別優化出了重組融合蛋白在E. coli BL21(DE3)和E. coli Rosetta菌株中的最佳表達
條件。重組融合蛋白在E. coli Rosetta中的最佳表達條件是1.5 mmol/L IPTG,35 ℃誘導表達2 h,在最佳條件下其產率為13.22 mg/L;在E. coli BL21(DE3)中的最佳表達條件是0.5 mmol/L IPTG,30 ℃誘導表達2 h,在最佳條件下產率為9.55 mg/L。
2.4 重組融合蛋白的鑒定
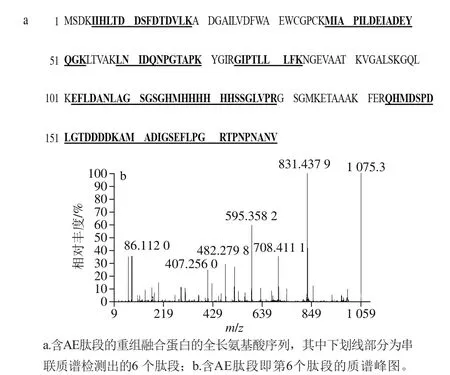
圖5 重組融合蛋白的質譜鑒定Fig. 5 Identification of the recombinant fusion protein by mass spectrometry
為進一步確定含pET32a(+)-AE E. coli誘導表達的蛋白條帶是否包含了所設計的AE肽段,E. coli Rosetta(pET32a(+)-AE)在最適誘導條件下培養后分離可溶性總蛋白,經SDS-PAGE分離,所誘導表達蛋白的大小處在22.1 kD的位置,與重組融合蛋白的理論分子質量20.29 kD比較接近但并不一致。切出22.1 kD蛋白條帶后送生工生物工程(上海)有限公司進行串聯質譜分析。質譜分析共檢測到6 個胰蛋白酶酶解肽段(圖5)。這6 個肽段的氨基酸殘基序列與目的融合蛋白的6 段氨基酸殘基序列完全一致(圖5a)。串聯質譜結果證明,誘導表達的重組融合蛋白含有AE肽段的12 個氨基酸殘基序列。
2.5 Igy抗AE肽段和草莓SMyEV的活性分析

圖6 純化的AE肽段免疫雞后制備的IgY進行的Dot-Blot分析Fig. 6 Dot-Blot analysis of the immunogenicity of AE peptide
AE是理論預測的SMyEV外殼蛋白中具有較強抗原表位活性的肽段。但目前抗原表位的預測方法或算法并不100%準確。因此,通過實驗驗證AE的抗原活性則是必須工作。鎳離子親和色譜純化的上述重組融合蛋白經腸激酶消化、葡聚糖分子篩分離AE肽段,再免疫產蛋雞。以首次免疫21 d后的雞卵黃中制備的Igy為一抗,辣根過氧化物酶標記的鼠抗雞抗體為二抗,以Dot-Blot分析3 種來源的蛋白混合物(含有pET32a(+)載體的E. coli Rosetta的總可溶性蛋白、含有pET32a(+)-AE載體的E. coli Rosetta的總可溶性蛋白和感染了SMyEV病毒草莓葉片的總可溶性蛋白)。結果表明(圖6),以AE肽段免疫雞產生的Igy可以專一性地結合SMyEV的外殼蛋白。也就是說,所預測的SMyEV外殼蛋白抗原表位AE確實具有免疫原性。
3 討 論
隨著生命科學與數學、計算機科學的交叉融合,產生了一門新的學科——生物信息學。生物信息學理論、方法已廣泛應用于大分子結構預測、藥物設計、大分子相互作用等研究領域[23]。以生物信息學理論和方法為基礎的蛋白質抗原表位預測對于疫苗的設計、抗體的制備以及分子檢測試劑盒的研制等都是必不可少的工具[23]。運用多種生物信息學方法/軟件綜合預測蛋白質分子抗原表位成功率已達到86%[24]。目前預測多肽抗原表位,主要的生物信息學方法綜合考慮了蛋白質氨基酸殘基序列的親水性、可及性、柔韌性、抗原性及二級結構等參數[25]。對于上述參數,其選擇的原則是:親水性高,疏水性低,可及性高,選擇的氨基酸序列不存在α-螺旋、β-折疊,在β-轉角附近,且易于或有可能形成不規則卷曲結構[26]。抗原表位序列以8~20 個氨基酸殘基為宜,應避免出現4 個以上連續相鄰的疏水性氨基酸殘基,并且疏水性殘基數目不超過6 個,序列中帶電荷的氨基酸殘基越多越好[25]。為提高預測的準確性,本研究在對SMyEV外殼蛋白抗原表位預測時,在采用多種生物信息學工具軟件對相關參數進行綜合分析的基礎上,進一步以位于蛋白質分子C端或N端的肽段為優先選擇。因為這類肽段通常位于蛋白質分子的表面且多為無規卷曲。由此確定了SMyEV的最終候選抗原表位肽段AE,由12 個氨基酸殘基組成,位于SMyEV外殼蛋白分子的N端。
AE肽段的DNA編碼序列雖以E. coli的偏好密碼子反轉譯而來,而且排除了稀有密碼子的出現,但在兩個常用的E. coli表達株系中重組融合蛋白的表達量存在明顯差異。E. coli Rosetta為E. coli BL21(DE3)的衍生菌,是針對稀有密碼子的優勢表達菌株。雖然本研究所設計的抗原表位已經排除了E. coli稀有密碼子的存在,但是,實驗結果表明,重組融合蛋白在E. coli Rosetta中的表達量明顯高于E. coli BL21(DE3)。為何在E. coli Rosetta中的誘導表達量明顯較高其原因還有待于進一步研究。
于翠梅等[27]報道了草莓輕型黃邊病毒CP基因的克隆、序列分析及原核表達工作。與他們的研究工作相比,本研究工作省去了草莓病毒苗的尋找、種植,總RNA的制備以及RT-PCR擴增目的蛋白的編碼基因(即CP的編碼基因)。其次,直接擴增自草莓SMyEV的CP基因在原核細胞中表達時由于密碼子偏好性或稀缺性往往表達效率低或不表達。而本研究通過抗原表位預測、密碼子優化、化學合成一較小的DNA片段(編碼12 個氨基酸殘基的AE肽段),不僅大大簡化了實驗程序,而且降低了實驗成本,最重要的是該方案極易實現外源片段在原核系統中的高效表達。雖然抗原表位預測目前還不能做到百分之百的準確,但這條途徑的上述優越性肯定了這是一個值得探索并不斷提高其準確性的方案。在本研究中,純化的AE肽段免疫雞后,其卵黃免疫球蛋白對細菌表達的含AE肽段的重組蛋白(未進行分離純化)和來自感染SMyEV的草莓可溶性蛋白均表現了較好的結合活性。這也表明,本研究所使用的抗原表位簡單、快速、低成本的制備策略確實可行。
[1] THOMPSON J R, JELKMANN W. Strain diversity and conserved genome elements in strawberry mild yellow edge virus[J]. Archives of Virology, 2004, 149(10): 1897-1909. DOI:10.1007/s00705-004-0353-4.
[2] MARTIN R R, TZANETAKIS I E. Characterization and rencent advances in detection of strawberry virus[J]. Plant Disease, 2006, 90(4): 384-396.
[3] SPIEGEL S. Double-stranded RNA in strawberry plants infected with strawberry mild yellow-edge virus[J]. Phytopathology, 1987, 77(10): 1492-1494. DOI:10.1094/Phyto-77-1492.
[4] THOMPSON J R, WETZEL S, KLERKS M M, et al. Multiplex RT-PCR detection of four aphid-borne strawberry viruses in Fragaria spp. in combination with a plant mRNA specific internal control[J]. Journal of Virologica Methods, 2003, 111(2): 85-93. DOI:10.1016/S0166-0934(03)00164-2.
[5] JELKMANN W, MARTIN R R, LESEMANN D E, et al. A new potexvirus associated with strawberry mild yellow edge disease[J]. Journal of General Virology, 1990, 71: 1251-1258. DOI:10.1099/0022-1317-71-6-1251.
[6] KING A, ADAMS M, CARSTENS E, et al. Virus taxonomy: ninth report of the international committee on taxonomy of viruses[M]. Amsterdam: Elsevier Academic Press, 2012: 639-650.
[7] ARBABI-GHAHROUDI M, TANHA J, MACKENZIE R. Prokaryotic expression of antibodies[J]. Cancer & Metastasis Reviews, 2005, 24(4): 501-519. DOI:10.1007/s10555-005-6193-1.
[8] GONG M, ZHANG J, LIU D, et al. The study of BL21 (DE3)/pET-11 as an expression system of human IL-2[J]. Acta Academiae Medicinae Sinicae, 1996, 18(1): 60-65.
[9] CLINTON J. Expression and purification of ATF3 in E. coli Rosetta and HaCaT cells for further studies, and the induction of ATF3 by UVA and anisomycin in HaCaT cells[J]. Synfacts, 2010, 9(1): 86.
[10] SAHA S, RAGHAVA G P. Prediction of continuous B-cell epitopes in an antigen using recurrent neural network[J]. Proteins: Structure Function & Bioinformatics, 2006, 65(1): 40-48. DOI:10.1002/ prot.21078.
[11] 劉寶全, 孫惠蘭, 李俊寶, 等. 獸醫免疫學實驗指導[M]. 上海: 科學技術出版社, 1985: 74-77.
[12] GARNIER J. Analysis of the accuracy and implications of simple methods for predicting the secondary structure of globular proteins[J]. Journal of Molecular Biology, 1978, 120(1): 97-120. DOI:10.1016/0022-2836(78)90297-8.
[13] LESLIE G A, CLEM L W. Phylogeny of immunoglobulin structure and functions. Ⅲ. Immunoglobulins of the chicken[J]. Journal of Experimental Medicine, 1969, 130: 1337-1352. DOI:10.1084/ jem.130.6.1337.
[14] CHOU P Y, FASMAN G D. Prediction of the secondary structure of proteins from their amino acid sequence[J]. Advances in Enzymology and Related Areas of Molecular Biology, 1978, 47(2): 145-148. DOI:10.1016/0968-0004(77)90440-6.
[15] HOPP T P, WOODS K R. Prediction of protein antigenic determinants from amino acid sequences[J]. PNAS, 1981, 78(6): 3824-3828.
[16] KARPLUS P A, SCHULZ G. Prediction of chain flexibility in proteins[J]. Naturwissenschaften, 1985, 72(4): 212-213. DOI:10.1007/ BF01195768.
[17] EMINI E A, HUGHES J V, PERLOW D S, et al. Induction of hepatitis A virus-neutralizing antibody by a virus-specific synthetic peptide[J]. Journal of Virology, 1985, 55(3): 836-839.
[18] JAMESON B, WOLF H. The antigenic index: a novel algorithm for predicting antigenic determinants[J]. Computer Applications in the Biosciences, 1988, 4(1): 181-186. DOI:10.1093/ bioinformatics/4.1.181.
[19] 吳玉章, 朱錫華. 一種病毒蛋白B細胞表位預測方法的建立[J]. 科學通報, 1994, 93(24): 2275-2279.
[20] 薩姆布魯克. 分子克隆實驗指南[M]. 黃培堂, 等, 譯. 2版. 北京: 科學出版社, 1999.
[21] 王孝平, 邢樹禮. 考馬斯亮藍法測定蛋白含量的研究[J]. 天津化工, 2009, 23(3): 40-42.
[22] 皮晉魁, 張煥容. 對兩種雞卵黃免疫球蛋白兩種提取方法的比較[J]. 四川畜牧獸醫, 2011, 38(10): 26-28. DOI:10.3969/ j.issn.1001-8964.2011.10.012.
[23] 閆紅, 徐發良, 顧長國, 等. MD22的B細胞抗原表位預測[J]. 免疫學雜志, 2006, 22(1): 17-19. DOI:10.3969/j.issn.1000-8861.2006.01.005.
[24] 萬濤, 孫濤, 吳加金. 蛋白順序性抗原決定簇的多參數綜合預測[J].中國免疫學雜志, 1997, 13(6): 329-333.
[25] ODORICO M, PELLEQUER J L. BEPITOPE: predicting the location of continuous epitopes and patterns in proteins[J]. Journal of Molecular Recognition, 2003, 16(1): 20-22. DOI:10.1002/jmr.602.
[26] YAO B, ZHENG D D, LIANG S D, et al. Conformational B-cell epitope prediction on antigen protein structures: a review of current algorithms and comparison with common on binding site prediction methods[J]. PLoS ONE, 2013, 8(4): e62249. DOI:10.1371/journal. pone.0062249.
[27] 于翠梅, 張志宏, 劉月學, 等. 草莓輕型黃邊病毒CP基因的克隆、序列分析及原核表達[J]. 華北農學報, 2012, 27(1): 30-34. DOI:10.3969/j.issn.1000-7091.2012.01.006.
Epitope Prediction, Cloning, Expression and Immunogenicity of the Coat Protein of Strawberry Mild Yellow Edge Virus
YANG Jumei1, MA Jianzhong1,*, WANG Yonggang1, DENG Zibing1, WEI Yan1, PAN Bo2, LI Yinwu2
(1. School of Life Science and Engineering, Lanzhou University of Technology, Lanzhou 730050, China; 2. Gansu Qimingxing Energy-Saving Company, Baiyin 730913, China)
The amino acid sequence of epitope peptides of the coat protein of strawberry mild yellow edge virus was predicted by bioinformatic tools including IEDB, DNAStar, DNAMAN and SnapGene. The peptide from the 27thto 38thamino acid residues with predicted high antigenic activity, referred to as AE, was reversely translated into its encoding DNA sequence according to the codon bias of E. coli. The encoding DNA fragment AE was then synthesized and cloned into the expression vector pET32a (+) between the EcoRⅠ and XhoⅠ sites. The open reading frame (ORF) containing the AE fragment was 561 bp, which encoded a fusion protein of 187 amino acid residues. The theoretical molecular mass of the recombinant fusion protein was 20.29 kD. Expression of the target gene in the recombinant plasmid pET32a (+)-AE was induced and optimized in E. coli BL21 (DE3) and E. coli Rosetta, respectively. The optimal expression conditions of the fusion protein in E. coli Rosetta were 1.5 mmol/L IPTG at 35 ℃ for 2 h, resulting in a yield of 13.22 mg/L, while those for expression in E. coli BL21(DE3) were 0.5 mmol/L IPTG at 30 ℃ for 2 h, giving a yield of 9.55 mg/L. The recombinant fusion protein was isolated by SDS-PAGE and identified by tandem mass spectrometry. The results showed that the recombinant fusion protein contained the designed peptide AE. The recombinant fusion protein was purified by Ni2+-affinity chromatography, digested by EK enzyme, and separated from the fusion protein tag by molecular sieve. Chicken immunoglobulin (Igy) was extracted from chicken eggs collected on the 21stday after the first injection. The binding activity of the Igy to AE and SMyEV was analyzed by the Dot-Blot method. The results showed that the Igy could specifically recognize both AE peptide and SMyEV indicating that the predicted AE peptide has desired immunogenicity.
strawberry mild yellow edge virus; coat protein; epitope prediction; prokaryotic expression; tandem mass spectrometry; immunogenicity
10.7506/spkx1002-6630-201716011
Q785;Q786
A
1002-6630(2017)16-0071-08
2016-09-19
甘肅省科技計劃項目(1504WKCA020);國家大學生創新創業訓練項目(DC2015085);甘肅省橫向科技項目(10-2203);蘭州理工大學實驗室管理處教研項目
楊菊梅(1990—),女,碩士研究生,研究方向為分子檢測。E-mail:cherry_yjm@sina.cn
*通信作者:馬建忠(1963—),男,教授,博士,研究方向為植物基因工程。E-mail:majz@lut.cn
楊菊梅, 馬建忠, 王永剛, 等. 草莓輕型黃邊病毒外殼蛋白抗原表位預測、克隆、表達及其免疫原性分析[J]. 食品科學, 2017, 38(16): 71-78. DOI:10.7506/spkx1002-6630-201716011. http://www.spkx.net.cn
YANG Jumei, MA Jianzhong, WANG Yonggang, et al. Epitope prediction, cloning, expression and immunogenicity of the coat protein of strawberry mild yellow edge virus[J]. Food Science, 2017, 38(16): 71-78. (in Chinese with English abstract)
10.7506/spkx1002-6630-201716011. http://www.spkx.net.cn

