基于COI基因對混養緬甸蟒個體來源及遺傳多樣性分析
樊翠平段玉寶田秀華白素英
(1.東北林業大學野生動物資源學院,哈爾濱,150040;2.西南林業大學林學院,云南省森林災害預警與控制重點實驗室,昆明,650224)
基于COI基因對混養緬甸蟒個體來源及遺傳多樣性分析
樊翠平段玉寶田秀華1*白素英1
(1.東北林業大學野生動物資源學院,哈爾濱,150040;2.西南林業大學林學院,云南省森林災害預警與控制重點實驗室,昆明,650224)
稿件運行過程
緬甸蟒; 個體來源; COI; 遺傳多樣性; 遺傳距離
緬甸蟒(Pythonbivittatus)是世界上最巨型的6種無毒蛇類之一[1],隸屬于蛇亞目(Serpentes)、蟒科(Pythonidae),被列為 《中國物種紅色名錄》極危物種,為國家Ⅰ級和 CITES附錄Ⅱ保護動物[2]。緬甸蟒在中國分布于福建(南平、金門)、廣東、香港、海南、廣西、云南、貴州(望謨、羅甸、紫云)、西藏(墨脫)、江西、四川(內江)等地[1-4]。
分子系統學是通過分子技術,從分子水平研究生物分類和生物進化關系的一門學科[5],這項技術需要大量特定標記的遺傳數據,通過對遺傳數據進行統計整理,分析出個體間的遺傳差異,達到對結果進行解釋的目的。分子系統學的高速發展,使分子標記被運用在生物學領域研究的方方面面[6]。線粒體(mitochondrial)是存在于真核生物細胞內的一種可以進行有氧呼吸的細胞器,同時也是動物體細胞質遺傳信息的唯一載體[7]。線粒體遺傳是嚴格的細胞質母系遺傳,它具有分子質量小,結構簡單,不易發生插入和缺失等特點,是研究低級分類單元較好的分子標記方式,在多種動物類群的系統進化研究中得到廣泛應用[8]。細胞色素c氧化酶亞單元I(Cy-tochrome oxidase subunitⅠ,COI)基因是線粒體蛋白質編碼基因之一[3],該基因序列雖然進化速率較低,序列相對保守,但卻也能夠發生充足的基因突變,提供豐富的DNA信息[9-10],被認為是一種非常好的分子生物學標記基因,這也是COI DNA條形編碼基因常被選用作為研究基因的主要原因[4]。海內外學者通過對線粒體控制區基因分析成功判定緬甸蟒屬于本土物種,通過對微衛星標記分析緬甸蟒遺傳多樣性。臺灣師范大學教授You等利用線粒體Cytb和COI對33條來自臺灣金門、福州動物園以及越南的緬甸蟒進行了分子系統發育研究,單倍型網絡顯示了金門種群與大陸種群親緣關系較近,證實了金門島的緬甸蟒并非外來種,是臺灣首例被列為本土種的蟒蛇[11];劉雅芳等對海南和越南2個已知來源的緬甸蟒種群,基于控制區II序列進行建樹、并計算兩群體的固定指數(Fst),得出海南緬甸蟒與越南緬甸蟒已經出現明顯分化的結果[12]。段玉寶等人通過微衛星標記得出緬甸蟒海南養殖群體在近期沒有經歷過瓶頸效應,群體數量沒有出現過明顯下降[10]。楊寶山等通過COI基因序列變異對不同地理種群的銀杏大蠶蛾(DictyoplcajaponicaButler)構建系統發育樹,分析結果表明南方組和北方組種群間已經按地理位置形成了一定的地理格局,AMOVA分析顯示北方組和南方組之間已經具有明顯的遺傳分化,因此可以根據COI基因序列變異將這13個地理種群劃分為北方和南方2個組的進化分枝[13]。張迪等人在基于COI基因序列的太湖新銀魚(Neosalanxtaihuensis)遺傳多樣性中認為線粒體COI基因非常適合銀魚科(Salangidae)魚類的物種鑒別和系統發育研究,在同種不同地理種群間遺傳關系研究中也同樣適宜,但若結合Cytb等基因一并分析,可以獲取更多的遺傳信息[14]。鄭文娟等人基于線粒體COI基因序列探討泥蚶(Tegillarcagranosa)的遺傳分化,得出我國沿海地區7個泥蚶群體已經分化形成了二大類群:福建以北(包括福建)的5個群體(江蘇鹽城、浙江奉化、浙江樂清養殖和自然群體、福建福鼎)形成一個類群,福建以南的2個群體(廣東湛江、海南海口)形成一個類群[15]。You基于Cytb和COI基因序列的蟒蛇分子鑒定中,表明福州動物園的蟒蛇是福建本地種群和東南亞種群混養的,其中金門種群和Fuzhou1和Fuzhou4組成福州本地種群,Fuzhou2、Fuzhou3、Fuzhou5與海南緬甸蟒蛇及越南蟒蛇等組成東南亞種群。該文章表明金門蟒蛇非來源東南亞,與金門原生種群與福建本地種同源[11]。
近些年,中國境內蟒蛇野外數量急劇減少,緬甸蟒人工養殖的成功繁育和馴化,為野外種群的擴增提供了可能。而國內對于該物種原人工繁育種群的遺傳學、分子生物學等相關研究十分缺乏。海南東盛弘蟒業科技股份有限公司飼養的緬甸蟒其產地與來源不同,由于沒有規范的來源記錄,且不同地理種群的蟒蛇在外形特征上不易區分,以至不能準確鑒定。為了更好地對蟒蛇進行科學的鑒別和保護,本研究首次對人工混養養殖種群基于COI基因序列進行了遺傳多樣性、遺傳距離較為詳盡研究,并重新確定人工混養養殖種群的物種來源,為人工繁育的緬甸蟒種群健康發展和野生資源的遷地保護提供了分子生物學參考。
1 材料與方法
1.1實驗材料
在海南東盛弘蟒業科技股份有限公司,采用無損傷取樣法收集緬甸蟒蛇蛻樣本,共采集28條緬甸蟒剛剛褪去的蛇蛻,冰凍帶回實驗室,-80℃保存。
1.2實驗方法
1.2.1基因組DNA的提取
利用經典的苯酚-氯仿方法提取緬甸蟒的蛇蛻基因組DNA:緬甸蟒基因組DNA通過酚、氯仿多次抽提,后用無水乙醇和70%的乙醇沉淀DNA;室溫干燥后加50 μL TE溶液溶解,-4℃保存或-20℃儲存。用紫外分光光度法檢測DNA的濃度,然后稀釋成50 ng備用。
1.2.2引物的擴增
本研究使用You等[11]報道的COI引物,引物由博仕生物技術股份有限公司合成。
F:5′-CCCTTATGAGTAGATTTACAGCCTA-3′
R:5′-GGATTGGGGCGTACATATTGTTTAGT-3′
PCR擴增的目的片段為1 500 bp,擴增體系為50 μL,其配比為:8×PCR Buffer 5 μL,dNTP Mixture 5 μL,rTaq酶0.5 μL,Primer 1和Primer 2均是1 μL(最終濃度),模板5~8 μL,然后補增滅菌的去離子水至50 μL。
在Eppendorf梯度PCR儀上進行PCR擴增,PCR反應條件:依次預變性溫度95℃時長5 min,變性溫度95℃時長28 s,退火溫度55℃時長28 s,延伸溫度72℃時長88 s,35個循環后72℃延伸8 min,4℃保存。將PCR擴增產物在濃度為1%的瓊脂糖凝膠中進行電泳檢測后,采用愛思進生物技術(杭州)有限公司的AxyPrep DNA凝膠回收試劑盒對PCR產物進行純化回收,純化回收后的樣品送博仕生物公司使用ABI3728全自動測序儀進行雙向測序。
1.2.3數據處理
將測得的基因序列,用Seqman軟件進行拼接,人工進行峰圖矯正后,使用MEGA 5.05與Genbank中的基因序列進行比對、剪切;使用DnaSP 5.8.01軟件統計多態位點數、核苷酸多樣性(Pi)、單倍型多樣性(Hd)和核苷酸平均差異數(K),確定單倍型。在NCBI中下載已知的緬甸蟒COI序列,用MEGA 5.05采用最大似然法(Maximum Likelihood,ML),以 Modeltest 3.7中h LRT篩選出的最優模型GTR+I+G 在 PAUP*4.0 b8中構建ML樹。用MEGA 5.05計算不同種間的遺傳距離。
表1 GenBank下載序列詳情

Tab.1 GenBank download sequence for details
2 結果
2.1 COI序列基因片段特征及遺傳多樣性
測序得到的28條緬甸蟒mtDNA COI基因序列,經過比對剪切得到COI基因全序列,片段長度為1 428 bp。其中T、C、A和G的含量分別為29.8%、15.5%、27.9%和26.9%,A+T的含量(54.8%)高于C+G的含量(45.2%)。保守位點(C)占89.6%,變異位點(V)占9.6%,簡約信息位點(Pi)占1.7%。緬甸蟒混養種群存在的單倍型數為8個,單倍型多樣性(Hd)為(0.726±0.068),核苷酸多樣性Pi為(0.003 57±0.000 56),平均核苷酸差異數k是5.099。
2.2 系統發育樹的構建
在GenBank分別下載3條越南緬甸蟒、1條海南緬甸蟒和12條金門緬甸蟒與1條印度蟒(表1)的COI基因序列,與本研究所檢測的28條養殖緬甸蟒樣本的COI同源基因序列采用鄰接法構建ML(圖1)。
由圖1的ML樹拓撲結構可以看出,12條金門緬甸蟒聚成一小支,與8條養殖緬甸蟒與海南緬甸蟒HN(KF293729.1)聚在一起,和其余20條養殖緬甸蟒聚與3條越南緬甸蟒(PM008(JX40187.1)、PM007(JX40186.1)、PM08(JX40189.1))聚成的一支共同形成姐妹支,然后與印度蟒(PMM(HM581978.1)聚在一起。
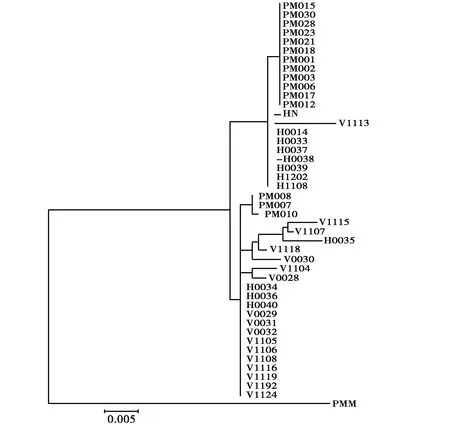
圖1 基于COI基因序列構建的全部個體ML系統進化樹Fig.1 Phylogenetic tree based on COI gene sequences of all individuals
2.3 遺傳距離
用MEGA 5.05計算不同種間的遺傳距離,首先對單個個體間遺傳距離進行分析,發現全部緬甸蟒個體間遺傳距離在0.000 7~0.012 75之間;緬甸蟒個體與印度蟒之間的遺傳距離為0.069 02~0.073 80。又根據已知地理分布和系統發育樹所顯示的差異將樣本分為4大類群,分別為海南類群(HN:包括海南個體與8條養殖個體)、金門類群(JM)、養殖群體1(YZ1:包括其余20條聚在一起的養殖個體)和越南群體(YN)。計算4個群體間遺傳距離(表2),根據其遺傳距離遠近判斷其歸屬。結果發現4個緬甸蟒群體內的遺傳距離都很接近(0.0~0.002 95)。4個群體間遺傳距離在0.002 11~0.006 99之間。其中YZ1群體與YN群體間遺傳距離為0.002 52,與海南群體間遺傳距離為0.006 99。
表2 基于COI基因序列計算的4個越南緬甸蟒群體間的遺傳距離
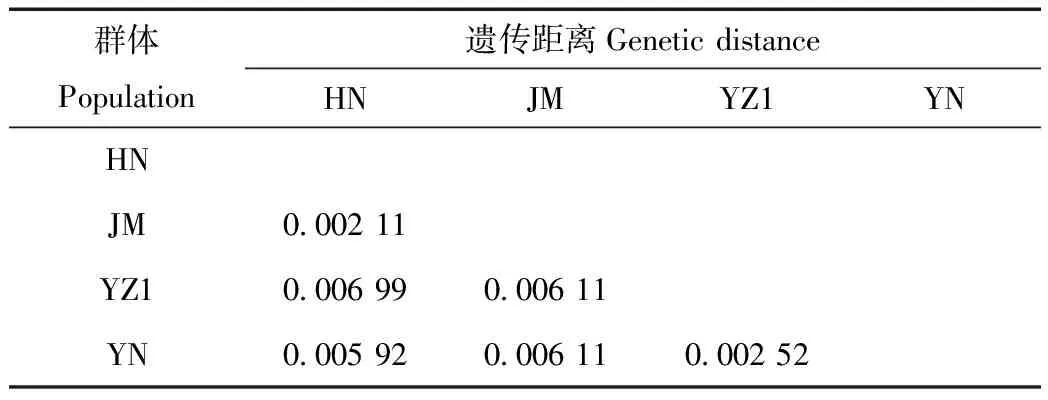
Tab.2 Genetic distance of partial COI gene between four Python bivittatus populations
3 討論
3.1 COI序列的多態性分析
通過對COI堿基序列的分析可以發現,C堿基在各堿基中所占的比例要低于其他堿基,其中A+T的含量(54.8%)明顯高于C+T的含量(45.2%),說明COI的堿基組成具有偏向性,這與脊椎動物線粒體DNA輕鏈(light strand)COI區核苷酸的堿基組成特點相一致[15]。已有研究表明,線粒體不同基因的進化速率存在差異。本文中檢測的28條養殖緬甸蟒COI基因中保守位點(C)占86.9%,變異位點(V)占4.6%,證明COI基因序列相對保守,變異速率慢。COI基因是線粒體13種編碼基因之一,因此其進化速率較慢,這與其他動物相關序列的研究結果相同。
人工混養養殖緬甸蟒群體單倍型多樣性相對比較高(>0.7),核苷酸多樣性較低(<0.007),這表明該群體遺傳多樣性并不高。已有的研究通過對線粒體DNA序列的遺傳變異進行分析,通過單倍型多樣性與核苷酸多樣性的高低組合將其分為4種類型:第一類是前者較低(< 0.5)同時后者也低(< 0.005);第二類是前者較高但后者較低;第三類是前者較低但后者卻較高;第四類是前者與后者同時相對較高[16]。本研究中養殖緬甸蟒的結果屬于第二種類型。物種種群數量大和生長環境的不均性是維持自然種群內單倍型多樣性較高的基礎,種群數量的增長速度也是影響遺傳多樣性的重要因素[17]。劉雅芳等對緬甸蟒線粒體控制區遺傳多樣性的研究結果顯示緬甸蟒總體上群體遺傳特征與本文一致[12]。一個種群能否健康持續的發展,遺傳多樣性是關鍵,它關乎著整個種群的長期進化和繁衍,這一點對于人工混養緬甸種群的人工馴養及繁殖也同樣重要[18]。遺傳多樣性低,同樣會為人工馴養帶來阻礙,因此在人工養殖過程中,我們更應加強遺傳管理,明確物種內種群的劃分關系,并建立明確的譜系關系,防止近親衰退。
3.2 系統發育樹分析
劉雅芳等人通過對海南和越南已知的緬甸蟒種群的線粒體控制區基因建樹并計算兩群體間的固定指數Fst,證明海南與越南緬甸蟒在分子水平上已出現明顯的分化[12]。本文基于COI核苷酸序列構建的系統發生樹顯示,緬甸蟒與外群完全分開,ML法系統發育樹基部分為2支:由金門緬甸蟒種群與8條養殖蟒蛇個體和海南緬甸蟒個體聚在一起形成1支,3條越南緬甸蟒與其余20條養殖緬甸蟒個體聚在一起形成1支。基于以上建樹方法所產生的結果,28條養殖緬甸蟒中有8條緬甸蟒判斷其來源地為海南,另外20條緬甸蟒的來源地為越南。由此看出基于COI氨基酸序列重建的系統發育樹能夠用于緬甸蟒種源劃分。
3.3 種群遺傳距離分析
Avise認為同一物種的個體間遺傳距離一般會保持在0.05之內,當差異超過0.06,該個體已經與其他個體之間發生了明顯的亞種或者種的分化[19]。從本研究的結果顯示,海南、越南、金門與養殖個體的個體間的遺傳距離在0.000 7~0.012 75之間,證明養殖個體與其他野生群體間未發生明顯亞種或種的分化。同時做群體間遺傳距離,發現YZ1群間與越南群體間的遺傳距離為0.002 52,與HN緬甸蟒群體間遺傳距離為0.006 99,與JM緬甸蟒群體間遺傳距離為0.006 11。YZ1群體與越南緬甸蟒之間遺傳距離最近,這也與ML系統發育樹結果一致,從而進一步證明了YZ1群體來自越南的結果的準確性。HN群體與JM群體間的遺傳距離較近(0.002 11),這也與其物種來源的地理位置遠近和系統發育樹結果一致。
4 結論
本研究首次通過對28條人工混養養殖緬甸蟒COI基因序列進行擴增,成功準確判斷了混養個體的個體來源,28條養殖緬甸蟒樣品中有8條來自海南,20條來自越南。養殖場可根據本研究的結果,在以后的種群選育過程中根據不同的繁殖需求選取不同個體進行配對繁殖,同時,根據不同群體建立譜系管理制度,有效避免近交衰退,從而保持緬甸蟒群體內較豐富的遺傳多樣性。本研究成果為人工繁育的緬甸蟒種群健康發展提供了分子生物學參考,同時對保護蟒蛇資源、人工飼養的種群管理以及明確市場上的產品來源等方面將具有重要的理論參考價值和實際應用價值。
[1] 銀星果.蟒蛇幼體對人工飼料的消化率及其生長發育規律研究[D].南寧:廣西大學,2014.
[2] 陳英苗,易蘭,段成,等.武漢市蛇類市場初步調查[J].資源開發與市場,2010,26(2):132-134.
[3] 張娟,張利平,宗卉,等.家鴨細胞色素C氧化酶Ⅲ(COⅢ)基因的克隆及序列分析[J].中國生物工程雜志,2007,27(3):54-59.
[4] 肖金學,王文強,廉振民.淺議分子生態學的概念[J].延安大學學報:自然科學版,2008,27(1):69-71,75.
[5] 茅云翔,吳菲菲,杜國英.紅藻DNA條形碼分析技術研究進展[J].中國海洋大學學報:自然科學版,2014,44(8):48-53.
[6] 王利,邢世巖,王正華,等.銀杏分子系統學研究進展[J].山東林業科技,2005(6):58-59.
[7] 顧志良,吳常信.牦牛線粒體ND1和ND2基因序列和物種系統發育分析[G]//中國動物遺傳育種研究進展—第十三次全國動物遺傳育種學術討論會論文集.北京:中國農業科學技術出版社,2005:33-34.
[8] 潘敏慧,萬永繼,魯成,等.用rRNA序列分析家蠶病原性微孢子蟲Endoreticulatusbombycis的進化[J].蠶業科學,2003,29(2):196-199.
[9] Barker D G,Barker T M.The distribution of the burmese python,Pythonmolurusbivittatus[J].Bulletin of the Chicago Herpetological Society,2008,43(3):33-38.
[10] 段玉寶,王英樹,馬建章,等.緬甸蟒養殖種群的遺傳多樣性分析[J].西南林業大學學報,2016,36(3):163-168.
[11] You C W,Lin Y P,Lai Y H,et al.Return of the pythons:first formal records,with a special note on recovery of the Burmese python in the demilitarized Kinmen islands[J].Zoological Studies,2013,52(1):1-11.
[12] 劉雅芳,侯冠彧,于萍,等.海南與越南緬甸蟒線粒體控制區遺傳多樣性及差異性分析[J].湖北農業科學,2015,54(2):398-401,408.
[13] 楊寶山,候慶君,王歡,等.不同地理種群銀杏大蠶蛾COI基因序列變異與遺傳分化[J].昆蟲學報,2009,52(4):406-412.
[14] 張迪,雷光春,龔成,等.基于COI基因序列的太湖新銀魚遺傳多樣性[J].湖泊科學,2012,24(2):229-306.
[15] 鄭文娟,朱世華,沈錫權,等.基于線粒體COI基因序列探討泥蚶的遺傳分化[J].動物學研究,2009,30(1):17-23.
[16] 李大命,張彤晴,唐晟凱,等.太湖大銀魚(Protosalanxchinensis)細胞色素b基因序列多態性分析[J].江蘇農業學報,2015,31(4):840-845.
[17] Ziegler T,Hendrix R,Vogt M,et al.The diversity of a snake community in a karst forest ecosystem in the central Truon Son,Vietnam,with an identification key[J].Zootaxa,2007,1493:1-40.
[18] 楊博,陳小勇,楊君興.鱇白魚線粒體DNA控制區結構和種群遺傳多樣性分析[J].動物學研究,2008,29(4):379-385.
[19] Avise J C.Molecular markers,natural history and evolution[M].Springer Science & Business Media,2012.
Pythonbivittatus; Individual sources; COI; Genetic diversity; Genetic distance
采用PCR技術首次用緬甸蟒人工養殖混養種群作為實驗樣本,進行mtDNA COI基因序列擴增,得到28條mtDNA COI基因序列,片段長度為1 428 bp,其中T、C、A和G的含量分別為29.8%、15.5%、27.9%和26.9%,A+T的含量(54.8%)高于C+G的含量(45.2%)。保守位點(C)占89.6%,變異位點(V)占9.6%,簡約信息位點(Pi)占1.7%。根據個體系統發育進化樹與群體間遺傳距離重新判定緬甸蟒人工養殖混養養殖種群的個體來源,并確定28條養殖個體中8條來自海南,20條來自越南。從遺傳距離結果可以看出,緬甸蟒養殖群體與野生緬甸蟒相比,并沒有出現明顯的遺傳分化。
Source of Origin and Genetic Diversity of Polyculture Burmese Python (Python bivittatus)Based on COI Gene
Fan Cuiping1Duan Yubao2Tian Xiuhua1*Bai Suying1
(1.College of Wildlife Resource,Northeast Forestry University,Harbin,150040,China;2.College of Forestry,Southweat Forestry University,Key Laboratory of Forest Disaster Warning and Control of Yunnan Province,Kunming,650224,China)
We obtained 28 Cytochrome oxidase subunitⅠ (COI)gene sequences of polyculture burmese python (Pythonbivittatus)using PCR technology.The length of the COI gene fragment was 1428 bp,of which the content of T,C,A and G is 29.8%,15.5%,27.9%,and 26.9%,respectively.Content of A+T (54.8%)was higher than that of C+G (45.2%).Conservative sites (C)accounted for 89.6%,mutation loci (V)accounted for 9.6%,and simple information site (Pi)accounted for 1.7%.Based on phylogeny trees and genetic distance between individuals,the 28 burmese pythons were considered to be 8 individuals from Hainan Province,China,and 20 individuals from Vietnam.According to genetic distance,there was no obvious genetic differentiation between the domestic and wild populations of burmese python.
樊翠平,女,25歲,碩士研究生;主要從事特種經濟動物飼養、分子生物學研究。E-mail:1391577155@qq.com 段玉寶,男,33歲,博士,講師;主要從事保護生物學、分子生物學研究。E-mail:boyciana@163.com
*通訊作者:田秀華,E-mail:tianxiu-hua@163.com
2016-12-06
Q953
A
修回日期:2016-02-24
發表日期:2017-05-10
2310-1490(2017)02-295-05

